粉红螺旋聚孢霉67-1短链脱氢酶基因CrSdr功能研究
陈莹莹,王亚楠,吕斌娜,谭晓东,李世东,孙漫红*,马桂珍
(1.江苏海洋大学江苏省海洋生物资源与生态环境重点实验室,连云港 222000;2.中国农业科学院植物保护研究所,北京 100193;3.北京市土肥工作站,北京 100029)
粉红螺旋聚孢霉Clonostachys rosea是一类分布广泛的丝状真菌,可以寄生多种植物病原真菌,如核盘菌Sclerotinia sclerotiorum、立枯丝核菌Rhizoctonia solani和灰葡萄孢Botrytis cinerea,还可以分泌抗菌和植物生长素类活性物质,显示出良好的生防潜力[1-4]。近年来,对粉红螺旋聚孢霉功能基因的挖掘也不断深入,其中对细胞壁降解酶和物质运输与代谢相关蛋白编码基因的研究最为广泛。生防真菌在寄生过程中,其分泌的几丁质酶、纤维素酶和葡聚糖酶等细胞壁降解酶可直接破坏、消解病原真菌细胞壁,为其后续的侵入、定殖和扩展创造条件,并最终抑制病原菌的扩展与对植物的侵染[5,6]。Tzelepis等[7]发现敲除几丁质酶基因chiC2后,粉红螺旋聚孢霉对灰葡萄孢和立枯丝核菌的抑制能力显著减弱,而过表达几丁质酶基因Chi67‑1能够显著提高其对核盘菌菌核的寄生能力和对大豆菌核病的防治效果[8]。Mamarabadi等[9]发现粉红螺旋聚孢霉与黄色镰刀菌Fusarium culmorum互作时会诱导N-乙酰-β-D-氨基葡萄糖苷酶基因cr-nag1上调表达。粉红螺旋聚孢霉生长和代谢过程中一些物质运输与代谢相关蛋白也会影响其寄生和拮抗能力。研究表明,热激蛋白70编码基因crhsp能够影响粉红螺旋聚孢霉菌落形态,基因缺失后对核盘菌的寄生能力显著降低,敲除ABCG29转运蛋白基因可导致该类生防菌对灰葡萄孢和禾谷镰刀菌的抑制作用下降[10,11]。Dubey等[12]在粉红螺旋聚孢霉中发现了2个含有细胞溶解酶结构域的蛋白LysM1和LysM2,能够影响自身孢子的萌发和菌丝生长,同时降低其对植物真菌病害的生防作用。
短链脱氢酶(short-chain dehydrogenase,SDR)广泛存在于各类生物体中,是一类重要的NAD(P)(H)依赖型氧化还原酶,也是迄今为止发现的最古老和最大的蛋白质超家族之一[13]。短链脱氢酶催化功能多样,可通过参与物质代谢及氧化还原反应[14,15],调节生物体内多种生理生化反应[16,17]。有研究表明,SDR通过参与植物的初级和次生代谢影响植物的生长、发育和对逆境的反应[18]。SDR还可以诱导植物产生系统抗性,阻碍细菌性病菌对马铃薯的侵染[19]。在丝状真菌中,短链脱氢酶基因参与了病原真菌的致病过程。研究发现,稻瘟病菌Magnaporthe oryzae短链脱氢酶基因MoSDR1能够影响分生孢子的形成、孢子萌发和附着胞的发育,降低稻瘟病菌的致病力[20]。在生防真菌中,氧化还原酶类基因通过调控寄生关键酶的合成以及一些次级代谢产物,如毒素和抗生素的合成与代谢,从而影响对寄主细胞的抑制作用与寄生能力。Malmierca等[21]发现,细胞色素P450基因缺失后苇状木霉Trichoderma arundinaceum对灰葡萄孢和立枯丝核菌的抑制作用明显减弱。在哈茨木霉T.harzianum中过表达NADPH氧化酶基因nox1能够诱导蛋白酶、纤维素酶和几丁质酶等几种细胞壁降解酶基因表达水平上调,并显著提高对终极腐霉Pythium ultimum的拮抗作用[22]。但迄今,短链脱氢酶基因在生防真菌中的生物学功能尚不明确。
粉红螺旋聚孢霉67-1是实验室分离到的一株高效生防菌株,前期从寄生核盘菌转录组中获得1个差异表达的短链脱氢酶基因CrSdr[23]。本研究通过基因敲除与回补及生物学测定,探讨其在粉红螺旋聚孢霉生防中的作用,该研究将为深入揭示粉红螺旋聚孢霉生防作用机制奠定理论基础。
1 材料与方法
1.1 供试菌株、质粒及植物材料
粉红螺旋聚孢霉Clonostachys rosea 67-1分自海南省菜园土壤。大豆菌核病菌Sclerotinia sclerotiorum分自黑龙江省大豆田病株,番茄灰霉病菌 Botrytis cinerea、黄瓜枯萎病菌 Fusarium oxysporum f.sp.cucumerinum和甘蓝枯萎病菌F.oxysporum f.sp.conglutinans分自河北廊坊温室病株。以上菌株由中国农业科学院植物保护研究所土传病害组保存。质粒pKH-KO和pKN由中国农业科学院植保所细菌病害研究组惠赠。供试大豆:中黄13号,购买于北京中蔬种业公司。
1.2 培养基
PDA培养基:新鲜马铃薯200 g,葡萄糖20 g,琼脂15 g,水1 L。PD培养基:新鲜马铃薯200 g,葡萄糖20 g,水1 L。LB培养基:胰蛋白胨10 g,酵母浸粉5 g,NaCl 10 g,水1 L,调节pH 7.0。胡萝卜培养基:新鲜胡萝卜洗净去皮,切成1 cm3左右小块,置于三角瓶中,装量为1/3。TB3培养基:酵母浸粉3 g,酸水解酪素3 g,蔗糖200 g,水1 L。以上培养基121 ℃湿热灭菌25 min。
1.3 CrSdr基因生物信息学分析
根据菌株67-1全基因组数据[23]获得CrSdr基因DNA序列。使用NCBI BlastP(https://blast.ncbi.nlm.nih.gov)分析氨基酸序列,使用ExPASy计算预测蛋白的分子量、等电点和亲疏水性,分别使用SignalP 4.1和TMHMM预测信号肽位点和跨膜区域,应用CDD预测CrSdr基因功能域,采用MEGA 7.0构建系统发育树[24]。
1.4 菌核诱导下CrSdr基因表达量监测
1.4.1 菌核的培养 将核盘菌接种于PDA上,26 ℃培养7~10 d,使用打孔器将菌落打成直径为3 mm的菌饼,接种至胡萝卜培养基中,每瓶10~15块。室温光照培养20 d,待培养基中产生大量的黑色颗粒状菌核后,用大量水反复冲洗培养物,去除胡萝卜残渣。将菌核自然晾干后保存备用。
1.4.2 RNA样品制备 将67-1接种于PDA平板上培养7~10 d,加入无菌水洗脱孢子,调整菌悬液浓度至1×107孢子/mL。吸取100 µL均匀涂布于铺有灭菌玻璃纸的PDA培养基上,26 ℃培养48 h。选取大小一致的菌核,75%酒精浸泡30 s,再加入2.5% NaClO溶液消毒3 min,无菌水冲洗3次,置于灭菌滤纸上吸去多余水分,超净工作台中吹干。将菌核铺满67-1平板,26 ℃下继续培养,分别于8、24和48 h时移去菌核,收集67-1菌丝,-80 ℃低温冰箱保存。以没有放置菌核的67-1纯培养为对照。每个处理5个重复。
1.4.3 RNA提取及反转录 采用RNeasy Plant Mini Kit试剂盒(QIAGEN,德国凯杰生物公司)提取样品RNA,DNase I(TransGen Biotech,北京全式金生物技术有限公司)去除基因组 DNA,反转录试剂盒(TIANGEN,天根生化科技有限公司)进行第一链cDNA的合成。
1.4.4 CrSdr基因表达量的定量监测 以延伸因子EF1作为内参基因[25],使用NCBI Primer-BLAST设计CrSdr基因及内参基因的荧光定量PCR引物CrSdr-qF/qR和EF1-F/R(表1)。采用Bio-Rad IQ 5实时荧光定量PCR检测仪和SYBR Green Ⅰ对CrSdr基因表达水平进行定量分析。反应体系25 µL,其中2×T5 Fast qPCR Mix(SYBR Green Ⅰ)12.5 µL,cDNA 2 µL,上下游引物各 1 µL,RNase-free ddH2O 10 µL。反应程序为:95 ℃预变性2 min;95 ℃变性10 s,55 ℃退火30 s,40个循环;程序运行后,从55 ℃到95 ℃之间每隔0.5 ℃采集一次荧光,共81个循环。采用2-∆∆Ct法计算CrSdr基因的相对表达水平[26]。每个处理3个重复。
1.5 CrSdr基因敲除与回补
1.5.1 敲除与回补载体的构建 利用同源重组原理,将CrSdr基因的上下游同源臂连接到带有潮霉素B抗性标记的pKH-KO载体上,构建敲除载体pKH-KO-CrSdr。设计相应CrSdr基因的回补引物CrSdr-Com-F/R(表1),PCR扩增获得CrSdr基因的全长序列(包括启动子、蛋白质编码区和终止子),将其克隆到pKN载体获得回补载体pKN-CrSdr-Com。
1.5.2 原生质体转化 采用PEG-CaCl2介导的原生质体转化法[27]将得到的基因敲除载体pKH-KO-CrSdr和回补载体pKN-CrSdr-Com分别转入67-1和基因敲除突变株ΔCrSdr的原生质体,得到基因敲除和回补转化子。分别以潮霉素B和G418为筛选标记,从PDA平板上筛选敲除转化子和回补转化子。将长出的菌落转接到不含抗生素的PDA平板上,连续培养3代,获得稳定遗传的敲除转化子和回补转化子。
1.5.3 基因敲除与回补菌株的筛选、鉴定 收集敲除和回补转化子菌丝,提取DNA。设计引物(表1),进行PCR和DNA测序验证。本研究所用引物合成及序列测定均由北京擎科生物科技有限公司完成。
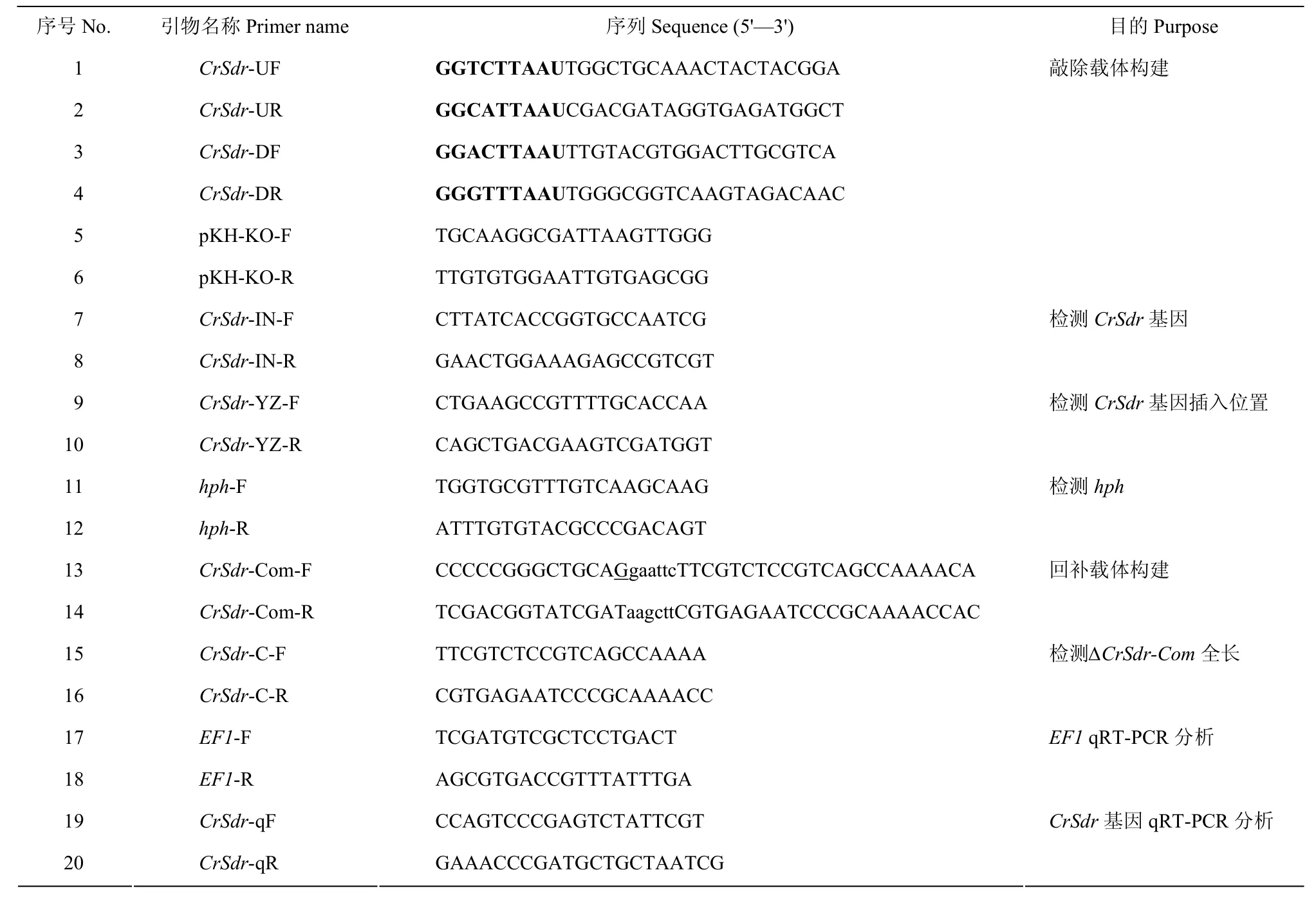
表1 本研究所用的引物Table 1 Primers used in this study
1.6 转化子生物学特性测定
1.6.1 生长速率、产孢量和孢子萌发率 分别打取野生型菌株、敲除突变株和回补菌株的菌饼(直径3 mm)接种于PDA平板中央,26 ℃培养,定期观察菌落大小和形态,从第3 d开始测量菌落直径,连续测定7 d,计算菌丝生长速率。培养10 d后,加入5 mL无菌水洗脱孢子,显微镜(BX41)下测定产孢量。调节各菌株孢子悬液浓度为1×107孢子/mL,接种于PD培养基中,26 ℃、180 r/min振荡培养,分别于8和16 h检测孢子萌发情况。
1.6.2 对不同胁迫压力的敏感性 PDA培养基中分别加入1 mol/L NaCl、1 mol/L KCl、1 mol/L山梨醇、20 mmol/L H2O2、0.2 mol/L CaCl2、0.03%十二烷基硫酸钠(SDS)和0.3 mg/mL刚果红(CR)。其中H2O2于培养基灭菌后添加,其余均在灭菌前添加。将野生型菌株和突变菌株的菌饼(直径3 mm)接种于不同胁迫压力的平板中,26 ℃恒温培养,7 d后测量菌落直径,并观察菌落形态的变化。每个处理5个重复。
1.6.3 对几种病原真菌的抑制作用 平板对峙法测定粉红螺旋聚孢霉菌株对大豆菌核病菌、番茄灰霉病菌、黄瓜枯萎病菌和甘蓝枯萎病菌的拮抗能力。打取不同菌株的菌饼(直径3 mm)放置在距离PDA平板边缘2 cm处,26 ℃下培养5 d,在平板另一端相同的位置接种直径为3 mm的病原真菌菌饼,26 ℃培养20 d后测量生防菌株菌丝的扩展距离,计算抑菌活性。每个处理5个重复。
1.6.4 对菌核的寄生能力 选取大小一致的菌核,75%酒精和2.5% NaClO消毒后分别浸于粉红螺旋聚孢霉孢子悬液中,10 min后取出吸干多余菌液,置于直径9 cm的无菌滤纸上,26 ℃培养,分别于8、24和48 h在体视显微镜(SZX12,OLYMPUS)下观察菌核寄生情况,并计算寄生率。7 d后,切片观察菌核内部侵染情况。寄生等级:0=菌核表面无寄生菌菌丝;1=菌核表面有稀疏菌丝;2=菌核被寄生菌菌丝覆盖,但表皮未脱落;3=菌核被寄生菌菌丝覆盖,菌核表皮脱落;4=菌核被菌丝覆盖,菌核内部软腐。每个菌株测定30个菌核,每个处理3个重复。
1.6.5 对大豆菌核病的防治效果 温室盆栽测定粉红螺旋聚孢霉野生型菌株和突变菌株对大豆菌核病的防治作用。选取籽粒饱满的大豆种子,1% NaClO表面消毒3 min,无菌水冲洗3次,晾干,播种于直径11 cm的塑料花盆中,每盆3粒种子。待大豆长到9片复叶时,在每片叶子的正面和背面均匀喷施浓度为1×107孢子/mL的粉红螺旋聚孢霉孢子悬液,2 h后接种培养5 d的核盘菌发酵液,以只接种病原菌的处理为对照。温室温度25 ℃~28 ℃,相对湿度为60%,花盆随机摆放。7 d后调查大豆叶片发病情况。分级标准:0=无病斑;1=病斑面积小于10%;2=病斑面积10%~30%;3=病斑面积30%~50%;4=病斑面积50%以上。每3盆(共9株)为1个处理,每个处理3个重复。病情指数=∑(各级病苗数×各级代表值)/调查总数×最高级代表值×100,防病效果(%)=(CK病情指数-处理病情指数)/(CK病情指数)×100。
1.7 数据统计与分析
试验数据采用Excel 2011和SPSS 24软件进行统计分析。
2 结果与分析
2.1 CrSdr基因的生物信息学分析
生物信息学分析表明,基因CrSdr(GenBank No.MW661068)全长768 bp,无内含子,编码255个氨基酸,预测的蛋白分子量为27.04 kDa,等电点6.51,为疏水性蛋白,无信号肽和跨膜结构域,该蛋白具有SDR结构域。系统发育分析表明,CrSdr基因与镰刀菌F.albosuccineum短链脱氢酶基因(KAF4471804.1)亲缘关系最近(图1)。
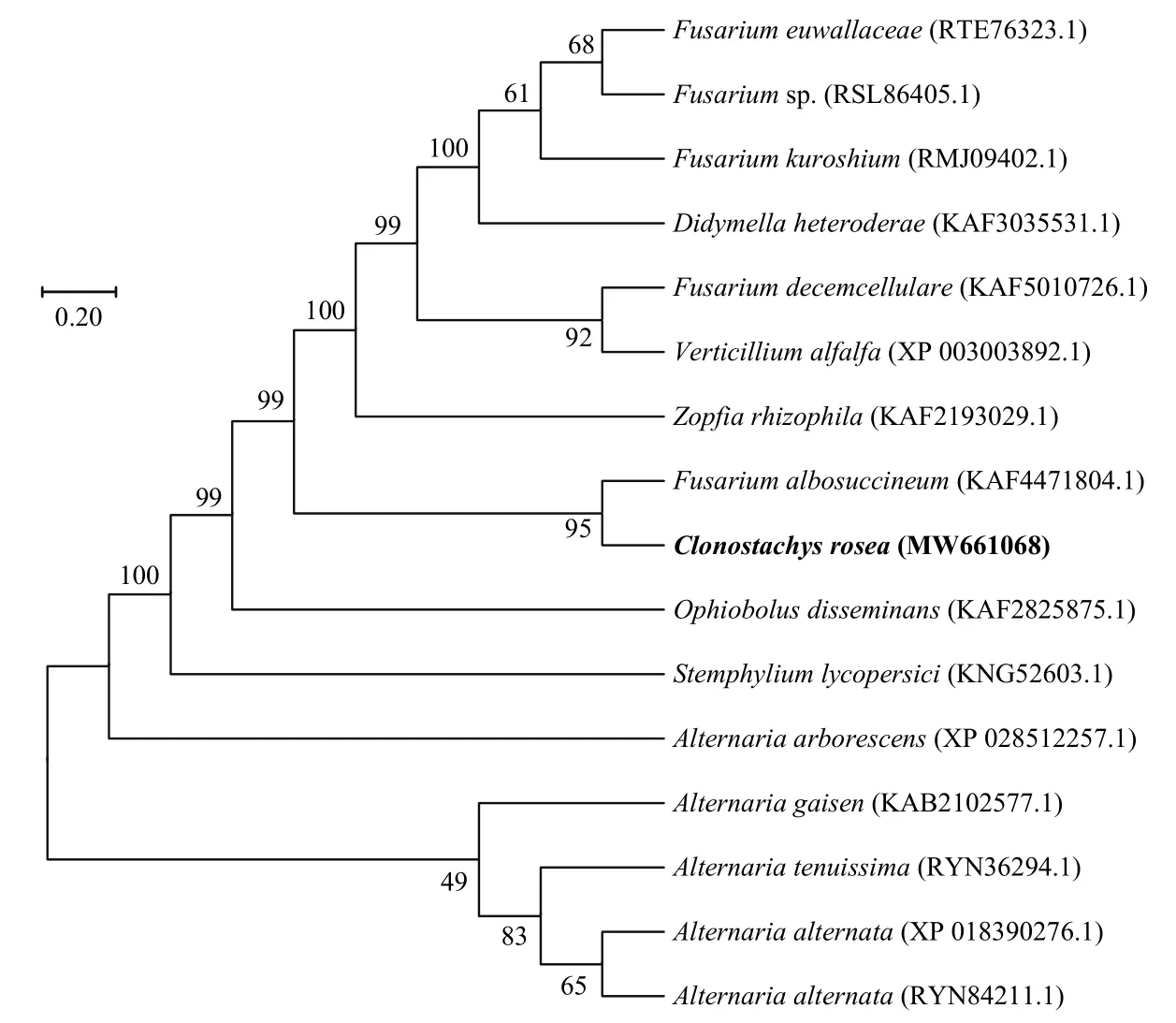
图1 粉红螺旋聚孢霉CrSdr基因的系统发育分析Fig.1 Phylogenetic analysis of CrSdr of C.rosea
2.2 CrSdr基因的表达量分析
荧光定量对粉红螺旋聚孢霉67-1 CrSdr基因的表达水平监测表明,菌核诱导下CrSdr基因表达趋势为先上调再下调,24 h时,CrSdr表达水平比对照提高4倍(图2),与67-1寄生核盘菌转录组数据一致[23]。
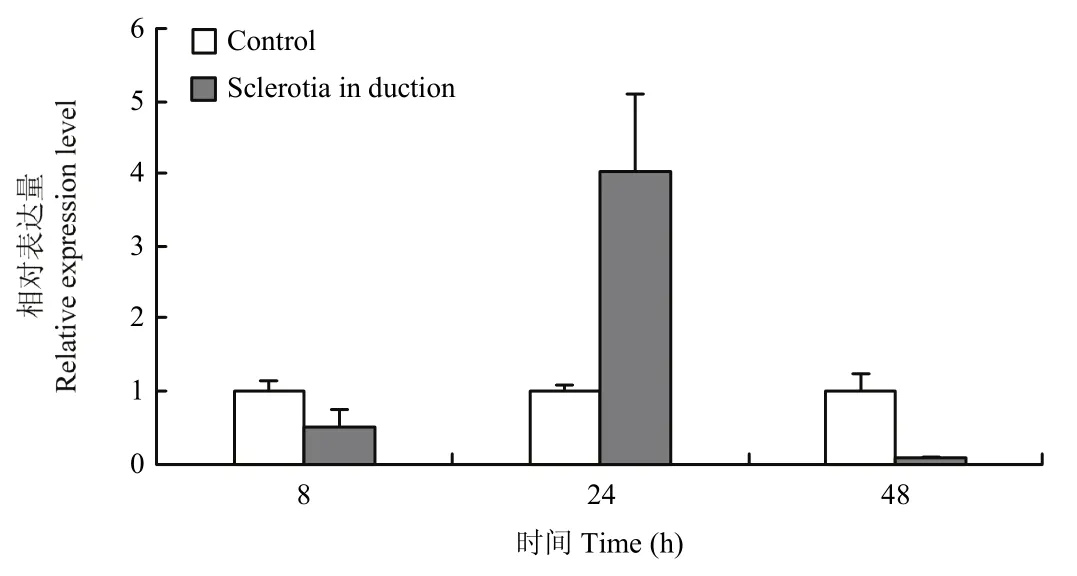
图2 菌核诱导下粉红螺旋聚孢霉67-1菌株CrSdr基因的表达水平Fig.2 Expression levels of CrSdr in C.rosea 67-1 under the induction of S.sclerotiorum sclerotia
2.3 CrSdr基因敲除和回补验证
筛选获得248个潮霉素抗性转化子,PCR验证得到3个敲除突变株∆CrSdr-1、∆CrSdr-2和∆CrSdr-3。PCR验证结果显示,敲除突变株中插入潮霉素抗性基因(1479 bp)。经CrSdr-YZ-F/R可扩增出3500 bp左右的片段,与预期片段一致。对扩增片段测序表明,CrSdr基因已被hph成功替换且未发生异位插入。将回补载体转入ΔCrSdr-1,PCR验证回补菌株ΔCrSdr-Com中存在目的基因片段(3016 bp)。测序结果表明,CrSdr基因成功插入突变株中(图3)。
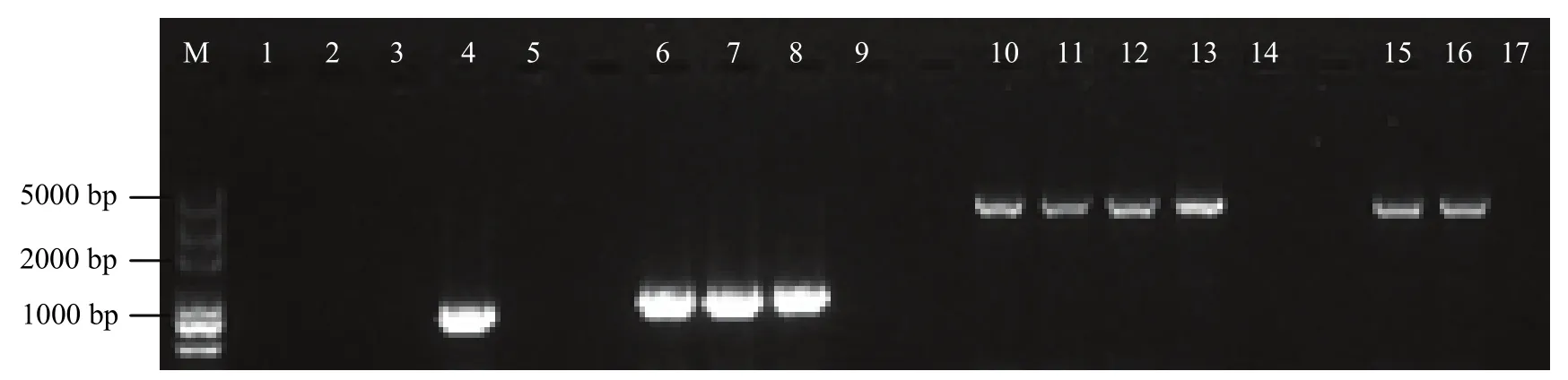
图3 粉红螺旋聚孢霉67-1突变株ΔCrSdr和ΔCrSdr-Com的PCR验证Fig.3 PCR verification of ΔCrSdr and ΔCrSdr-Com mutants of C.rosea 67-1
2.4 CrSdr基因对粉红螺旋聚孢霉生长和产孢的影响
粉红螺旋聚孢霉CrSdr基因敲除后,菌株菌落大小、形态、菌丝生长速率和孢子萌发率均无明显变化(图4),但敲除突变株∆CrSdr的产孢量比野生菌株增加了43.2%(表2)。
图4 敲除CrSdr基因对粉红螺旋聚孢霉67-1菌落形态的影响Fig.4 Effect of CrSdr deletion on the colony morphology of C.rosea 67-1
2.5 CrSdr基因对粉红螺旋聚孢霉胁迫反应的影响
与67-1野生菌株和回补菌株∆CrSdr-Com相比,敲除突变株∆CrSdr在含有NaCl、KCl和山梨醇的PDA培养基中生长缓慢,对渗透压胁迫表现出更强的敏感性,并且菌落形态发生了改变,菌丝较为疏松平坦。而CrSdr基因缺失后粉红螺旋聚孢霉对CaCl2的敏感性有所降低(图5)。
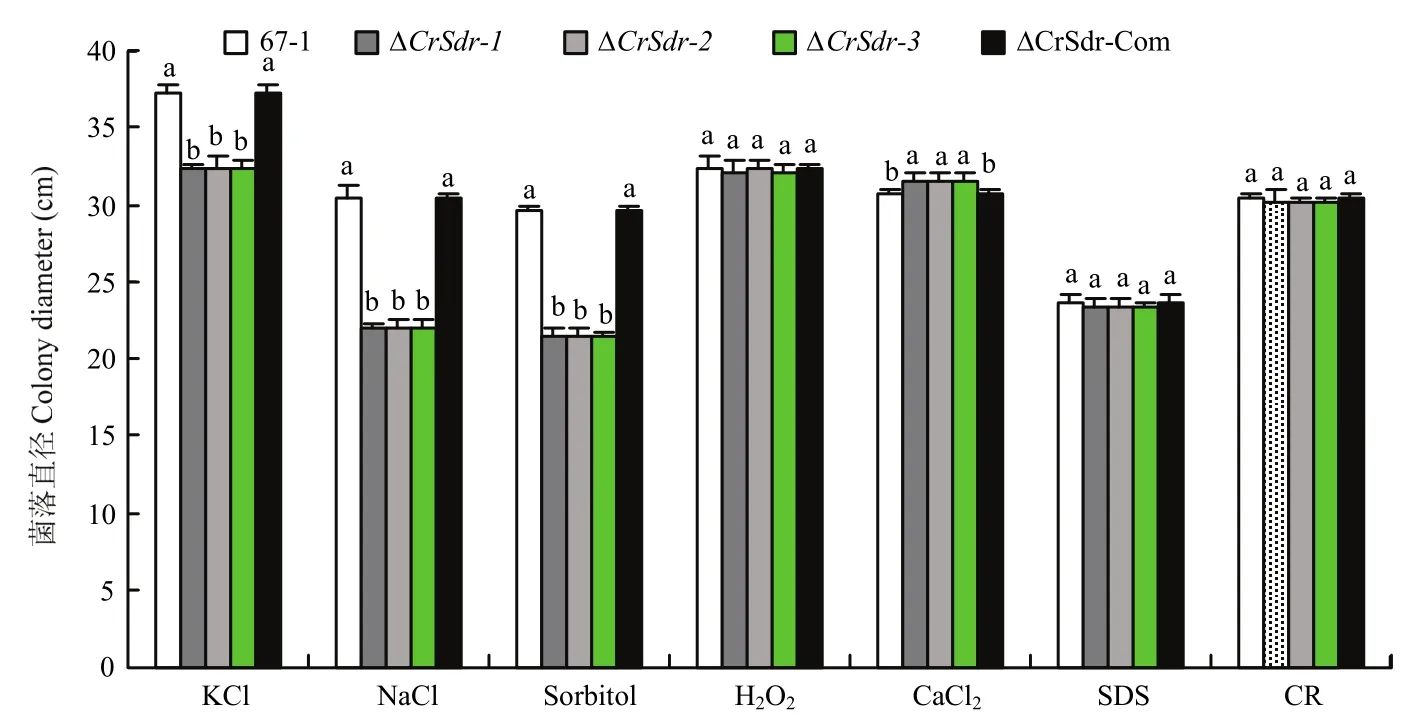
图5 不同胁迫压力下粉红螺旋聚孢霉转化子的菌落直径Fig.5 Colony diameters of C.rosea mutants under different stresses
2.6 CrSdr基因对粉红螺旋聚孢霉抗病原真菌活性的影响
敲除CrSdr基因后,粉红螺旋聚孢霉对番茄灰霉病菌和大豆菌核病菌的扩展距离较野生型分别减少了23.1%和13.6%,抗菌活性显著降低,基因回补后菌株的抗菌活性与野生型67-1一致(图6,表3)。

表3 粉红螺旋聚孢霉转化子向4种病原真菌的扩展距离Table 3 Extended distances of C.rosea mutants towards 4 pathogenic fungi
2.7 CrSdr基因对粉红螺旋聚孢霉寄生核盘菌菌核能力的影响
接种后8 h粉红螺旋聚孢霉67-1菌丝开始在菌核表面生长,24 h时寄生率达到86.7%。敲除CrSdr基因后寄生率下降了44.9%,而回补菌株对菌核的寄生能力恢复。培养7 d后可以看到,野生型和回补菌株处理组菌核内部完全腐烂和软化,而敲除突变株侵染菌丝稀疏,菌核仍较为坚硬。67-1和∆CrSdr-Com对菌核的寄生能力达到4级,而∆CrSdr的寄生能力仅为2级(图7)。
2.8 敲除CrSdr基因对粉红螺旋聚孢霉防治大豆菌核病作用的影响
接种核盘菌7 d后,对照组大豆叶片出现大面积黄褐色病斑,茎部发生褐变,植株病情多达到4级,粉红螺旋聚孢霉67-1孢子液处理组病斑面积显著减少,对大豆菌核病的防效达到68.9%。而敲除CrSdr基因后对病害的防效仅为34.1%,比野生型下降了50%以上。基因回补后菌株的生防作用恢复到原来的水平(图8,表5)。

图8 粉红螺旋聚孢霉CrSdr突变株对大豆菌核病的防治效果Fig.8 Control efficacy of CrSdr mutants of C.rosea against soybean Sclerotinia stem rot
3 讨论
本研究从粉红螺旋聚孢霉 67-1寄生核盘菌转录组中筛选到了一个显著上调表达的编码短链脱氢酶基因CrSdr,发现CrSdr基因缺失能够提高生防菌分生孢子的数量、降低对菌核的寄生能力和生防效果。研究首次证明了CrSdr基因在粉红螺旋聚孢霉重寄生过程和病害防控中发挥着重要作用。
氧化还原酶可以影响丝状真菌的生长和产孢,进而影响其定殖和重寄生水平[21,28,29]。本研究中,我们发现CrSdr基因缺失不影响粉红螺旋聚孢霉的正常生长,但是能显著增加分生孢子的数量,证明CrSdr基因参与调控了粉红螺旋聚孢霉的产孢过程。但前期也有对病原真菌的研究发现,敲除稻瘟菌 M.oryzae MoSDR1和禾谷镰刀菌F.graminearum FgPex3基因会导致产分生孢子能力下降[20,28],表明在不同的真菌中氧化还原酶基因调控产孢的方式不同。
真菌可以通过多种方式响应外源胁迫,维持细胞形态和正常的生理过程[30,31]。有研究发现,丝状真菌受到外界高渗压力后,可通过激活甘油合成相关酶基因的表达,在胞内积累高浓度甘油,提高细胞抵抗高渗胁迫反应的能力[32,33]。本试验中,在高渗胁迫(NaCl、KCl和山梨醇)条件下短链脱氢酶基因敲除突变株表现出更强的敏感性,这与前人对禾谷镰刀菌的研究结果一致[28,34]。我们还发现,基因缺失突变株在盐胁迫下菌丝明显减少,对丛枝菌根真菌Glomus intraradices的研究也表明,高渗胁迫使菌体生物量减少并使菌丝生长状态发生改变[35],但其作用机制仍需进一步探讨。在后续的研究中,可以尝试通过检测生防菌细胞中甘油含量的变化,明确其在胁迫应答反应中的作用。
研究发现,粉红螺旋聚孢霉67-1对供试4种病原真菌均有较好的抑制作用,能够在病原菌菌落上扩展,而敲除突变株∆CrSdr对番茄灰霉病菌、大豆菌核病菌和黄瓜枯萎病菌的定殖能力明显减弱,但对甘蓝枯萎病菌的抑制没有发生明显变化,这说明CrSdr基因参与不同病原菌的防控机制有可能存在差异。我们还发现,接种8 h后粉红螺旋聚孢霉孢子开始萌发,菌丝开始在核盘菌菌核表面生长,随着时间的推移对菌核表现出越来越强烈的寄生作用。在识别和侵染过程中,短链脱氢酶通过参与不同的信号传递途径影响对宿主的寄生[36,37]。本研究结果表明,敲除CrSdr基因显著降低了粉红螺旋聚孢霉67-1对核盘菌菌核的寄生能力及对大豆菌核病的防治效果,推测短链脱氢酶基因的缺失可能影响了相关信号的传递,进而影响其生防能力。
通过本研究我们明确了短链脱氢酶基因CrSdr在粉红螺旋聚孢霉寄生核盘菌和生防过程中发挥着重要作用。后期将在此基础上,对粉红螺旋聚孢霉不同侵染阶段进行显微形态和生物学研究,结合分子生物学分析明确CrSdr基因在菌寄生过程中的作用,为揭示粉红螺旋聚孢霉生防机制奠定基础。

