遗传多样性小鼠资源及其遗传学特点
王欣佩,张伶燕,劳兰兰,叶凡,刘云鹏,李丽,秦川
(中国医学科学院医学实验动物研究所,北京协和医学院比较医学中心,卫健委人类疾病比较医学重点实验室,北京市人类重大疾病实验动物模型工程技术研究中心,北京 100021)
影响人类健康的疾病,包括肥胖症、糖尿病、肿瘤、心血管疾病等,多为个体遗传信息与外部环境相互作用的结果。不同个体在同一疾病中往往表现出不同的易感性和临床表征,造成这一现象的主要原因是个体间遗传信息的多样性和复杂性[1-6]。小鼠作为经典的模式生物之一,是人类疾病研究中理想的动物模型[7]。目前,已有超过12 000个小鼠品系被建立。然而经典的实验室小鼠C57BL/6J、BALB/C、A/J等由于具有相似的起源和单一的遗传背景,难以体现人类遗传信息的复杂性[8-9]。
为克服小鼠模型在生物复杂性和多样性方面的不足,以多亲本重组杂交建立的新型小鼠资源的策略应时而生。如图1所示,亲本中包括5个广泛使用的近交系(A/J、C57BL/6J、129S1/SvImJ、NOD/ShiLtJ和NZO/HlLtJ)和3个野生近交系PWK/PhJ、WSB/EiJ以及CAST/EiJ,覆盖了小家鼠种群90%的遗传变异[10-12]。8个亲本通过连续三代杂交,获得携带全部亲本遗传信息的F3代杂交小鼠。F3代再经过20代以上近交传代,最终获得稳定遗传的重组近交系小鼠[13-15]。随后,与这些重组近交系小鼠拥有同样亲本的远交系小鼠也被建立[16]。新建立的近交系和远交系群体源自共同的亲本,通过重组杂交携带多个亲本的遗传信息,共同组成遗传多样性小鼠资源(Genetic Diversity mice,GD小鼠)[17-18]。
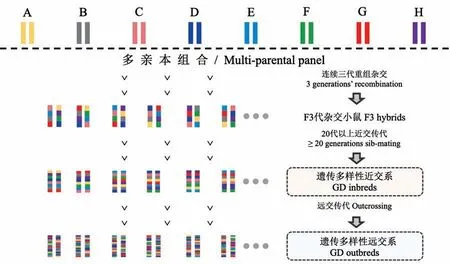
图1 遗传多样性近交系和远交系小鼠的育种策略Figure 1 Cultivation strategy of GD inbred and outbred mice
作为具有遗传多样性特点的新型资源,每个GD近交系小鼠品系具有固定的基因型,而每个GD远交系小鼠是独一无二的个体,拥有更多的等位基因组合可能[19]。由远亲繁殖策略发展而来的GD远交系小鼠,其遗传信息中大约存在4500万个单核苷酸多态性(single nucleotide polymorphism,SNP),是传统实验室小鼠品系的四倍[16]。因此,GD远交系小鼠是实现高分辨率基因定位的理想资源,而GD近交系小鼠可以为GD远交系小鼠的研究提供预测,也可以为复杂性状遗传学机制的研究提供可重复的基因型来源。本文系统介绍了遗传多样性小鼠的繁育策略,同时,借助全外显子组学方法分析了GD远交系和近交系小鼠在遗传信息多样性方面的特点。
1 材料与方法
1.1 材料
1.1.1 实验动物
无特定病原体(Specific Pathogen Free,SPF)遗传多样性近交系和远交系小鼠,雌雄各3只,8周龄后进行配对繁殖,体重15~25 g。小鼠源自中国医学科学院北方实验动物资源中心【SCXK(京)2019-0011】,饲养于中国医学科学院医学实验动物研究所SPF级环境设施中【SYXK(京)2019-0039】。设施环境温度20~26°C,相对湿度40%~70%,12 h/12 h固定明暗周期,自由进食饮水。所需的垫料、鼠盒、水瓶、均经过高温高压灭菌处理。实验操作过程中符合实验动物伦理学要求(伦理审批号:IACUC2019-QC007)。
1.1.2 主要试剂与仪器
DNA提取试剂盒(DP304)购自天根生化科技(北京)有限公司;直接电泳PCR试剂TaKaRa TaqTMVersion 2.0 plus dye(RR901Q)购自宝日医生物技术(北京)有限公司。TGrade加热型五段程控金属浴(天根);电泳仪(Bio-Rad);PCR仪(Bio-rad)。
1.2 方法
1.2.1 GD小鼠种群的培育
GD近交系各品系小鼠以全同胞兄妹进行传代扩繁。GD远交系小鼠由32对GD近交系小鼠建立,每对小鼠分别标记为固定的笼位号,从1到32号(图2A)。F1代杂交中,雌鼠留在原笼位不动,将雄鼠转移至下一笼位进行配对。即将1号笼位的雄鼠转移至2号笼位,与2号笼的雌鼠配对。2号笼位的雄鼠转移与3号笼的雌鼠配对,以此类推。按照这样的远交策略,目前GD远交系小鼠已经繁育至第34代。
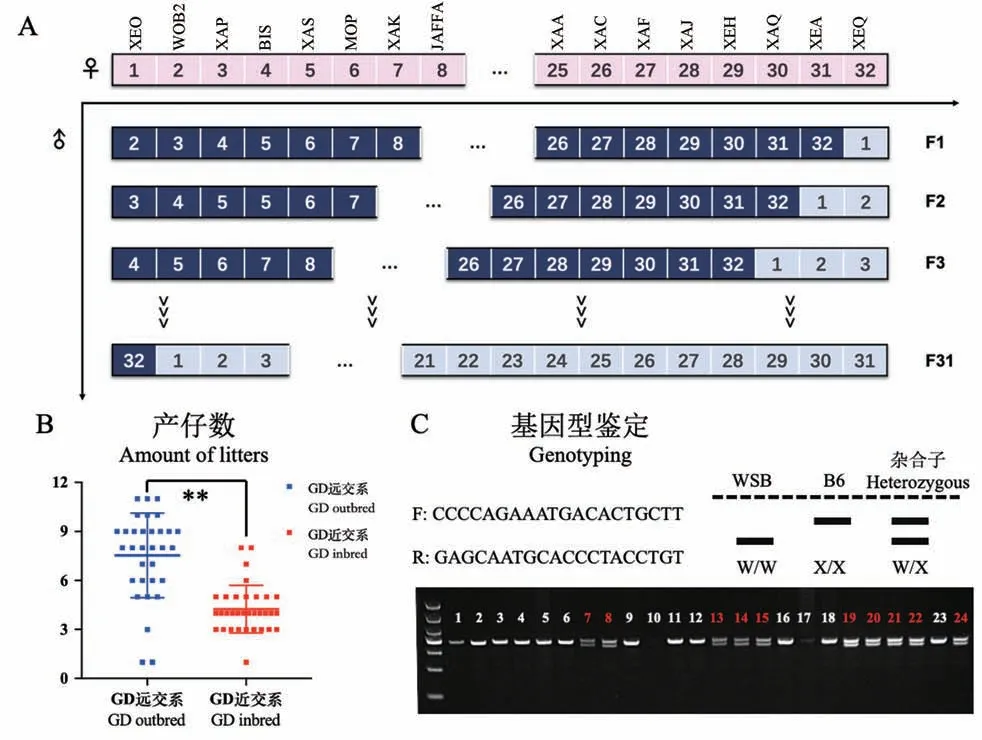
图2 GD远交系小鼠的培育策略与生殖表型Note.A.Breeding of GDoutbred mice.B.Comparison of the number of offspringper fetus in GDinbred and outbred mice.C.Genotyping of GD outbred,primers F-CCCCAGAAATGACACTGCTT,R-GAGCAATGACCCTACCTGT.DNA gel electrophoresis(3%)was performed after PCR amplification.The product fragment is between 280~500 bp.Compared with the average litter amount in GD outbed,**P<0.001.Figure 2 Strategy of cultivation of GD outbreds and their performance in reproduction
1.2.2 基因组DNA提取
使用组织/细胞基因组DNA提取试剂盒(DP304)提取基因组DNA。取鼠尾组织(30~50 mg)加入组织裂解液消化过夜后,按照试剂盒说明书以柱提法进行基因组DNA提取。
1.2.3 全外显子组学测序(Whole Exome Sequencing,WES)
小鼠基因组DNA样本由华大基因(深圳,中国)质量检测后进行全外显子组学测试。基因组DNA样品通过Covaris超声波样本处理系统被随机打断成200~300 bp的片段。随后对这些片段进行DNA片段末端修复、接头连接和线性扩增,制备成杂交文库。能被外显子芯片捕获的片段随后被扩增并经安捷伦2100生物分析仪和qPCR质控,质控合格的片段使用Illumina HiSeq系列平台进行高通量测序,并保证每个样品的数据量均达标。产生的原始图像数据由Illumina碱基识别软件(Base calling)转化为原始序列数据并以FASTQ文件格式存储。
1.2.4 WES数据分析
基于前一步骤获得的下机数据进行处理分析,通过质量控制流程,去除接头序列以及低质量和未测出的碱基数据。过滤后的片段通过序列比对软件BWA(Burrows-wheeler aligner)与参考基因组(mm10)进行比对,获得BAM格式文件。依据GATK(Genome analysis toolkit)官方推荐,使用最优变异检测分析流程处理比对结果。使用Picard工具去除比对结果中的重复序列片段,对每个样品的测序深度、覆盖度和对比率等评价指标进行统计。使用GATK v3.6的HalpotypeCaller工具同时检测数据中的SNPs和InDels,将所获得的高质量变异结果存储为VCF格式文件,并通过SnpEff(http://snpeff.sourceforge.net/SnpEff_manual.html)软件进行注释。
相关数据库及文件格式说明的网址如下:(1)UCSC build mm10:http://hgdownload.cse.ucsc.edu/goldenPath/mm10/bigZips;(2)RefGene database for mm10:http://hgdownload.cse.ucsc.edu/goldenPath/mm10/database/;(3)dbSNP:http://www.ncbi.nlm.nih.gov/snp;(4)SAM/BAM file format,Sequence Alignment/Map Format Specification: http://samtools.github.io/hts-specs/SAMv1.pdf;(5)VCF format: http://samtools. github. io/hts-specs/VCFv4.2.pdf。
1.2.5 染色体的物理重组图谱
利用BWA软件把每个样本的reads分别与参考基因组mm10进行比对,借助Picard工具去除重复reads,使用GATK软件的HaplotypeCaller工具来检测SNP集。子代样本与亲本样本的gvcf文件采用GATK的GenotypeGVCFs工具整合,采用GATK官网推荐的参数进行过滤(详见www.broadinstitute.org/gatk/gatkdocs/,Variant Annotations部分),获得高质量的Genotype数据集。随后,使用滑动窗口的方法来寻找重组位点:比较每个位点子代基因型与8个亲本基因型的一致性,从1开始递增滑动窗口(递增步数为1),同时采用基因型连续一致的计数方法,比较子代与亲本样本在滑动窗口内的一致性数量。由于测序错误与比对错误可能导致极少数的假阳性SNP,对小于100 kb的bin可以根据前后bin的亲本进行校正,最后利用R绘制子代品系每条染色体的物理重组图谱。
1.3 统计学方法
使用SPSS 22.0统计学软件对实验数据进行分析,以平均值±标准差(±s)来表示均值,采用方差t检验,以P<0.05为差异具有统计学意义。
2 结果
2.1 遗传多样性小鼠资源的培育
由多个亲本建立的GD小鼠资源依照不同的繁殖方式形成近交系和远交系两个群体。通过对两个小鼠群体生殖发育表型数据的记录,我们观察到两个小鼠群体在繁殖能力上具有较大差异。如图2B所示,GD近交系小鼠平均每胎产仔3~5只,而远交系小鼠每胎产仔7~9只(P<0.001)。按照GD小鼠近交系培育的策略理论推算,通过连续三代的重组杂交,理应获得4万余种近交系小鼠。然而实际繁育过程中,在经历20代近交传代后,最终获得1000余个近交系品系。此外,近交衰退现象还可能导致一部分GD近交系小鼠生殖能力下降,最终逐渐灭绝。相比近交系,GD远交系小鼠拥有杂交优势,体格健壮、繁殖力强,有利于群体扩大。
在GD远交系小鼠的培育过程中,发现作为亲本之一的WSB/EiJ品系在2号染色体75~90 Mb片段上存在有丝分裂驱动的现象。即该位点上如携带有来自WSB/EiJ的遗传信息,在多次传代后,种群内所有个体在该位点上逐渐被WSB/EiJ的遗传信息占据。为了防止这种遗传信息不平衡传播现象的出现,对GD远交系小鼠进行了基因型鉴定。如图2C所示,在每一代远交系小鼠配种中,仅选取4只在2号染色体75~90 Mb片段上携带有杂合子基因型的雌鼠和雄鼠进行繁育,以维持群体基因遗传稳态。
2.2 遗传多样性小鼠的遗传学特点
以多亲本策略建立遗传多样性小鼠的目的是利用尽可能多的基因型资源,通过多次的重组杂交,提高小鼠遗传信息的多样性。为从基因组水平探讨GD小鼠种群的多样性,分别对遗传多样性小鼠的近交系和远交系小鼠进行全外显子测序。全基因组测序(Whole Genome Sequence,WGS)和WES是遗传学研究中常用的技术,其中WGS技术可以获得研究样本的完整遗传信息,同时面临时间和经济成本高昂的局限。相比WGS,WES技术仅仅捕获基因组外显子区域DNA序列,数据量虽仅占全基因组数据的1%,但包含大约85%的致病遗传突变和大部分与表型相关的遗传信息[20]。在群体遗传学特点的研究中,WES数据可以经济高效的实现资源关联分析。
如图3所示,通过对WES数据进行遗传变异分析,分别获得GD近交系和远交系小鼠的单核苷酸多态性(SNPs),及小片段的插入缺失(insertions and deletions,Indels)两种遗传变异数据。如图3A所示,在GD远交系小鼠中发现了295 955个SNPs,在GD近交系小鼠中检测到218 953个SNPs。图3B中,GD远交系小鼠外显子序列中存在48 002个Indels,而在GD近交系小鼠中存在38 128个Indels。以上结果表明,GD远交系小鼠在SNPs和InDels总数方面分别是GD近交系小鼠的1.35倍和1.26倍,也就是说GD远交系小鼠携带有更为丰富的遗传变异。从图3A、3B中,还可以看到,在SNPs和InDels两种遗传变异的检测中,远交系小鼠在杂合变异(heterozygous)的数量方面更具优势,近交系小鼠则在纯合变异(homozygous)的数量方面更具优势。
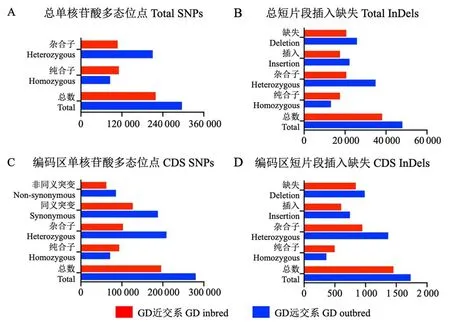
图3 GD小鼠遗传变异SNPs和InDels的检测Note.A.The number of total SNPs in GD mice,including heterozygous and homozygous variants.B.The number of total Indels in GD mice,including deletions and insertions,heterozygous and homozygous variants.C.The number of CDS SNPs in GD mice,including non-synonymous and synonymous,heterozygous and homozygous variants.D.The number of CDSInDels in GD mice,including deletions and insertions,heterozygous and homozygous variants.Figure 3 Call and detections of SNPs and InDels in GD mice
在编码区(coding sequence,CDS)SNPs和InDels的识别和统计中(图3C、3D),发现,在GD远交系小鼠CDS区拥有280 198个SNPs和1734个InDels,而GD近交系小鼠仅存在196 205个SNPs和1445个InDels,GD远交系小鼠在编码区序列中同样携带有更丰富的遗传变异。由此可知,相比于GD近交系小鼠,通过繁育策略获得的GD远交系小鼠提供了更多样的遗传信息组合方式。更加丰富多样的基因型数据为提高数量性状基因定位(quantitive trait locus,QTL)的精度提供了可能。
2.3 染色体物理图谱的绘制
比较GD远交系和近交系小鼠基因型和原始亲本基因型的一致性,以窗口滑动的方法寻找染色体上的重组位点,在滑动窗口内,一致性最高的亲本被判定为待检测片段的来源。通过这一方法,分别绘制了GD远交系和近交系小鼠的染色体物理图谱。如图4A、4B所示,相比GD近交系小鼠,GD远交系小鼠在染色体上表现出“马赛克”样式的特点。这一结果表明,GD远交系小鼠在遗传重组方面更具有优势,而GD近交系小鼠则在稳定遗传方面更具优势。

图4 GD小鼠染色体物理图谱Figure 4 Chromosome physical map of GD mice
2.4 亲本贡献
由于GD近交系小鼠具有稳定遗传的特点,可以更好的从群体角度反映亲本在子代小鼠中的遗传特点。在完成染色体物理图谱的绘制后,对GD近交系小鼠的亲本贡献进行了比较分析。如图5A所示,8个亲本对其子代近交系小鼠遗传信息的贡献并非按照1/8比例均等分布。亲本贡献的中位数分别为:A/J(12.3%),C57BL/6J(12.8%),129S1/SvImJ(14.4%)、NOD/ShiLtJ(13.7%)、NZO/HlLtJ(14.94%),PWK/PhJ(7.88%)、WSB/EiJ(11.95%)、CAST/EiJ(7.92%)。相比其他6个亲本,亲本CAST/EiJ和PWK/PhJ被稳定遗传的信息最少。如图5C所示,在对亲本贡献进行统计的工程中,发现在59个稳定遗传的GD近交系小鼠中,有7个品系仅仅获得了7个亲本的遗传信息,其中分别有2个品系丢失亲本PWK/PhJ遗传信息、2个品系丢失了WSB/EiJ,3个品系丢失了CAST/EiJ。此外,统计了59个GD近交系小鼠的每条常染色体上片段交换的次数。如图5B所示,从1至19号染色体,每条染色体上发生重组交换的次数不等,从各条染色重组次数的中位数来看,1号染色体上发生重组次数最多(69次),19号染色体上发生重组次数最少(18次)。造成亲本遗传偏好和染色体重组偏好的原因是否与自然选择等因素相关,目前还不明确。
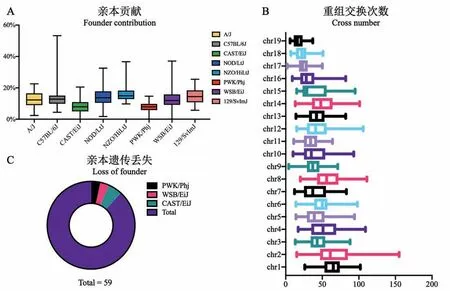
图5 亲本贡献Note.A.Founder contribution to Genetic Diversity Inbred mice.B.The frequency of recombination on autosomes of GD inbred mice.C.Loss of founder genetic information in GD inbred mice.Figure 5 Founder contributions
3 讨论
经典遗传学的研究工作,很大程度上依赖于传统的近交系小鼠。全世界各个科研机构保存有成千上万的小鼠资源,然而,这些小鼠在遗传变异方面具有高度的同源性和相关性,难以体现出人类遗传的复杂性,成为限制复杂性状疾病研究的瓶颈[9]。有别于经典近交系小鼠单一的遗传背景,遗传多样性小鼠资源采用多亲本策略建立的具有生物复杂性的小鼠资源。已有多个研究工作表明,遗传多样性小鼠在生长发育[21]、生理代谢[22-23]、免疫应答[24-27]等方面表现出丰富的表型特点。通过表型数据的差异分析,结合遗传信息对比分析,可获得与小鼠表型相关的候选基因[28-29]。
本文系统介绍了遗传多样小鼠资源的建立,包括近交系和远交系两个群体的建立。进一步,借助WES测序和数据分析,比较分析了GD近交系小鼠和远交系小鼠的遗传学特点,从遗传变异SNPs和InDels层面证明两个小鼠群体在遗传信息多样性的优势。通过染色体物理图谱的绘制和亲本贡献的分析,证明了原始亲本对GD小鼠遗传异质性的贡献。就GD远交系和近交系两个群体来说,近交系小鼠具有稳定遗传的特点,随着高通量、低成本基因分型和基因测序技术的发展,可以进行小鼠遗传变异与表型的关联分析,适用于识别和验证与复杂性状疾病相关的遗传因素。而GD近交系小鼠还存在一定局限性,缺乏杂交优势引起某些隐形等位基因效应,导致小鼠在活力方面显著低于远交繁殖小鼠。同时,对于大多哺乳动物来说,近交繁殖是一种不同常规的遗传状态,难以准确反应人类的遗传组成,远交系小鼠则可克服这一局限性。GD远交系小鼠由于的遗传多样性来自于自然发生的等位基因变异,可以更细微的反映某个已知基因编码区或调节区遗传变异引起的表型的差异[17]。因此,GD近交系和远交系两个群体在复杂性状遗传学机制的研究方面具有优势互补的特点。随着遗传多样性小鼠资源的发展,与之相关的信息学工具和数据资源正在迅速发展,将为解释复杂性状的遗传学机制提供新的挑战和机遇[30]。