结核分枝杆菌Hrp1蛋白的生物信息学分析
刘 静,吴 芳,张 杰,董江涛,柳小玲,梁 粟,王 菊,张春军,吴江东,章 乐,赵海军,张万江
全世界约有1/3的人口感染了结核分枝杆菌(Mycobacteriumtuberculosis,MTB),其中大多数是潜伏性感染(Latent Tuberculosis Infection,LTBI),目前无有效治疗LTBI患者的疫苗和药物,早发现结核杆菌潜伏感染患者,并对其进行诊断治疗,可以控制后期结核病患者人数[1]。Rv2626c基因的表达是MTB处于休眠期的重要标志之一,Rv2626c抗原受控于休眠调节子DosR(dormancy regulon),编码缺氧反应蛋白1(hypoxic response protein 1, Hrp1),Hrp1蛋白具有较好的抗原性和免疫原性,能有效刺激机体产生一定的细胞免疫及较高的抗体水平,并且该蛋白易被LTBI患者所识别[2],多项研究提示其很有可能会成为一种新型诊断MTB潜伏感染的候选抗原和治疗潜伏性感染的候选疫苗。为此,本文对Hrp1蛋白进行生物信息学研究分析,对其结构功能进行全面分析预测,为进一步了解MTB长期潜伏感染机制提供理论依据,以期为筛选 MTB 诊断标志物和研制新型结核疫苗奠定基础。
1 材料与方法
通过NCBI网站查询,Hrp1蛋白由143个氨基酸序列组成,检索的氨基酸序列如下:MTTARDIMNAGVTCVGEHETLTAAAQYMREH-DIGALPICGDDDRLHGMLTDRDIVIKGLAAG-LDPNTATAGELARDSIYYVDANASIQEMLN-VMEEHQVRRVPVISEHRLVGIVTEADIARH-LPEHAIVQFVKAICSPMALAS。
1.2 方 法
1.2.1 Rv2626c基因开放阅读框架分析及其编码蛋白的理化性质及亲疏水性分析 分析Rv2626c基因的开放阅读框架,采用NCBI网站中的ORF Finder软件(http://www.ncbi.nlm.nih.gov/orffinder)。上传Rv2626c基因编码的氨基酸序列至Protpapram生信软件(https://web.expasy.org/protparam)及ProtScale生信软件(http://web.expasy.org/protscale),分析其编码蛋白Hrp1蛋白的理化性质,包括氨基酸数目、分子式、分子质量、氨基酸组成、等电点、亲(疏)水性等。
1.2.2 Hrp1蛋白的信号肽、跨膜区、糖基化及磷酸化位点预测 运用信号肽分析系统SOSUI(http://harrier.nagahama-i-bio.ac.jp/sosui/)在线软件及Signal IP(http://www.cbs.dtu.dk/services/SignalP/)分析Hrp1的信号肽及其位置;运用跨膜区预测软件Tmpred(https://embnet.vital-it.ch/software/TMPRED-form.ht)分析Hrp1蛋白跨膜区;TMHMM(http://www.cbs.dtu.dk/services/TMHMM/)软件输入Hrp1蛋白的氨基酸序列,对该蛋白的拓扑结构进行预测分析[3]。利用分析Hrp1的糖基化位点采用NetNGlyc软件(http://www.cbs.dtu.dk/services/NetNGlyc/);预测Hrp1蛋白的磷酸化位点采用NetPhos 3.1 Server磷酸化分析软件(http://www.cbs.dtu.dk/services/NetPhos/)。
1.2.3 Hrp1蛋白的亚细胞定位分析 联合运用3种在线软件:PSORT(https://www.genscript.com/psort.html)、WoLF PSORT(https://wolfpsort.hgc.jp/)和TargetP-2.0-CBS(http://www.cbs.dtu.dk/services/TargetP/)对Hrp1蛋白进行亚细胞定位分析;预测Hrp1蛋白的核定位信号,运用NLStradamus在线软件(http://www.moseslab.csb.utoronto.ca/NLStradamus/)。
1.2.4 Hrp1蛋白结构预测 运用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/secpred_sopma.pl)预测 Hrp1蛋白的二级结构;SWISS-MODEL在线软件(https://swissmodel.expasy.org)对 Hrp1蛋白进行三级结构同源性建模分析。
1.2.5 Hrp1蛋白与人类蛋白的同源性分析 采用EXPASY进入主页(https://web.expasy.org/blast/),将Hrp1蛋白的氨基酸序列输入后,点击Homo sapiens,确定Run BLAST后,可对Hrp1蛋白氨基酸序列与人类蛋白的同源性进行BLAST分析。
随着时代发展,无人艇技术已得到深入发展。无人艇不仅可用于民用领域,比如海上搜救、垃圾清理和环境监测等方面,还可用于军事上的海上巡逻、侦查、监视和扫雷等方面,具有十分广泛的应用领域。[1]同时,无人艇路径规划对于舰船实现自动化航行和航线优化具有重要意义,要求在复杂的海洋环境中,根据已知的地理信息数据,寻找出一条从起点到终点的最安全且航程最短的航线。[2]目前已有多种算法被应用于路径规划,例如粒子群算法[3]、神经网络算法[4]和人工势场法。[5]其中遗传算法较为灵活,使用相对广泛,已被应用于众多无人设备的路径规划。
1.2.6 Hrp1蛋白的B细胞表位、辅助性T细胞表位、细胞毒性T淋巴细胞表位预测 在IEDB(http://www.iedb.org/home-v3.php)网站,输入氨基酸序列,通过Karplus&Schulz分析柔韧性、Kolaskar&Tongaonkar预测抗原性、Emini预测抗原表面可及性、Parker 预测亲水性及Chou&Fasman 法预测氨基酸编码蛋白质的β转角,对B细胞表位进行联合预测分析;β转角与无规则卷曲多出现在蛋白表面,为凸出疏松结构,与抗体更有利结合,成为B细胞抗原表位可能性较大[4];最后选取亲水性与表面可及性良好、柔韧性强、抗原性好、最好为卷曲和转角这种可能性大的区域作为候选B细胞表位,尽量避开α螺旋、β折叠结构。通过SYFPEITHI在线生信软件(http://www.syfpeithi.de/0-Home.htm)对Hrp1抗原蛋白Th表位进行预测[5],进入SYFPEITHI首页,选择HLA-DRB1*0101、0401、0701、0801、1101、1501这6种HLA基因分型进行预测辅助T细胞表位,选取得分为22分以上的氨基酸序列,综合分析后筛选出候选的辅助T细胞表位。利用RANKPEP在线生信软件进行预测,进入预测界面(http://imed.med.ucm.es/Tools/rankpep.html),将Hrp1氨基酸序列输入,选择SYFPEITHI所预测的HLA基因分型,找出在线软件红色标记的结果,筛选出结果较好的多肽序列作为候选的细胞毒性T淋巴细胞表位。最后采用DNAStar软件中Protean模板得到关于Hrp1蛋白表位预测的位置图,该图可直观看到Hrp1蛋白表位富集的区域,具体在图中表现为红色方块标记的区域。
1.2.7 Hrp1蛋白的相互作用蛋白预测及富集分析 将Hrp1全长氨基酸序列上传至STRING在线软件(http://string-db.org/cgi/input.pl),对Hrp1的相互作用蛋白进行预测,其生物过程进行功能富集分析。
2 结 果
2.1 Rv2626c基因开放阅读框架分析及其编码蛋白的理化性质及亲疏水性分析结果
2.1.1 Rv2626c基因开放阅读框架分析 结果显示Rv2626c基因(ID:888576)位于MTB(H37Rv)的全基因组(NC-000962.3)的2952562-2952993区域,全长432 bp,起始密码子为ATG,终止密码子为TAG,Rv2626c基因含有5个开放阅读框,其中最长的一个开放阅读框显示Rv2626c基因基本被通读,全长432 bp,与预测的编码Hrp1蛋白长度基本相似。
2.1.2 Rv2626c基因编码蛋白质的理化性质及亲疏水性分析 根据Protpapram预测结果,该蛋白的理论等电点PI为4.96,分子量为15 517.72;该蛋白由143个氨基酸构成,包含19种氨基酸,其中以丙氨酸(A)(13.3%)、异亮氨酸(I)(8.4%)、缬氨酸(V)(8.4%)3种氨基酸的组成比例较高,不包括色氨酸(W)、吡咯赖氨酸(O)和硒半胱氨酸(U)。带负电荷的残基(Asp+Glu)总数为21,带正电荷的残基(Arg+Lys)总数为11。Hrp1蛋白分子式为C667H1084N194O211S10,原子总数2 166,Rv2626c基因编码蛋白的消光系数为4 595,280 nm处的吸光度为0.296。当蛋白的N端残基为Met时,该蛋白在哺乳动物网状细胞中的半衰期为30 h,在酵母体内的半衰期大于20 h,在大肠杆菌体内的半衰期大于10 h。Hrp1蛋白不稳定性指数为19.62,为稳定蛋白,脂肪族氨基酸指数为100.35,平均亲水为0.042。ProtScale预测进一步分析显示Rv2626c基因编码蛋白为亲水性蛋白,第99位氨基酸亲水性得分最高为-1.767;第132为氨基酸疏水性得分最高为1.9(图1)。
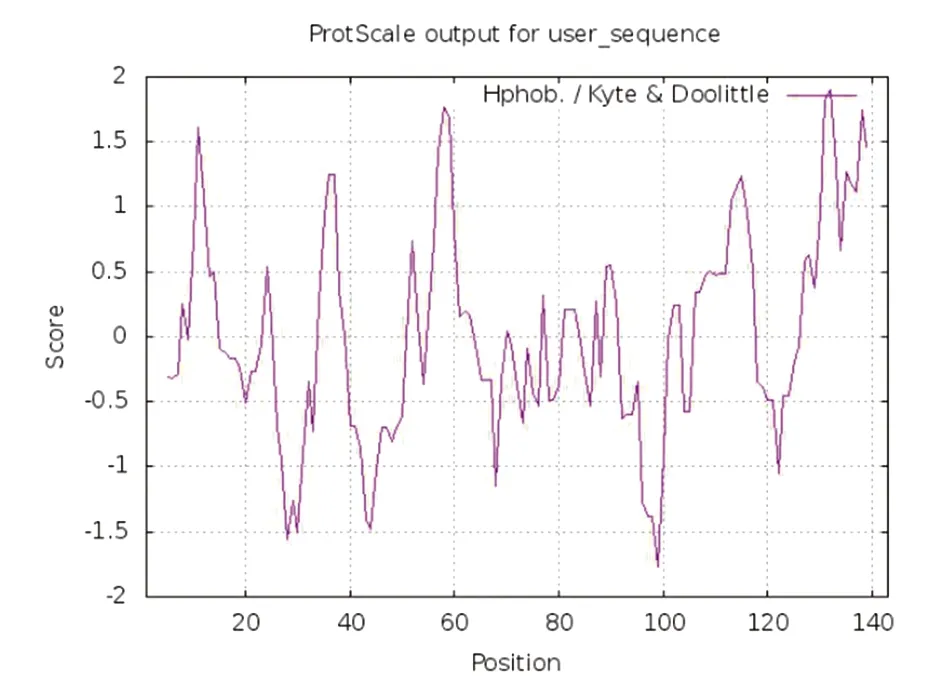
Note:Abscissa for the amino acid position, the vertical axis for the hydrophobic score(pro,hydrophobic,respectively,with negative values,positive values)
2.2 Hrp1蛋白的信号肽、跨膜区、糖基化及磷酸化位点分析 运用信号肽分析系统SOSUI结果显示该蛋白为可溶性蛋白,无信号肽,该蛋白是具有一个跨膜区长度为23个氨基酸序列的跨膜螺旋,N端氨基酸位于第1位,C端位于第16位,跨膜区氨基酸序列为 MTTARDIMNAGVTCVG;Signal IP结果显示该蛋白max.C值为0.131,max.Y值为0.117,max.S值为0.143,mean S值为0.107,其中mean S值若大于0.5,则预测为分泌蛋白,存在信号肽;故可知该蛋白没有信号肽,不是分泌蛋白而是膜蛋白。TMpred分析Hrp1蛋白在17-33、127-143氨基酸之间可能存在跨膜结构,无信号肽(图2)。
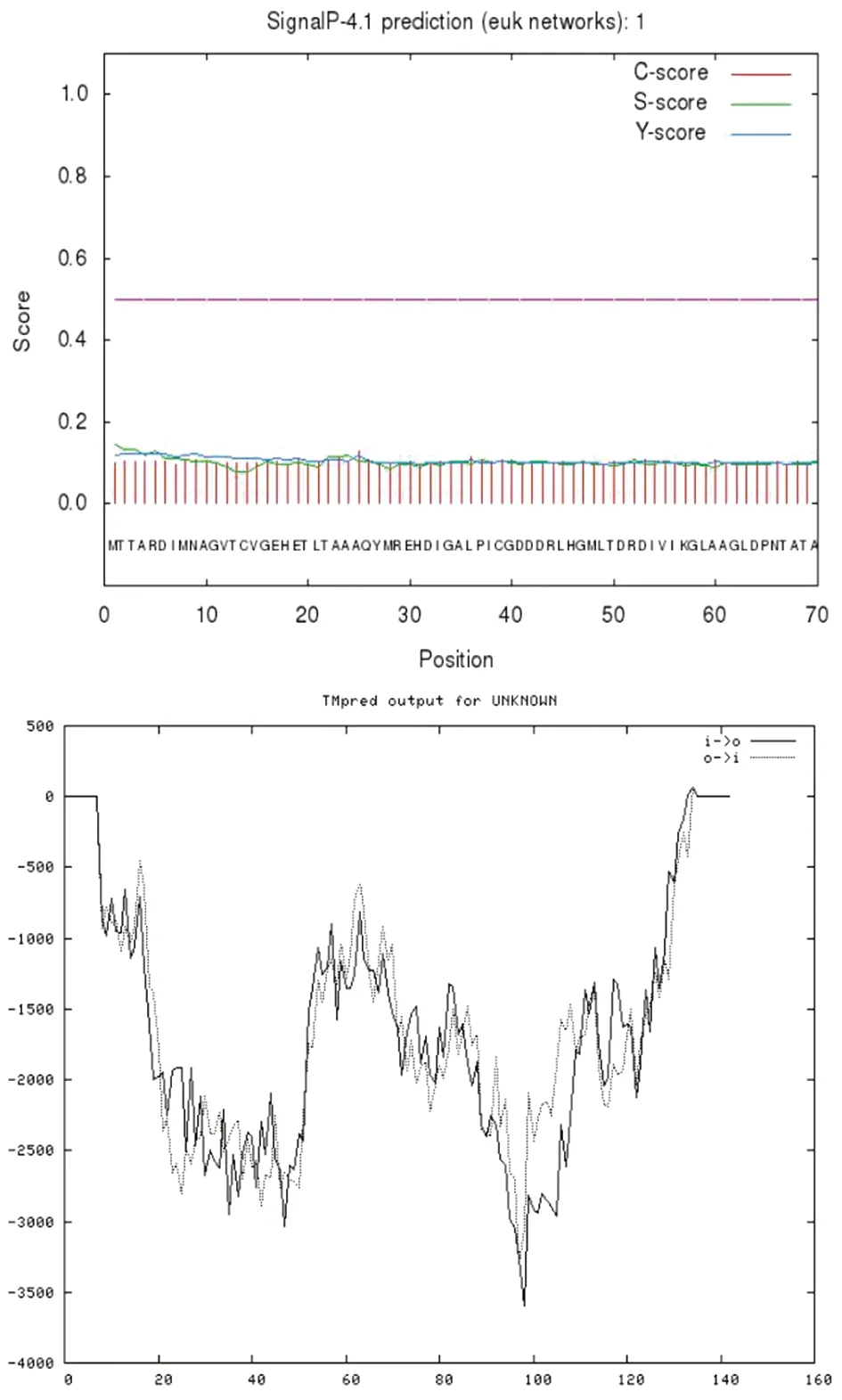
图2 Hrp1蛋白跨膜结构预测(左)与信号肽预测(右)
TMHMM提交蛋白序列的全部氨基酸残基共143个,结果预测分析得到该蛋白(图3)跨膜螺旋氨基酸数为0.007 25(该指标>18提示很有可能为跨膜蛋白),N端前60个氨基酸中跨膜氨基酸数为0.003 25,N端位于膜胞质侧的总概率为0.469 45。综合分析表明Hrp1蛋白位于膜外,为非跨膜蛋白。

The axis of represents the amino acid residue number(1-210)corresponding to the submitted protein sequence.The vertical axis values are the probabilities that each amino acid is inside the membrane, outside the membrane, and the helix region.
在线软件NetNGlyc分析Hrp1的糖基化位点存在于84位(图4);采用NetPhos 3.1 Servera在线软件预测Hrp1蛋白有6个磷酸化位点,其中磷酸化丝氨酸位点有2个,分别位于77、106位氨基酸;磷酸化苏氨酸位点有2个,分别位于3、50位氨基酸;2个磷酸化酪氨酸位点,分别位于79、80位氨基酸(图5)。
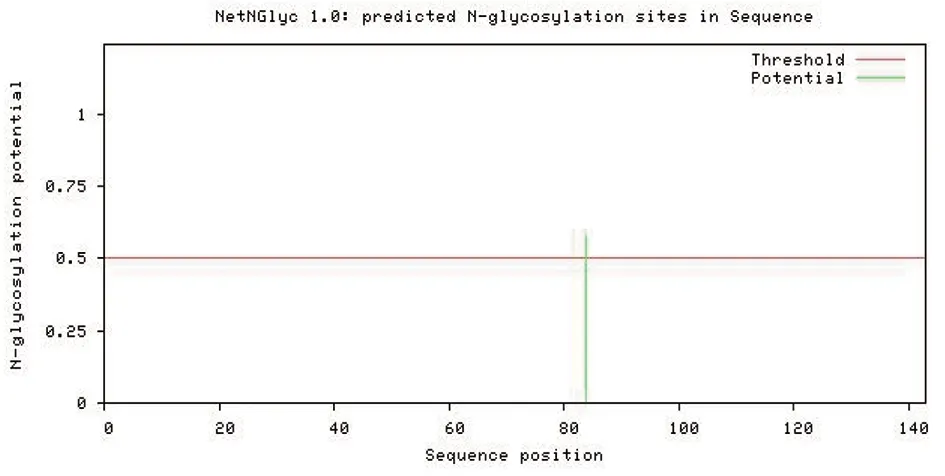
图4 Hrp1蛋白亚基糖基化位点分析

图5 Hrp1蛋白磷酸化位点分析
2.3 Hrp1蛋白的亚细胞定位分析 PSORT结果显示该蛋白其亚细胞定位于细胞质,可靠性为94.1%;在线软件WoLF PSORT分析结果得出Cysk为12, cyto为9.5, mito为4, nucl为1.5,cyto-nucl为6,表明Hrp1蛋白可能定位于细胞质;TargetP-2.0-CBS结果显示该蛋白属于分泌蛋白的可能性为0.000 1,定位在其他位置的可能性为0.999 7;NLStradamus在线软件预测Hrp1蛋白无NLS,可推测此蛋白位于细胞核外。综合4种在线软件分析可得Hrp1蛋白亚细胞定位于细胞质。
2.4 Hrp1蛋白结构预测分析 利用在线分析软件SOPMA预测Hrp1蛋白的二级结构,α-螺旋(Hh)63个,占44.06%;β-转角(Tt)11个,占7.69%;β-折叠(Ee)30个,占20.98%;无规则卷曲(Cc)39个,占27.27%(图6)。该蛋白结构较稳定。

Blue red green and purple stand for alpha helix extended strand beta turn and random coil respectively
利用SWISS-MODEL软件,对Hrp1蛋白的三级结构进行预测分析,选择同源性最高的1xkf.1为模板,基于Hrp1蛋白的X射线分析进行同源三级建模(图7)。其GMQE评分为0.91,QMEAN评分为1.47,手势向上,说明该模型结果可靠,质量较好。
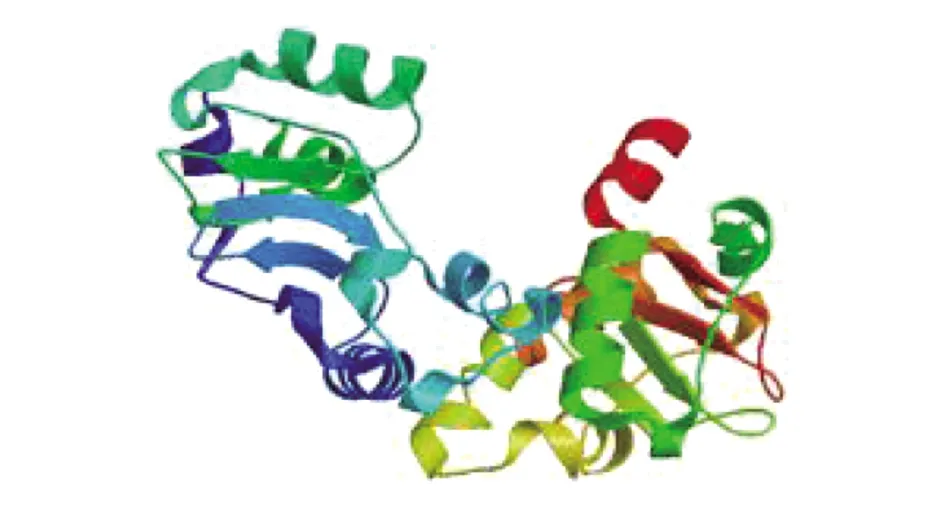
图7 Hrp1蛋白三级结构的预测
2.5 同源性分析结果 BLAST结果得出,Hrp1中氨基酸序列在第11-70位,与人类蛋白质锌指蛋白440(ZNF440)的96-153位氨基酸相似性较高,但二者的相似度仅为25%,因此,Hrp1与人类蛋白相比,同源性较低,交叉反应发生的概率较低,可以作为候选的新型结核疫苗和结核病诊断靶点,该抗原蛋白的表位可以进行生信分析,对结核病诊断与候选表位疫苗有一定的参考研究价值。在研究分析中,即在预测和筛选表位时,应注意避免选择位于11-70的氨基酸序列,以降低交叉反应的概率[6]。
2.6 Hrp1蛋白的B细胞表位、辅助性T细胞表位、细胞毒性T淋巴细胞表位预测
2.6.1 Hrp1抗原蛋白的B细胞表位预测 通过IEDB软件对Hrp1蛋白的B细胞表位进行预测,结果显示Hrp1蛋白的可塑性、表面可及性、线性表位、β-转角、亲疏水性、抗原6个方面(图8),柔韧性越强越有利与抗体结合,柔韧性参数以基线1.0为参照,高于1.0的氨基酸区段柔韧性强,是易形成抗原表位的区域,可及性高于基线1.0的区段易形成B细胞表位,再通过结合线性表位、β-转角、抗原性这3个方面筛选,筛选得出B细胞表位(表1)。

Note: A:Linear Epitope Prediction 2.0 Results; B:Beta-Turn Prediction Result; C:Fexibility Prediction Results; D:Surface Accessibility Prediction Results; E:Hydrophlicity Prediction; F:Antigenicity Results
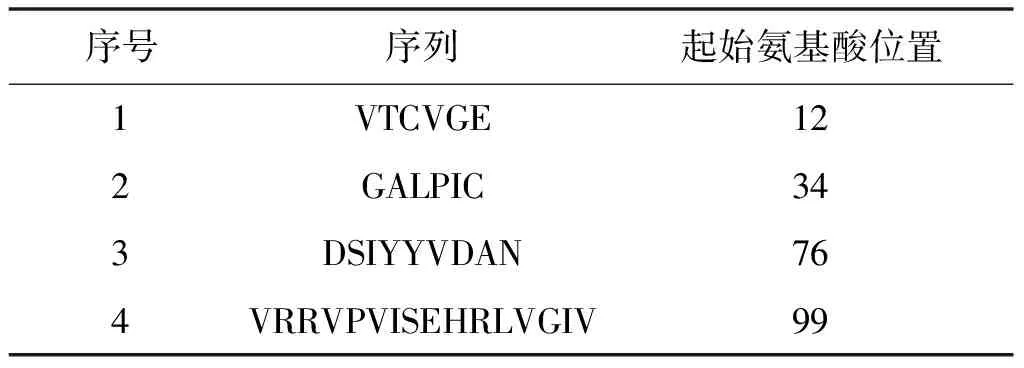
表1 Hrp1蛋白B细胞抗原表位预测结果
2.6.2 预测Hrp1蛋白的辅助性T细胞表位结果 使用辅助性T细胞表位分析软件预测Hrp1蛋白的限制性Th细胞表位,对候选意义的每一种HLA 亚型相应的表位数目情况进行统计,其中HLA-DRB1 * 0401、HLA-DRB1*0701表型的表位数量最多,结果如表2所示;对于某个表型预测分值较高的氨基酸序列进行下一步分析选择,以作为候选Th表位,如表3为最终结果。
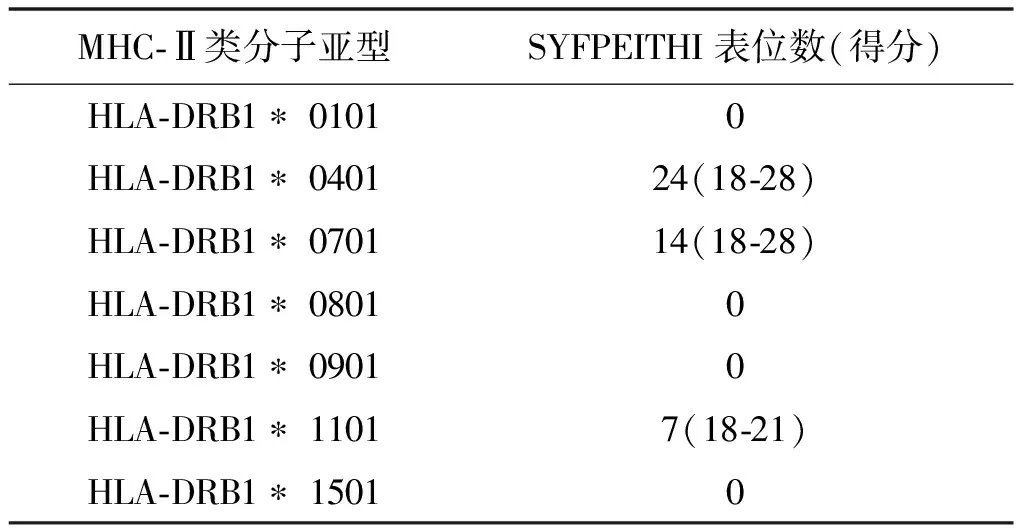
表2 SYFPEITHI软件预测的Hrp1蛋白Th细胞表位信息
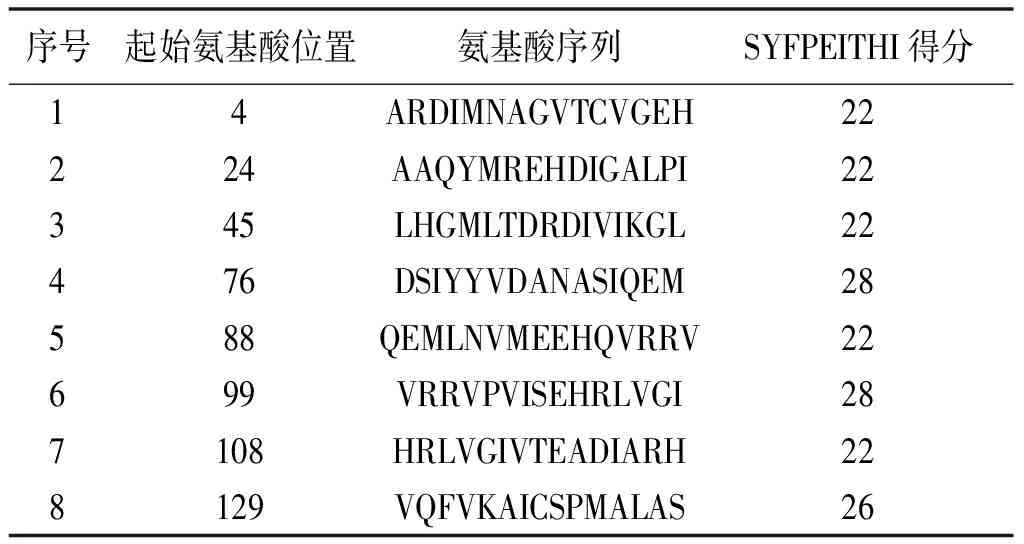
表3 Hrp1蛋白Th细胞表位综合分析
2.6.3 Hrp1蛋白的细胞毒性T淋巴细胞预测分析结果 根据预测软件结果,选择出每一种表型分值较高的多肽序列,其可成为候选CTL表位,具体结果见表4。

表4 Hrp1蛋白CTL表位综合分析
结合Hrp1蛋白的B细胞表位、辅助性T细胞表位、细胞毒性T淋巴细胞表位预测结果,根据DNAStar软件中Protean模板得到关于Hrp1蛋白表位预测的位置图,该图可直观看到Hrp1蛋白表位富集的区域,具体的表位富集表现在红色方块标记的区域(图9)。
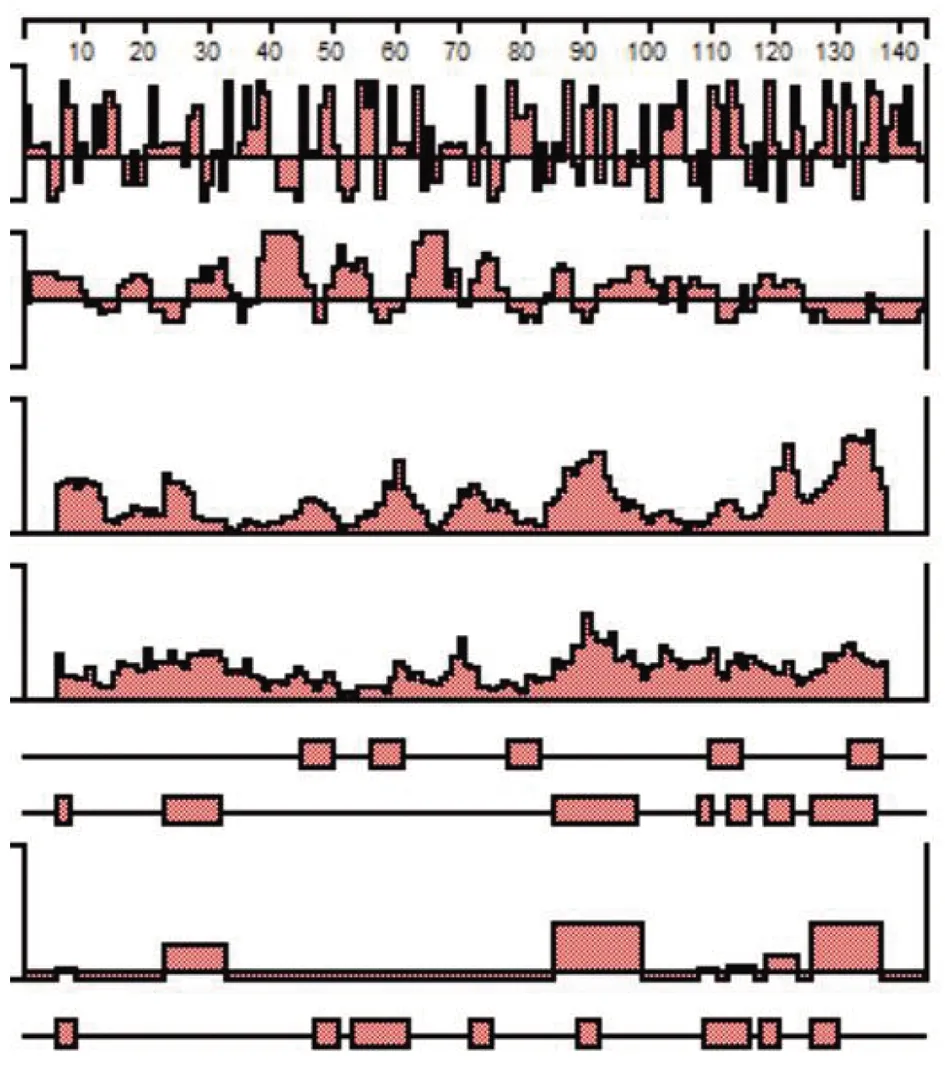
图9 Hrp1蛋白表位预测位置图
2.7 Hrp1蛋白的相互作用网络预测结果 STRING在线分析软件预测了与Hrp1蛋白相互作用的蛋白有Rv2627c、Rv2628、Rv1738、devR、Rv2624c、TB31.7、rip3、pfkB、guaA、hspX(图10)。通过分析Hrp1的相互作用蛋白,可得Hrp1可能在活化的巨噬细胞内产生大量 NO,然后作为细胞间的信号分子杀伤被吞噬的微生物,调节体内细胞一系列重要的功能;该蛋白可能与抑制蛋白质合成有关,刺激巨噬细胞和外周血单个核细胞分泌重要的细胞因子,然后减少细菌生长,该蛋白的相互作用网络主要涉及NF-kB信号通路。

图10 MTB H37Rv Hrp1相互作用蛋白
3 讨 论
目前在临床上唯一预防结核病的疫苗是卡介苗(BCG),但卡介苗基因组发生的众多突变导致相当一部分人群丧失保护效力,其对不同人群的保护效率差异较大,机体保护水平范围从0%~80%不等,且对潜伏感染MTB没有保护效果[7]。在临床上约有90%的结核杆菌感染者,其身体基本上处于健康状态,但是如果机体免疫系统缺乏抵抗力,结核杆菌会重新复制,病灶被打破,随之大量的细菌释放,首先进入肺部,随后至全身,发展为活动性肺结核,故结核病疫情控制工作者一直关注结核潜伏期感染诊断试剂和治疗性疫苗的研究开发。结核杆菌可以在细胞内潜伏数十年,因此需要一个可以诱导具有长期记忆的综合免疫保护应答疫苗,在持续感染人群中,检测到对持续感染期抗原的具有一定的特异性反应,而没有在卡介苗免疫人群检测到,因此猜想以潜伏感染相关蛋白设计相关疫苗,有希望研究出针对持续感染期抗原的保护性免疫反应,这有助于在潜伏感染期间清除MTB的复发。对结核分枝杆菌相关潜伏感染蛋白以生信软件为基础进行结构功能的生物信息学分析,可为结核病潜伏感染致病机制的阐述及新型药物结核疫苗的开发提供一定的研究理论基础,而关于抗原表位的研究也成为最近MTB诊断预防治疗的重点方向。在本文研究中,联合运用生物信息学的技术方法对Hrp1蛋白的相关信息进行分析、处理、模拟和预测,本文预测抗原表位的模式,可提高该蛋白用于表位疫苗的研发及诊断和实验试剂开发的水平,避免耗费大量的人力和物力,对免疫学的发展非常有益,使免疫学数据更加丰富。
缺氧反应蛋白1是由Rv2626c基因编码,Bashir等在研究中发现实验鼠的巨噬细胞表面结合重组Hrp1蛋白后可以引起小鼠体内发生Ⅰ型免疫反应,并且小鼠体内的NO及iNOS分泌量增加,小鼠巨噬细胞B7-1、B7-2和CD40的表达随之增强,经重组Hrp1蛋白刺激后的小鼠巨噬细胞和活动性结核病患者的外周血单个核细胞可分泌高水平的IL-12及TNF-α[8]。Azzurri等应用ELISA方法对结核病患者血清中抗Hrp1抗体水平进行检测,结果发现未经治疗的活动性结核病患者高于结核病密切接触者及健康社区对照人群,结核病患者化疗后抗Hrp1抗体水平下降[9]。Roupie等将Hrp1 DNA疫苗免疫小鼠,发现小鼠IFN-γ和IL-2分泌增加,Hrp1 DNA疫苗可以诱导较强的体液免疫反应及Th1型细胞免疫反应的效力优于其他潜伏感染抗原Rv1733c、Rv1738、Rv2029c、Rv2031c、Rv2023、Rv2627c及Rv2628[10]。由此可知Hrp1具有较好的免疫原性,其通过固有免疫及获得性免疫两种方式来调节巨噬细胞的效应能力,且该抗原蛋白很容易被结核分枝杆菌潜伏患者所识别,多项研究提示其很有可能会成为一种新型诊断MTB潜伏感染的候选抗原和潜伏性感染的候选疫苗。
本研究利用多种工具和生物信息学资源对结核分枝杆菌Hrp1蛋白的性质、结构、功能等进行分析。结核分枝杆菌Hrp1由143个氨基酸组成,该蛋白分子式为C667 H 1084 N194 O211 S10,理论等电点PI为4.96,分子量为15 517.72,不稳定性指数为19.62,为稳定疏水性蛋白。ORF Finder发现Rv2626c基因全长432 bp,含有5个开放阅读框,其中最长的一个开放阅读框显示Rv2626c基因基本被通读,全长432 bp,与预测的编码Hrp1蛋白长度基本相似。综合利用Protpapram、ProtScale、SOSUI工具预测Hrp1蛋白无信号肽,并通过在线生信软件可知该蛋白不属于分泌蛋白,主要定位在结核分枝杆菌的细胞质,在细胞质内发挥功能[11]。其二级结构主要为α-螺旋和无规则卷曲,共占71.33%,生物信息学分析Hrp1蛋白含有6个磷酸化位点,具有良好的抗原优势表位和氨基酸磷酸化位点,推断猜想以该蛋白设计疫苗,有助于在潜伏感染期间清除MTB的复发,是探索结核分枝杆菌持久性感染机制的重要研究靶标。该蛋白无规则卷曲占27.27%,表明其容易与抗体嵌合,并预测Hrp1含有多个T细胞的抗原优势表位。蛋白抗原的二级结构与B细胞表位关系密切[12],对Hrp1二级结构、亲水性、表面可能性、柔韧性和抗原性等进行全面分析,最后结果可知12-18、34-40、76-85、99-114位等4个B细胞表位。目前预测辅助性T细胞表位效果较好且常用的在线生信软件是SYFPEITHI,本研究筛选中国人群常见的MHC-Ⅱ类分子亚型DRB1* 0101、0401、0701、0801、1101、1501[13],综合分析得到的结果对于研究中国人群具有一定的实际意义。经过结果分析发现,本研究对Hrp1抗原蛋白的Th 细胞、CTL 和B细胞抗原表位进行生物信息学预测分析,并应用生物信息学预测分析Hrp1蛋白的结构和功能,发现Hrp1蛋白含有较丰富的B、T细胞抗原表位,为进一步研究该蛋白的功能和明确其在MTB潜伏感染机制中作用提供理论依据,以期为筛选 MTB 诊断标志物和研制新型表位结核疫苗奠定基础[14]。STRING数据库分析显示Hrp1蛋白与Rv2627c、Rv2628、Rv1738、devR、Rv2624c、TB31.7、rip3、pfkB、guaA和hspX有相互作用,该蛋白可能刺激产生NO及细胞因子的过程受转录因子NF-kB调控,NO可通过MTB的休眠调节子启动Rv2626c基因在内的多个下游基因的表达,使MTB进入休眠状态,抵御宿主免疫系统的杀伤。与抑制蛋白质合成有关,还可能会刺激巨噬细胞和外周血单个核细胞分泌重要的细胞因子[15]。
目前对MTB缺氧相关潜伏期抗原蛋白Hrp1的机制尚不十分清晰明确[16],深入研究Hrp1的免疫学特性及结构功能特征,对控制结核潜伏感染、预防结核发病和结核免疫治疗具有一定意义。本研究对 Hrp1蛋白进行了全面的生物信息学分析,采用多种方式在多个角度进行了分析和预测,有效地规避了单一软件预测的缺陷,为进一步研究Hrp1蛋白的结构、功能、理化性质提供参考,为进一步揭示结核分枝杆菌的致病机制奠定理论基础。
利益冲突:无

