sRNA STnc440分子特征及对鼠伤寒沙门菌毒力的影响
李 静,宁程程,李 娜,季春晖,张星星,才学鹏,乔 军,孟庆玲,夏咸柱
(1 石河子大学 动物科技学院,新疆 石河子 832003;2 新疆农垦科学院畜牧兽医研究所,新疆 石河子 832000;3 中国农业科学院 兰州兽医研究所,甘肃 兰州 730046)
鼠伤寒沙门菌(Salmonellatyphimurium,STM)是引起畜禽和人沙门菌病的重要革兰氏阴性致病菌[1]。STM在家畜和野生动物中广泛分布,从不同动物和食物中均可分离得到,主要存在于家禽[2]、猪[3]和牛肉[4]中,被认为是典型的宽宿主范围沙门菌血清型[5]。在我国,STM是一种常见的食源性病原菌,免疫力低下的婴幼儿和老年人易感染该菌而发病[6],会引起肠道黏膜上皮出现严重的炎症,导致严重的胃肠炎[7],给食品安全造成严重的威胁。
小RNAs(small RNAs,sRNAs)是一类长度40~500 nt、可执行多种功能的非编码RNA(non-coding-RNAs,ncRNAs)[8]。近年来,人们已在沙门菌中发现并鉴定了百余种具有调控功能的非编码sRNAs[9]。现有的研究发现,一些sRNA可与靶基因mRNA结合,通过转录后水平调节细菌靶基因的表达,以增强细菌在不利环境中的适应性[10]。STM进入宿主细胞后通过调节因子以增加自身毒力和对环境的耐受性,然后与细胞粘附并通过内化作用进入宿主细胞而在胞内长时间生存,这是许多因子相互作用的结果,这些因子中就包含有sRNA[11]。
研究表明,在STM侵染巨噬细胞过程中,sRNASTnc440的表达显著上调[12],然而对STnc440的生物学功能至今尚不清楚。为此,本研究对sRNASTnc440进行了克隆与分子特征分析,并构建其STM缺失株和回补株,利用细胞侵染和小鼠感染试验对缺失株细胞粘附力、侵袭力及小鼠毒力、肝脏和脾脏载菌量进行检测,旨在揭示sRNASTnc440的生物学功能,探讨其对STM毒力的调控作用,为进一步阐明sRNASTnc440对STM毒力调控的分子机制提供科学依据。
1 材料与方法
1.1 试剂、菌株、质粒和细胞
质粒小提试剂盒,购自美国OMEGA Bio-Tek;DL2000 DNA Marker和DL5000 DNA Marker,购自北京全式金生物技术有限公司;DMEM培养基和胰酶,购自Gibco;限制性内切酶BamH Ⅰ和Hind Ⅲ,购自宝生物工程(大连)有限公司;电击杯,购自Bio-Rad公司;SL1344鼠伤寒沙门菌、大肠杆菌DH5α、RAW264.7细胞和pBR322质粒,均由新疆石河子大学预防兽医学实验室保存;质粒pKD46、pKD3和pCP20,由江苏扬州大学兽医学院提供;pMD19-T质粒,购自宝生物工程(大连)有限公司;6周龄BALB/c小鼠,购自郑州医科大学。
1.2 引物设计与合成
根据GenBank中已发布的STM SL1344基因组序列(GenBank登录号:FQ312003.1),设计用于sRNASTnc440扩增及缺失验证的特异性引物F1/R1(F1:5′-GATTGTTTGATTCGTTGATT-3′,R1:5′-GGCATTCGGTAAGTGTTG-3′),用于扩增sRNASTnc440同源片段的特异性引物F2/R2(F2:5′-TGTTTGATTCGTTGATTCATTGTTG-GGGATATTTATGTTTTACTTACCTCGTGTA-GGCTGGAGCTGCTTC-3′,R2:5′-ACAGAATAA-TATTCACTGTCTGTTAATTATTACAGAGAG-AGTTAATTTATATGAATATCCTCCTTAGTT-3′(含同源臂和氯霉素引物),以及用于扩增sRNASTnc440片段的特异性引物F3/R3(F3:5′-AAGCTTGATTGTTTGATTCGTTGATT-3′(下划线为Hind Ⅲ位点)),R3:5′-GGATCCGGCATTCGGTAAGTGTTG-3′(下划线为BamH Ⅰ位点))。引物均由北京华大基因研究中心有限公司合成。
1.3 STM STnc440基因的克隆与分子特征分析
以SL1344基因组为模板,以F1/R1为引物,PCR扩增STnc440基因。PCR反应体系为20 μL: 2×PCR Mix 10 μL, ddH2O 7 μL,模板2 μL,上、下游引物各0.5 μL。阴性对照的模板更换为同等体积ddH2O。反应条件为:94 ℃预变性 5 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 45 s,35个循环;72 ℃延伸10 min。PCR产物用2%琼脂糖凝胶进行电泳回收,并与pMD19-T载体连接,转化入大肠杆菌DH5α感受态细胞中,对菌液进行PCR,筛选阳性克隆。将筛选的阳性克隆于37 ℃扩大培养后,送至北京华大基因研究中心有限公司测序。对测序结果进行启动子序列和转录因子预测分析,并构建基因遗传进化树。
1.4 STM STnc440缺失株和回补株的构建与鉴定
1.4.1 缺失株 通过λ-Red重组酶的方法[13]构建STMSTnc440缺失株。以pKD3质粒为模板,用F2/R2引物扩增STnc440基因同源左右臂序列,PCR反应体系同1.3节,阴性对照模板更换为同等体积ddH2O。反应条件为:94 ℃预变性 5 min;95 ℃ 30 s,57 ℃ 30 s,72 ℃ 45 s,35个循环;72 ℃延伸10 min。扩增产物用1.5%琼脂糖凝胶进行电泳检测。
制作SL1344感受态细胞,将pKD46质粒电转化入SL1344感受态细胞中,然后将扩增片段电转化入经阿拉伯糖诱导的含有pKD46质粒的SL1344感受态细胞中,氯霉素抗性基因cat与STnc440基因发生同源重组,cat替换了靶基因STnc440,以F1/R1为引物,PCR反应体系同1.3节,阴性对照模板更换为同等体积ddH2O,SL1344基因组为模板作为对照(亲本处理组),对重组子进行PCR和测序验证,验证正确的重组子命名为△STnc440::cat。将质粒pCP20电转化入△STnc440::cat中,进行第2次重组,消除cat,以F1/R1为引物对重组子进行PCR和测序验证,验证正确的重组子即为STMSTnc440缺失株,命名为△STnc440。
1.4.2 回补株 用F3/R3引物扩增STnc440基因序列,PCR反应体系同1.3节。反应条件为:94 ℃预变性 5 min;95 ℃ 30 s,58℃ 30 s,72 ℃ 45 s,35个循环;72 ℃延伸10 min。扩增产物经1.5%琼脂糖凝胶电泳检测后,纯化回收目的片段,将其与PBR322载体相连,构建质粒PBR322-STnc440。将质粒PBR322-STnc440转化大肠杆菌感受态细胞DH5α,涂布在有氨苄抗性(氨苄青霉素100 μg/mL)的平板上,挑取单菌落培养过夜,提取转化子的质粒PBR322-STnc440进行BamH Ⅰ和Hind Ⅲ双酶切验证。将验证正确的PBR322-STnc440质粒电转化入△STnc440株感受态细胞中,构建STnc440回补株△STnc440/STnc440,以F1/R1为引物对其进行PCR鉴定,设置阴性对照和SL1344亲本处理组,PCR产物用1%琼脂糖凝胶进行电泳检测。
1.5 STM △STnc440和△STnc440/STnc440株生长曲线及侵染能力检测
挑取验证正确的STM SL1344、△STnc440和△STnc440/STnc440单菌落分别接种于液体LB中,37 ℃ 180 r/min振荡培养过夜,第2天调整菌液在600 nm处的光密度值(OD600)为0.1,将菌液按照1∶100的比例(体积比)接种于LB液体培养基中,每小时测定1次OD600值,每次重复测定3个平行,连续测定14 h,绘制生长曲线。
用含体积分数10%新生牛血清的DMEM培养基复苏培养RAW264.7细胞,待细胞密度为2×105mL-1时,分别用STM SL1344、△STnc440和△STnc440/STnc440菌液(细菌密度为2×106CFU/mL)在37 ℃下按照感染复数(multiplicity of infection,MOI)为10感染细胞1 h,重复3次。吸弃多余菌液,细胞用无菌PBS洗涤后,加入体积分数0.5%的Triton-X100,裂解细胞后2 000 r/min离心5 min,弃上清,取1 mL无菌PBS重悬,选择10-2、10-3和10-4梯度稀释涂板,进行细菌计数。计算细菌粘附率:细菌粘附率=粘附细菌数/每孔中加入的细菌总数×100%。
侵袭力测定试验方法基本同上,细菌与细胞37 ℃作用1 h,无菌PBS洗涤,然后加入含有庆大霉素(100 μg/mL)的培养基于37 ℃作用1 h,杀死胞外细菌。裂解细胞于2 000 r/min离心3 min,相同梯度(10-2~10-4)稀释并涂板计数。计算细菌侵袭率:细菌侵袭率=细胞内细菌数/每孔加入的细菌总数×100%。
1.6 STM △STnc440和△STnc440/STnc440株的致病性检测
将STM SL1344、△STnc440和△STnc440/STnc440株,分别以2×103,2×104,2×105,2×106,2×107,2×108和2×109CFU/只 的剂量感染6周龄BALB/c小鼠,以PBS代替STM菌液为对照,重复3次,连续10 d监测小鼠存活情况,统计存活率,并采用改良寇氏法计算小鼠半数致死量(median lethal dose,LD50)。以2×104CFU/只 的剂量,用STM SL1344、△STnc440和△STnc440/STnc440株分别感染6周龄BALB/c小鼠,重复3次,参照文献[14]的方法,在感染1,3,5,7和9 d后分别收集小鼠的肝脏和脾脏,称质量,在PBS中匀浆后连续稀释涂板,统计肝脏和脾脏的载菌量(CFU/g)。
1.7 STM STnc440调控靶基因的分析
用sRNA靶点预测网站http://cs.wellesley.edu/~btjaden/TargetRNA2/userguide.html[15]对sRNASTnc440下游调控靶点进行预测分析,并根据预测结果进一步分析STnc440的调控靶基因。
2 结果与分析
2.1 STM sRNA STnc440基因的克隆与分子特征分析
用F1/R1引物扩增获得了长度约为269 bp的片段,与预期结果相符(图1)。测序分析结果(图2)显示,F1/R1引物扩增产物的长度为269 bp, 其中sRNASTnc440的序列长度为81 bp,在基因组中位于SL1344_4247和SL1344_4248基因之间;在sRNASTnc440基因上游5′-UTR含有-10 box和-35 box启动子区域,有转录调节因子rpoD16结合位点。由于rpoD16是促进RNA聚合酶附着于特定起始位点的因子,推测在细菌侵染细胞时,因rpoD16的特异性结合促进了STnc440的转录。
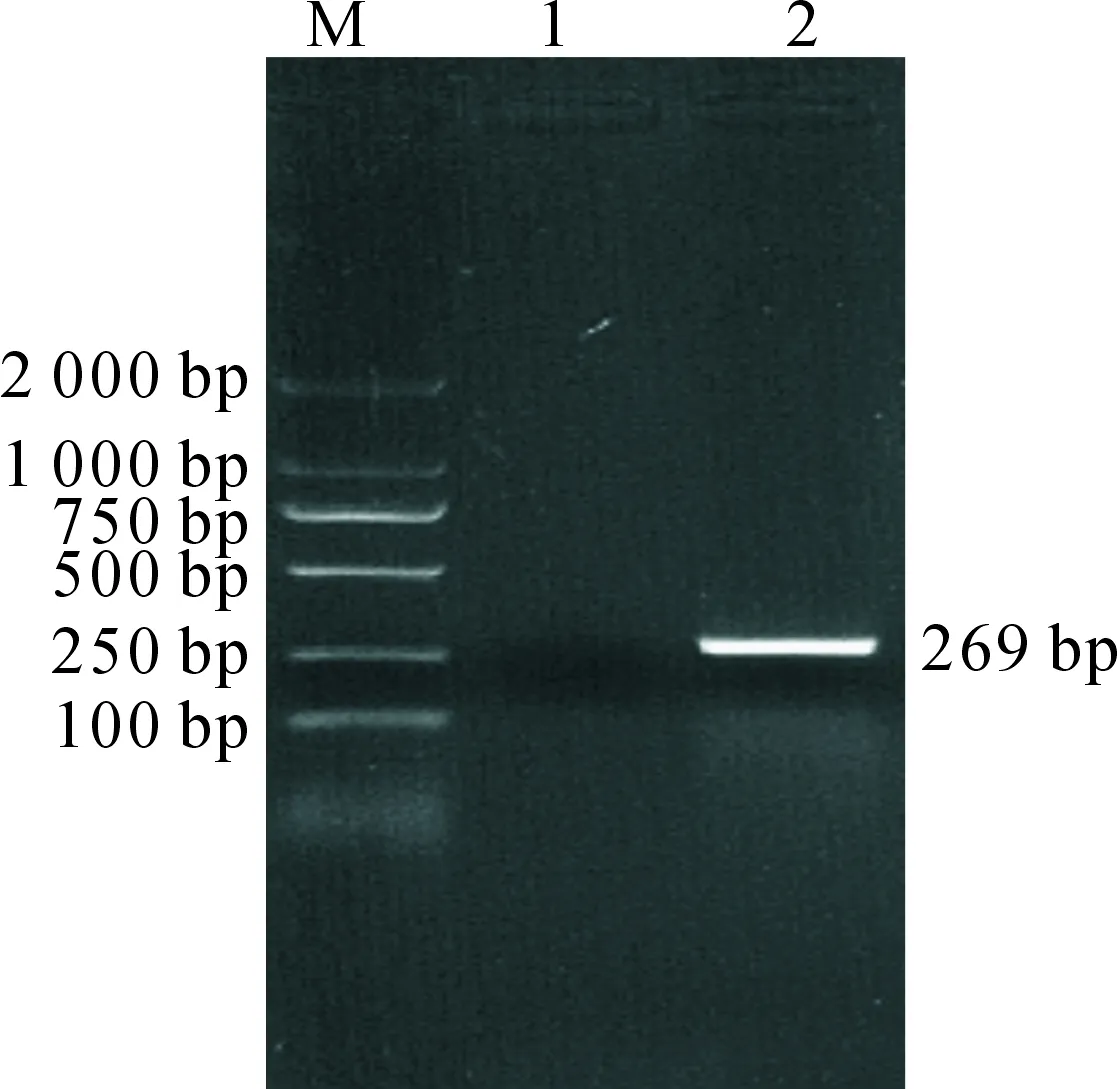
M.DL2000;1.阴性对照;2.sRNA STnc440基因M.DL2000;1.Negative control;2.sRNA STnc440 gene
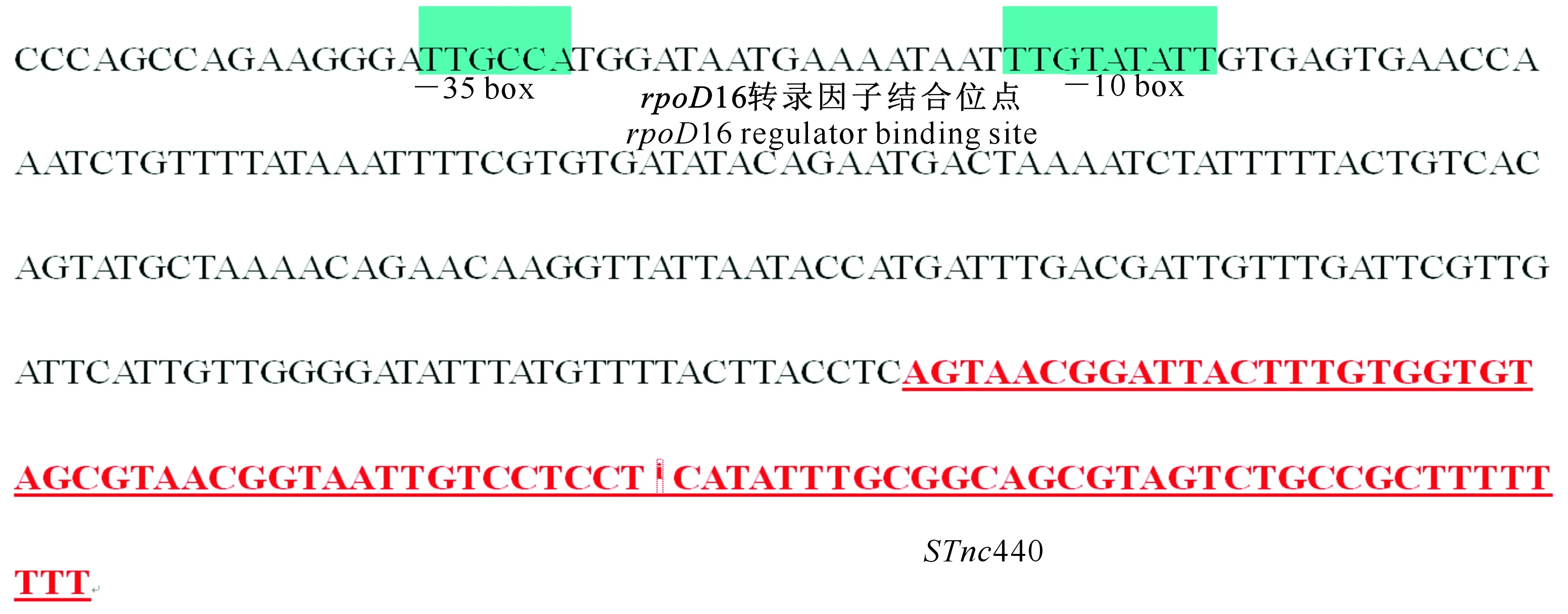
图2 STM sRNA STnc440基因的测序分析Fig.2 Sequencing analysis of STM sRNA STnc440 gene
2.2 STM sRNA STnc440基因序列的遗传进化分析
sRNASTnc440基因与其他肠道亚型沙门菌基因的同源性高达100%。基因遗传进化树分析结果(图3)表明,sRNASTnc440在沙门菌肠道亚种中高度保守。
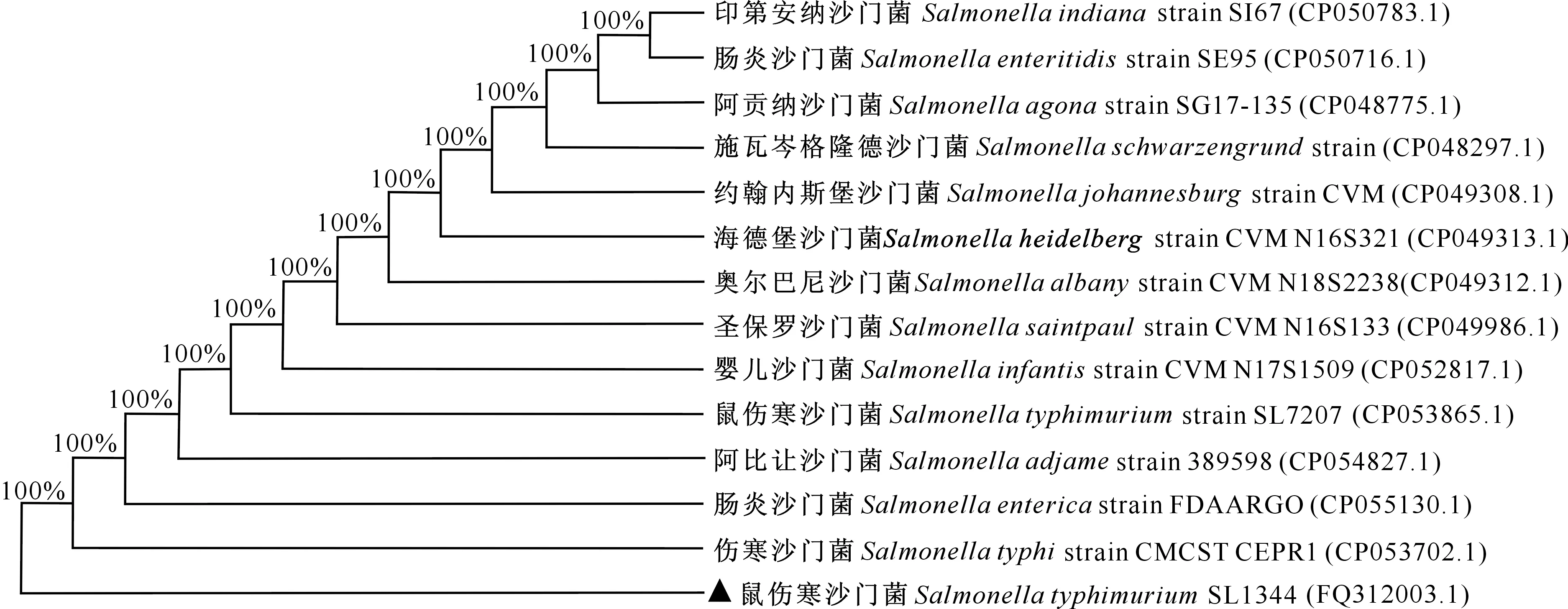
图3 STM sRNA STnc440的遗传进化分析Fig.3 Genetic evolution analysis of STM sRNA STnc440
2.3 STM sRNA STnc440缺失株和回补株的构建与鉴定
PCR扩增sRNASTnc440基因同源左右臂序列,扩增片段长度为1 114 bp(图4-A),与预期结果相符,测序验证结果正确。经pBR322-STnc440双酶切鉴定,可得到4 363和279 bp的酶切条带(图4-B),与预期结果相符。以F1/R1为引物进行PCR验证, SL1344STnc440、△STnc440::cat、△STnc440、△STnc440/STnc440的扩增产物大小分别为269,1 202,266和269 bp(图4-C),均与预期大小相符,测序验证结果均正确。△STnc440基因组的PCR验证表明,sRNASTnc440基因成功缺失。
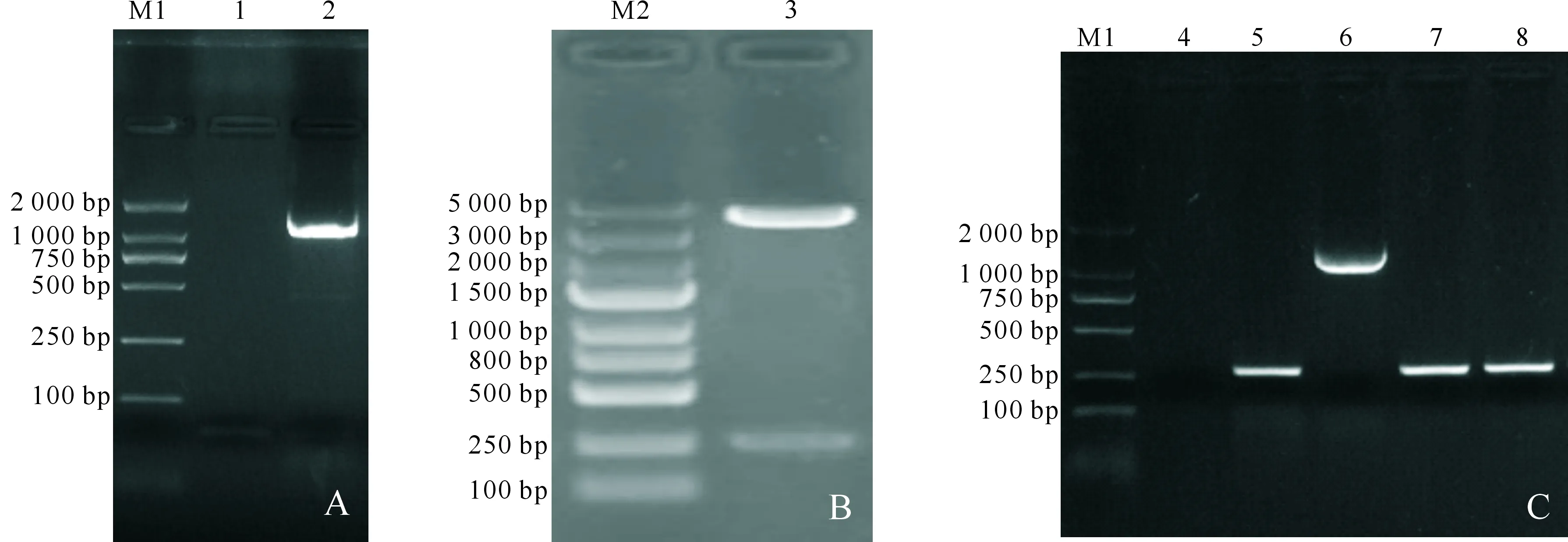
A.sRNA STnc440同源片段的PCR扩增,M1.DNA Marker DL2000,1.阴性对照,2.sRNA STnc440同源片段;B.sRNA STnc440双酶切鉴定,M2.DNA Marker DL5000,3.PBR322-STnc440双酶切产物;C.△STnc440和△STnc440/STnc440株的鉴定,4.阴性对照,5.SL1344 STnc440扩增产物,6.△STnc440::cat重组子扩增产物,7.△STnc440 STnc440扩增产物,8.△STnc440/STnc440 STnc440扩增产物A.PCR amplification of homologous fragments of sRNA STnc440,M1.DNA Marker DL2000,1.Negative control,2.sRNA STnc440 homologous fragment;B.sRNA STnc440 double restriction digestion identification,M2.DNA Marker DL5000,3.PBR322-STnc440 double digestion;C.Identification of △STnc440 and △STnc440/STnc440 strains,4.Negative control,5.SL1344 STnc440 amplification product,6.△STnc440::cat recombinant amplification product,7.△STnc440 STnc440 amplification product,8.△STnc440/STnc440 STnc440 amplification product
2.4 STM sRNA STnc440缺失株和回补株生长曲线及侵染能力测定
由图5的生长曲线可知,与SL1344相比,△STnc440和△STnc440/STnc440在对数期(2~7 h)和稳定期(8~14 h)生长速率无明显差异,表明STM sRNASTnc440基因不影响STM的生长特性。
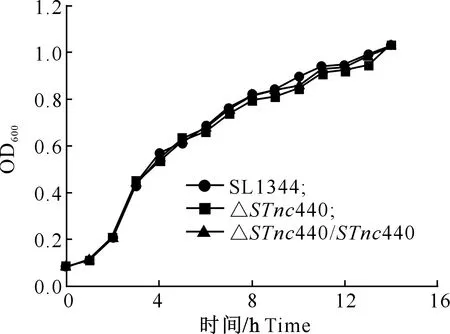
图5 STM SL1344、△STnc440和△STnc440/STnc440的生长曲线Fig.5 Growth curves of STM SL1344,△STnc440 and △STnc440/STnc440
细胞侵染试验测定结果(图6)表明,△STnc440对RAW264.7细胞的粘附率(图6-A)显著低于亲本株,侵袭率(图6-B)低于亲本株(P<0.05),但差异不显著。而回补株△STnc440/STnc440的粘附率和侵袭率均有所恢复,与亲本已无显著差异,提示STM sRNASTnc440在STM粘附和侵染细胞过程中发挥了调控作用。
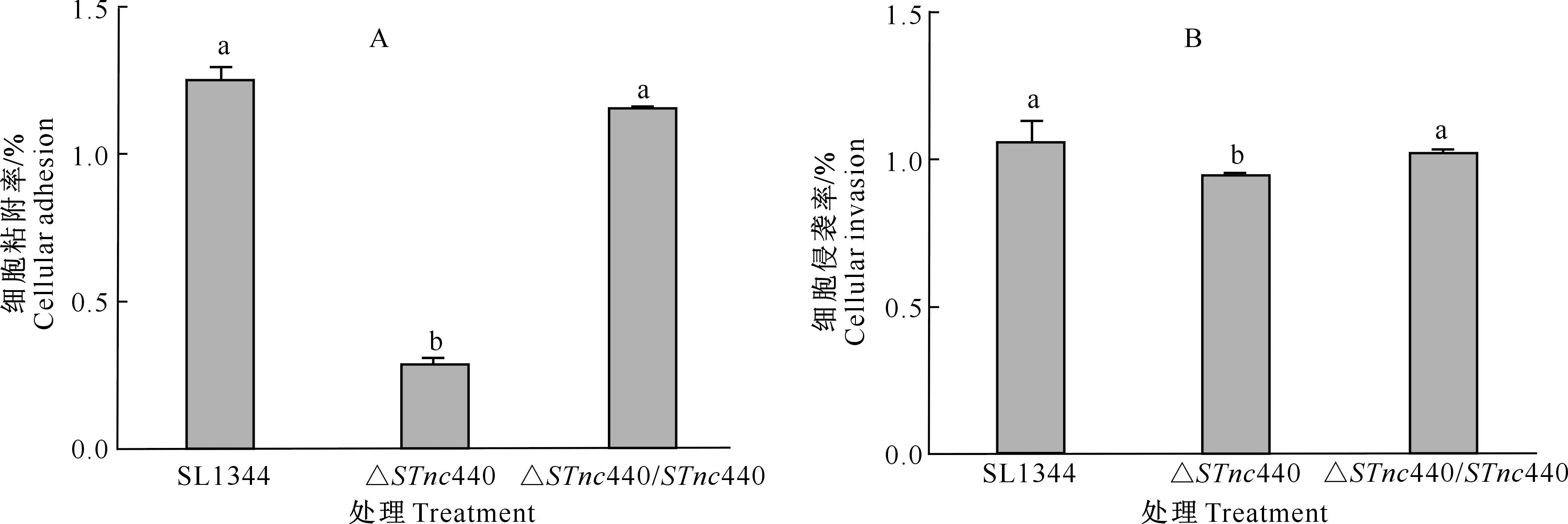
图柱上标不同小写字母表示处理间差异显著(P<0.05)Different lowercase letters indicate significant differences between treatments (P<0.05)
2.5 STM △STnc440和△STnc440/STnc440株对小鼠的致病性
小鼠存活率分析结果(表1)显示,与SL1344和△STnc440/STnc440组相比,△STnc440组小鼠的存活率从第3天开始显著升高,表明△STnc440的致病性显著减弱;SL1344、△STnc440和△STnc440/STnc440组的小鼠LD50分别为5.92×104,1.87×105和8.75×104CFU/mL,表明△STnc440毒力低于亲本株,提示sRNASTnc440对STM毒力有重要调控作用。
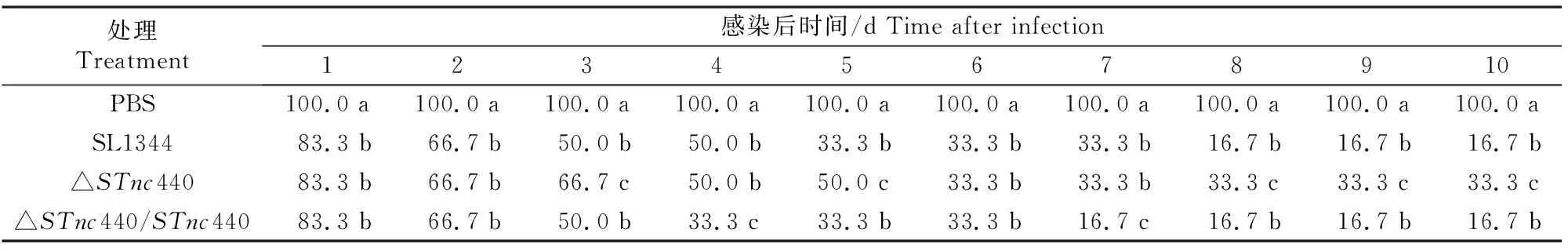
表1 小鼠感染后存活率的变化Table 1 Changes of survival rate of mice after infection %
由图7可知,与SL1344和△STnc440/STnc440组相比,在感染后第3,5和7天,△STnc440组小鼠肝脏(图7-A)和脾脏载菌量(图7-B)均显著降低(P<0.05),提示sRNASTnc440对STM在宿主体内的生存增殖具有调控作用。
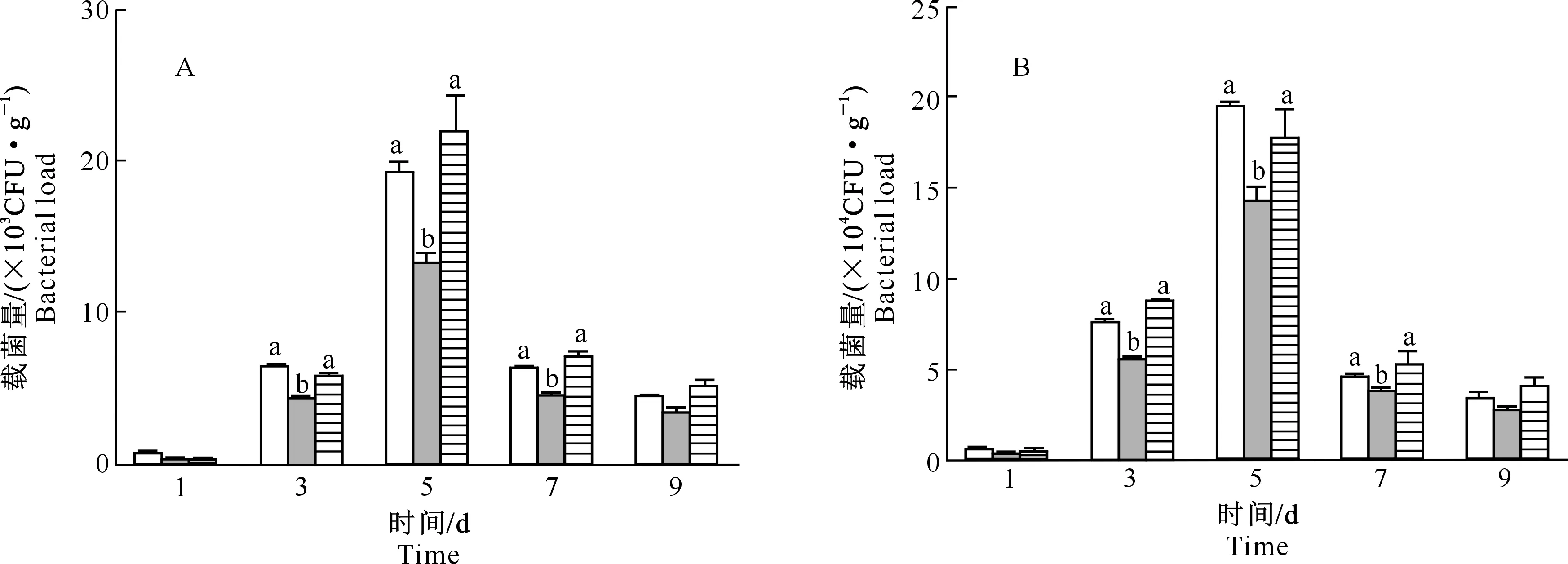
图柱上标不同小写字母表示同一时间不同处理相比差异显著(P<0.05)Different lowercase letters indicate significant differences(P<0.05)
2.6 STM sRNA STnc440调控靶基因的预测分析
用Mfold(http://unafold.rna.albany.edu)进行sRNASTnc440的二级结构预测,sRNA靶点网站预测结果(图8)显示,SL1344_2880(Ⅰ型毒力岛蛋白)为sRNASTnc440调控的靶基因,与SL1344_2880 5′-UTR -44~-59位(图8-A)互补配对的sRNASTnc440的60~63和66~74碱基位于RNA二级结构的茎-环上(图8-B),推测sRNASTnc440通过调控SL1344_2880 Ⅰ型毒力岛蛋白进而调控STM的毒力。

A.sRNA STnc440与SL1344_2880的互补配对位置;B.sRNA STnc440基因的二级结构A.sRNA STnc440 and SL1344_2880 complementary pairing position;B.sRNA STnc440 gene secondary structure
3 讨 论
尽管细菌的ncRNAs对靶基因有多种多样的调控方式,但目前人们一般认为,sRNAs主要通过转录后水平对靶基因的表达进行调控[16]。sRNAs与靶基因的5′-UTR结合可阻碍核糖体结合位点,导致翻译被抑制[17],或者阻止形成抑制性的二级结构,从而激活翻译[18];同时也能与靶基因直接互补配对影响核糖体结合、翻译、终止或总核糖核酸的稳定性[19]。此外,sRNA还可以与调节蛋白相互作用,阻断调节蛋白与靶mRNA的结合,直接影响蛋白的翻译[20]。近年来,许多学者对STM 4/74株的sRNAs开展了研究工作,已检测到约280个在各种条件下表达的sRNAs[21]。分子特征分析发现,STnc440为一种顺式编码sRNA,并且在沙门菌肠道亚种中高度保守,该sRNA的表达调控可能受到-10 box、-35 box和其他转录因子的调控[22]。rpoD16是sigma70 RNA聚合酶基因,该基因是促进RNA聚合酶附着于特定起始位点的转录因子[23]。因此预测表明,STnc440转录过程中可能受到rpoD16转录因子的调控。
2000年,Julio等[24]报道了一种与沙门菌毒力有关的sRNA。Shabarinath等[12]通过对STM感染小鼠巨噬细胞转录组的研究发现,有31个sRNAs的表达显著上调。部分sRNAs在体外生长过程中表达水平非常低,但在巨噬细胞中表达水平显著上调,提示这些sRNAs可能与STM的毒力及其胞内生存相关[9]。目前,虽已在STM SL1344株的基因组中发现并鉴定了280种sRNAs[25],然而大部分STM sRNAs的生物学功能及其致病性尚不清楚。Shabarinath等[12]研究发现,sRNASTnc440在STM侵染细胞后表达显著上调,但目前对STnc440在STM感染细胞中的生物学功能仍缺乏深入研究。本研究通过λ-Red重组技术成功构建了缺失株及回补株,并且比较了缺失株和回补株对细胞的侵染能力及对小鼠的致病性,结果证实,△STnc440对巨噬细胞的侵染能力显著减弱,而回补株的侵染能力明显恢复;缺失STnc440基因后其对小鼠的致病性显著减弱,提示STnc440对STM毒力发挥着重要的调控作用。
目前,关于sRNA对靶基因调控作用的研究均是通过构建缺失株进行表型分析,然后利用在线软件预测可能调控的靶基因,最后通过试验验证sRNA对靶基因mRNA的调控作用[15]。本研究通过生物信息学预测分析发现,STnc440的60~63和66~74位碱基与SL1344_2880 Ⅰ型毒力岛蛋白5′-UTR -44~-59位碱基序列可互补配对,提示STnc440可能通过调控SL1344_2880 Ⅰ型毒力岛蛋白而调控STM的毒力。综上所述,本研究结果为深入揭示sRNASTnc440在调控STM毒力中的分子机制奠定了基础。

