基于KASP标记的葡萄品种鉴定
王富强,张建,温常龙,樊秀彩,张颖,孙磊,刘崇怀,姜建福
基于KASP标记的葡萄品种鉴定
王富强1,张建2,温常龙2,樊秀彩1,张颖1,孙磊1,刘崇怀1,姜建福1
1中国农业科学院郑州果树研究所,郑州 450009;2北京市农林科学院北京蔬菜研究中心,北京 100097
【】开发一组能够区分中国主栽葡萄品种的竞争性等位基因特异性PCR(KASP)分子标记,为中国葡萄品种保护、品种登记和市场维权等提供技术支撑。基于前人筛选的60个葡萄高多态性SNP位点,转化为KASP标记。分别使用23份代表性品种和76份主要栽培品种对转化成功的KASP标记进行初筛、复筛、验证,获得一组高质量的KASP标记,并用于构建76份葡萄品种的DNA指纹图谱。60个SNP位点有3个不具备基因组特异性,6个无法设计KASP-PCR引物,最终51个SNP成功转化为KASP标记,转化率达到89.47%。利用LGC-SNPline平台对23份代表性品种进行基因分型,51个KASP标记全部成功分型。基于次要等位基因频率(MAF)大于0.25、多态性信息含量(PIC)大于0.35、缺失率小于0.2、杂合率小于0.6等参数,初步筛选出27个优质KASP标记。使用构建指纹图谱的76份品种复筛出22个高质量KASP标记。22个KASP标记的缺失率均小于0.12,PIC均大于0.3,杂合率在0.4—0.6的标记占77.27%,MAF大于0.3的标记占95.45%。此外,利用这22个标记对同一品种不同树体提取的23份代表性品种的DNA扩增检测,前后两次检测分型结果稳定一致,表明这22个标记具有较好的重复性和稳定性。进一步将基于22个标记获得的76份葡萄品种分型结果转化为二元编码数据,得到76份葡萄品种的指纹图谱。经邻接聚类分析和群体结果分析,可将76份葡萄品种分为3类,并能正确区分二倍体和多倍体。仅用10个标记(VIT_15_18567587、VIT_6_4258638、VIT_8_3320936、VIT_12_22228357、VIT_11_19390306、VIT_16_17950801、VIT_18_11138668、VIT_12_739916、VIT_16_13454358、VIT_16_21202286)就能区分70份品种,其中有54份品种达到品种鉴定的标准(差异位点数≥2)。从60个葡萄SNP中成功转化51个KASP标记,并筛选出22个高质量KASP标记,构建了76份葡萄品种的SNP指纹图谱,首次验证了KASP技术在我国葡萄品种鉴定中的可行性。
葡萄;KASP标记;品种鉴定;指纹图谱;遗传多样性分析
0 引言
【研究意义】葡萄为葡萄科(Vitaceae)葡萄属(L.)多年生藤本植物[1],全世界至少有23 000个品种(http://www.vivc.de/),其适应性强,适栽范围广。不同地区间葡萄品种交流频繁,而部分引种或地方品种又没有确切的科学名称,难免发生同物异名或同名异物现象[2]。另外,当前葡萄苗木市场管理暂不完善,随意更改品种名称、炒作品种等不良现象时有发生,极大损害了育种家的利益,不利于葡萄产业的健康、可持续发展。因此,寻找一种经济、高效、准确的品种鉴别方法,对于提高我国葡萄种质资源管理效率和品种保护能力均具有重要意义。【前人研究进展】DNA分子标记具有周期短、受环境影响小、可实现高通量检测等优点,为葡萄品种鉴定提供了新的手段。其中SSR(simple sequence repeat)和SNP(single nucleotide polymorphism)分子标记已被国际植物新品种权保护联盟(UPOV)BMT分子测试指南[3]和国内《植物品种鉴定DNA指纹方法总则》(NY/T 2594-2016)[4]定为优先推荐的两种标记方法。目前,国内已研发出一套用于中国葡萄品种鉴定的30个SSR标记体系[5],并利用其中8个引物区分国内290份葡萄品种[6]。但因SSR标记数量和检测通量有限、检测成本偏高、数据读取费时耗力等因素[7],限制了更大范围的葡萄品种鉴定工作。SNP分子标记作为最新一代标记,具有数量多、具二态性、稳定遗传等优点[8],加上各种SNP高通量检测平台的出现,可以很好弥补SSR标记的技术缺陷[9],已在生物学、农业、医学和生物进化等领域得到了广泛应用[10]。其中,高通量SNP检测技术主要有基因芯片和LGC公司推出的KASP(kompetitive allele specific PCR)技术,适用于不同检测需求[9]。基因芯片技术主要适用于少样本多位点检测,如我国自主研发的Wheat 660K[11]、Rice SNP50[12]、Maize 50K[13]基因芯片,单次位点检测量达上万个,不足之处是当前使用成本偏高,开发难度较大;KASP技术则适于多样本、少位点的检测,具有高效、灵活、准确、低成本的特点[14],国内外研究者已利用KASP技术分别构建小麦[15-16]、玉米[17]、水稻[10]、棉花[18]、甘蓝[19]、黄瓜[20]等作物的核心标记体系,为相应作物的品种鉴定和品种保护等提供了便利。而SNP标记在葡萄品种鉴定中,主要使用成本较高的基因芯片和测序技术,如Cabezas等[21]已构建了针对欧亚种葡萄的SNP鉴定体系,并确定了48个SNP为鉴定品种的标准位点;LAUCOU等[22]基于18K SNPs葡萄基因芯片从945份材料中筛选出14个高多态性SNP标记用于欧洲地区栽培品种鉴定。【本研究切入点】目前国际上筛选出的SNP标记仅在二倍体欧亚种中得到应用,而我国主栽的葡萄多为多倍体品种,因此有必要开发适用范围更广的SNP标记。虽然李贝贝等[23]和LIANG等[24]利用不同重测序技术获得上万个SNP位点,分别对304份和472份葡萄种质进行了遗传多样性分析,但没有筛选出用最少的SNP位点组合来鉴别葡萄品种的具体结果。在实际的葡萄品种鉴定工作中,研究者希望用最少的位点区分更多的样本,以达到高效、准确、廉价的鉴定目的,比较而言,KASP技术更适合SNP分子标记开发。【拟解决的关键问题】本研究基于前人开发的葡萄SNP标记,以国内主栽葡萄品种、新育成品种为试材,首次尝试利用KASP技术筛选出一套能够鉴别中国主栽葡萄品种的核心KASP标记,并构建葡萄品种SNP指纹图谱,为我国葡萄新品种保护、市场维权等提供技术支持。
1 材料和方法
1.1 试验材料
试验材料于2020年6月采自中国农业科学院郑州果树研究所国家果树种质郑州葡萄圃。
KASP标记初筛:使用23份葡萄代表性品种,其具备不同倍性、不同种性、不同用途等特点,如二倍体欧亚种酿酒品种‘赤霞珠’‘黑比诺’,二倍体欧美杂种鲜食品种‘阳光玫瑰’‘金手指’,三倍体欧美杂种鲜食品种‘夏黑’,四倍体欧美杂种鲜食品种‘巨峰’‘巨玫瑰’等(表1)。KASP标记复筛:使用76份葡萄品种,其中13份国内主栽品种、63份近年新育成品种(表2)。
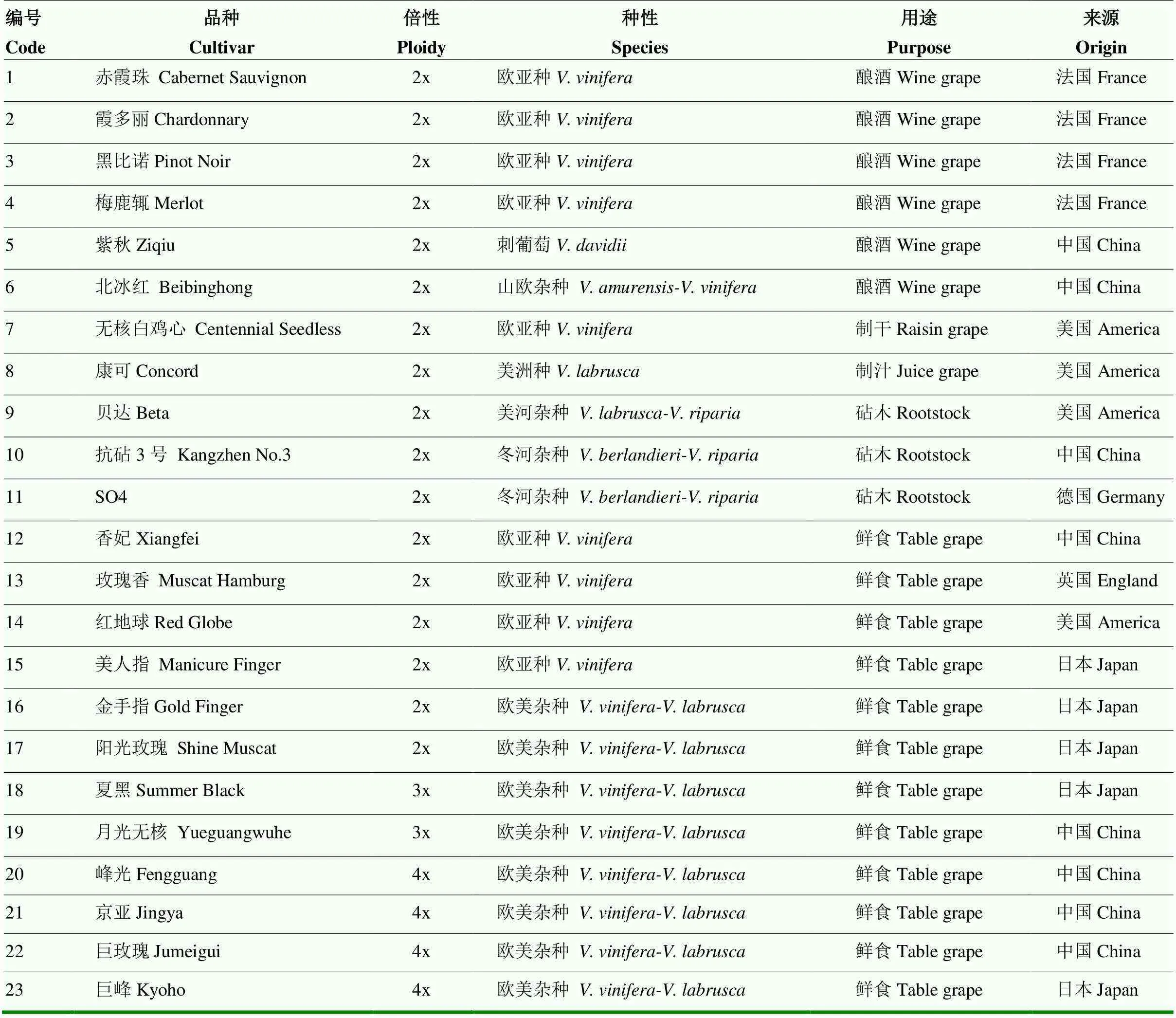
表1 KASP标记初筛使用的23份葡萄代表品种信息

表2 KASP标记复筛使用的76份葡萄品种信息
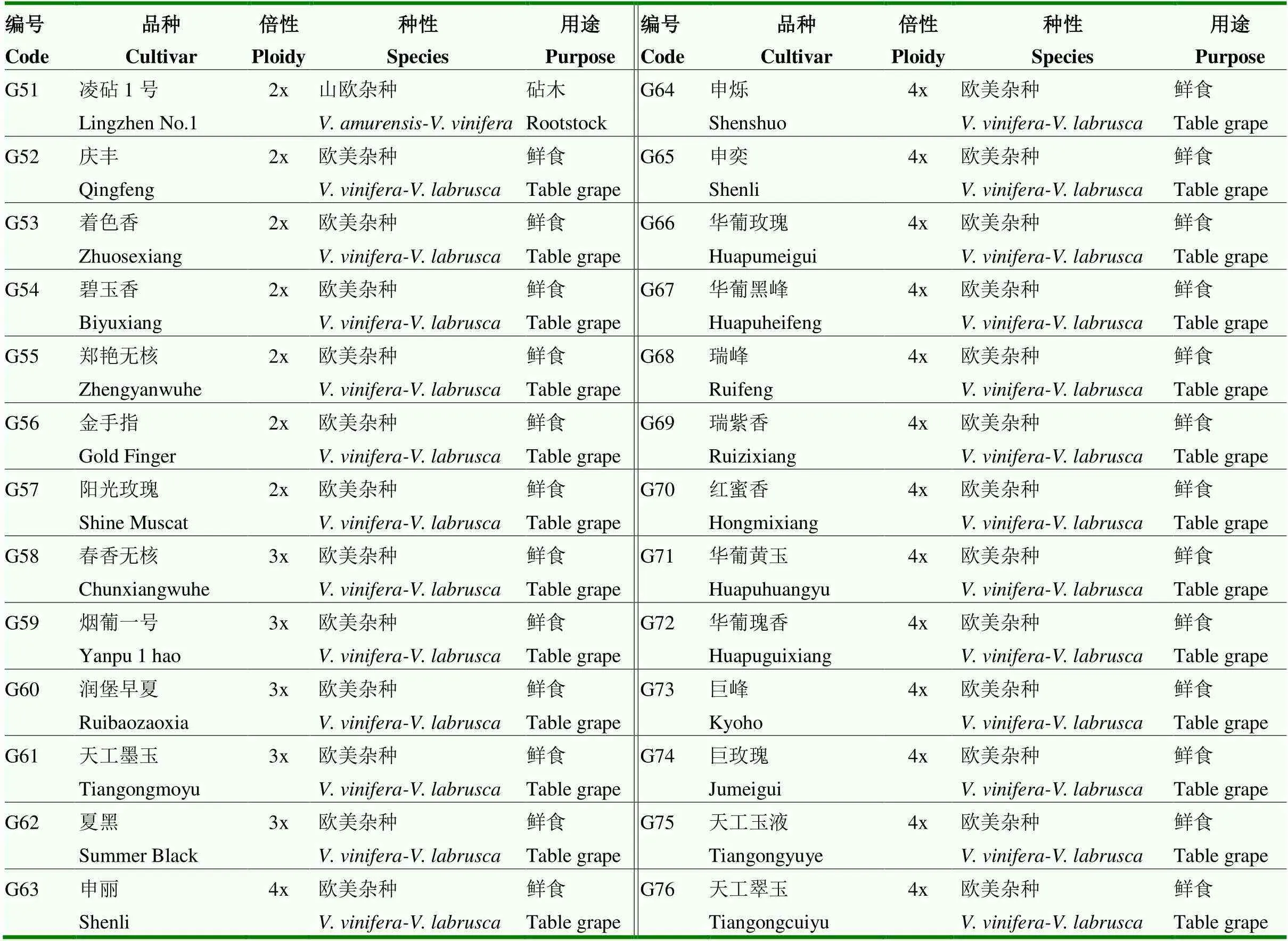
续表2 Continued table 2
1.2 DNA提取与检测
使用爱森生物科技有限公司的植物基因组DNA提取试剂盒,提取葡萄叶片DNA,采用NanoDrop 1000 spectrophotometer(Themo Scientific)紫外分光光度计检测DNA浓度和纯度(OD260/OD280=1.7—2.0),并用超纯水将浓度稀释至50 ng·μL-1,按编号装入96孔PCR板中,用于高通量扩增检测[8]。
1.3 KASP标记转化和引物合成
根据前人研究结果,共选用60个SNP位点作为KASP标记的筛选库,分别来自CABEZAS等[21]的46个和LAUCOU等[22]的14个SNP位点。为保证筛选的SNP位点具有基因特异性,从Ensemblplants网站下载SNP位点前后各100 bp的葡萄DNA序列(https://plants.ensembl.org/Vitis_vinifera/Gene),首先在NCBI中进行比对(https://blast.ncbi.nlm.nih.gov/ Blast.cgi?PROGRAM=blastn&PAGE_TYPE=BlastSearch&LINK_LOC=blasthome;Standard databases为RefSeq Representative genomens;Organism为wine grape (taxid: 29760)),去除非特异性的位点。然后,将SNP位点信息前后各100 bp的特异性DNA序列在建有LGC公司KASP检测技术平台的北京市农林科学院蔬菜改良中心设计并合成引物。其中引物设计要求两条正向引物的变性温度在62.5℃左右,温差不超过1℃,GC含量在50%—55%;反向引物的变性温度在66℃,GC含量不超过60%。
1.4 PCR扩增与标记检测
PCR反应体系(10.14 µL)为KASP Master mix 5 μL、KASP Primer mix 0.14 μL和模板DNA(50 ng·μL-1)5 μL。PCR反应条件:第一轮94℃ 15 min;94℃ 20 s,61—55℃ 60 s,10个Touch Down循环(每个循环降低0.6℃);第二轮94℃ 20 s,55℃ 60 s,26个循环。扩增产物使用荧光微孔板检测仪检测,然后采用LGC公司开发的SNP viewer 2.0软件读取检测数据[20]。
1.5 数据统计与分析
根据SNP分型结果数据计算相关遗传多样性参数,包括MAF(次要等位基因频率)、PIC(多态信息含量)[20,25]、Heterozygosity(杂合度)、Missing rate(缺失率)等,均在Excel 2019中使用公式进行计算:

式中,P和P是SNP的两个等位基因在所有被测品种中的发生频率,是样本数量[20]。
依据SNP标记具有的二态性特点,将分型数据转化为二元编码数据,野生型(与葡萄基因组一致)表示为(1、0),突变体表示为(0、1),杂合基因型表示为(1、1),缺失位点记为(999、999)[8],然后利用PowerMarker V3.25软件的邻接算法(Neighbor- joining)计算76个葡萄品种的遗传距离[26],并用Figtree v1.4.4构建聚类图。
使用STRUCTURE 2.3.4进行贝叶斯聚类[27],并确定最佳的类群分组。设定等位变异频率特征数(遗传群体数)K=1—15,Burn-in周期为100 000,MCMC的重复次数为100 000次,采用混合模型和相关等位基因频率,对不同的K值进行15次重复运行,然后将后缀为“_f”的结果文件压缩,上传到“STRUCTURE HARVESTER”网站(http://taylor0.biology.ucla.edu/ struct_harvest/),根据Evanno等[28]的方法计算得到∆K和似然值的对数函数lnp(D),分别针对基因库数(K)建模,确定最佳K值。
采用北京市农林科院蔬菜改良中心提供的Perl脚本计算出最优组合标记[20]。
2 结果
2.1 KASP标记转化结果
对60个SNP位点进行特异性分析,与葡萄参考基因组(Standard databases:RefSeq Representative genomens;Organism:wine grape (taxid: 29760))序列比对后,发现3个非特异性标记,最终57个SNP位点成功设计出51个KASP标记,转化率为89.47%(附表1)。
2.2 KASP标记的初筛
利用KASP技术对23份葡萄品种进行基因分型,51个KASP标记全部成功分型。其中,MAF值大于0.25的标记有31个,PIC值大于0.35的标记有35个,缺失率小于0.2的标记有44个,杂合率小于0.6的标记有50个,同时满足以上4个条件的标记有27个,并用于下一步的复筛(图1)。
2.3 KASP标记的复筛及DNA指纹图谱构建
使用初筛出的27个KASP标记对76份葡萄品种进行基因分型。结果表明,VIT_3_5724485、VIT_4_680574、VIT_11_5406647、VIT_15_18031506、VIT_18_3829207等5个标记分型结果不具备差异性,并舍弃;其余22个KASP标记在76份样本中均分型成功(图2)。其中,22个标记的缺失率均小于0.12,杂合率在0.4—0.6的占77%,PIC值在0.4—0.5的占95%,MAF值大于0.3的占95%(图3)。此外,使用筛出的22个KASP标记对同一品种不同树体提取的23份代表性品种的DNA扩增检测,前后两次检测的分型结果一致,表明筛选的22个KASP标记具有较好的重复性和稳定性,可以初步作为一组核心标记用于葡萄的品种鉴定。将这22个标记获得的76份葡萄品种分型结果转化为二元编码数据,得到76份中国主要栽培葡萄品种的SNP指纹图谱(图4)。
2.4 76个葡萄品种的聚类分析和群体结构分析
基于22个KASP标记的分型结果,利用PowerMarker V3.25和Figtree v1.4.4软件绘制76份葡萄品种的聚类图(图5-a)。76份葡萄品种可以分为3大类:第Ⅰ大类由19份多倍体欧美杂交品种单独聚为一大类(图5-a中标红色的分枝),其中‘春香无核’‘润堡早夏’‘天工墨玉’同为‘夏黑’的芽变品种,和‘夏黑’聚在一起;亲本之一同为‘红富士’的‘天工翠玉’‘天工玉液’2份品种也在该大类中,但因另一亲本不同,没有紧密聚在一起;‘巨玫瑰’‘华葡黄玉’‘华葡瑰香’同为‘巨峰’和‘沈阳玫瑰’的杂交后代,和‘巨峰’聚在一起;其他含有‘巨峰’亲缘的多倍体品种也与‘巨峰’聚在一起。第Ⅱ大类由39份二倍体品种组成,包含35份欧亚种、3份欧美杂种、1份毛葡萄(图5-a中标绿色的分枝),其中‘早康宝’和‘丽红宝’、‘翠香宝’和‘秋黑宝’、‘郑艳无核’和‘庆丰’、‘云楚无核’和‘岳秀无核’、‘瑞都红玫’‘瑞都脆霞’‘瑞都香玉’‘瑞都早红’5组分别出自同一杂交组合后代群体的品种,各自聚类在一起;具有‘玫瑰香’亲缘关系的‘脆红宝’‘晨香’‘卓越玫瑰’‘华葡翠玉’‘华葡早玉’5份品种与‘玫瑰香’聚类在一起;同样,具有‘红地球’亲缘关系的‘玉波一号’‘玉波黄地球’‘云楚无核’‘岳秀无核’‘华葡翠玉’‘华葡紫峰’‘红艳无核’7份品种也与‘红地球’聚类在一起;‘红特沙’与亲本之一‘里扎马特’紧密聚在一起。第Ⅲ大类主要由11份欧亚种、3份欧美杂种、3份山欧杂种、1份刺葡萄,共18份品种组成,同为二倍体品种(图5-a中标蓝色的分枝),其中‘赤霞珠’‘北冰红’‘爱格丽’‘媚丽’‘泰美’‘玉玲珑’6份酿酒葡萄品种聚在该大类中;‘北冰红’‘凌丰红’2份出自同一育种单位的山欧杂交品种,和‘惠良刺葡萄’紧密聚类在一起;‘玉波二号’‘短枝玉玫瑰’同为‘达米娜’和‘紫地球’的杂交后代,紧密聚在一起;由‘金手指’和‘紫地球’杂交获得的‘玉珍香’也与‘金手指’聚在该类群中。

A:51个KASP标记的名称(红色字体为初筛出的27个高质量KASP标记);B:葡萄染色体号;C:缺失率;D:杂合率;E:多态性信息含量;F:次要等位基因频率
用STRUCTURE 2.3.4进行种群遗传结构相关性分析,根据EVANNO等[28]的方法,用最大似然值∆K来确定K值。本研究中当K=3时,∆K取得最大值,推断76份葡萄品种分为3个类群(图5-b、图5-c),Ⅰ、Ⅱ、Ⅲ类分别占总群体数的39.47%、38.16%、22.37%,其中Ⅰ类群包含18个欧美杂种品种,占总欧美杂种群体数(25份)的72.00%,Ⅱ和Ⅲ群体中共包含36份欧亚种品种,占总欧亚种群数(46份)的78.26%,与聚类分析结果具有一定的一致性。
2.5 KASP标记在品种鉴定中的应用
对76份葡萄品种的指纹数据比对分析,发现3组基因型一致的品种分别为:‘夏黑’和‘润堡早夏’(‘夏黑’的芽变品种),‘天工翠玉’和‘天工墨玉’(同一家育种单位选育的品种),‘里扎马特’和‘红特沙’(亲本之一为‘里扎马特’)。采用北京市农林科学院蔬菜改良中心提供的Perl脚本计算出一组仅用10个标记就能区分70份品种的最优组合标记(图6),有54份葡萄品种达到品种鉴定的标准(差异位点数≥2)[29]。其中,中国主栽的‘红地球’‘玫瑰香’‘美人指’‘香妃’‘红宝石无核’‘碧香无核’‘金手指’‘阳光玫瑰’‘赤霞珠’9份葡萄品种含有至少2个KASP标记的基因型可区别于其他品种。新育成的品种中,‘脆红宝’‘翠香宝’‘早康宝’‘丽红宝’‘秋黑宝’‘无核翠宝’6份“宝”系列品种,‘瑞都无核怡’‘瑞都科美’‘瑞都脆霞’‘瑞都香玉’‘瑞都早红’‘瑞都红玫’6份“瑞都”系列品种,‘玉波一号’‘玉波二号’‘玉珍香’‘短枝玉玫瑰’‘玉波黄地球’5份“玉”系列品种,‘华葡紫峰’‘华葡翠玉’‘华葡早玉’‘华葡黄玉’‘华葡玫瑰’5份“华葡”系列品种,同一亲本后代的‘云楚无核’‘岳秀无核’及‘庆丰’‘郑艳无核’、单亲后代的‘MCS2’‘泰美’、山欧杂种的‘北冰红’‘凌丰红’‘凌砧1号’、刺葡萄种的‘惠良刺葡萄’以及多倍体‘天工玉液’‘烟葡一号’等品种间均有不少于2个KASP标记的差异。

图2 基于76份葡萄品种,VIT_6_4258638(A)和VIT_16_21202286(B)KASP标记荧光检测结果

图3 22个KASP标记基于76份葡萄品种获得的缺失率、杂合度、多态性信息含量、次要等位基因频率值在不同范围内所占百分比

第1列:KASP标记名称;第2列:KASP标记对应的突变碱基类型;其余列:依次为76个主要栽培葡萄品种的基因型
3 讨论
中国作为东亚种群的集中分布区,包含至少38个种[1],保存各类葡萄种质资源达3 000多份[30],且中国新育成的葡萄品种数量还以每年至少20份的速度增长[31]。但由于人们频繁的引种交流和长期的葡萄传播过程,同物异名或同名异物的现象时有发生[2],如云南‘水晶’、贵州‘水晶’、关口葡萄实为尼加拉的同物异名品种,盐井‘黑珍珠’和茨中‘教堂’葡萄实为‘巴柯’的同物异名品种[32]。此外,葡萄无性繁殖能力强,成活率高,育种家的成果一旦被窃取,极易被扩繁、易名、炒作,不仅侵害育种家的利益,而且加大了葡萄苗木市场的管理难度。
构建DNA指纹图谱技术(DNA-Fingerprinting)对于快速准确的品种鉴定和纯度鉴定、维护育种家的利益等具有重大意义[8]。当前,研究者已在水稻[29]、玉米[17]、棉花[33]、甘蓝[34]等多种作物中进行了以SSR和SNP核心标记为主的指纹图谱构建。在葡萄作物上,李贝贝等[6]利用8对引物构建国内最大群体量的SSR指纹图谱。但SSR标记在实际应用中还存在数量和检测通量有限、位点存在一定突变率、数据准确读取费时耗力、无法有效区分芽变品种等不足[15]。与SSR相比,SNP是基于单核苷酸的突变,突变频率更低,遗传稳定性更高,批量检测时平均每个孔位的使用成本比SSR的低1/10—1/20;同时,SNP也是构建DNA指纹数据库的推荐标记之一,但目前国内还没有利用SNP标记构建葡萄指纹图谱的研究[35]。而构建SNP指纹图谱首先要选择合适的SNP分型技术。目前,至少有20种SNP分型技术,其中SEMAGN等[14]以玉米为材料,从转化率、准确性、成本、灵活性等角度对比分析了KASP和Illumina GoldenGate两种技术,认为KASP基因分型技术在少位点检测大样本群体时具有更大的优势。赵勇等[36]基于53份大豆材料,使用4个SNP比较GBS、CAPS、KASP三种分型方法,也认为KASP技术更具有灵活实用性。刘丽华等[9]同样认为KASP技术可实现高通量的少量位点检测大量群体的目的,更适合作物品种鉴定工作。马丽等[17]、李志远等[8]、刘宝平等[37]和LI等[38]基于KASP技术分别筛选出22个、59个、48个和50个核心KASP标记,用于玉米、甘蓝、番茄和白菜等作物的品种鉴定中。

A:邻接聚类分析;B:K值与∆K趋势变化图;C:K=3时群体遗传结构分析图

1: VIT_15_18567587; 2: VIT_6_4258638; 3: VIT_8_3320936; 4: VIT_12_ 22228357; 5: VIT_11_19390306; 6: VIT_16_17950801; 7: VIT_18_ 11138668; 8: VIT_12_739916; 9: VIT_16_13454358; 10: VIT_16_21202286
本研究基于KASP技术,利用前人筛选的SNP位点,首次尝试构建我国76份主要栽培葡萄品种的SNP指纹图谱。基于KASP分型技术的检测原理,选择的SNP位点需要在群体中稳定遗传、具备高多态性。同时,为保证分型结果准确,筛选的标记应具有基因组特异性。本研究使用的SNP位点分别来自CABEZAS等[21]对1 324份葡萄材料有效分型验证的46个SNPs和LAUCOU等[22]基于18K SNPs葡萄基因芯片对945份材料筛出的14个SNPs,保证了选择的位点具有足够的区分能力。
为保证构建的76份葡萄指纹图谱的真实性,本研究基于检测获得的基因型数据进行聚类分析和群体分析,两种分析结果一致。聚类分析结果表明所有的多倍体欧美杂种起源于二倍体,但又区别于二倍体,在聚类图中单独聚为一类。其他二倍体品种分为两大枝,符合对应的亲缘关系,与王富强等[5]基于30对核心SSR引物对52份葡萄品种聚类分析结果具有一致性,首次验证了KASP分子标记可用于研究多倍体和二倍体葡萄之间的遗传进化关系,使用KASP标记构建葡萄指纹图谱具有可行性。
在品种鉴定分析中,因‘春香无核’‘润堡早夏’‘天工墨玉’同为‘夏黑’的芽变品种,以及亲本之一为‘里扎马特’的‘红特沙’,遗传相似度极高,使用22个KASP标记无法有效区分。只有‘无核白鸡心’的芽变品种‘科玉无籽’与‘无核白鸡心’含有6个纯合基因型的差异,说明芽变品种的鉴定依然是当前品种鉴定的难点,需要用更多的标记进行区分。而按照SSR分子标记的品种有效区分标准,本研究获得的10个标记(VIT_15_18567587、VIT_6_4258638、VIT_8_3320936、VIT_12_22228357、VIT_11_19390306、VIT_16_17950801、VIT_18_11138668、VIT_12_739916、VIT_16_13454358、VIT_16_21202286)可完全区分54份品种,包含了我国主栽的‘红地球’‘红宝石无核’‘金手指’‘阳光玫瑰’等品种,以及亲缘关系较近的“宝”系列、“瑞都”系列、“玉”系列、“华葡”系列品种,这与使用SSR标记区分相关品种具有一致性[5],表明使用KASP标记对葡萄进行品种鉴定的实用性,且比SSR标记具有更快更准、更易判读等优势。
4 结论
从60个葡萄SNP位点,成功转化51个KASP标记,并筛选出22个高质量KASP标记,构建了76份中国主要栽培葡萄品种的SNP指纹图谱。经邻接聚类分析和群体结果分析,可将76份葡萄品种分为3类,并能正确区分开二倍体和多倍体。仅用10个标记就能区分70份品种,其中有54份品种达到品种鉴定的标准(差异位点数≥2),首次验证了KASP技术在我国葡萄品种鉴定中的可行性。
[1] 孔庆山. 中国葡萄志. 北京: 中国农业科学技术出版社, 2004: 16.
KONG Q S. Chinese Ampelography. Beijing: Chinese Agricultural Science and Technology Press, 2004: 16. (in Chinese)
[2] LI B B, JIANG J F, FAN X C, ZHANG Y, SUN H S, ZHANG G H, LIU C H. Molecular characterization of chinese grape landraces (L.) using microsatellite DNA markers. Hortscience, 2017, 52(4): 533-540.
[3] BUTTON P. The international union for the protection of new varieties of plants (UPOV) recommendations on variety denominations. Proceedings of the Fifth International Symposium on the Taxonomy of Cultivated Plants, 2008, 799: 191-200.
[4] 植物品种鉴定-DNA指纹方法-总则. 中华人民共和国农业行业标准. NY/T 2594—2016.
General guideline for identification of plant varieties using DNA markers. Agricultural Industry Standard of the People’s Republic of China, NY/T 2594—2016. (in Chinese)
[5] 王富强, 李贝贝, 樊秀彩, 张颖, 刘崇怀, 姜建福. 葡萄品种SSR分子鉴定体系的建立及应用. 果树学报, 2020, 37(9): 1281-1293.
WANG F Q, LI B B, FAN X C, ZHANG Y, LIU C H, JIANG J F. Establishment and application of SSR molecular identification system in grapevine.Journal of Fruit Science, 2020, 37(9): 1281-1293. (in Chinese)
[6] 李贝贝, 姜建福, 张颖, 樊秀彩, 孙海生, 张国海, 刘崇怀. 葡萄品种DNA指纹数据库的构建及遗传多样性分析. 植物遗传资源学报, 2018, 19(2): 338-350.
LI B B, LIU C H, JIANG J F, ZHANG Y, FAN X C, SUN H S, ZHANG G H, LIU C H. DNA fingerprinting and genetic diversity analysis of grape (L.) cultivars based on SSR markers. Journal of Plant Genetic Resources, 2018, 19(2): 338-350. (in Chinese)
[7] RASHEED A, HAO Y F, XIA X C, KHAN A, XU Y B, VARSHNEYR K, HE Z H. Crop breeding chips and genotyping platforms: Progress, challenges, and perspectives. Molecular Plant, 2017, 10(8): 1047-1064.
[8] 李志远, 于海龙, 方智远, 杨丽梅, 刘玉梅, 庄木, 吕红豪, 张扬勇. 甘蓝SNP标记开发及主要品种的DNA指纹图谱构建. 中国农业科学, 2018, 51(14): 2771-2787.
LI Z Y, YU H L, FANG Z Y, YANG L M, LIU Y M, ZHUANG M, LV H H, ZHANG Y Y. Development of SNP markers in cabbage and construction of DNA fingerprinting of main varieties. Scientia Agricultura Sinica, 2018, 51(14): 2771-2787. (in Chinese)
[9] 刘丽华, 庞斌双, 刘阳娜, 李宏博, 王娜, 王拯, 赵昌平. 基于SNP标记的小麦高通量身份鉴定模式. 麦类作物学报, 2018, 38(5): 529-534.
LIU L H, PANG B S, LIU Y N, LI H B, WANG N, WANG Z, ZHAO C P. High-throughput identification mode for wheat varieties based on SNP markers. Journal of Triticeae Crops, 2018, 38(5): 529-534. (in Chinese)
[10] YANG G L, CHEN S P, CHEN L K, SUN K, HUANG C H, ZHOU D H, HUANG Y T, WANG J F, LIU Y Z, WANG H, CHEN Z Q, GUO T. Development of a core SNP arrays based on the KASP method for molecular breeding of rice. Rice, 2019, 12(1): 21.
[11] SUN C W, DONG Z D, ZHAO L, REN Y, ZHANG N, CHEN F. The wheat 660K SNP array demonstrates great potential for marker- assisted selection in polyploid wheat. Plant Biotechnology Journal, 2020, 18(6): 1354-1360.
[12] CHEN H D, XIE W B, HE H, YU H H, CHEN W, LI J, YU R B, YAO Y, ZHANG W H, HE Y Q, TANG X Y, ZHOU F S, DENG X W, Zhang Q F. A high-density SNP genotyping array for rice biology and molecular breeding. Molecular Plant, 2014, 7(3): 541-553.
[13] XU C, REN Y H, JIAN Y Q, GUO Z F, ZHANG Y, XIE C X, FU J J, WANG H W, WANG G Y, XU Y B, LI P, ZOU C. Development of a maize 55 K SNP array with improved genome coverage for molecular breeding. Molecular Breeding, 2017, 37(3): 1-12.
[14] SEMAGN K, BABU R, HEARNE S, OLSEN M. Single nucleotide polymorphism genotyping using kompetitive allele specific PCR (KASP): Overview of the technology and its application in crop improvement.Molecular Breeding, 2014, 33(1): 1-14.
[15] RASHEED A, WEN W E, GAO F M, ZHAI S N, JIN H, LIU J D, GUO Q, ZHANG Y J, DREISIGACKER S, XIA X C, HE Z H. Development and validation of KASP assays for genes underpinning key economic traits in bread wheat. Theoretical and Applied Genetics, 2016, 129(10): 1843-1860.
[16] GREWAL S, HUBBART-EDWARDS S, YANG C Y, DEVI U, BAKER L, HEATH J, ASHLING S, SCHOLEFIELD D, HOWELLS C, YARDE J, ISAAC P, KING I P, KING J. Rapid identification of homozygosity and site of wild relative introgressions in wheat through chromosome-specific KASP genotyping assays. Plant Biotechnology Journal, 2020, 18(3): 743-755.
[17] 马丽, 钟敬, 应继锋, 沈志成, 刘法新, 张立阳, 郑秀婷, 张先文, 阮祥经, 林海艳, 傅军, 刘欢. 基于KASP技术开发的玉米核心SNP标记及其应用. ZL201910623802.0. 2019-09-20.
MA L, ZHONG J, YING J F, SHEN Z C, LIU F X, ZHANG L Y, ZHENG X T, ZHANG X W, YUAN X J, LIN H Y, FU J, LIU H. Corn core SNP markers based on KASP technology and their application. ZL201910623802.0. 2019-09-20. (in Chinese)
[18] 匡猛, 王延琴, 周大云, 马磊, 方丹, 徐双娇, 杨伟华, 魏守军, 马峙英. 基于单拷贝SNP标记的棉花杂交种纯度高通量检测技术. 棉花学报, 2016, 28(3): 227-233.
KUANG M, WANG Y Q, ZHOU D Y, MA L, FANG D, XU S J, YANG W H, WEI S J, MA Z Y. High-throughput genotyping assay technology for cotton hybrid purity based on single-copy SNP markers. Cotton Science, 2016, 28(3): 227-233. (in Chinese)
[19] LI Z Y, YU H L, LI X, ZHANG B, REN W J, LIU X P, FANG Z Y, YANG L M, ZHUANG M, LV H H, ZHANG Y Y. Kompetitive allele-specific PCR (KASP) genotyping and heterotic group classification of 244 inbred lines in cabbage (L. var.). Euphytica: International Journal of Plant Breeding, 2020, 216: 1086-1099.
[20] ZHANG J, YANG J J, ZHANG L K, LUO J, ZHAO H, ZHANG J N, WEN C L. A new SNP genotyping technology target SNP-seq and its application in genetic analysis of cucumber varieties. Scientific Reports, 2020, 10(1): 275-305.
[21] CABEZAS J A, IBÁÑEZ J, LIJAVETZKY D, VÉLEZ D, BRAVO G, RODRÍGUEZ V, CARREÑO I, JERMAKOW A M, CARREÑO J, RUIZ-GARCÍA L, THOMAS M R, MARTINEZ-ZAPATER J M. A 48 SNP set for grapevine cultivar identification. BMC Plant Biology, 2011, 11(1): 153.
[22] LAUCOU V, LAUNAY A, BACILIERI R, LACOMBE T, ADAM- BLONDON AF, BERARD A, CHAUVEAU A, DE-ANDRES M T, HAUSMANN L, IBANEZ J, LE PASLIER M C, MAGHRADZE D, MARTINEZ-ZAPATER J M, MAUL E, PONNAIAH M, TOPFER R, PEROS J P, BOURSIQUOT J M. Extended diversity analysis of cultivated grapevinewith 10K genome-wide SNPs. PloS ONE, 2018, 13(2): e0192540.
[23] 李贝贝, 张恒, 姜建福, 张颖, 樊秀彩, 房经贵, 刘崇怀. 基于SLAF-seq技术的葡萄种质遗传多样性分析. 园艺学报, 2019, 46(11): 2109-2118.
LI B B, ZHANG H, JIANG J F, ZHANG Y, FAN X C, FANG J G, LIU C H. Analysis of genetic diversity of grape germplasms using SLAF-seq technology. Acta Horticulturae Sinica, 2019, 46(11): 2109-2118. (in Chinese)
[24] LIANG Z C, DUAN S C, SHENG J, ZHU S S, NI X M, SHAO J H, LIU C H, NICK P, DU F, FAN P G, MAO R Z, ZHU Y F, DENG W P, YANG M, HUANG H C, LIU Y X, DING Y Q, LIU X J, JIANG J F, ZHU Y Y, LI S H, HE X H, CHEN W, DONG Y. Whole-genome resequencing of 472accessions for grapevine diversity and demographic history analyses. Nature Communications, 2019, 10(1): 511-519
[25] BOTSTEIN D, WHITE R L, SKOLNICK M, DAVIS R W. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. American Journal of Human Genetics, 1980, 32(3): 314-331.
[26] 赵久然, 李春辉, 宋伟, 王元东, 张如养, 王继东, 王凤格, 田红丽, 王蕊. 基于SNP芯片揭示中国玉米育种种质的遗传多样性与群体遗传结构. 中国农业科学, 2018, 51(4): 626-634.
ZHAO JR, LI C H, SONG W, WANG Y D, ZHANG R Y, WANG J D, WANG F G, TIAN H L, WANG R. Genetic diversity and population structure of important chinese maize breeding germplasm revealed by SNP-chips. Scientia Agricultura Sinica, 2018, 51(4): 626-634. (in Chinese)
[27] FALUSH D, STEPHENS M, PRITCHARD J K. Inference of population structure using multilocus genotype data: linked loci and correlated allele frequencies. Genetics, 2003, 164(4): 1567-1587.
[28] EVANNO G, REGNUAT S, GOUSET J. Detecting the number of clusters of individuals using the software structure: A simulation study. Molecular Ecology, 2005, 14(8): 2611-2620.
[29] 水稻品种鉴定-DNA指纹方法. 中华人民共和国农业行业标准. NY/T 1433—2007.
Identification of rice (L.) varieties using microsatellite markers. Agricultural Industry Standard of the People’s Republic of China, NY/T 1433—2007. (in Chinese)
[30] 段长青, 刘崇怀, 刘凤之, 王忠跃, 刘延琳, 徐丽明. 新中国果树科学研究70年—葡萄. 果树学报, 2019, 36(10): 1292-1301.
DUAN C Q, LIU C H, LIU F Z, WANG Z Y, LIU Y L, XU L M. Fruit scientific research in new China in the past 70 years: grape. Journal of Fruit Science, 2019, 36(10): 1292-1301. (in Chinese)
[31] 孟聚星, 姜建福, 张国海, 孙海生, 樊秀彩, 张颖, 吴久赟, 刘崇怀. 我国育成的葡萄新品种系谱分析. 果树学报, 2017, 34(4): 393-409.
MENG J X, JIANG J F, ZHANG G H, SUN H S, FAN X C, ZHANG Y, WU J Y, LIU C H. Pedigree analysis of grape cultivars released in China. Journal of Fruit Science, 2017, 34(4): 393-409. (in Chinese)
[32] 李贝贝, 张颖, 樊秀彩, 李民, 刘崇怀, 姜建福. ‘巴柯’和‘尼加拉’葡萄疑似同物异名品种的SSR及形态学分析. 果树学报, 2019, 36(4): 393-400.
LI B B, ZHANG Y, FAN X C, LI M, LIU C H, JIANG J F. Analysis of suspected synonyms of ‘Baco Noir’ and ‘Niagara’ by SSR markers and morphology. Journal of Fruit Science, 2019, 36(4): 393-400. (in Chinese)
[33] 匡猛, 杨伟华, 许红霞, 王延琴, 周大云, 冯新爱. 中国棉花主栽品种DNA指纹图谱构建及SSR标记遗传多样性分析. 中国农业科学, 2011, 44(1): 20-27.
KUANG M, YANG W H, XU H X, WANG Y Q, ZHOU D Y, FENG X A. Construction of DNA Fingerprinting and analysis of genetic diversity with SSR markers for cotton major cultivars in China. Scientia Agricultura Sinica, 2011, 44(1): 20-27. (in Chinese)
[34] 结球甘蓝品种鉴定技术规程-SSR分子标记法. 中华人民共和国农业行业标准. NY/T 2473—2013.
Protocol for identification of cabbage varieties SSR marker method. Agricultural Industry Standard of the People’s Republic of China, NY/T 2473—2013. (in Chinese)
[35] 王富强, 樊秀彩, 张颖, 刘崇怀, 姜建福. SNP分子标记在作物品种鉴定中的应用和展望. 植物遗传资源学报, 2020, 21(5): 1308-1320.
WANG F Q, FAN X C, ZHANG Y, LIU C H, JIANG J F. Application and prospect of SNP molecular markers in crop variety identification. Journal of Plant Genetic Resources, 2020, 21(5): 1308-1320. (in Chinese)
[36] 赵勇, 刘晓东, 赵洪锟, 袁翠平, 齐广勋, 王玉民, 董英山. 大豆SNP分型方法的比较. 分子植物育种, 2017, 15(9): 3540-3546.
ZHAO Y, LIU X D, ZHAO H K, YUAN C P, QI G X, WANG Y M, DONG Y S. Comparison of methods for SNP genotyping in soybean. Molecular Plant Breeding, 2017, 15(9): 3540-3546. (in Chinese)
[37] 刘宝平, 王均帅, 焦珍珍, 邢海星, 张东峰, 马睿, 赵小翠, 尹增奇, 吴坤生. 一套适用于番茄DNA指纹库构建的KASP引物组合及其应用. ZL201910916095.4. 2019-12-24.
LIU B P, WANG J S, JIAO Z Z, XING H X, ZHANG D F, MA R, ZHAO X C, YIN Z Q, WU K S. A set of KASP markers combinations suitable for the construction of tomato DNA fingerprint library and their application. ZL201910916095.4. 2019-12-24. (in Chinese)
[38] LI P R, SU T B, WANG H P, ZHAO X Y, WANG W H, YU Y J, ZHANG D S, WEN C L, YU S C, ZHANG F L. Development of a core set of KASP markers for assaying genetic diversity insubsp.Makino. Plant Breeding, 2019, 138: 309-324.
Identification of Grape Cultivars Based on KASP Markers
WANG FuQiang1, ZHANG Jian2, WEN ChangLong2, FAN XiuCai1, ZHANG Ying1, SUN Lei1, LIU ChongHuai1, JIANG JianFu1
1Zhengzhou Fruit Research Institute, Chinese Academy of Agricultural Sciences, Zhengzhou 450009;2Beijing Vegetable Research Center, Beijing Academy of Agricultural and Forestry Sciences, Beijing 100097
【】This study was aimed to develop a set of kompetitive allele specific PCR (KASP) markers that can be used to distinguish the main cultivated grape cultivars in China, so as to provide the technical support for domestic grape cultivar protection, cultivar registration and market right protection. 【】60 highly polymorphic SNP loci from previous studies in grapes were selected to design KASP markers. 23 representative grape cultivars and 76 main cultivated grape cultivars in China were used for preliminary screening, re-screening, and verification of the successfully transformed KASP markers. A set of high-quality KASP markers was further selected to establish the DNA fingerprint database for the 76 grape cultivars. 【】Among the 60 SNP loci, 3 were not genome-specific, 6 could not be used to design KASP-PCR markers, and 51 could be successfully transformed to KASP markers with a conversion rate of 89.47%. Next, 23 representative grape cultivars were successfully genotyped by using the 51 KASP markers on LGC-SNP line platform. Then, 27 KASP markers of high quality were selected successfully based on the minor allele frequency (MAF) greater than 0.25, polymorphism information content (PIC) greater than 0.35, deletion rate less than 0.2, and heterozygosity rate less than 0.6. Finally, 22 KASP markers were successfully re-screened in the 76 main cultivated grape cultivars. The missing rate of the 22 markers was less than 0.12, the PIC value of them was greater than 0.30, the heterozygosity rate of 0.40-0.60 was 77.27%, and the MAF of greater than 0.30 was 95.45%. In addition, the consistent genotype data were acquired with different trees from each of the 23 representative cultivars by using the 22KASP markers, indicating that these 22 KASP markers had good reproducibility and stability for grape cultivar identification. The SNP genotyping results of 76 grape cultivars based on 22 markers were converted to binary coding data, and the fingerprints of 76 grape cultivars were obtained. Based on neighbor-joining cluster analysis, the 76 grape cultivars could be divided into 3 populations. Diploid and polyploidy cultivars were also correctly identified. The 10 markers (VIT_15_18567587, VIT_6_4258638, VIT_8_3320936, VIT_12_22228357, VIT_11_19390306, VIT_16_17950801, VIT_18_11138668, VIT_12_739916, VIT_16_ 13454358, VIT_16_21202286) were enough to be able to distinguish 70 of the 76 cultivars, of which 54 cultivars met the criteria for variety identification (the number of different loci ≥2). 【】From 60 grape SNPs, 51 KASP markers were successfully transformed, and 22 high-quality KASP markers were selected successfully for developing the fingerprints of the 76 main grape cultivars in China. It was the first time to verify the feasibility of KASP genotyping in the identification of grape cultivars in China.
grape; KASP marker; cultivar identification; fingerprint; genetic diversity analysis

10.3864/j.issn.0578-1752.2021.13.012
2020-08-26;
2021-01-23
国家重点研发计划(2019YFD1001401)、国家现代农业产业技术体系(CARS-29-yc-1)、中国农业科学院科技创新工程(CAAS-ASTIP- 2019-ZFRI)
王富强,E-mail:82101185031@caas.cn。通信作者姜建福,E-mail:jiangjianfu@caas.cn
(责任编辑 赵伶俐)

