宁都黄鸡粪便基因组DNA提取方法比较及盲肠微生物组成分析
朱 祺,罗 雯,*,艾佐佐,柴学文,王樟凤
(1.南昌师范学院 生物系,江西 南昌 330032;2. 江西省地方鸡种遗传改良重点实验室/南昌师范学院 生物技术研究所,江西 南昌 330032)
宁都黄鸡原名“宁都三黄鸡”,原产于江西省宁都县黄石、对坊等南部乡镇[1],至今已有1 500多年的饲养历史[2]。目前对于宁都黄鸡的研究多是关于肉质品质、外貌特征和生产性能等方面,尚未见宁都黄鸡肠道菌群的系统分析研究。
肠道微生物是一个相当复杂的微生态系统[3],而禽类肠道微生物对微生物资源的开发具有重要意义[4]。禽类为肠道微生物提供了生存生长的环境和营养物质[5],而肠道微生物则通过合成供给宿主所需的营养,辅助消化吸收,抵制潜在有害菌[6],调节宿主的免疫机能,维持宿主的健康,因此彻底了解肠道微生物组对宿主的影响是非常重要的[7]。动物消化道中微生物群落的多样性能够保证肠道微生物区系平衡,维持胃肠道环境相对稳定,并且结肠、盲肠中微生物最多[8],因此本研究以盲肠为研究对象。
高通量测序技术已成为当前微生物群落多样性研究的主流方法[9]。要获得高质量的测序结果,一个重要的条件是提供高质量的DNA、RNA样品。目前提取样本基因组的试剂盒很多,但各自的提取效果缺少对比分析。本研究将对基因组质量提取的不同预处理方法和不同试剂盒的提取物质量进行比较,并基于扩增子测序结果对宁都黄鸡的盲肠菌群组成结构进行初步探究,从而为进一步探索肠道微生物在宁都黄鸡生长中的作用、乃至未来禽微生态制剂的研发奠定基础。
1 材料与方法
1.1 材料与试剂
雄性72日龄宁都黄鸡,同一天出壳,同一条件饲养,均产自于江西省地方鸡种遗传改良重点实验室家禽分子遗传育种实践基地(江西省南昌市南昌县武阳镇)。
从同时期饲养的200羽笼养宁都黄公鸡中随机选取10羽,在室外解剖,用灭菌的细线扎住嗉嚢以上和结肠两个部位,将消化道取出放入装有75%乙醇的烧杯,再转移入超净工作台中,无菌采集盲肠内容物。将10个盲肠内容物样品各取相等湿重,混合均匀,放入-70 ℃冰箱中备用。
试验选用试剂盒包括:QIAamp Fast DNA Stool Mini Kit(QIAGEN),Stool gDNA Miniprep Kit(BIOMIGA),粪便基因组DNA快速提取试剂盒(BioTeke)。
1.2 方 法
1.2.1 不同试剂盒提取效果比较 取宁都黄鸡盲肠内容物混合样品150 mg,分别使用QIAGEN、BioTeke、BIOMIGA 3个公司的试剂盒提取基因组DNA,每种试剂盒重复提取3份。QIAGEN公司试剂盒提取产物标记为Qia,BioTeke公司试剂盒提取产物标记为Ke,BIOMIGA公司试剂盒提取产物标记为GA。将提取的基因组DNA用1%琼脂糖凝胶电泳检测,并置于-20 ℃保存备用。
以上述提取的基因组DNA为模板,以细菌16S rDNA扩增引物N8(5'-AGAGTTTGATGGCTCAG-3')和N1487(5'-ACGGTTACCTTGACGACTT-3')进行PCR 扩增。采用25 μL扩增体系:上、下游引物各1 μL,12.5 μL Taq green mix(2×),0.5 μL的样品DNA模板和10 μL的dd H2O。PCR反应条件为94 ℃预变性5 min;95 ℃变性1 min,55 ℃退火1 min,72 ℃延伸2 min,30个循环;72 ℃延伸10 min。1%琼脂糖凝胶电泳检测PCR产物。
1.2.2 扩增子测序及分析 以上述3种试剂盒提取的基因组DNA为模板,使用16S rDNA V3-V4区特异引物341F(5'-CCTAYGGGRBGCASCAG-3')和806R(5'-GCACTACHVGGGTWTCTAAT-3')进行PCR扩增,扩增程序:98 ℃变性1 min,扩增30个循环(98 ℃ 10 sec;50 ℃ 30 sec;72 ℃ 30 sec),72 ℃延伸5 min。PCR产物使用2%琼脂糖凝胶电泳分离,对目的条带使用Thermo Scientific公司的GeneJET胶回收试剂盒回收纯化。采用TruSeq○RDNA PCR-Free Sample Preparation Kit建库试剂盒进行文库构建,构建好的文库经过Qubit和Q-PCR定量,文库合格后,使用HiSeq2500平台进行测序。原始数据去除Barcode和引物序列后经拼接、过滤、去除嵌合体后得到有效数据。再对有效数据在97%水平上用UPARSE软件进行操作分类单元(operational taxonomic unit,OTU)聚类,并进行物种注释。使用QIIME软件进行Alpha多样性和Beta多样性分析。以上试验由北京诺禾致源生物信息有限公司协助完成。
1.2.3 LEfSe分析组间差异菌 使用LEfSe软件进行LDA Effect Size检测,将对数线性判别分析(LDA,Linear discriminative analysis)评分阈值设为4,Kruskal-Wallis秩和统计检验分析获得组间具有统计学差异的生物标识。
1.2.4 数据统计分析 采用SPSS 19.0统计分析软件对试验数据进行处理,非参数Kruskal-Wallis秩和统计进行显著性分析。
2 结果与分析
2.1 不同试剂盒基因组DNA提取效果比较
2.1.1 3种试剂盒提取样本的琼脂糖凝胶电泳 如图1所示,BioTeke公司的试剂盒提取样本的基因组DNA条带稍暗,但较清晰、整齐;BIOMIGA公司的试剂盒提取的样本条带与QIAGEN公司试剂盒提取的样本条带差别不明显。虽三种试剂盒基因组DNA的提取效果有区别,但无法清晰对比出三者之间的优劣。

图1 3种试剂盒提取基因组DNA的琼脂糖凝胶电泳图M.Marke Q;1~2.QIAGEN试剂盒;3~4.BioTeke试剂盒;5~6.BIOMIGA试剂盒Fig.1 Agarose gel electrophoresis image of genomic DNAextracted by 3 different kinds of kitsM. Marke Q; 1~2. QIAGEN kit;3~4. BioTeke kit;5~6. BIOMIGA kit
2.1.2 3种试剂盒提取基因组DNA样本的PCR扩增效果比较 如图2所示,QIAGEN、BioTeke、BIOMIGA 3个公司的试剂盒所提取的基因组DNA都能成功扩增出16S rDNA产物,且扩增出的条带都清晰处于同一水平。
2.1.3 扩增子测序序列数和OTU数量统计 对GA、Ke、Qia 3组样品分别进行16S rDNA V3-V4区扩增子测序分析。如表1所示,各组样品经过滤嵌合体后,有效数据均不低于6×104条。虽然样本Qia组有效数据均值最高,Ke组有效数据均值最低;而OTU聚类结果则为GA组均值最高,Qia组均值最低。但根据Kruskal-Wallis秩和检验结果显示,各组数据差异不显著(P>0.05)。对3组样本进行均一化处理,后续的Alpha多样性分析和Beta多样性分析都是基于均一化处理后的数据(均一化时选取的数据量:cutoff=32158)。

图2 不同试剂盒提取基因组DNA的16S rDNA PCR扩增产物图M.Marke Q;1~2.QIAGEN试剂盒;3~4.BioTeke试剂盒;5~6.BIOMIGA试剂盒Fig.2 16S rDNA PCR amplification products of genomicDNA extracted from different kitsM. Marke Q; 1~2. QIAGEN kit;3~4. BioTeke kit; 5~6. BIOMIGA kit

表1 样品序列数和OTU数量统计Table 1 Statistics of sample sequence number and OTU quantity
2.1.4 Alpha多样性比较 如图3所示,各组样品随机抽取的数据量达到30 000以上时,曲线趋于平坦,说明所得序列可基本反映真实环境中细菌群落结果[10]。采用Shannon、Simpson、Chao1、ACE等指数来表征各组样品中微生物的Alpha多样性,如表2所示。对各组间Alpha多样性指数值进行Kruskal-Wallis秩和检验,结果表明,各组间Chao1、ACE指数值均无显著差异,但Ke-GA和Qia-GA两组间Shannon指数和Simpson指数差异显著,而Qia-Ke间无显著差异(见表3)。说明BioTeke公司和QIAGEN公司提取样本的群落多样性较为接近,BIOMIGA公司提取样本的群落多样性与前两者差异显著。

表2 各组样品间Alpha多样性统计Table 2 Alpha diversity statistics of different groups

表3 各组样品间Alpha多样性统计差异显著性检测结果Table 3 Difference significance test results of Shannonindex between different groups

图3 各组样品稀释曲线Fig.3 Rarefaction curve of each group
2.1.5 物种分布情况分析 选取3组样品在门、纲、目、科或属分类水平上平均丰度排名前10的物种,生成三元相图(ternaryplot),可以直观查看3组样本在各级分类水平上优势物种的差异[11]。从图4可以看出,3组样品在各级分类水平上优势物种均有不同。引起差异的原因推测是试剂盒种类不同,对各微生物类群的裂解效率不同。
2.1.6 Beta多样性分析 Beta多样性组间差异分析的箱形图见图5,Kruskal-Wallis秩和检验结果表明,各组间物种Beta多样性差异不显著(P>0.05)。进一步通过MRPP分析进行组间群落结构差异显著性检验,Observe Delta值越小说明组内差异小,Expect delta值越大说明组间差异大;A值大于0说明组间差异大于组内差异,A值小于0说明组内差异大于组间差异。由表4可知,3种试剂盒提取物分析获得的盲肠微生物群落结构具有组间差异,但差异不显著(P>0.05)。
2.1.7 组间差异菌分析 用LDA值作分布柱状图和进化分支图(图6)。柱状图的长度揭示差异菌群影响的大小。进化分支图由内至外辐射的圆圈代表由门至属(或种)的分类级别,每个小圆圈代表该水平下的一个分类,其直径大小与相对丰度大小呈正比;无显著差异的统一着色为黄色,差异生物标识跟随组进行着色。通过LEfSe算法进行的组间微生物差异分析结果表明,LDA为4时,组间具有统计学差异(P<0.05)的生物标识有5个,均与Qia组相关,生物标识影响由大至小分别为拟杆菌门(Bacteroidetes)、拟杆菌纲(Bacteroidia)、拟杆菌目(Bacte-roidales)、理研菌科RC9肠菌群(Rikenellaceae RC9 gut group)、理研菌科(Rikenellaceae)。

图4 各级分类水平三元相图Fig.4 Ternaryplot under each classification levels

图5 Beta多样性箱形图A. Weighted Unifrac;B. Unweighted UnifracFig.5 Box-plot of Beta diversity

表4 MRPP 组间差异分析Table 4 Difference analysis between MRPP groups

图6 组间微生物差异效应判别分析柱状图(A)和进化分支图(B)Fig.6 Histogram(A) and cladogram(B) based LEfSe analysis of different groups
2.2 宁都黄鸡肠道菌群组成分析
综合3种试剂盒提取物的扩增子分析结果,获得宁都黄鸡盲肠微生物在各级分类水平上的菌群组成特征。
2.2.1 门水平下盲肠菌群组成分析 宁都黄鸡肠道微生物群落构成多样性丰富。将3组样本注释到门水平,共检测到21个菌门,其中18个门在3组样本中均可检出,而芽单胞菌门(Gemmatimonadetes)和纤维杆菌门(Fibrobacteres)只在GA组中检出,黏胶球形菌门(Lentisphaerae)只在Ke和Qia组中被检出,且这3个菌门占比均不足0.01%。相对丰度前10的菌门见图7,结果表明宁都黄鸡盲肠菌群以拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)、广古菌门(Euryarchaeota)和变形菌门(Proteobacteria)4个菌门为主,占比达90%以上。
2.2.2 纲水平下盲肠菌群组成分析 在纲的分类水平共鉴定出50个纲,相对丰度前10的菌纲见图8。其中拟杆菌纲(Bacteroidia)、甲烷杆菌纲(Methanobacteria)和梭状芽胞杆菌纲(Clostridia)是相对丰度最高的3个纲,占比超过75%。相对丰度前10的菌纲占比已达总菌的90%以上。

图7 基于门水平菌群分布Fig.7 Distribution of intestinal microflora at phylum level

图8 基于纲水平菌群分布Fig.8 Distribution of intestinal microflora at class level
2.2.3 目水平下盲肠菌群组成分析 在目水平上,综合3组样本共鉴定出80个目,相对丰度前10的菌目见图9。其中拟杆菌目(Bacteroidales)、甲烷杆菌目(Methanobacteriales)和梭状芽胞杆菌目(Clostridiales)总比例已超过75%。
2.2.4 科水平下盲肠菌群组成分析 将3组样本注释到科水平,共检测到134个科。在注释的宁都黄鸡肠道微生物菌群中,甲烷杆菌科(Methanobacteriaceae)是主要的优势菌群;相对丰度排名前十的还有理研菌科(Rikenellaceae)、瘤胃菌科(Ruminococcaceae)、毛螺菌科(Lachnospiraceae)、线粒体科(Mitochondria)、拟杆菌科(Bacteroidaceae)、肉杆菌科(Carnobacteriaceae)、Bacteroidales S24-7 group、脱硫弧菌科(Desulfovibrionaceae)和普雷沃氏菌科(Prevotellaceae)。

图9 基于目水平菌群分布Fig.9 Distribution of intestinal microflora at order level
2.2.5 属水平下盲肠菌群组成分析 对3组样本注释到属水平,共鉴定出268个菌属。从图11可知,相对丰度较高的前3个属分别是甲烷杆菌属(Methanobrevibacter)、理研菌科RC9肠菌群(RikenellaceaeRC9gutgroup)和拟杆菌属(Bacteroides)。

图10 基于科水平菌群分布Fig.10 Distribution of intestinal microflora at family level
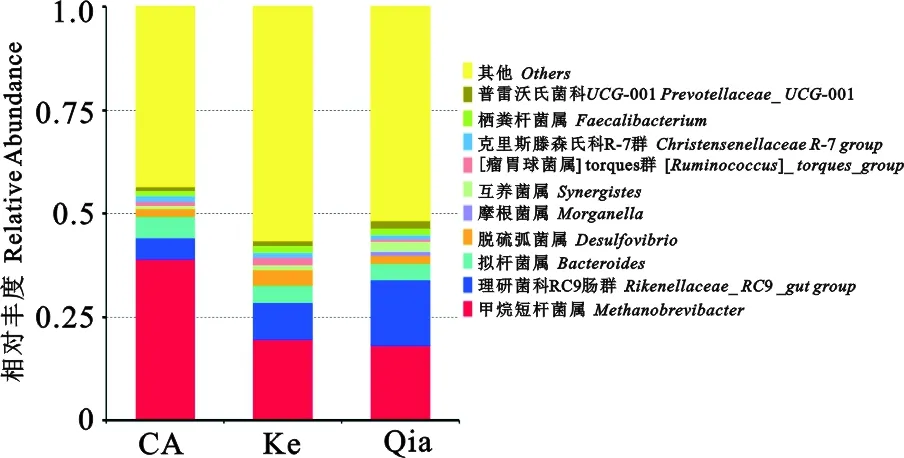
图11 基于属水平菌群分布Fig.11 Distribution of intestinal microflora at genus level
3 讨 论
3.1 不同试剂盒提取基因组DNA效果比较
通过扩增子测序分析,虽然不同试剂盒提取的基因组DNA分析所得组间群落结构确有差异,但使用BIOMIGA、BioTeke和QIAGEN的粪便基因组DNA提取试剂盒获得的3组样本间,群落结构差异不显著。同时,代表物种丰富度的Chao1、ACE指数值在各组间也无显著差异。但是,值得关注的是,兼有群落多样性和均匀性因素的Shannon和Simpson指数值在Ke-GA和Qia-GA两组间差异显著,揭示菌群多样性和均匀度分析结果差异显著,即BIOMIGA公司试剂盒提取物分析所得菌群多样性和均匀度与BioTeke及QIAGEN公司试剂盒提取物分析结果相比相对较低;而BioTeke及QIAGEN公司试剂盒提取物在肠道微生物物种丰富度分析中无显著差异。提示不同试剂盒的微生物裂解效率差异可能对高通量测序分析结果产生影响。LEfSe分析结果显示,LDA为4时,组间具有统计学差异的5个生物标识均与Qia组相关,且均属于拟杆菌门,提示QIAGEN公司试剂盒在拟杆菌门微生物裂解效率方面具有明显优势。
3.2 宁都黄鸡肠道菌群组成分析
迄今关于禽类不同物种肠道微生物组成的研究表明,尽管食性相同,但不同禽类肠道菌群依然存在部分分化的现象,说明宿主系统发生地位对肠道菌群结构起到了重要的作用[12-13]。虽然不同研究对鸡同一肠段菌群组成特征分析结果不尽相同[14-20],但拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)在鸡肠道中,占比均较高,为主要优势菌群。综合3组样本的结果,本研究在宁都黄鸡盲肠内容物中共鉴定出21个门、50个纲、80个目、134个科、268个属的微生物,物种丰富度高。虽然拟杆菌门、厚壁菌门和变形菌门相对丰度也较高,但与同类研究结果[21]不同的是,广古菌门(Euryarchaeota)丰度显著高于变形菌门;且在属分类水平上,第一优势属为甲烷杆菌属(Methanobrevibacter),而已报道的鸡盲肠优势属——梭状芽孢杆菌(Clostridium)、瘤胃球菌(Ruminococcus)和乳杆菌(Lactobacillus),在本研究中占比均相对较低。本研究中发现的菌群组成和物种分布差异是饲养环境或饲养条件引起的偶然现象,还是宁都黄鸡肠道菌群的独有特征,值得进一步验证。
大量研究已经证明,肠道细菌可以为宿主提供养料,但也有一些肠道细菌与宿主竞争营养,因此分析肠道中的细菌类群对于阐明肠道菌群在促进宿主生长方面的作用机理以及为禽微生态制剂研究提供理论和实践依据具有重要意义。本文充分发挥Illumina HiSeq测序方法的大数据优势[22],最大限度客观还原肠道菌群结构以及丰度比例,为研究宁都黄鸡肠道微生物与宿主之间的相互关系进一步优化宁都黄鸡的饲养方式提供了前期基础。

