三浅裂野牵牛MADS-box基因家族全基因组鉴定与组织特异性表达分析
姚旭峰,董静静,刘世芳,张 毅,唐锐敏,王文斌,解红娥,吴宇浩,武宗信,贺立恒,李润植,贾小云
(山西农业大学 a.生命科学学院,太谷 030801;b.农学院,太谷 030801;c.棉花研究所,运城 044000)
MADS-box是一类广泛存在于动物、植物和真菌等真核生物中的基因家族,其名称来自于酿酒酵母的MCMI基因(酵母微染色体维持基因)、拟南芥的AGAMOUS基因(花发育调控基因,C功能基因)、金鱼的DEFICIENS基因(花发育调控基因,B功能基因)和人的SRF4基因(血清应答因子基因)的首字母。MADS-box基因家族的蛋白序列在N端均含有一个由58~60个氨基酸组成的保守结构域,即MADS-box结构域[1-2]。依据MADS-box蛋白所含有结构域的类型可将其分为2类:Type Ⅰ(M-type)和Type II(MIKC-type),其中Type Ⅰ包含Mα、Mβ、Mγ、Mδ 4个亚家族,Type II可进一步分为MIKC*与MIKCC2个亚家族[3]。Type II型MADS-box包括4个特征结构域 :MADS-box(M)、Intervening(I)、Keratin(K)和 C-terminal(C),所以又称为MIKC类型[4]。MIKC*亚家族与MIKCC相比,缺少特征性K结构域[5]。在植物中,Type Ⅰ型的MADS-box基因通常含有1~2个外显子;而Type II型含有多个内含子和外显子,一般包括6个内含子和7个外显子[6]。
关于MADS-box基因的研究,最早是从金鱼草(Antirrhinum majusL.)和拟南芥(Arabidopsis thaliana)花形态的突变体开始的,随着研究的深入,人们发现绝大多数植物的MADS-box基因都与花的形态发生密切相关[7],如Ⅰ型MADS-box基因与植物配子分化、胚和胚乳的发育相关[8-10]。Kotoda等[11]从苹果(Malus domestica)中克隆得到MdMADS5,发现其与拟南芥AP1是同源基因,将MdMADS5转化拟南芥后可使其开花时间提前、花序变短、簇生叶减少。Kitahara等[12]从野生玫瑰中克隆了2个基因MASAKO C1和MASAKO D1,研究发现它们与雄蕊和心皮的形成有关。
甘薯[Ipomoea batatas(L.) Lam]又名红薯,属旋花科,是一年生或者多年生的蔓生草本植物,是重要的粮食、饲料和能源作物[13]。甘薯是六倍体作物(2n= 6x=90),因其基因组比较复杂,故其研究相比于其他农作物相对滞后[14]。有研究表明,三浅裂野牵牛(Ipomoea trifida)是甘薯的近缘野生种[15-17],所以了解三浅裂野牵牛基因组信息将有助于解析六倍体甘薯基因组和二倍体基因组的进化模式,揭示不同物种基因组间的关系,为研究甘薯的起源与进化奠定基础。关于MADS-box基因的研究,已经陆续在拟南芥、水稻、番茄等模式植物中报道[18-20]。目前,三浅裂野牵牛中MADS-box基因家族的鉴定和功能研究的报道较少。本文将首次从全基因组水平鉴定三浅裂野牵牛中MADS-box(Ipomoea trifida MADS-box,ItfMADS)基因家族成员,并对其进行生物信息学分析,为甘薯MADS-box基因功能的研究奠定基础。
1 材料与方法
1.1 ItfMADS基因家族的鉴定及蛋白理化性质分析
三浅裂野牵牛全基因组数据在密歇根州立大学建立的甘薯资源库(Sweetpotato Genomics Resource,http://sweetpotato.plantbiology.msu.edu/)中下载获得。根据Sweetpotato Genomics Resource提供的三浅裂野牵牛转录组数据的注释信息,挑选出MADS-box蛋白序列,对得到的蛋白序列进行去冗余、去重复,并将候选序列提交至NCBI Conserved Domain Search(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)和SMART(http://smart.embl.de/)进行结构域分析,进一步确定候选成员是否含有MADS-box保守结构域,鉴定得到ItfMADS家族成员。
利用ExPASy-ProtParam tool(https://web.expasy.org/protparam/)在线分析工具对ItfMADS蛋白的氨基酸数目、分子量和等电点等理化性质进行分析。利用在线程序SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)对ItfMADS蛋白进行二级结构分析。利用在线程序PSORT Prediction(http://psort.hgc.jp/form.html)预测ItfMADS蛋白亚细胞定位。
1.2 系统进化分析
在Phytozome11.0数据库(http://phytozome.jgi.doe.gov/pz/portal.html#)中下载92个拟南芥MADS-box(Arabidopsis thalianaMADS-box,AtMADS-box)蛋白序列,利用MEGA7.0构建ItfMADS蛋白和At-MADS蛋白的系统进化树。采用最大似然法(maximum likelihood)的P-距离(P-distance)模型建树,参数选择部分删除(partial deletion)空位(gap),校验参数(Bootstrap)取值1 000,其他运行参数为默认值。
1.3 多序列比对与保守结构域基序分析
利用clustalW软件对鉴定所得的ItfMADS成员进行多序列比对,将特征结构域序列提交至WebLogo(http://weblogo.threeplusone.com)进行结构域保守基序分析。
1.4 保守基序分析及基因结构分析
采用TBtools中的内置MEME工具进行保守基序分析,基序查找的最大数目设置为5,其他参数为默认[21]。MEME查找得到的ItfMADS蛋白保守基序用在线程序SMART (http://smart.embl-heidelberg.de/)进行功能注释分析。利用TBtools分析ItfMADS基因家族成员的基因结构,并绘制基因结构图。
1.5 染色体定位分析
通过TBtools软件获得ItfMADS各成员的染色体位置信息,利用MG2C在线软件(http://mg2c.iask.in/mg2c_v2.0/)绘制染色体定位图。
1.6 启动子顺式作用元件预测分析
提取ItfMADS基因CDS序列上游2 000 bp区段,提交到PlantCare网站(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)进行顺式作用元件预测,筛选并分析激素响应元件、抗逆响应元件。
1.7 表达模式分析及实时荧光定量多聚核苷酸链式反应(qRT-PCR)分析
下载甘薯基因组学资源网站中的转录组数据,利用TBtools绘制表达热图。为进一步验证ItfMADS基因的表达模式,采用多聚核苷酸链式反应(quantitative Real-time PCR,qRT-PCR)的方法分析ItfMADS基因在三浅裂野牵牛中不同组织部位的表达情况。三浅裂野牵牛的种子由徐州甘薯中心提供,种植于山西农业大学生命科学院试验田,取1月龄的叶片、茎和根进行后续试验。设计ItfMADS基因的定量引物,并以GAPDH基因为内参基因。利用BIO-RAD CFX 96荧光定量仪(美国伯乐公司)进行试验,试验重复3次,结果用2–△△CT法分析数据[22]。试验所用引物委托生工生物工程(上海)股份有限公司合成。引物如表1所示。

表1 qRT-PCR扩增引物序列Tab.1 Primers used for qRT-PCR amplification
2 结果与分析
2.1 ItfMADS基因家族的鉴定及蛋白理化性质分析
在三浅裂野牵牛基因组中,共发现51个Unigene序列被注释为MADS-box基因。对51个候选MADS-box基因的蛋白序列进行结构域鉴定,结果表明,有1个候选成员不具备MADS-box保守结构域,其余50个候选成员均具有MADS-box保守结构域,去除12条冗余序列,共得到38个MADS-box家族成员,根据它们的编码基因在染色体上的相对位置,命名为ItfMADS01~ItfMADS38。
采用ProtParam在线工具分析ItfMADS基因编码蛋白质的理化性质,包括氨基酸数目(number of amino acids)、分子量(molecular weigh,MW)、等电点(pI)、不稳定指数(instability index)和脂溶指数(aliphatic index)等(表2)。结果可知:ItfMADS各成员的氨基酸数目差异很大,氨基酸数目小于200的有9个,占比23.7%,氨基酸数目在200~300之间的有28个,占比73.7%,大于300个氨基酸的序列仅有1个,占比2.6%;其中最短的ItfMADS01仅由70个氨基酸组成,最长的ItfMADS18有461个氨基酸,依次对应的ItfMADS蛋白分子量介于7 980.31~51 802.82 kD之间;ItfMADS蛋白大多显碱性,少部分显酸性,其中,12个成员的pI小于7,呈酸性,占比31.6%,其余的26个成员均呈碱性;不稳定指数分析表明,38个ItfMADS蛋白的稳定指数均大于40,均为不稳定蛋白;脂溶指数分析表明,38个ItfMADS蛋白的脂溶指数均小于100,均为亲水性蛋白。
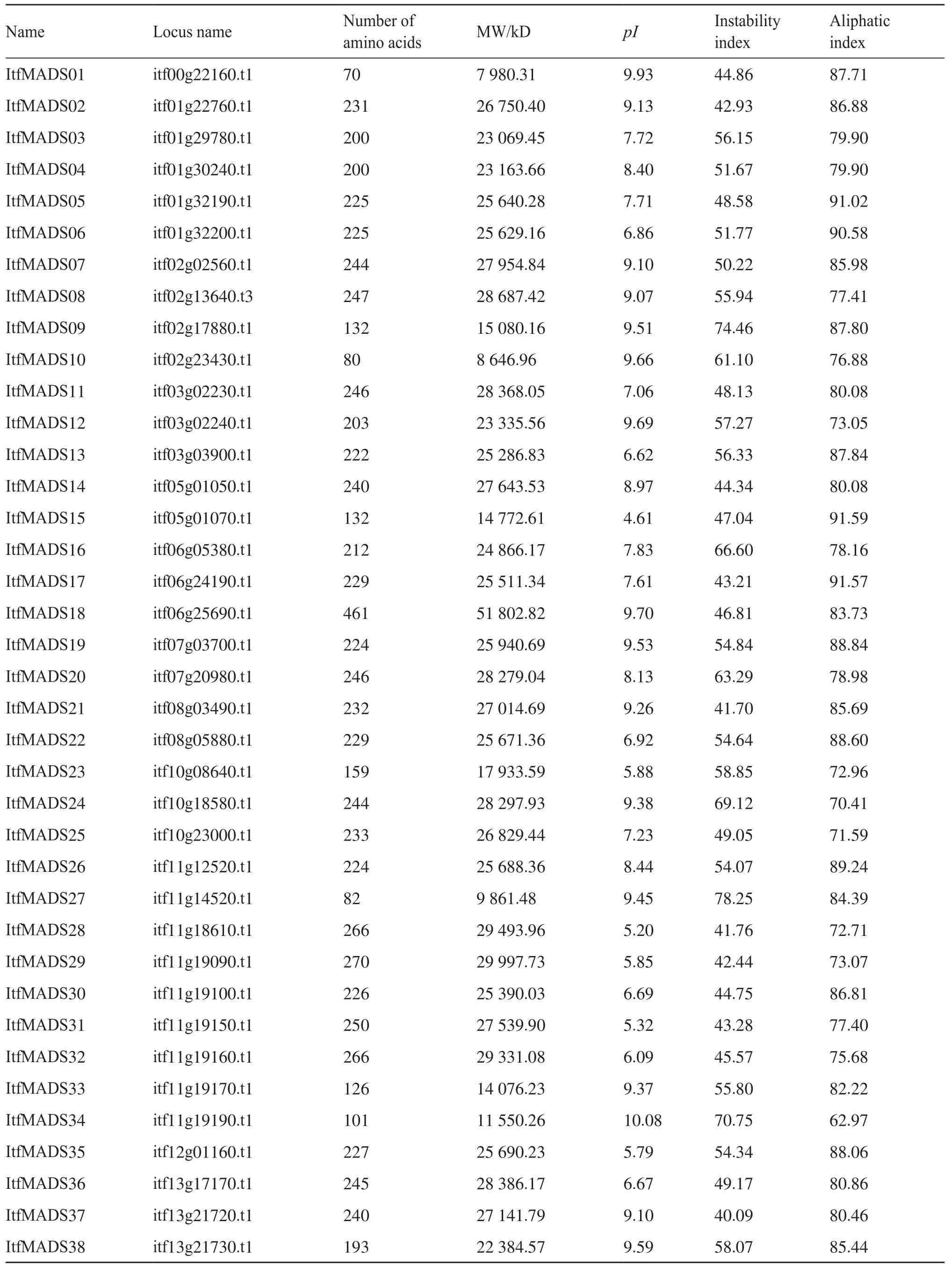
表2 ItfMADS蛋白的理化性质Tab.2 The chemicophysical properties of ItfMADS proteins
氨基酸残基间通过范德华力、氢键等作用发生折叠缠绕形成的高级结构称为蛋白质二级结构,其中主要包括α-螺旋(α-helix)、β-转角(β-turn)、扩展链(extended strand)和无规则卷曲(random coil)等结构[23]。利用SOPMA在线工具分析ItfMADS蛋白质的二级结构(表3),结果表明:ItfMADS27没有β-转角结构;ItfMADS01的β-转角结构所占比例最低;其余的36个ItfMADS蛋白中,所占比例最高的是α-螺旋结构,其次是无规则卷曲结构,所占比例最低的是β-转角结构。
利用PSORT Prediction在线软件预测ItfMADS成员的亚细胞定位。结果如表3所示:38个ItfMADS蛋白中,32个成员定位于细胞核(cell nucleus),占比84.2%;ItfMADS30和ItfMADS31定位于线粒体基质(mitochondrial matrix),ItfMADS15定位于细胞质(cytoplasm),ItfMADS09定位于线粒体内膜(mitochondrial inner membrane),ItfMADS10定位于叶绿体类囊体(chloroplast thylakoid space),ItfMADS23定位于细胞质膜(plasma membrane)。
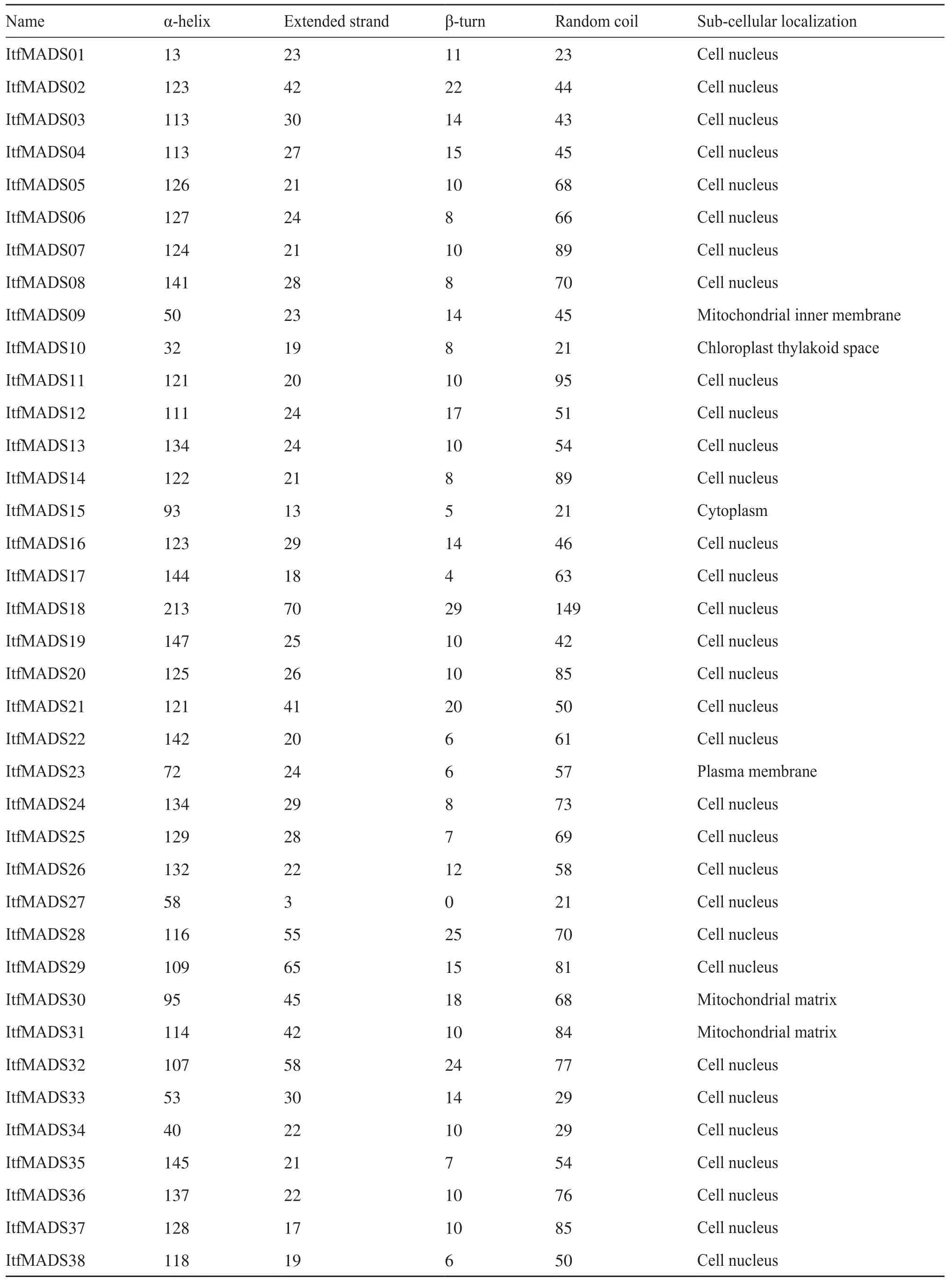
表3 ItfMADS蛋白质二级结构与亚细胞定位Tab.3 The secondary structure and subcellular localization of ItfMADS proteins
2.2 系统进化分析
采用Maximum Likelihood法对来源于拟南芥和三浅裂野牵牛的MADS-box蛋白进行系统进化分析(图1)。参照Parenicová等[18]对拟南芥MADS-box家族成员系统进化分析的结果,ItfMADS-box蛋白可分为Ⅰ型和Ⅱ型,属于Ⅰ型的成员有9个,属于Ⅱ型的有29个。其中,9个Ⅰ型成员全部属于Mα亚家族,Ⅱ型成员为MIKC亚家族。
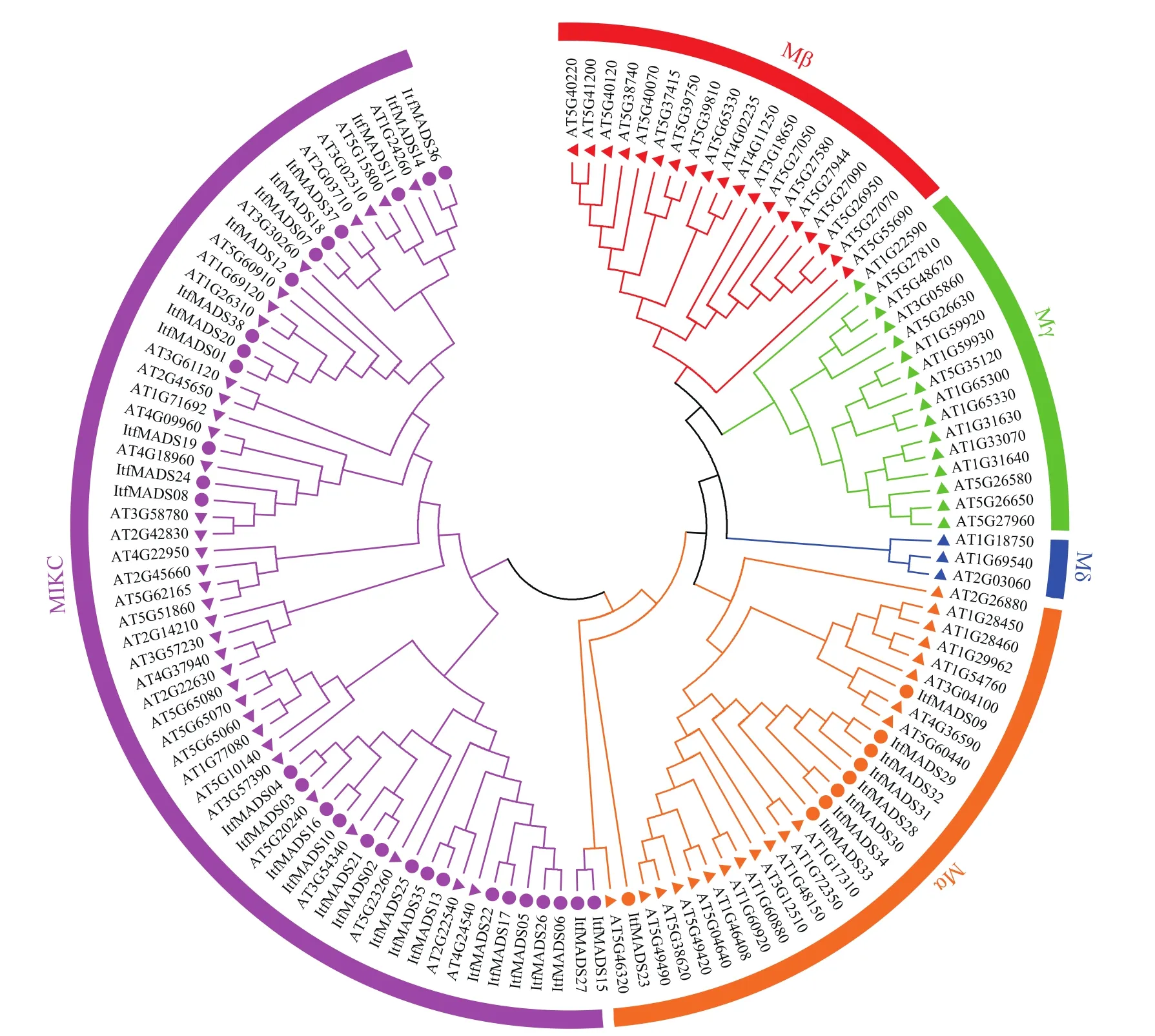
图1 ItfMADS-box和AtMADS-box蛋白系统进化分析Fig.1 Phylogenetic analysis of ItfMADS-box and AtMADS-box proteins
2.3 多序列比对与保守结构域基序分析
在植物中,Ⅰ型MADS-box成员只具有MADS-box结构域,而Ⅱ型MADS-box成员除此之外还具有一个比较保守的K结构域[24-25]。我们利用clustalW软件对38个ItfMADS进行多序列比对,得到其特征结构域序列,然后将其提交到Weblogo,获得结构域保守基序图(图2)。由图2可知:MADS-box结构域中3、17、24位置的精氨酸以及位置23的赖氨酸高度保守;K结构域中位置18的甘氨酸高度保守。

图2 结构域保守基序分析Fig.2 Sequence analysis of conserved domains
2.4 保守基序分析及基因结构分析
利用MEME软件分析得到ItfMADS蛋白的5个保守基序(图3),包含motif 1的ItfMADS蛋白的比率为92.1%,motif 3所占的比率为89.5%,说明motif 1跟motif 3是ItfMADS蛋白中重要的保守基序;另外,包含motif 2的ItfMADS蛋白有31个,包含motif 5的成员最少,仅有5个。SMART功能注释分析表明,motif 1是MADS-box结构域,motif 3是K结构域。
利用软件TBtools对ItfMADS的基因结构进行分析(图3)。结果表明:有6个ItfMADS成员包括6个内含子和7个外显子,占比15.8%;8个成员没有内含子,占比21.1%;ItfMADS18的外显子和内含子数量最多,分别为11个和10个。
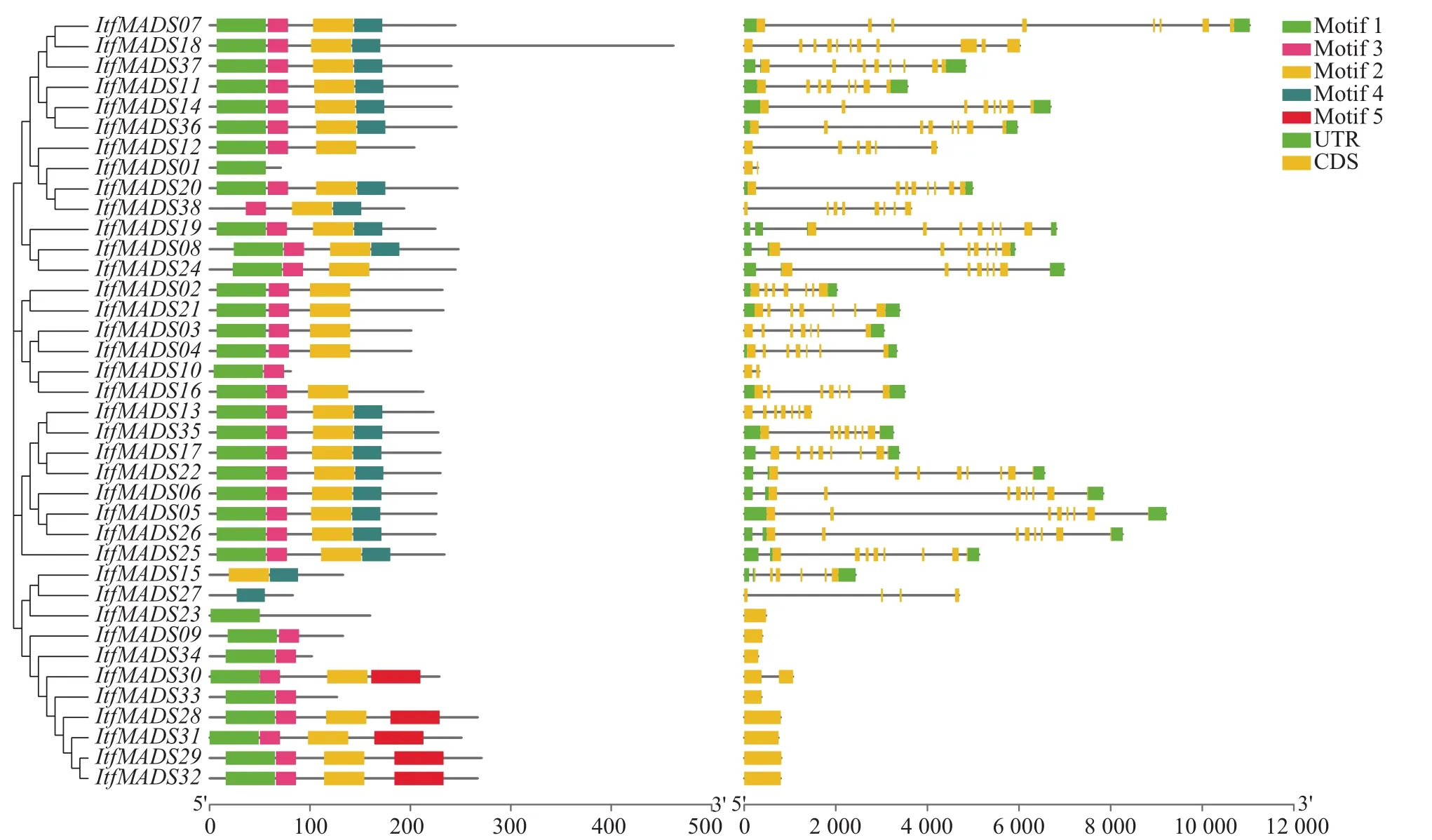
图3 ItfMADS基因结构和保守基序分析Fig.3 Analysis of structure and conserved motif of ItfMADS genes
2.5 染色体定位分析
通过MG2C在线软件对ItfMADS基因进行染色体定位分析(图4)。其中Chr00是由无序支架组成的[26]。通过基因定位发现:MADS-box基因家族中,Chr11染色体上的成员最多,有9个;其次是Chr01染色体,有5个;Chr02有4个成员,而Chr12和Chr00各仅有1个成员,分别为ItfMADS35和ItfMADS01。
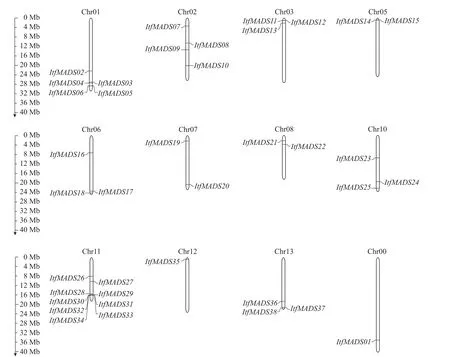
图4 ItfMADS基因家族的染色体定位Fig.4 Chromosomal locations of the ItfMADS gene family
2.6 启动子顺式作用元件预测分析
MADS-box基因的功能多样,可调节植物根、叶、花和果实的发育,且在生殖发育中控制花分生组织和花器官中的基因表达[27]。有研究发现,抑制或超表达胁迫应答的MADS-box基因都能够影响转基因植株的抗逆性,说明MADS-box基因不仅在植物生长发育中发挥至关重要的作用,还参与了胁迫响应过程[28]。因此,启动子顺式作用元件预测中,我们主要关注植物生长发育、逆境胁迫等相关元件。通过对ItfMADS基因启动子的顺式作用元件进行预测,并且对10个以上成员中都存在的顺式作用元件进行统计和分析(表4),发现38个成员中,除了普遍存在的启动子区域作用元件(CAAT-box和TATA-box)外,光响应元件(Box 4)和厌氧诱导元件(anaerobic response element,ARE)是在各成员中存在最多的两个顺式作用元件,分别与光响应和胁迫响应有关。另外,光响应类顺式作用元件的数量最多,有10个;激素响应类的顺式作用元件有5个;胁迫响应类顺式作用元件有3个。
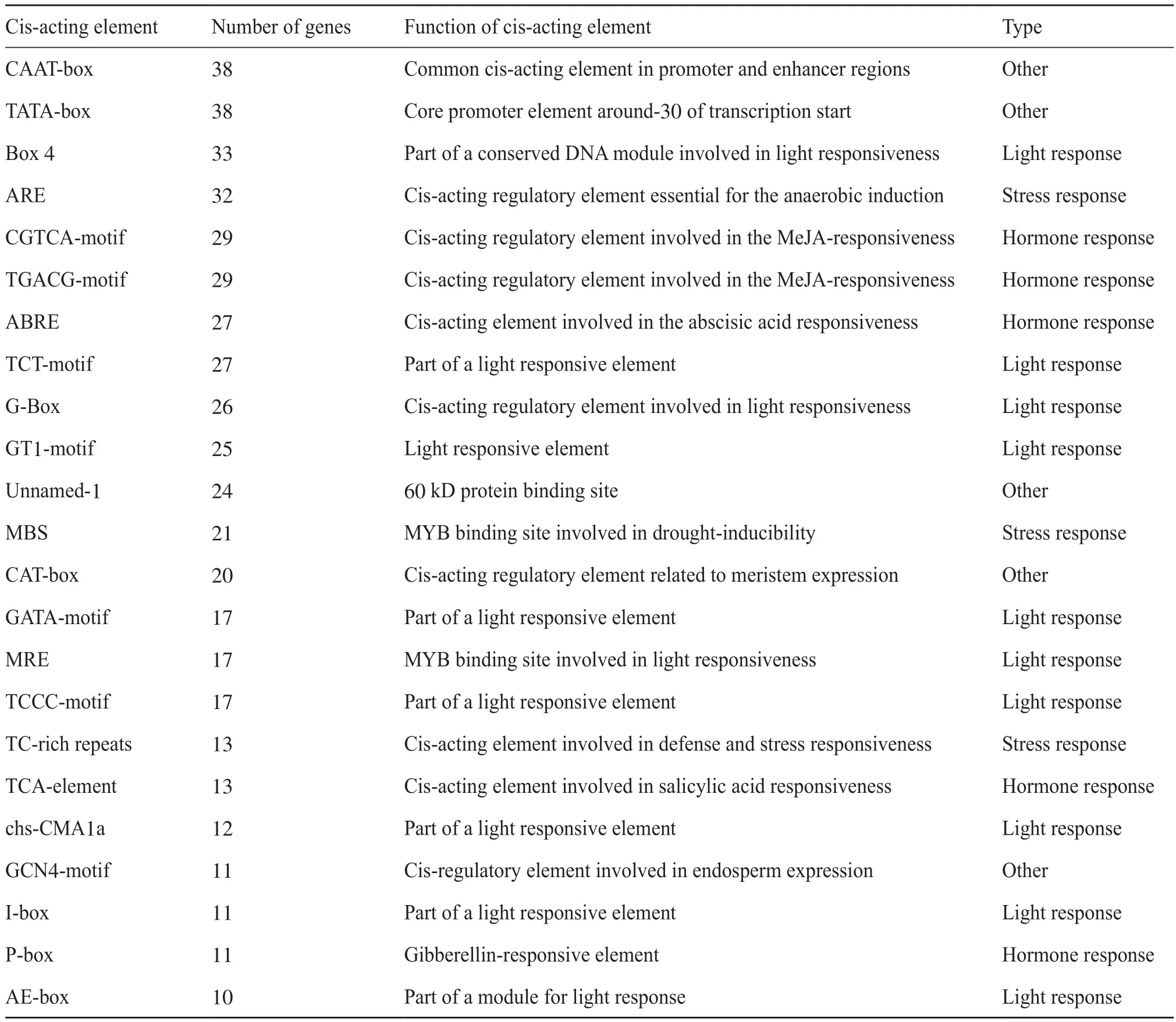
表4 启动子顺式作用元件预测分析Tab.4 Prediction analysis of promoter cis-action elements
2.7 组织特异性表达分析及qRT-PCR分析
利用RNA-seq数据,通过TBtools建立了由不同组织和器官(根、茎、叶)中的38个ItfMADS基因的表达热图,每千个碱基的转录每百万映射读取的碎片(fragments per kilobase of exon model per million mapped reads,FPKM)取log2值后作图(图5)。结果表明,13个ItfMADS成员(It-fMADS01、ItfMADS09、ItfMADS10、ItfMADS12、ItfMADS19、ItfMADS23、ItfMADS28、ItfMADS29、ItfMADS30、ItfMADS31、ItfMADS32、ItfMADS33和ItfMADS34)几乎不在根、茎、叶中表达,其余25个成员至少在一种组织中表达。一些ItfMADS成员在根、茎、叶中显示相似的表达模式,其中,3个ItfMADS成员(ItfMADS17、ItfMADS22和ItfMADS27)在根中表达量较高,ItfMADS17和ItfMADS22在根、茎、叶中均表达并且表达量较高,ItfMADS05和ItfMADS35在茎、叶中的表达量远高于根。综上,ItfMADS基因在三浅裂野牵牛的根、茎、叶中的表达存在差异,说明ItfMADS基因的表达具有组织特异性。
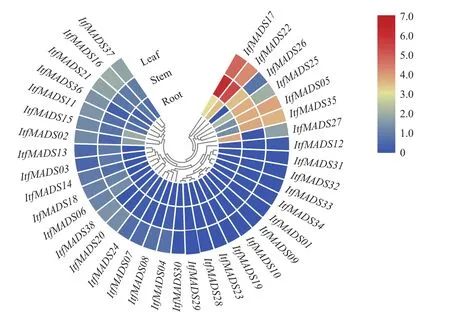
图5 ItfMADS基因在不同组织中的表达谱Fig.5 Expression profile of ItfMADS gene in different tissues
为了进一步验证转录组数据的可靠性,本试验进行了qRT-PCR验证。根据组织特异性表达分析,随机挑选9个ItfMADS基因(ItfMADS02、ItfMADS07、ItfMADS08、ItfMADS11、ItfMADS14、ItfMADS16、ItfMADS21、ItfMADS24和ItfMADS36),设计特异性引物,利用qRT-PCR分析它们在根、茎和叶中的表达情况(图6)。结果表明,部分基因的qRT-PCR与RNA-seq数据不一致,可能是由于转录组的假阳性造成的[29]。
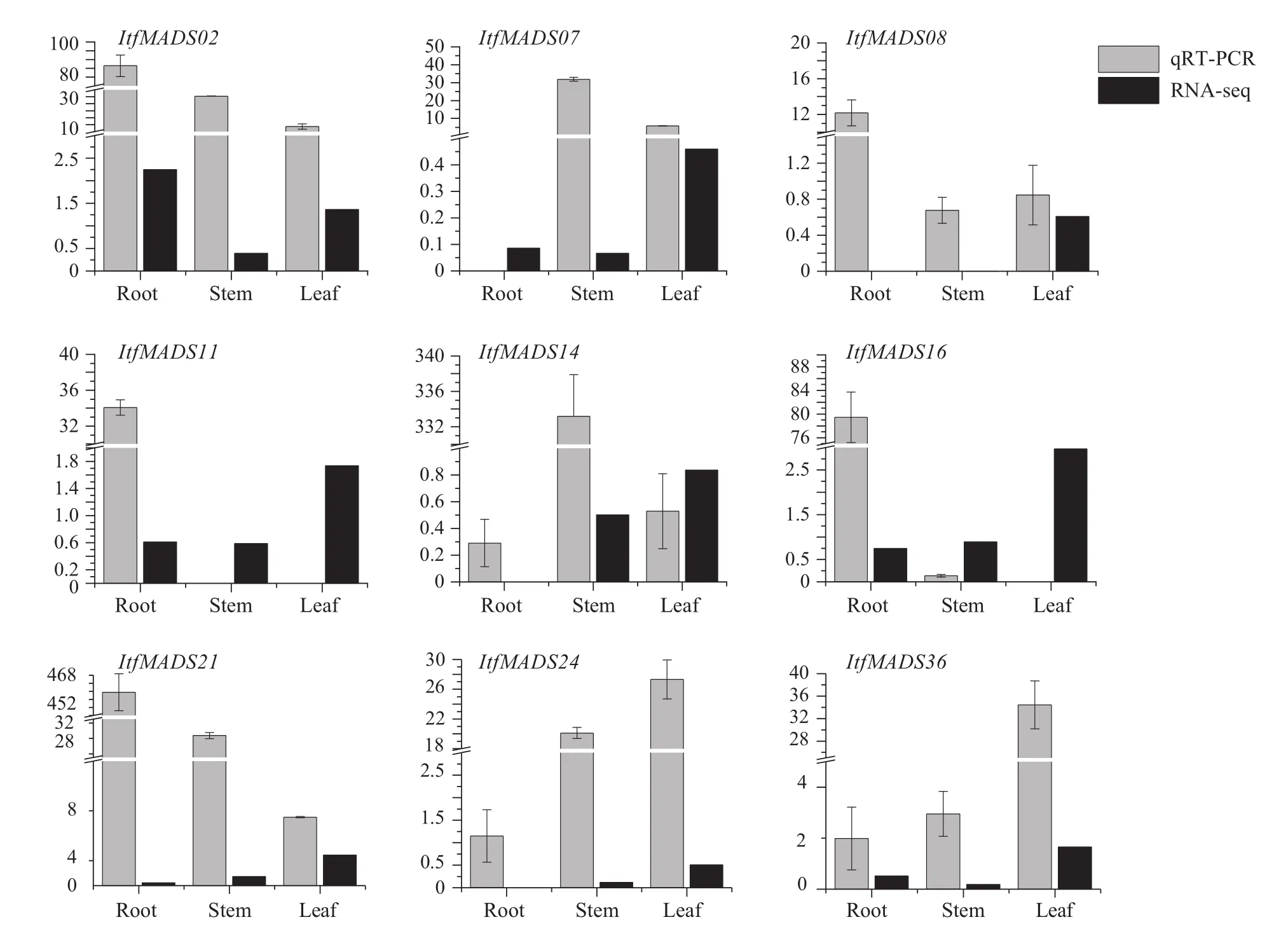
图6 qRT-PCR和RNA-seq结果Fig.6 The results of qRT-PCR and RNA-seq
2.8 ItfMADS基因在不同胁迫下的表达模式分析
我们利用下载到的生物和非生物胁迫的RNA-seq数据,通过TBtools构建了不同胁迫处理下38个ItfMADS基因的表达热图,以FPKM取log2值后作图(图7)。生物胁迫包括β-氨基丁酸(beta-aminobutyric acid)和苯并噻二唑(benzothiadiazoles-methylester)2种,分别用BABA,BTHT表示;非生物胁迫包括冷胁迫(cold stress)、热胁迫(heat stress)、干旱胁迫(mannitol drought stress)和盐胁迫(NaCl salt stress)4种,分别用COLD,HEAT,MANN,NACL表示。结果表明:2个ItfMADS成员(ItfMADS17和ItfMADS22)在各种胁迫下表达量均较高;ItfMADS05除了在生物胁迫中的β-氨基丁酸没有表达外,在其余胁迫下表达量均高;ItfMADS35在冷胁迫下的表达量相比于其他胁迫低;有17个ItfMADS成员在各种胁迫下均不表达。这说明,在生长受到胁迫时,古老的MADS-box基因能够响应胁迫以适应环境,但抵御胁迫的调控机理目前仍不清楚[30]。
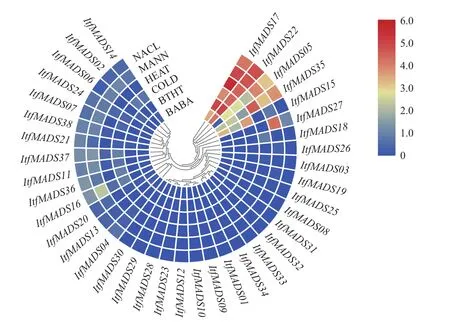
图7 ItfMADS基因在不同胁迫时的表达谱Fig.7 Expression profile of ItfMADS gene under different stresses
3 讨论
目前,许多学者已经对拟南芥[18]、葡萄[31]、芝麻[32]、苹果[33]和油菜[34]等多个物种的MADS-box基因家族进行了研究,但植物的生长发育和形态建成是一个极其复杂的过程,仍然还有许多难题尚未解决[7]。因此,深入研究MADS-box家族基因的功能对甘薯分子育种以及品种遗传改良具有重要意义。
本研究利用生物信息学方法在三浅裂野牵牛的基因组中鉴定到38个MADS-box基因,并对它们编码的蛋白质进行理化性质分析,获得ItfMADS各成员的基因结构、蛋白质二级结构、亚细胞定位、保守基序等信息,为甘薯MADS-box基因家族成员的克隆和功能研究提供了理论基础。基因结构分析表明,38个成员中有6个符合植物MADS-box基因的典型结构。蛋白质的二级结构是判断蛋白质稳定性的重要因素,其中,α-螺旋和β-折叠是蛋白质的有序结构,具有稳定性,而无规则卷曲为蛋白质的无序结构,具有不稳定性,该结果与理化性质中预测的蛋白质不稳定系数结果相一致。
参照模式物种中MADS-box家族的分类情况,在系统进化关系的基础上,可将这38个ItfMADS成员分为2类:Ⅰ型和Ⅱ型,其中,属于Ⅰ型的ItfMADS成员有9个,属于Ⅱ型的ItfMADS成员有29个。ItfMADS的分类情况与拟南芥和水稻不同,说明重复基因在不同物种进化过程中的保留情况不同,因此这些不同物种中同一分类的MADS-box基因在进化过程中受到的约束不同[35-36]。值得注意的是,从三浅裂野牵牛和拟南芥的系统进化树中,我们发现在ItfMADS家族中,Ⅰ型的9个成员均属于Mα亚家族,Mβ、Mγ、Mδ亚家族中均没有ItfMADS成员,ItfMADS成员空缺的原因还有待进一步研究。
植物中,Ⅱ型MADS-box基因的结构和功能研究比较清楚,但Ⅰ型MADS-box基因在植物生命过程的作用和机理还知之甚少。近年来,有研究表明Ⅰ型MADS-box基因是植物生长发育和繁殖过程的重要调控因子,在拟南芥的胚和胚乳等器官发育过程中起重要作用[37]。目前,研究最多且最为清楚的是著名的“ABCDE”模型,它揭示了MADS-box基因与花器官发育之间的关系。人们对“ABCDE”模型通俗解释为A+E控制萼片的发育,A+B+E控制花瓣的发育,B+C+E控制雄蕊的发育,C+E控制心皮的发育,D+E控制胚珠发育[30]。除少部分基因外,大多数A、B、C、D、E类同源基因都是Ⅱ型中MIKCC类的MADS-box基因。前人已经证明在水稻和拟南芥等模式植物中,A、B、C、D和E一共5类基因都参与调控植物的花器官发育,根据系统进化分析,三浅裂野牵牛的MADS-box同源基因也可能具有相似的功能。此外, 还有研究发现MADS-box基因参与落叶果树花芽休眠与休眠解除, 但其调控机制仍不清楚[38]。

