呼吸道菌群负担和多样性与危重患者预后的相关性分析*
魏立娜,李真玉,张 华,刘易峰
(1.天津医科大学第二医院,天津 300211;2.天津医科大学护理学院,天津 300070)
随着人们对微生态的认识加深,不同器官部位微生态变化与相应疾病进展的相关性逐渐得到关注,许多有价值的发现给临床工作带来了宝贵的启示[1]。呼吸道菌群由共生和病原微生物组成,年龄、病情严重程度、机体生理功能可影响肺部菌群的负荷、丰度和多样性[2],菌群特点变化在许多肺部疾病发生、进展、急性发作中起到重要作用[3-4],但具体机制尚不明确。研究表明,呼吸道菌群与慢性阻塞性肺疾病(COPD)进展、急性呼吸窘迫综合征(ARDS)发生和住院病死率相关,微生态平衡对疾病预后的作用不可忽视[5]。重症监护室(ICU)患者原发病重、全身情况差、常伴有感染,导致肺部微生物群落改变,后者反过来促进病情进展和不良预后发生[6]。然而,国内目前在这一领域少有相关研究,本文旨在深入探索ICU患者呼吸道菌群与死亡率相关性,丰富人体微生态研究,为ICU临床工作带来启示,现报道如下。
1 资料与方法
1.1 一般资料
选取2016年1月至2020年4月天津医科大学第二医院ICU收治的232例患者为研究对象。纳入标准:(1)因呼吸、心血管、神经系统疾病,以及肿瘤、中毒等引起的器官功能障碍、呼吸衰竭、顽固性心力衰竭等病情危重者;(2)年龄大于18岁;(3)ICU住院时间大于3 d至小于28 d;(4)因肺部感染、肺间质性疾病、痰液滞留等原因必要或自愿行支气管肺泡冲洗者;(5)患者或家属已签署知情同意书。排除标准:(1)具有近期活动性大咯血、活动性肺结核等支气管肺泡灌洗禁忌证者;(2)妊娠、近期有糖皮质激素或免疫抑制剂应用史者;(3)预计生存时间小于或等于24 h者;(4)严重创伤者。根据是否在ICU死亡分为存活组和死亡组。所有患者中男125例,女107例,平均年龄(49.78±17.26)岁;入ICU病因:呼吸系统疾病95例(40.95%),神经系统疾病31例(13.36%),心血管系统疾病43例(18.53%),消化系统疾病24例(10.34%),其他39例(16.81%)。本研究经过医院伦理委员会审核批准。
1.2 方法
1.2.1病例采集
排除临床资料不全和自动出院病例,收集患者入院病历中相关资料,包括:年龄、性别、既往史、急性生理功能和慢性健康状况评分系统Ⅱ(APACHEⅡ)评分、序贯器官衰竭估计(SOFA)评分、血生化检查结果等。所有患者于入ICU第1天行支气管肺泡冲洗,回收冲洗液以行DNA提取。
1.2.2APACHEⅡ和SOFA评分[7]
根据患者年龄、血压、肝肾功能、手术史等资料进行APACHEⅡ评分,评分越高表示全身情况越差。SOFA评分涉及呼吸、凝血、肝脏、神经、泌尿、循环系统,每个系统0~4分,总分24分,评分越高则器官功能越差。
1.2.3呼吸道菌群标本采集与DNA提取
入ICU 24 h内应用生理盐水对患者行支气管肺泡冲洗操作,回收冲洗液,4 ℃保存。将冲洗液于14 000 r/min离心10 min,弃去上清液,沉淀物用200 μL的无菌磷酸盐缓冲液(PBS)重悬。应用DNA提取试剂盒(德国QIAGEN公司)和PCR扩增试剂盒(加拿大Fermentas公司)抽提悬浮物中DNA,操作依据说明书。
1.2.4引物设计和PCR扩增
细菌基因组16S rDNA的V3~V5区序列由GenBank数据库[8]提供,片段长度550 bp,引物由生工生物工程(上海)股份有限公司合成。应用上述引物进行PCR扩增,PCR反应程序为预变性(95 ℃、3 min),变性(95 ℃、30 s),退火(56 ℃、30 s),延伸(72 ℃、30 s),行35个循环,于4 ℃下保存。
1.2.5测序结果
行16S rDNA拷贝数、丰度、多样性分析,扩增产物经纯化后由深圳华大基因研究院完成16S rDNA宏基因组测序与定量,在PCR产物中加入样品标签序列,然后行混合测序,根据基因表达标签分类细菌物种、分析群落丰度、多样性等。菌落多样性采用Mothur软件计算,应用Alpha多样性分析根据实际观测到的OTU数目评估,群落丰度观察指标选取chao、sobs、ace指数,数值越大则丰度越高;群落多样性采用simpson和shannon指数评估,前者数值越大则多样性越低,后者反之。
1.3 统计学处理
2 结 果
2.1 两组一般资料比较
存活组和死亡组各排除资料不完整或自动出院者7例和2例,最终纳入223例。58例(26.01%)于ICU内死亡,其中11例直接死因为感染,其余死于原发病进展;死亡组APACHEⅡ评分、SOFA评分、急性肾损伤比例均高于存活组(P<0.05),见表1。
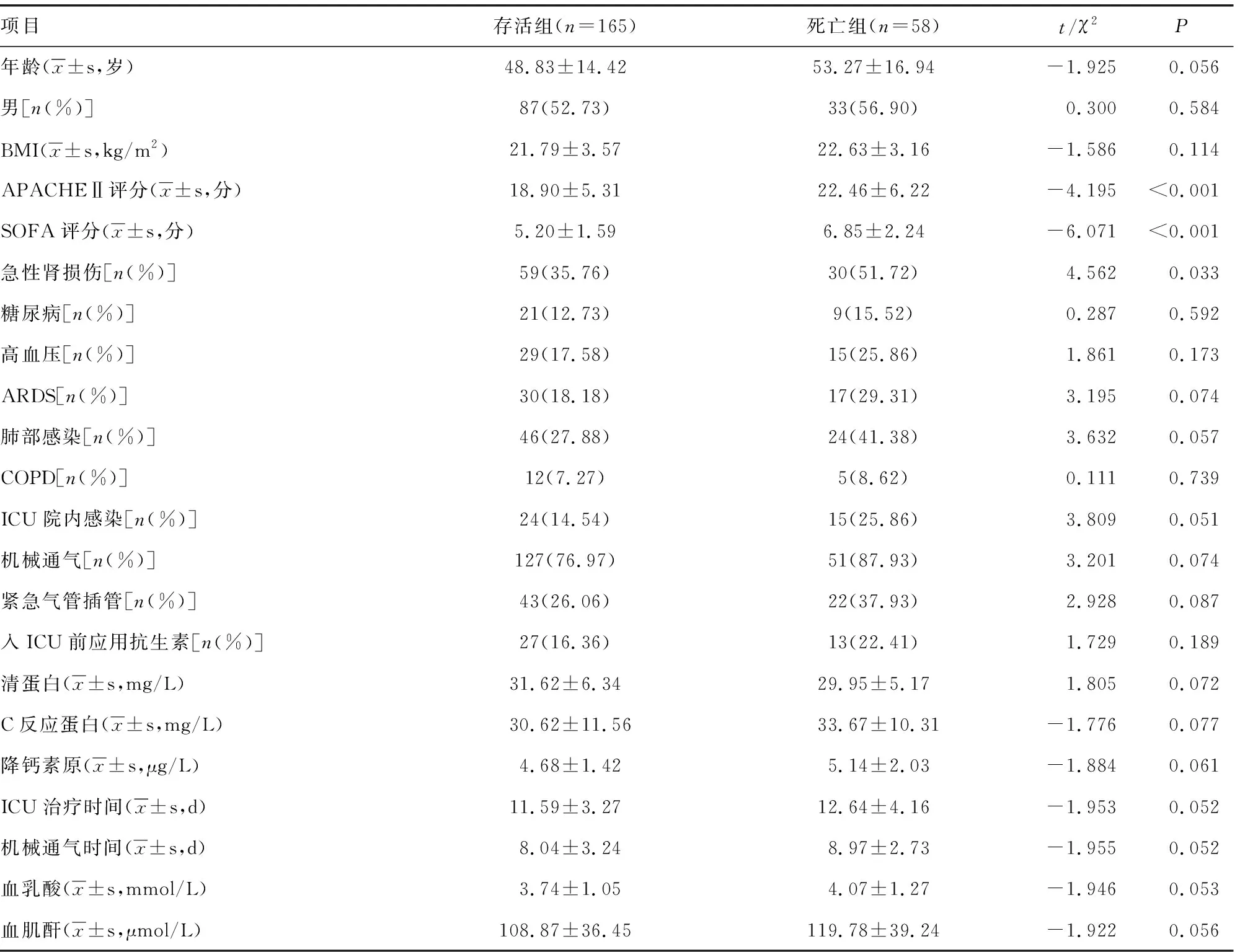
表1 两组一般资料比较
2.2 两组呼吸道菌群负荷和多样性比较
死亡组16S rDNA拷贝数、simpson指数高于存活组,chao、sobs、shannon、ace指数低于存活组,差异有统计学意义(P<0.05),见表2。
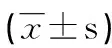
表2 两组呼吸道菌群负荷和多样性比较
2.3 ICU患者死亡相关因素logistic回归分析
APACHEⅡ评分、SOFA评分、16S rDNA拷贝数为ICU患者死亡危险因素(P<0.05);chao指数、sobs指数、shannon指数、ace指数为保护因素(P<0.05),见表3。
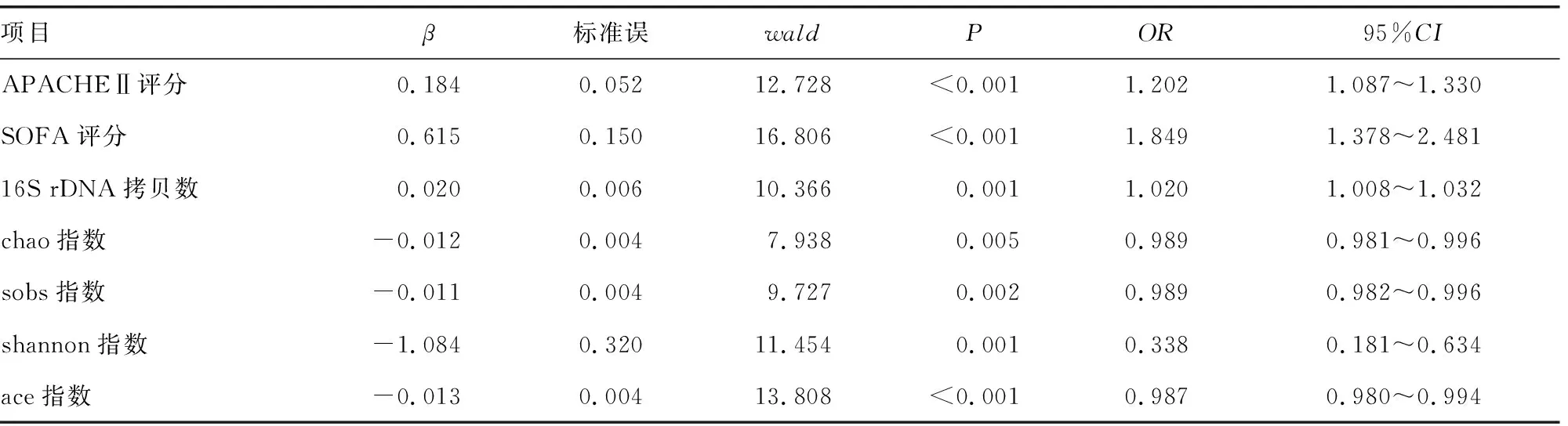
表3 ICU患者死亡相关因素logistic回归分析
3 讨 论
呼吸道拥有复杂的菌群系统,越来越多的学者发现肺部微生态平衡遭到破坏后,促进了肺部疾病和不良结局的发生[9-10]。ICU患者作为一个预后普遍较差的群体,所具有的病情不稳定、生理紊乱严重、致病机制复杂等因素严重破坏了人体微生态平衡,既往研究多集中于肠道菌群,而失衡的肺部菌群环境推动了ARDS、呼吸机相关性肺炎、不良预后的发生[11-12],呼吸道微生态或在促进患者死亡中扮演着特殊的角色[13]。因此,本文基于此点,挖掘ICU患者呼吸道菌群与死亡率的相关性,旨在为干预患者预后带来新的思路。
良好的生理状态和器官功能是支撑ICU患者好转的基础,本研究中死亡组APACHEⅡ评分和SOFA评分均高于存活组,APACHEⅡ评分和SOFA评分为ICU患者死亡危险因素。APACHEⅡ评分和SOFA评分可准确反映患者机体状态和器官功能,与预后相关,两者已被广泛应用于许多疾病和危重病患者的预后评估中[14-15]。王力鹏等[16]报道APACHEⅡ和SOFA评分与急诊重症患者死亡风险相关,可有效预测预后,支持了本研究结论。
本研究对比ICU内死亡患者和存活患者的呼吸道菌群负荷,观察到死亡组16S rDNA拷贝数高于存活组,16S rDNA拷贝数为ICU患者死亡危险因素。ICU患者在身体机能欠佳、生理状态改变、感染风险高的基础上长时间接触大量病原体,这些都可能是呼吸道细菌数量增多的原因,不论是哪一者都将影响患者预后。DICKSON等[17]报道在脓毒症、ARDS、重症疾病状态下,肺部菌群负荷增多、群落组成改变,其中最突出的变化是肺内肠杆菌科成为优势菌群,而本研究未对ICU患者呼吸道菌群成分进行分析、未明确呼吸道菌群负荷增多原因,有待完善。
肺部微生态的稳定主要取决于菌群的组成、丰度和多样性,本文中死亡组simpson指数高于存活组,chao、sobs、shannon、ace指数均低于存活组;chao指数、sobs指数、shannon指数、ace指数为ICU内死亡的保护因素。死亡组入ICU 24 h内菌群丰度和多样性较低,可能是由致病菌繁殖和共生菌减少引起,并与预后相关。BUDDEN等[18]报道发生肺炎、肺囊性纤维化、ARDS等疾病时,患者呼吸道优势菌群和菌群多样性发生改变,这种改变与感染、病情进展可能存在相互作用的关系。另有研究表明肺部菌群负荷、组成成分、多样性与ICU患者未使用呼吸机时间相关,肺内肠杆菌的富集与患者发生ARDS有关,肺部菌群负荷、组成成分、多样性可预测ICU患者预后[19]。这些结论均给本研究提供了一定理论支持。本文通过细菌载量、丰度、多样性证实ICU死亡患者肺部菌群变化,升高的细菌载量和降低的多样性反映了菌群组成变动,可能伴随着优势菌群的改变,但本文未探索患者肺部菌群组成,此乃不足之处。同时本研究论证了危重病患者肺内菌群状态与预后相关,关于人体微生态的研究往往集中于肠道菌群,下呼吸道菌群由于采样困难常被忽视,但越来越多的证据指出肺部微生态平衡可能是改善许多疾病患者预后的重要途径[20]。然而,呼吸道菌群在ICU患者发病、进展、死亡过程中的具体机制尚未明确,可能是通过推动感染和ARDS发生加重器官功能负担,亦或是危重病情与肺部菌群相互作用的结果,需更多研究加以证实。
综上所述,呼吸道微生态平衡和ICU患者病死率相关,深入对肺部菌群变化的探索,有助于为危重病患者提供更多改善方案。

