基于生物信息学耐吉非替尼非小细胞肺癌细胞的差异表达基因分析▲
黄溢问 吴 凤 阳 联 张文峰 贾家宝 孙 易 庞伟毅
(桂林医学院公共卫生学院,广西桂林市 541000,电子邮箱:1278769309@qq.com)
据《全球癌症数据2018》[1]的统计结果,肺癌的全球发病率和全球死亡率在所有癌症中均居首位,其中2018年有210万例新发肺癌患者,180万例患者死于肺癌,由此可见该病严重增加了全球卫生体系的负担。肺癌可分为小细胞肺癌和非小细胞肺癌(non-small cell lung cancer,NSCLC),以后者最为常见。以吉非替尼为代表的肺癌靶向药物出现后,患者中位生存期显著延长,不良反应也大大减少[2-3]。吉非替尼是一种表皮生长因子受体(epidermal growth factor receptor,EGFR)酪氨酸激酶抑制剂(tyrosine kinase inhibitor,TKI),可抑制肿瘤生长,但由于肿瘤的异质性等众多因素,吉非替尼也出现了耐药现象,但其机制至今仍未完全阐明[4]。近年来,基因芯片和测序亦作为新兴技术之一,可以在极短的时间内产生大量的数据;但同时这些数据也夹杂着大量的错误信息,传统的逐个基因的探索方法也因此显得耗时且低效[5]。这使得学者们开始重视信息的规范化,以及对数据的管理及分析,因此传统的生物化学实验室逐渐向生物信息学领域发展,也由此衍生了计算生物学、生物信息学等新型分析方法;这些方法已被用于肿瘤的分类和其相关分子机制的探索,其有效性及高效性在一定程度上规避了传统生物化学方法的弊病。本研究利用基因芯片数据,运用生物信息学工具分析肺癌耐药相关的通路和基因,为吉非替尼耐药机制的研究提供线索和依据。
1 资料与方法
1.1 数据来源 在GEO数据库(https://www.ncbi.nlm.nih.gov/geo/)中下载基因表达谱数据集GSE122005。该数据集以人NSCLC细胞HCC827作为样本,一共6份样本,由3份吉非替尼敏感细胞样本(Gefitinib1、Gefitinib2、Gefitinib3)和3份获得性吉非替尼耐药细胞样本(Acquired1、Acquired2、Acquired3)构成,该基因表达谱数据集采用Affymetrix公司的平台GPL570[HG-U133_Plus_2]Affymetrix Human Genome U133 Plus 2.0 Array进行测定。
1.2 数据质量检测 使用R语言3.5.2处理原数据集,将基因探针ID转化为对应的基因名称后获得表达矩阵,绘制表达分布图、样本聚类图,并进行主成分分析,从而进行数据质量分析。
1.3 差异表达基因的筛选 使用R语言的Limma包对表达矩阵进行差异分析,并筛选出差异表达基因。
1.4 差异表达基因的富集及通路分析 使用在线工具DAVID 6.7(https://david.ncifcrf.gov/)和KOBAS 3.0(https://kobas.cbi.pku.edu.cn/kobas3),导入差异表达基因,查看并导出基因本体论(Gene Ontology,GO)富集分析和京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)信号通路分析结果[6]。
1.5 蛋白质互相作用网络的构建 将差异表达基因批量导入在线工具STRING 11.0(https://string-db.org/),生成并导出蛋白质互相作用(protein-protein interaction,PPI)网络,使用CytoScape 3.7.1中的cytoHubba插件计算每个节点的核心程度,根据评分进行排名,取排名前10的基因作为关键基因。
1.6 生存曲线分析 选取关键基因,以基因的中位表达水平作为分组界限,以总生存期作为统计指标,利用在线工具Kaplan-Meier Plotter(http://www.kmplot.com/)绘制不同基因表达水平的NSCLC患者的生存曲线。
2 结 果
2.1 数据质量的检测结果 数据集的表达分布图显示各样本的表达量恒定,样本聚类图、主成分分析可以清晰地区分出非替尼敏感细胞样本(Gefitinib1、Gefitinib2、Gefitinib3)和获得性吉非替尼耐药细胞样本(Acquired1、Acquired2、Acquired3)。见图1。

图1 吉非替尼敏感细胞样本和耐药细胞样本的芯片数据质量检测
2.2 差异表达基因的筛选结果 本研究以调整后P值≤0.01、|log2FC|≥1.5作为筛选差异表达基因的条件,最终获得604个差异表达基因,其中表达上调基因332个,表达下调基因272个,见图2。

图2 两个样本间差异表达基因的火山图
2.3 GO富集分析结果 以P<0.01作为检验水准,结果共富集到98项有统计学意义的GO条目。其中,涉及9种分子功能,包括生长因子结合、糖蛋白结合等;17种细胞组成,包括核小体、细胞外空隙、细胞外区域部分、质膜部分等; 72种生物过程,包括核小体装配、创伤反应等。见表1。
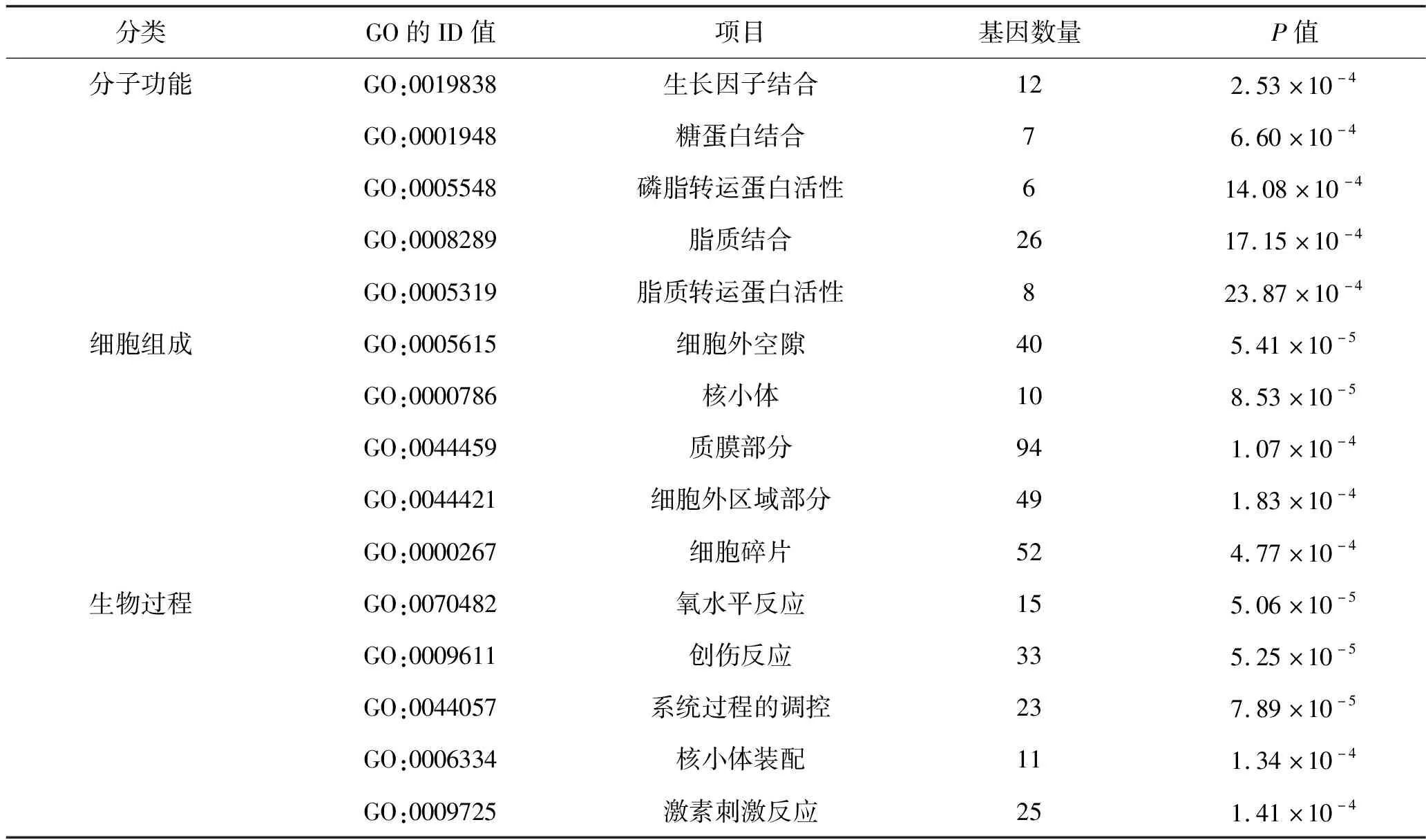
表1 差异表达基因的部分GO富集分析结果
2.4 KEGG信号通路分析结果 以调整后P值<0.01作为检验水准,共发现37条通路具有统计学意义,包括癌症中的转录失调、肿瘤坏死因子信号通路等。见表2。

表2 差异表达基因的部分KEGG信号通路分析
2.5 PPI网络 PPI网络中共有555个节点,1 446个相互作用,见图3;排名前10的基因包括C-X-C基序趋化因子配体[chemokine(C-X-C motif)ligand,CXCL]8、Erb-B2受体酪氨酸激酶(Erb-B2 receptor tyrosine kinase 2,ERBB2)、组织金属蛋白酶抑制剂1(tissue inhibitor of metalloproteinases 1,TIMP1)、分泌性磷蛋白(secreted phosphoprotein 1,SPP1)、CXCL1、钙黏着蛋白2(cadherin 2,CDH2)、C-C基序趋化因子配体[chemokine(C-C motif)ligand,CCL]2、内皮素1(endothelin 1,EDN1)、CCL20、集落刺激因子2(colony-stimulating factor 2,CSF2),见图4。
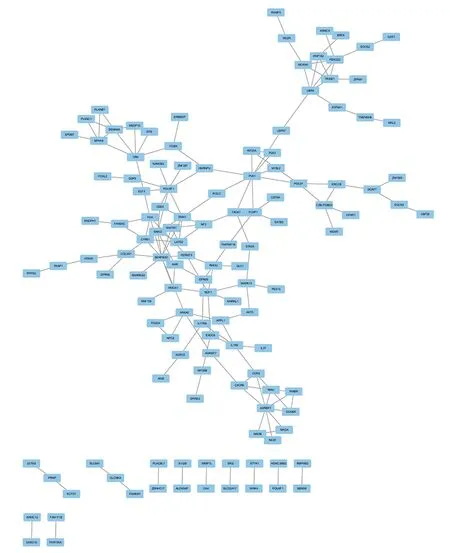
图3 差异表达基因的PPI网络
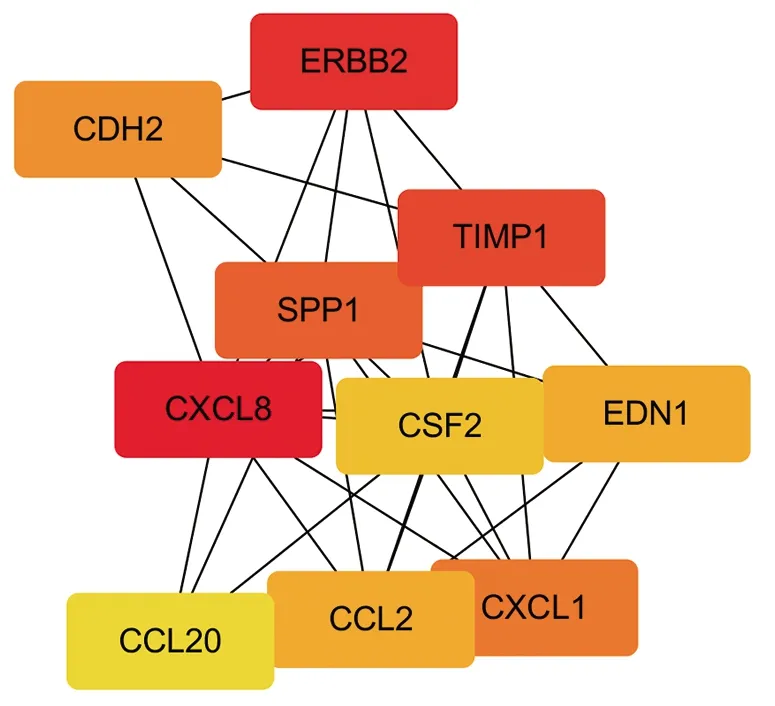
图4 关键基因
2.6 生存分析结果 Kaplan-Meier Plotter分析结果显示,与预后相关的基因包括CXCL8、EDN1、SPP1、TIMP1,log-rank检验提示均P<0.01,见图5。CXCL8、SPP1、TIMP1高表达或EDN1低表达的NSCLC患者,总生存期更短,预后较差;与差异分析结果进行对比,在耐药细胞中CXCL8、SPP1、TIMP1为表达上调基因,EDN1为表达下调基因。

图5 生存曲线
3 讨 论
吉非替尼等EGFR-TKI药物的出现极大地改善了EGFR变异的NSCLC患者的生存状况,然而如大多数肿瘤一样,NSCLC对吉非替尼也产生了耐药性。耐药的产生是一个多因素诱导的复杂过程,有文献表明,最早发现的耐药机制与EGFR二次突变(T790M突变等)有关,其中超过一半的EGFR-TKI变异所致耐药病例为T790M突变;此外,选择性旁路的激活、下游目标的激活和上皮间质转化也被证实与吉非替尼耐药存在一定相关性[7]。
本研究共筛选出与NSCLC对吉非替尼耐药有关的差异表达基因604个,其中表达上调基因332个,表达下调基因272个。利用筛选出的差异表达基因进行富集分析和通路分析,结果显示这些差异基因主要参与生长因子结合、糖蛋白结合、磷脂转运蛋白活性、脂质结合、脂质转运蛋白活性等分子功能,参与氧水平反应、创伤反应、系统过程的调控、核小体装配、激素刺激反应等生物过程,其细胞组分主要有细胞外间隙、核小体、质膜部分、细胞外区域部分、细胞碎片等;信号通路分析显示代谢途径、癌症通路、补体系统、癌症中的转录失调、肿瘤坏死因子等与吉非替尼耐药有关,但仍需进一步的实验研究以证实。
根据PPI网络获得10个核心基因,包括CXCL8、ERBB2、TIMP1、SPP1、CXCL1、CDH2、CCL2、EDN1、CCL20、CSF2。(1)CDH2与多种肿瘤进展、转移有关。肺鳞癌和肺腺癌有着完全不一样的预后结局,有学者对肺腺癌和肺鳞癌之间的表达差异蛋白质进行分析发现,CDH2在肺腺癌中显著上调,而在肺腺癌的血管内皮细胞中上调更为显著;肺腺癌血管生成现象明显,丝裂原活化蛋白激酶/胞外调节蛋白激酶和丝裂原活化蛋白激酶/c-Jun氨基末端激酶信号通路可能在CDH2诱导的低氧诱导因子1α和血管内皮生长因子介导的血管生成过程中扮演重要角色,这可能是肺腺癌总生存期相对较短的原因[8]。(2)ERBB2和EGFR类似,都是生长因子受体酪氨酸激酶亚家族的成员,也是乳腺癌和卵巢癌的高危基因。临床上约有10%的肺腺癌患者存在REBB2突变,该型患者的预后差,生存期短[9]。(3)SPP1又称骨桥蛋白,基因定位于人染色体4q上,主要参与调节生理过程,如发育、分化、炎症和伤口愈合[10],其表达对多种癌症的发生和转移有重大意义,如肺癌、卵巢癌、胃肠癌、前列腺癌等,且可以作为NSCLC分期和预后评估的指标,其中SPP1高表达的NSCLC患者预后较差[11]。(4)EDN1是促炎性细胞因子,具有血管收缩活性、促进细胞外基质合成的作用,在器官纤维化和氧化应激方面也起着很重要的作用[12-13]。由于EDN1能诱导黑色素瘤细胞凋亡,以及前列腺癌中EDN1受体沉默,故有学者推测EDN1表达量的降低有利于肿瘤细胞生长[14]。目前已有生物信息学分析结果提示EDN1与NSCLC的发生机制有密切关系[15],但尚无与吉非替尼耐药相关性的证据。(5)TIMP 是基质金属蛋白酶的抑制因子,作用于细胞外基质,减少基质金属蛋白酶的降解,增强细胞间粘连,与肿瘤的转移和患者预后相关[16]。TIMP1是其成员之一,其靶分子为基质金属蛋白酶1。在临床中高表达TIMP1常常是多种肿瘤预后不良的征象[17],但同属于一个家族的TIMP2的高水平表达却提示预后良好,恶性程度较低[18]。(6)CCL2、CCL20、CXCL1、CXCL8都是趋化因子的成员。CCL2是趋化因子CC亚家族成员之一,是一类在人体的生理机能中发挥着重要作用的小分子蛋白,多由免疫细胞和神经胶质细胞等分泌,具有化学趋化活性。近年来,有研究表明CCL2受到血小板源性生长因子的自分泌回路调控,导致肿瘤转移相关基因表达量的增加,从而增强肿瘤的侵袭性[19]。CCL20 可以自分泌的方式通过丝裂原活化蛋白激酶和磷脂酰肌醇 3-激酶信号通路,与肿瘤细胞增殖及扩散有关[20],高水平表达的CCL20与肺癌患者预后不良有关[21]。CXCL1 在肺癌组织中通常呈高表达,随着其表达量升高,肿瘤相关中性粒细胞数量增加并浸润肺癌组织,从而促使肿瘤生长[22]。有研究显示,在合并恶性胸腔积液的NSCLC患者中,T细胞数量和CXCL1水平显著增高;CXCL1的表达受到微小RNA-141调控,微小RNA-141通过微小RNA-141-CXCL1-CXCR2通路可调控T细胞向胸腔积液转移[23]。CXCL8 主要作用于C-X-C基序趋化因子受体1 和2、达菲抗原趋化因子受体,其也与多种肿瘤的发生、发展有关[24]。由此可见,上述差异表达的核心基因都与肺癌等多种肿瘤的发生、发展、预后相关。此外,我们还在生存分析中发现CXCL8、SPP1、TIMP1高表达和EDN1低表达的NSCLC患者总生存期短于相应的低表达组或高表达组,这4个基因在预后较差组的表达水平恰好与耐药样本的表达水平相一致(同为高表达或同为低表达),提示了CXCL8、SPP1、TIMP1和EDN1可能在吉非替尼耐药机制中发挥重要作用。但是目前尚无相关研究报告其中的作用机制;同时在吉非替尼耐药的机制中,有关这些基因或蛋白的研究也相对较少。因此本研究结果虽然提示这些差异表达的基因在吉非替尼耐药机制中可能扮演重要角色,但其中的作用机制还需进一步研究以探讨。
综上所述,本研究筛选出的差异表达基因中,CXCL8、SPP1、TIMP1、EDN1可能在NSCLC对吉非替尼耐药的机制中起重要作用,但具体的作用机制有待进一步研究。本研究结果存在一定局限性:所分析的样本仅来源于细胞,样本量少,且仅从计算生物学方法的角度进行分析,因而所得差异表达基因在NSCLC对吉非替尼耐药中的作用还需进一步研究验证。

