柯萨奇病毒B4型VP1蛋白的生物信息学分析①
陈泳蓓,陆俏岑,何韵怡,陈柏希,吴 岳,刘洪波③
(桂林医学院a.第二附属医院检验科;b.临桂临床医学院,广西 桂林 541199)
肠道病毒被认为是引起1型糖尿病(type 1 diabetes mellitus,T1DM)的主要致病因素之一,其中以柯萨奇病毒B4型(CVB4)最为常见[1]。研究表明,CVB4感染与T1DM发病存在相关性,但CVB4致T1DM的机制仍未明确。1979年,Yoon等[2]从1名T1DM患者的胰腺组织中分离出CVB4,随后在T1DM患者中发现了CVB4的结构蛋白VP1[3]。因此,认为VP1蛋白可能是CVB4引起T1DM的重要分子和CVB4功能和表位所依托的分子[4]。笔者以从胰岛中分离得到的CVB4株的VP1蛋白作为研究对象,对其进行生物信息分析,预测其基本理化性质、相关功能结构以及B细胞线性表位,为肠道病毒引起T1DM的机制研究提供生物信息学基础。
1 资料和方法
1.1 CVB4 VP1氨基酸序列获取和生物信息工具
从NCBI数据库(https:∥www.ncbi.nlm.nih.gov/)下载28条CVB4全长氨基酸序列,并以新发T1DM患者胰岛中分离得到的CVB4 Tuscany株[3](登录号为ABF19105.1)VP1蛋白为研究对象,利用Bioedit、DNAstar、B细胞线性表位预测在线服务器ABCpred(https:∥ webs.iiitd.edu.in/raghava/abcpred/ABC _submission.html)、免疫表位数据库网站IEDB(http:∥tools.iedb.org/bcell/)以及生物信息学门户ExPASy(https:∥www.expasy.org/proteomics)提供的多种在线工具对其进行分析[5]。
1.2 CVB4 VP1蛋白的基本理化性质分析
利用Bioedit对VP1氨基酸的组成进行分析。利用ProtParam预测VP1的分子组成、相对分子质量、等电点、消光系数、不稳定系数,以及在哺乳动物网织红细胞、酵母和大肠杆菌中的半衰期等。
1.3 CVB4 VP1蛋白的结构功能和B细胞线性表位预测
以VP1为研究对象,应用SignalP进行信号肽分析;Tmpred和TMHMM进行跨膜螺旋预测;NetPhos分析磷酸化位点;MotifScan分析乙酰化位点;Net-NGlyc分析糖基化位点;TargetP和NetNES分析亚细胞定位;SMOPA和Predictprotein分析二级结构;利用ABCpred和IEDB对线性B细胞表位进行综合预测,利用MEGA将预测到的表位序列与下载的28条CVB4全长氨基酸序列进行序列比对,分析表位保守性。
2 结果
2.1 CVB4 VP1 的氨基酸组成
应用Bioedit软件对VP1的氨基酸组成进行分析发现,VP1由284个氨基酸组成,其中Ser 10.18%、Thr 9.12%、Val 7.37%,这3种氨基酸含量最多。见图1。

图1 VP1氨基酸组成
2.2 CVB4 VP1的理化性质
利用ProtParam工具预测,VP1蛋白的分子组成为C1410H2173N395O434S15,理论相对分子质量为32 083.05,等电点为8.31。若全部胱氨酸残基以Cys形式形成二硫键,则其在水溶液中(A280 nm)的摩尔消光系数为46 090 L·mol-1·cm-1;若所有二硫键打开,其在水溶液中(A280 nm)的摩尔消光系数为45 840 L·mol-1·cm-1。若VP1的N端为Gly,在哺乳动物体外网织红细胞中的半衰期为30 h,在酵母体内>20 h,在大肠杆菌内>10 h,其不稳定系数为46.20,属于不稳定蛋白。
2.3 CVB4 VP1蛋白的功能相关结构
应用SignalP分析,提示VP1为非分泌性蛋白。应用Tmpred和TMHMM进行跨膜螺旋分析,发现VP1无跨膜螺旋结构。应用NetPhos分析,发现VP1有36个磷酸化位点。应用MotifScan分析,发现VP1无乙酰化位点。应用Net-NGlyc进行糖基化位点分析,显示VP1有2个糖基化位点。应用TargetP进行亚细胞定位分析,提示VP1定位于线粒体。应用NetNES核定位分析,发现VP1无核定位信号。
2.4 CVB4 VP1蛋白的二级结构及抗原表位
应用SMOPA和Predictprotein在线工具对VP1蛋白进行二级结构分析,发现VP1的螺旋、折叠、转角、无规则卷曲分别占29.82%、24.56%、10.18%、35.44%。运用Predictprotein分析,发现VP1的螺旋、折叠、无规则卷曲分别占8.77%、23.86%、67.37%。结合两种方法的预测分析,可知螺旋和折叠是VP1蛋白的刚性支撑,无规则卷曲是VP1的主要结构,是VP1抗原表位存在的可能位置。
2.5 VP1蛋白的B细胞线性表位
应用ABCpred预测VP1蛋白的B细胞线性表位,以0.85为阈值,结果有10条肽入选(表1)。在线预测网站IEDB分析,得到VP1蛋白的β-转角、表面可及性、柔韧性、抗原性、亲水性等抗原表位数据(图2)。综合ABCpred预测和IEDB分析,推测B细胞线性表位较可能位于33~49、120~136、141~157、200~216、235~251位氨基酸区段。利用MEGA对这5条预测到的优势表位序列进行型内保守性分析,显示这些表位均具有较高的保守性,其中141~157、200~216位氨基酸区段在CVB4内完全保守,33~49、120~136、235~251位氨基酸区段均仅存在1个氨基酸突变。见图3。
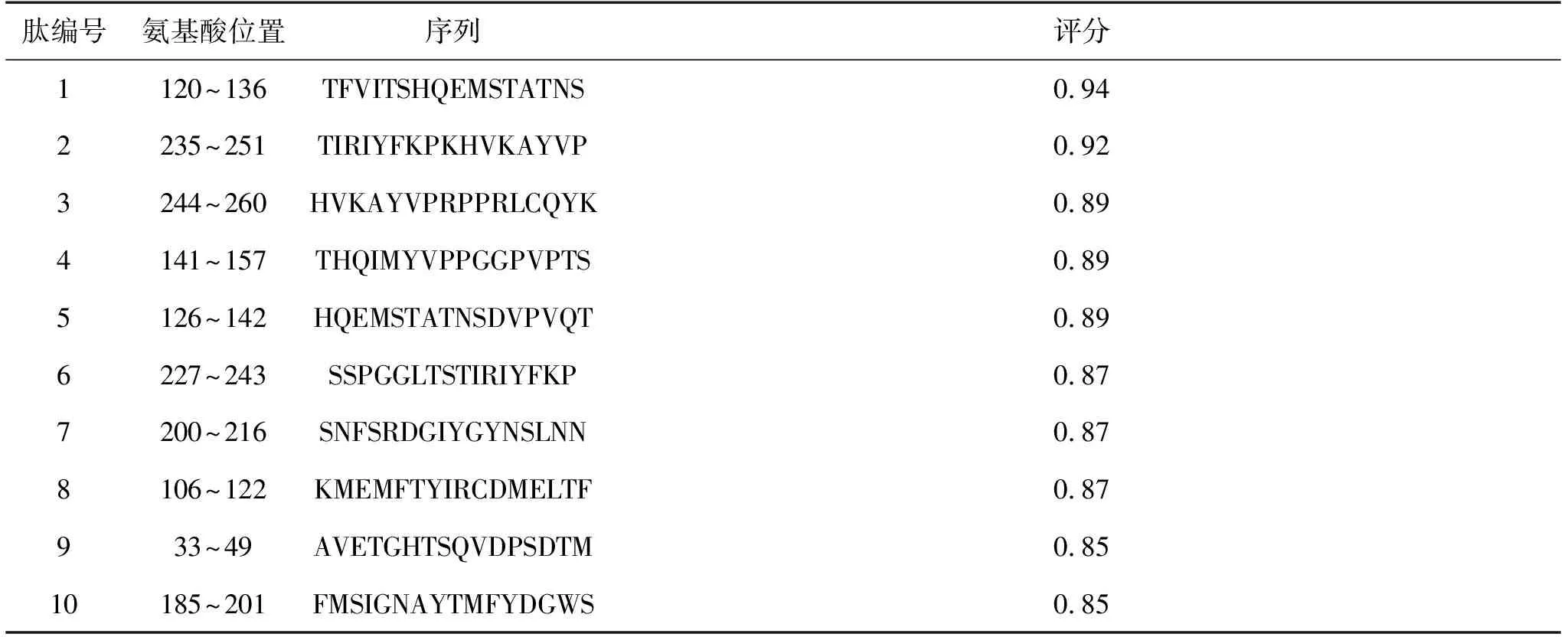
表1 ABCpred预测VP1的线性B细胞表位

A:β-转角;B:表面可及性;C:柔韧性区域;D:抗原性;E:亲水性图2 IEDB分析VP1的抗原表位
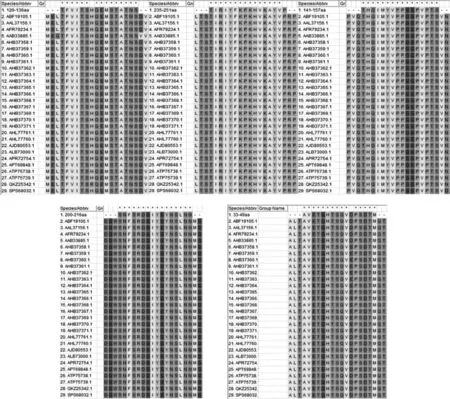
图3 MEGA分析优势表位序列的保守性
3 讨论
T1DM是一种受遗传和环境因素共同影响的自身免疫病,以T细胞介导胰岛β细胞损伤引起自身免疫破坏为特征。肠道病毒感染在T1DM发病中具有重要作用,特别是CVB4[6-7]。VP1是肠道病毒的结构蛋白,新发T1DM患者的胰岛组织中能检测出VP1蛋白,且其表达高于对照组[8]。提示VP1与T1DM发病可能存在相关性。本文以新发T1DM患者胰岛中分离的CVB4 Tuscany株为研究对象,通过多种在线工具分析CVB4 VP1蛋白的基本理化性质,结构功能特征、B细胞线性表位以及其三维结构,直观地显示了CVB4 VP1蛋白的生物学信息,为CVB4致T1DM的相关研究提供数据参考。
肠道病毒结构蛋白VP1与酪氨酸磷酸酶IA-2存在相同序列“ALTAV”[9]。本研究中,28条CVB4全长氨基酸序列的比对结果也显示了“ALTAV”序列在CVB4中完全保守。信息学分析发现,“ALTAV”只部分涵盖了预测到的优势表位33~49 aa(AVETGHTSQVDPSDTM),并不在主要抗原表位上。提示CVB4通过分子模拟机制引起T1DM的可能性不高,但有可能是通过表位扩展,在持续感染过程中暴露了该隐匿表位,引起糖尿病性自身免疫反应;这需要构建克隆表达“ALTAV”序列,并利用NOD小鼠加以验证[10]。二级结构分析显示CVB4 VP1蛋白以无规则卷曲为主,无规则卷曲往往是蛋白质功能和构象的基本结构,同时B细胞线性表位也多位于无规则卷曲所在的位置。目前,利用生物信息学技术分析筛选肠道病毒B细胞线性表位的报道较多[11-13],但关于CVB4鲜有报道。本研究利用ABCpred预测CVB4 VP1的线性B细胞表位,并通过免疫表位数据库网站IEDB提供的多种在线工具分析了VP1蛋白β-转角、表面可及性、柔韧性、抗原性以及亲水性5个参数,最后获得了5条保守的优势线性B细胞表位,为后期疫苗开发提供参考。
CVB4的VP1蛋白可能在病毒感染胰岛细胞过程中扮演重要的信号角色。亚细胞定位分析发现,CVB4 VP1存在线粒体定位信号,提示CVB4感染胰岛细胞后,可能通过影响线粒体功能而导致胰岛细胞死亡,引起T1DM的发生[14]。通过分析发现,CVB4 VP1蛋白存在多个磷酸化位点,而病毒的感染最重要的信号是一系列蛋白质的磷酸化[15]。提示CVB4在感染过程中,可通过对VP1特定位点进行磷酸化,从而开启或关闭下游的一系列信号,引起胰岛细胞死亡。
CVB4引起T1DM的机制尚未明确,本研究利用多种在线分析工具对CVB4 Tuscany分离株VP1蛋白进行了一系列分析,这些分析结果将有助于研究肠道病毒致T1DM的作用及机制。

