亚麻响应低钾胁迫转录谱分析
黄文功 姜卫东 姚玉波 宋喜霞 刘 岩 陈 思 赵东升 吴广文 袁红梅 任传英 孙中义 吴建忠 康庆华,*
专题
亚麻响应低钾胁迫转录谱分析
黄文功1姜卫东1姚玉波1宋喜霞1刘 岩1陈 思1赵东升1吴广文1袁红梅1任传英2孙中义3吴建忠4康庆华1,*
1黑龙江省农业科学院经济作物研究所, 黑龙江哈尔滨 150086;2黑龙江省农业科学院食品加工研究所, 黑龙江哈尔滨 150086;3黑龙江省农业科学院畜牧研究所, 黑龙江哈尔滨 150086;4黑龙江省农业科学院草业研究所, 黑龙江哈尔滨 150086
钾是亚麻生长发育必需的大量元素。本研究以钾高效利用亚麻品种Sofie为试验材料, 在低钾处理12 h和96 h下, 利用转录组测序及qRT-PCR进行低钾胁迫下差异基因表达调控的研究。结果表明, 低钾处理7 d的亚麻叶片边缘变黄, 与对照相比, 低钾处理植株矮化。筛选出对低钾响应强烈的3个钾运转蛋白基因(Lus K channel 1)、(Lus STELAR K+outward rectifier)和(Lus high affinity K+transporter 5), 低钾胁迫响应峰值时间为12 h和96 h; 与对照相比, 低钾处理12 h鉴定到差异表达基因1154个(508个上调, 646个下调), GO功能富集分析表明, 这些差异表达基因主要富集于代谢过程、细胞进程、单一生物过程、催化活性和结合功能五大类, KEGG通路富集分析表明, 这些差异表达基因涉及到能量代谢、碳水化合物代谢、碳代谢、氨基酸代谢、萜类化合物代谢和植物激素信号转导等通路。进而筛选出7个与钾直接相关基因(4个钾运输蛋白、2个钾通道蛋白及1个钠钾钙交换蛋白)、13个与激素相关基因以及6个与纤维素合成相关基因。7个与钾直接相关基因中, 2个基因表达量上调1.75倍和2.64倍, 5个基因表达量下调1.21~9.57倍。以上解析的差异基因初步揭示了亚麻低钾涉及的转录调控途径, 可为亚麻耐低钾相关基因的克隆与功能验证奠定基础。
亚麻; 低钾; 转录组; 差异表达基因
中国耕地土壤钾含量普遍低, 以土壤中速效钾含量70 mg kg-1为标准, 中国低钾土壤总面积约2.27×108hm2[1], 严重制约了作物的正常生长发育。中国钾肥施用量从2011年的6.05´109kg增加到2017年的1.95´1010kg[2-3]。大量施用钾肥, 不仅增加生产成本、加速钾资源枯竭, 并造成环境污染问题。研究表明, 不同物种甚至同一作物不同品种在土壤钾素利用上表现出显著差异[4]。最近, 转基因等方法被用来提高钾吸收和利用效率[5]。这些措施将缓解钾素资源的进一步枯竭, 并提高作物产量和品质。亚麻(L.)是一种广泛种植的经济作物, 有悠久的栽培历史。钾是亚麻生长发育必需的大量元素。对亚麻钾吸收和利用进行科学研究, 提高亚麻对低钾胁迫适应性, 对于中国农业化肥实现“双减”, 保护环境, 避免过量施用化肥造成的环境污染, 实现绿水青山理念, 实现中国生态农业绿色可持续发展有重要意义。
钾是植物生长的必需元素, 是植物体内含量最高的阳离子, 占植物干物质总量2%~10%, 在生长发育中起重要作用[6-8]。植物中的钾调节许多物理过程包括植株生长、产量和品质参数[9]。钾参与光合作用及叶绿素合成, 钾是植物体中重要的渗透调节物质。K+迁移对低钾土壤中的植物生长至关重要, 已发现许多钾通道, 如KT/KUP/HAK转运蛋白家族[10-12]和KCOs家族[13]。还发现其他具有双重功能的转运体, 包括具有Na+和K+转运体双重功能的HKT转运体[14]和CBL-CIPK, 后者受Ca2+信号通路调节, 并激活AKT1通道[15-17]。此外, ROS信号通路中的RCI3/RAP被认为激活转运体HAK。植物低钾表现明显症状为茎弱、容易倒伏、叶片失水、耐旱性和耐寒性降低、蛋白质和叶绿素分解、叶片变黄最终组织坏死[18]。
目前已发表的亚麻基因组序列图[19]极大地促进了亚麻的研究。亚麻的简化基因组测序结果表明, 遗传图谱总长度为1483.25 cM, 2339个标记均匀分布于15个连锁群, 分别对应于亚麻15条染色体, 相邻标记间平均距离为0.63 cM, 该图谱是目前为止密度最高的亚麻全基因组遗传连锁图谱[20]。有学者提出一种涉及激活蛋白的低钾反应机制的存在[21]。目前亚麻对低钾胁迫的分子机制及参与这一调控过程的基因仍不明晰。特定响应基因表达的变化决定了钾胁迫下亚麻形态及生理变化, 差异基因表达研究有利于揭示钾胁迫下亚麻内在的分子响应机制, 为分子辅助品种选育奠定基础。本研究以亚麻品种Sofie为研究材料, 深入挖掘低钾胁迫下Sofie中的差异表达基因, 通过揭示钾胁迫相关的分子及信号通路, 以期为亚麻耐低钾相关基因的克隆与功能验证奠定基础。
1 材料与方法
1.1 植物材料
钾高效利用亚麻品种Sofie由国家麻类种质改良中心亚麻分中心提供。
1.2 生长条件及胁迫处理
将Sofie种子播种于装有蛭石(已经过121℃湿热灭菌20 min)的纸杯中, 纸杯置于塑料托盘上, 每个托盘浇等体积水, 每个纸杯播种30粒种子。出苗至胁迫处理前每3天浇1/2 MS, 将播种后的亚麻置于相对湿度70%的植物培养箱中, 23℃, 16 h光照/18℃, 8 h黑暗。设MS培养基中的K+浓度(20 mmol L-1)和0 (0 mmol L-1) 2个钾处理梯度。
在Sofie生长至3对真叶时(出苗后7 d)进行2种浓度的钾处理, 每个组培瓶中放置10株均匀一致的亚麻植株, 每个组培瓶200 mL培养液, 处理重复3次。每3 d更换1次培养液, 直至出现低钾症状(叶片边缘变黄)为止。
生长一致的3周Sofie幼苗正常浇MS培养液(充足的K+供应, 含KCl)作为低钾对照(CK), 浇低钾培养液(无KCl)的作为低钾处理(KS)。CK营养液是改变的MS培养液, KCl代替KNO3。CK和KS胁迫12 h和96 h, 整株苗取样, 液氮冻后-80℃贮存, 供RNA提取用于后续测序分析。
1.3 qRT-PCR引物
根据拟南芥中的钾运转蛋白相关基因, 在网站(http://phytozome.jgi.doe.gov/pz/portal.htmL)设计亚麻、、、、、、、、共9个基因的引物, 每个基因设计3对引物。
1.4 转录组测序
采用TIANGEN RNA prep pure多糖多酚植物总RNA提取试剂盒提取RNA。用TIANGEN Tgem Spectrophotometen检测所提取RNA A260/A280比值及浓度。CK和KS处理12 h和96 h提取的Sofie整株植株RNA (3次重复)分别命名为12h-CK-1、12h-CK-2、12h-CK-3、12h-KS-1、12h-KS-2、12h-KS-3、96h-CK-1、96h-CK-2、96h-CK-3、96h-KS-1、96h-KS-2和96h-KS-3。总RNA的质量和数量分析、建库及Illumina测序由华大基因公司(BGI, Shenzhen, China)完成。RNA-seq相应数据(编号: SRP120129)上传至公共数据库NCBI (https://www.ncbi.nlm.nih.gov/)。
1.5 qRT-PCR分析
按上述方法提取总RNA。TIANGEN RNAprep pure Plant Kit已含有去基因组DNA试剂, 逆转录时省去该步骤。逆转录方法采用ReverTra Ace qPCR RT Master Mix with gDNA Remover (Code No. FSQ-301)。RNA的变性: 将RNA 65℃ 5 min, 立即冰上冷却。逆转录反应体系为10 µL, 包含RNA template 0.5 pg~0.5 µg、Nuclease-free Water、5×RT Master Mix II。轻轻搅拌反应溶液, 以下温度进行反应: 37℃ 15 min, 50℃ 5 min, 98℃ 5 min, 4℃保存。
将1 μg总RNA合成第1链cDNA。PCR反应体系为95℃ 3 min; 95℃ 5 s, 60℃ 40 s, 72℃ 10 s, 45个循环。为验证引物特异性, 45个循环后进行溶解曲线绘制。qRT-PCR试验设置3次生物学重复。qRT-PCR试验内参基因为。利用2−ΔΔCt计算基因相对表达量。利用Primer 3 (http://frodo.wi.mit. edu/primer3)设计引物。
1.6 统计分析
所有试验3次重复, 数据用平均值±标准差(SD)表示。采用SPSS 17.0软件分析数据, 各组间比较采用单因素方差分析。<0.05的值为显著差异。
2 结果与分析
2.1 亚麻低钾胁迫响应时间的确定
2.1.1 亚麻低钾胁迫处理前后亚麻表型 Sofie出苗7 d, 在蛭石中的生长状态及各植株生长情况见图1-A。由图1-B可看出, 各植株生长一致, 长势良好。处理时, 对照和处理的生长状态见图1-C。处理7 d, 低钾和对照的生长状态见图1-D和E。说明低钾处理7 d植株出现叶片边缘变黄症状, 与对照相比, 低钾处理植株矮化。
2.1.2 亚麻低钾胁迫标记基因筛选 以低钾处理48 h K0的cDNA稀释10倍为模板进行PCR, 筛选标记基因引物及内参。从图2-A~C可看出, 引物LusKC1-1、LusSKOR-1、LusHAK5-2、LusKEA5-2和LusActin特异性好。本试验从4个特异性好的标记基因引物LusKC1-1、LusSKOR-1、LusHAK5-2和LusKEA5-2中选取3个标记基因、、及内参进行qRT-PCR试验。
A: 蛭石中生长; B: 处理前的植株表型; C: 低钾胁迫对照和处理的生长状态; D: 处理后低钾和对照的植株表型; E: 处理后低钾和对照的单株表型。a: 低钾胁迫的植株表型; b: 对照的植株表型。
A: growth in vermiculite; B: plant phenotype before treatment; C: growth condition of control and treatment under low-K+stress; D: plant phenotypes of low-K+and control plants after treatment; E: single plant phenotype of low-K+and control after treatment. a: plant phenotype under low-K+; b: plant phenotype of control.
M: DL2000; 1: LusKC1-1; 2: LusKC1-2; 3: LusKC1-3; 4: LusSKOR-1; 5: LusSKOR-2; 6: LusSKOR-3; 7: LusAKT2-1; 8: LusAKT2-2; 9: LusAKT2-3; 10: LusHAK5-1; 11: LusHAK5-2; 12: LusHAK5-3; 13: LusKUP2-1; 14: LusKUP2-2; 15: LusKUP2-3; 16: LusKUP3-1; 17: LusKUP3-2; 18: LusKUP3-3; 19: LusKUP12-1; 20: LusKUP12-2; 21: LusKUP12-3; 22: LusKEA5-1; 23: LusKEA5-2; 24: LusKEA5-3; 25: LusCHX17-1; 26: LusCHX17-2; 27: LusCHX17-3; 28: LusEF1A; 29: LusUBI; 30: LusTUA; 31: LusEF2; 32: LusActin.
2.1.3 亚麻低钾胁迫响应时间筛选 在低钾胁迫6 h、12 h、24 h、48 h、96 h、192 h、10 d和15 d时, 利用筛出的标记基因引物LusKC1-1、LusSKOR-1、LusHAK5-2及内参引物进行qRT-PCR试验。从图3可看出,在12 h和96 h表达量显著高于其他时间(<0.05);在12 h和96 h表达量高, 这2个时间表达量显著高于其他时间(<0.05);在12 h和96 h表达量显著高于其他时间(<0.05)。综合3个标记基因、和qRT-PCR表达情况发现, 3个标记基因12 h开始高表达, 96 h表达处于另一个高峰。选取低钾处理及对照在12 h和96 h取样, 进行后续转录组测序。
同一基因中不同小写字母分别表示在0.05水平差异显著。
Different lowercase letters mean significant differences at the 0.05 probability level during the same gene.
2.2 亚麻低钾的RNA-seq
2.2.1 亚麻低钾转录组测序统计分析 利用低钾12 h和96 h Sofie及对应时间对照样品总RNA构建Illumina测序文库。每个文库平均得到38.07 Mb原始测序读数, 其中95.38%以上为过滤读数。将过滤读数与亚麻基因组序列比对, 至少86.17%读数成功与数据库参考基因组序列比对上(表2)。
2.2.2 亚麻低钾差异表达基因筛选 本研究将差异表达基因的差异倍数阈值设为≥2.0,-value阈值设为≤0.05。利用-value阈值<0.05和log2ratio的绝对值≥1来筛选出低钾的差异表达基因。低钾12 h鉴定到1154个(508个上调表达, 646个下调表达)低钾响应表达基因。低钾96 h差异表达基因明显减少, 共鉴定到247个(131个上调表达, 116个下调表达)低钾响应表达基因。图4-A展示了不同低钾时期的差异表达基因的异同情况, 18个基因在两时期均响应, 其中上调基因8个, 下调基因10个, 推测这些基因可能在整个低钾时期有重要功能。图4-B显示低钾对照组和处理组差异基因数。比较这些结果消除了基因表达与时间和生长相关的自然变异。在CK组和KS组中, 有886个基因共同表达, 其中343个基因上调, 543个基因下调。
2.3 GO功能富集
对差异表达基因(1154个)进行GO功能富集发现, 在12h-KS vs. 12h-CK对比组差异基因主要富集于代谢过程、细胞进程、单一生物过程、催化活性和结合功能五大类(图5)。

表2 亚麻12个转录组数据的主要特性
A: 亚麻低钾处理后12 h和96 h差异基因数; B: 低钾对照组和处理组差异基因数。
A: the number of DEGs at 12 h and 96 h after low-K+treatment in flax; B: the number of DEGs in control and low-K+treatment.
2.4 KEGG通路富集
为进一步理解差异表达基因的功能, 将12h-KS vs 12h-CK对比组所有差异基因(1154个)与KEGG数据库比对, 确定差异表达基因参与的最主要的能量代谢(光合有机物固碳、光合作用), 碳水化合物代谢(戊糖、果糖磷酸途径、甘露糖代谢)、碳代谢、氨基酸代谢、萜类化合物代谢和植物激素信号转导通路(图6)。差异表达基因中有本研究关注的pathway注释的基因为5个, 富集的途径有5条, 包括光合作用途径(photosynthesis) 1条、植物激素信号转导途径(plant hormone signal transduction) 3条及萜类主链生物合成途径(Terpenoids backbone biosynthesis) 1条。其中基因(MSTRG.20565.1)表达量上调2.64倍, 在光合作用途径中负责K+运输; 基因(MSTRG.7397.8)表达量上调8.52倍, 在生长素合成途径中负责生长素的合成; 基因(MSTRG.25736.1)表达量下调1.81倍, 在细胞分裂素合成途径中负责细胞分裂素的合成; 基因(MSTRG.16160.5)表达量下调6.23倍, 在乙烯合成途径中负责乙烯的合成; 基因(MSTRG.5636.3)表达量下调1.86倍, 在萜类主链生物合成途径中负责细胞壁的合成。
2.5 差异表达基因的功能分析
2.5.1 亚麻低钾下与钾直接相关基因筛选 本研究在亚麻低钾转录组数据的差异基因中筛选出7个与钾直接相关基因(表3), 包括4个钾运输蛋白(MSTRG.20565.1、MSTRG.24915.2、MSTRG.10817.1和MSTRG.6817.1), 2个钾通道蛋白(MSTRG.4695.1和MSTRG.30540.1)及1个钠钾钙交换蛋白(MSTRG.14498.2)。其中2个上调基因(MSTRG.20565.1和MSTRG.4695.1), 5个下调基因(MSTRG.24915.2、MSTRG.10817.1、MSTRG.30540.1、MSTRG.14498.2和MSTRG.6817.1), 这些基因可能在钾胁迫响应中起重要作用。
本研究发现, 许多差异表达基因在低钾胁迫信号转导和离子转运体调控通路中扮演重要角色, 如附图1。1个AKT通道蛋白(上调2.15倍), 参与钾通道转运, 响应于非生物胁迫; 19个CIPK信号转导蛋白(上调1.46~11.76倍), 参与钙信号传导, 响应于非生物胁迫; 1个CML信号转导蛋白(上调3.74倍), 参与钙信号传导, 影响植物生理代谢; 2个ARF信号转导蛋白(下调3.25倍和4.61倍), 参与生长素调控, 影响侧根发育; 5个NRT转运蛋白(下调1.14~4.69倍), 参与硝酸盐转运, 影响植物的生长发育。鉴于这些基因直接参与钾信号传导及K+运输, 与Wang等[22]报道一致。推测低钾胁迫将直接影响这些基因的表达, 进而影响亚麻中钾信号的传导及K+运输, 从而影响亚麻的表型。
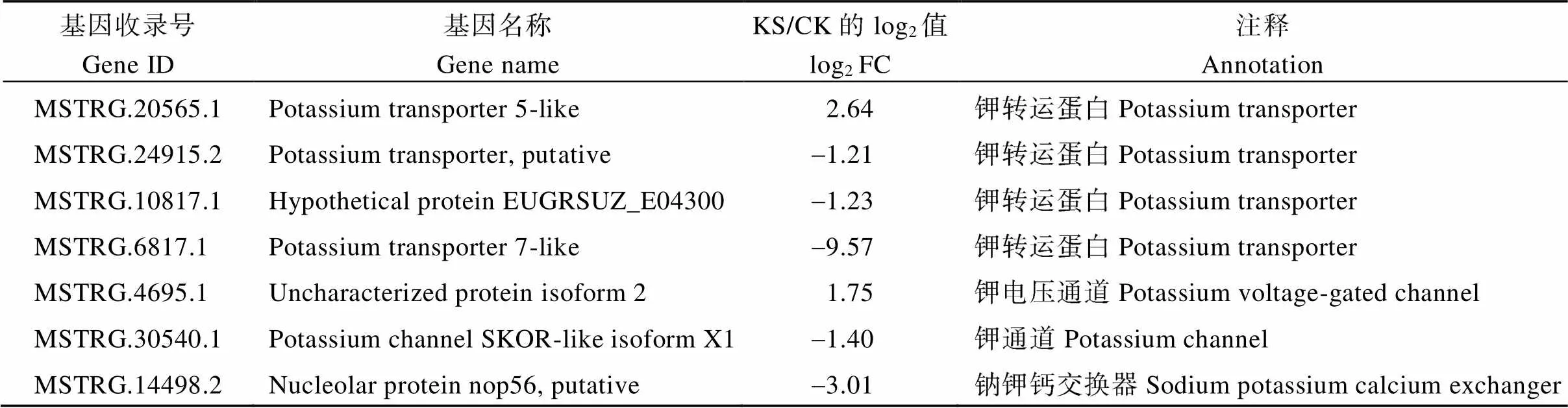
表3 亚麻低钾下7个与钾直接相关基因筛选
2.5.2 亚麻低钾下与激素相关基因筛选 在所有差异对比组中, 共103个基因注释到激素响应蛋白功能, 包括17个ABA响应蛋白、16个乙烯响应蛋白、22个生长素响应蛋白、10个JA响应蛋白以及38个细胞分裂素响应蛋白。本研究发现, 13个与激素直接相关的基因(表4), 其中1个(上调8.52倍), 一种植物生长素受体, 参与植物生长调控; 2个(下调1.44倍和1.51倍), 生长素响应基因, 应答生长素效应; 1个(下调9.62倍), 生长素初期响应基因; 2个(下调3.02倍和3.58倍), 植物生长素原初反应基因, 参与植物生长素调节; 1个(下调1.81倍), 一种组氨酸磷酸转移蛋白, 参与细胞分裂素的负向调控; 1个(下调10.02倍), 乙烯信号关键基因, 参与乙烯信号通路的正向调控; 2个(下调1.20倍和3.10倍), 负反馈调控, 参与乙烯信号转导; 3个(下调6.23倍、1.90倍和7.72倍), 介导乙烯受体的信号负调控乙烯反应。
本研究筛选出生长素合成通路6个基因, 包括1个、2个、1个、2个, 低钾胁迫下, 这些基因调控亚麻植株生长, 造成植株矮化; 细胞分裂素合成通路1个基因, 低钾胁迫下,调控细胞分裂, 造成亚麻植株矮化的表型; 乙烯合成通路6个基因, 包括3个、1个、2个, 低钾胁迫下, 这些基因引起亚麻植株衰老, 即叶片变黄(附图2)。这3个通路为揭示植物激素对低钾胁迫反应分子机制奠定基础[23]。低钾胁迫可能诱导这些基因差异表达, 调控激素合成, 进而影响亚麻的生长发育。
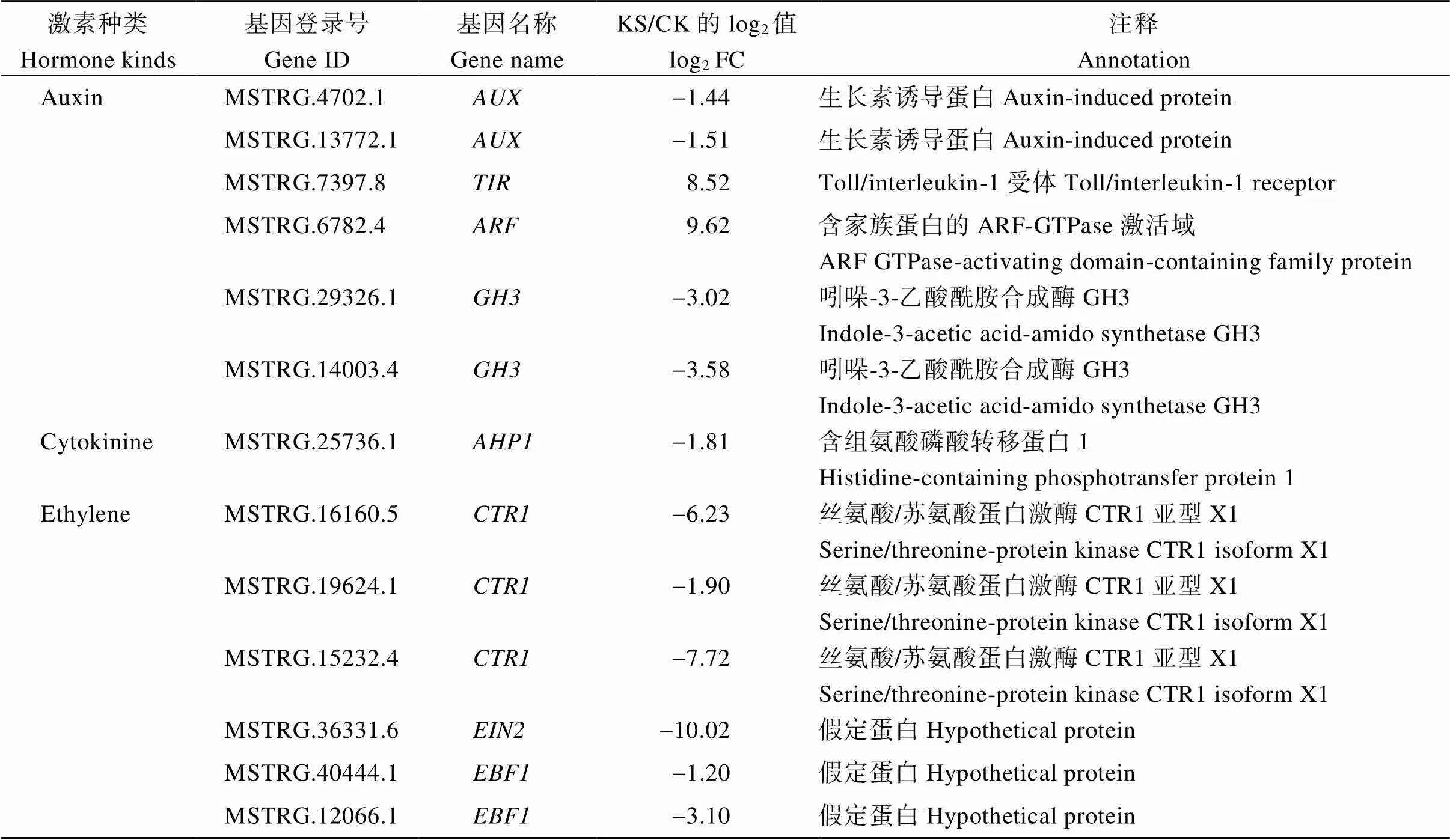
表4 亚麻低钾下13个与激素相关基因筛选
2.5.3 亚麻低钾下与纤维素相关基因筛选 本研究在差异表达基因中筛选出6个与纤维素相关基因(表5), 包括2个(下调8.70倍和2.13倍), 组成胞泌复合体的关键亚基, 参与复合体在靶膜组装; 2个(下调2.03倍和1.73倍)及1个(下调1.94倍), 植物糖磷脂酰肌醇锚定蛋白, 定向微纤丝, 突变后阻碍纤维素合成; 1个(下调1.86倍), 一种双特异性蛋白激酶, 参与纤维素合成。本研究筛选出2个、2个、1个和1个(附图3), 鉴于这些基因直接参与纤维素合成[24], 低钾胁迫可能诱导这些基因表达, 抑制亚麻纤维素合成, 进而调控产量形成。
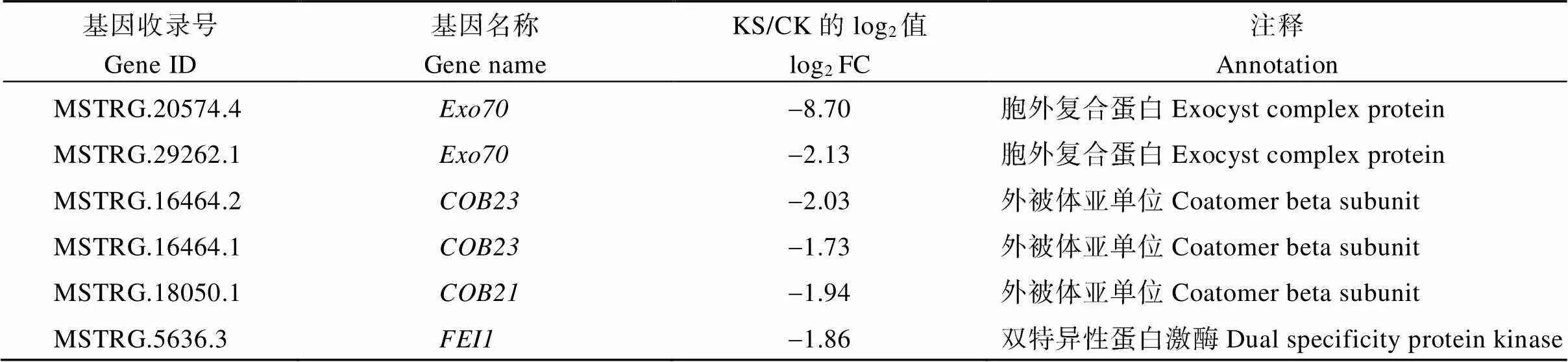
表5 亚麻低钾下6个与纤维素相关基因筛选
2.6 RNA-Seq和qRT-PCR相关性
为验证转录组测序分析结果, 随机选取、、、、、、和等8个基因用于qRT-PCR分析。2种方法得到的基因表达变化数据相关系数2达到0.91 (图7), 说明RNA-seq测序数据相对准确, 低钾相关基因表达量结果可信。
3 讨论
3.1 低钾对亚麻表型的影响及亚麻响应低钾的时间
植物长期受到低钾胁迫时, 会出现黄化病, 老叶先显现症状[25]。植物低钾影响茎叶及根的生长发育[26-27]。植株茎叶低钾早期老叶叶尖失绿。随低钾时间增加, 萎黄症状从老叶转移到新叶。严重低钾时, 植物叶子从叶尖到整个叶缘显现出萎黄病, 最终坏死。随腐胺在叶片中积累, 植物叶片萎黄并发生大量褐斑。与上述植物低钾表型相似, 低钾亚麻植株出现叶片边缘变黄症状, 并表现出矮化的表型。
研究表明, 西瓜低钾响应早期和后期的时间点分别为6 h和120 h[28]。而本研究通过亚麻低钾8个时间点筛选出12 h和96 h为亚麻低钾响应的早期和后期时间点。与西瓜低钾响应不同的是, 亚麻低钾响应早期时间(12 h)晚于西瓜(6 h), 但亚麻后期响应时间(96 h)早于西瓜(120 h)。
3.2 植物低钾胁迫的信号转导和离子转运体调控
本研究2个ARF信号转导蛋白(下调3.25倍和4.61倍), 参与生长素调控, 影响侧根发育; 5个NRT转运蛋白(下调1.14~4.69倍), 参与硝酸盐转运, 影响植物的生长发育。鉴于这些基因直接参与钾信号传导及钾离子运输, 与Wang等[22]报道一致。推测低钾胁迫将直接影响这些基因的表达, 进而影响亚麻中钾信号的传导及K+运输, 从而影响亚麻的表型。
钾是植物生长所需的主要营养物质。对低K+条件的适应性机制主要为钙调磷酸酶B样蛋白(Calcineurin B-like Calcium Sensors, CBL)和CBL相互作用激酶(CBL-Interacting Protein Kinases, CIPK)组成的Ca2+信号网络。低钾胁迫信号转导和离子转运体调控通路涉及大量膜上K+信号转导蛋白和离子通道基因及细胞质和核中一些信号转导蛋白及离子转运蛋白。低钾胁迫通过调控细胞质和细胞核中一些信号转导蛋白影响膜上相关信号转导蛋白和离子通道基因表达(附图1)[22]。本研究低钾胁迫发现大量差异表达基因, AKT通道蛋白在亚麻低钾处理后其表达量上调2.15倍, 而拟南芥中CIPK23及KC1协同调控AKT1介导低钾胁迫[29], 水稻中过表达AKT1可通过提高组织内K+含量提高植株对渗透和干旱胁迫抗性[30], 说明AKT通道蛋白在亚麻和拟南芥或水稻有类似功能; 发现CIPK信号转导蛋白在亚麻低钾处理后其表达量上调1.46~11.76倍, 而杨树中CBL-CIPK信号通路可通过调控Shaker-like钾通道增加对低钾胁迫耐受性[31], 说明CIPK信号通路在亚麻和杨树有类似功能; 发现ARF信号转导蛋白在亚麻低钾处理后其表达量下调3.25倍和4.61倍,而拟南芥中低钾胁迫下ARF2磷酸化可解除对K+转运基因抑制, 促进钾吸收[32], 说明ARF信号转导蛋白在亚麻和拟南芥有类似功能; 发现NRT转运蛋白在亚麻低钾处理后其表达量下调1.14~4.69倍, 而拟南芥中NPF7.3/NRT1.5介导的NO3–迁移可通过调控SKOR影响K+从根到芽迁移[33], 说明NRT转运蛋白在亚麻和拟南芥有类似功能。鉴于这些基因在低钾胁迫亚麻中的表达明显改变, 推测这些基因在亚麻对低钾胁迫的响应中发挥着重要作用。
3.3 植物激素在低钾胁迫中的作用
植物激素广泛参与逆境生理和生化反应。乙烯是植物对非生物胁迫应答中重要信号分子之一, 它与乙烯受体等主要转录因子结合, 激活下游基因表达, 触发乙烯应答[34]。生长素可诱导ARF/AUX/GH3等基因在植物胁迫应答中快速瞬时高表达[35]。脱落酸作为植物地下-地上部分之间信息传递的中心媒介, 通过激活K+进出细胞路径诱导细胞膨胀压变化[36]。植物激素也相互影响, 乙烯和生长素通过抑制主根生长而促进根毛伸长, 从而对低钾胁迫下根系形态发育起指导作用。生长在低钾条件的植物根形态类似于生长在响应外源乙烯和生长素处理的形态[37]。低钾条件下, 乙烯合成和信号转导相关基因表达增强[38]。
4 结论
利用qRT-PCR筛选出亚麻对低钾响应强烈的3个标记基因及内参分别为:、、和。钾胁迫相关基因对低钾响应高峰时间出现在12 h和96 h。通过低钾转录组数据的分析和挖掘, 确定7个与钾直接相关基因(4个钾运输蛋白、2个钾通道蛋白及1个钠钾钙交换蛋白), 低钾胁迫直接影响亚麻这些基因的表达, 进而影响亚麻中钾信号的传导及K+运输; 确定13个与激素相关基因(生长素合成通路6个基因、细胞分裂素合成通路1个基因和乙烯合成通路6个基因), 低钾胁迫诱导这些基因表达, 调控激素合成, 进而影响亚麻生长发育; 确定6个与纤维素合成相关基因(2个、3个和1个基因), 低钾胁迫诱导这些基因表达抑制亚麻纤维素合成, 进而调控产量形成。
[1] 中国农业科学院土壤肥料研究所. 中国化肥区划. 北京: 中国农业科技出版社, 1986. pp 13–15. Institute of Soil and Fertilizer, Chinese Academy of Agricultural Sciences. Fertilizer Regionalization in China. Beijing: China Agricultural Science and Technology Press, 1986. pp 13–15 (in Chinese).
[2] 国家统计局农村社会经济调查司. 中国农村统计年鉴. 北京: 中国统计出版社, 2012. pp 270–271. Department of Rural Social and Economic Investigation, National Bureau of Statistics. China Rural Statistical Yearbook. Beijing: China Statistics Press, 2012. pp 270–271 (in Chinese).
[3] 亓昭英, 屈小荣, 马锁立, 商立鹏. 2018年我国钾肥行业运行报告及发展预测. 磷肥与复肥, 2019, 34(2): 1–4. Qi S Y, Qu X R, Ma S L, Shang L P. Operation report and development prediction of Chinese potassium fertilizer industry in 2018., 2019, 34(2):1–4 (in Chinese with English abstract).
[4] 刘国栋, 刘更另. 籼稻耐低钾基因型的筛选. 作物学报, 2002, 28: 161–166. Liu G D, Liu G L. Screening of low potassium tolerance genotypes inrice., 2002, 28: 161–166 (in Chinese with English abstract).
[5] Wang Y, He L, Li H D, Xu J, Wu W H. Potassium channel a-subunit At KC1 negatively regulates AKT1-mediated K+uptake in Arabidopsis roots under low-K+stress., 2010, 20: 826–837.
[6] Schachtman D P, Shin R. Nutrient sensing and signaling: NPKS., 2007, 58: 47–69.
[7] Leigh R A, Wyn Jones R G. A hypothesis relating critical potassium concentrations for growth to the distribution and function of this ion in the plant cell., 1984, 97: 1–13.
[8] Clarkson D T, Hanson J B. The mineral nutrition of higher plants., 1980, 31: 239–298.
[9] Lester G E. Whole plant applied potassium: effects on cantaloupe fruit sugar content and related human wellness compounds., 2005, 682: 487–492.
[10] Rubio F, Santa M G E, Rodríguez N A. Cloning of, and barley cDNAs encoding HAK potassium transporters in root and shoot cells., 2010, 109:34–43.
[11] Bañuelos M A, Garciadeblas B, Cubero B, Rodríguez N A. Inventory and functional characterization of the HAK potassium transporters of rice., 2002, 130: 784–795.
[12] Wang Y H, Garvin D F, Kochian L V. Rapid induction of regulatory and transporter genes in response to phosphorus, potassium, and iron deficiencies in tomato roots. Evidence for cross talk and root/rhizosphere-mediated signals., 2002, 130: 1361–1370.
[13] Voelker C, Schmidt D, Czempinski K, Czempinski K. Members of theAtTPK/KCO family form homomeric vacuolar channels in planta., 2006, 48: 296.
[14] Munns R, Tester M. Mechanisms of salinity tolerance., 2008, 59: 651–681.
[15] Xu J, Li H D, Chen L Q, Wang Y, Liu L L. A protein kinase, interacting with two calcineurin B-like proteins, regulates K+transporter AKT1 in., 2006, 125: 1347–1360.
[16] Lan W Z, Lee S C, Che Y F, Jiang Y Q, Luan S. Mechanistic analysis of AKT1 regulation by the CBL-CIPK-PP2CA interactions., 2011, 4: 527–536.
[17] Mao J, Nuruzzaman M S M, Shi S, Chao J T, Jin Y R, Wang Q, Liu H B. Mechanisms and physiological roles of the CBL-CIPK networking system in., 2016, 7: 62.
[18] Munson R D. Potassium in Agriculture. Madison: ASA/CSSA/ SSSA. 1985. pp 754–794.
[19] Wang Z W, Hobson N, Galindo L, Zhu S L, Shi D H, McDill J, Yang L F, Hawkins S, Neutelings G, Datla R, Lambert G, Galbraith D W, Grassa C J, Geraldes A, Cronk Q C, Cullis C, Dash P K, Kumar P A, Cloutier S, Sharpe A G, Wong G K, Wang J, Deyholos M K. The genome of flax () assembled de novo from short shotgun sequence reads., 2012, 72: 461–473.
[20] Wu J Z, Zhao Q, Zhang L Y, Ma Y H, Pan L Y, Lin H, Wu G W, Yuan H M, Yu Y, Wang X, Yang X, Li Z G, Jiang T B, Sun D Q. QTL Mapping of fiber-related traits based on a high-density genetic map in flax (L.)., 2018, 9: 885–894.
[21] Huang W G, Zhang S Q, Wu G W, Yu Y, Ren C Y, Kang Q H, Liu Y, Liang C B, Zhang L G, Zhan Y G. Transcriptome profiling of potassium starvation responsiveness in flax (L.)., 2019, 51: 865–878.
[22] Wang Y, Wu W H. Regulation of potassium transport and signaling in plants., 2017, 39: 123–128.
[23] Ruben P F, Manuel B. A review of the effects of soil organisms on plant hormone signalling pathways., 2015, 114: 104–116.
[24] Joanna K P, Joseph J K. The regulation of cellulose biosynthesis in plants., 2019, 31: 282–296.
[25] Armengaud P, Breitling R, Amtmann A. The potassium- dependent transcriptome of Arabidopsis reveals a prominent role of jasmonic acid in nutrient signaling., 2004, 136: 2556–2576.
[26] Cakmak I, Hengeler C, Marschner H. Partitioning of shoot and root dry matter and carbohydrates in bean plants suffering from phosphorus, potassium and magnesium deficiency., 1994, 45: 1245–1250.
[27] Jung J Y, Shin R, Schachtman D P. Ethylene mediates response and tolerance to potassium deprivation in., 2009, 21: 607–621.
[28] Fan M L, Huang Y, Zhong Y Q, Kong Q S, Xie J J, Niu M L, Xu Y, Bie Z L. Comparative transcriptome profiling of potassium starvation responsiveness in two contrasting watermelon genotypes., 2014, 239: 397–410.
[29] Wang X P, Chen L M, Liu W X. AtKC1 and CIPK23 synergistically modulate AKT1-mediated low-potassium stress responses in., 2016, 170: 2264–2277.
[30] Ahmad I, Mian A, Maathuis F J M. Overexpression of the rice AKT1 potassium channel affects potassium nutrition and rice drought tolerance.2016, 67: 2689–2698.
[31] Zhang H, Yin W, Xia X. Shaker-like potassium channels in Populus, regulated by the CBL-CIPK signal transduction pathway, increase tolerance to low-K+stress., 2010, 29: 1007–1012.
[32] Zhao S, Zhang M L, Ma T L. Phosphorylation of ARF2 relieves its repression of transcription of the K+transporter gene HAK5 in response to low potassium stress., 2016, 28: 3005–3019.
[33] Drechsler N, Zheng Y, Bohner A, Nobmann B, Wirén N, Kunze R, Rausch C. Nitrate-dependent control of shoot K homeostasis by NPF7.3/NRT1.5 and SKOR in., 2015, 169: 2832–2847.
[34] Li W, Ma M, Feng Y, Li H J, Wang Y C, Ma Y T, Li M Z, An F Y, Guo H W. EIN2-directed translational regulation of ethylene signaling in., 2015, 163: 670–683.
[35] Wang S, Bai Y, Shen C, Wu Y R, Zhang S N. Auxin-related gene families in abiotic stress response in Sorghum bicolor., 2010, 10: 533–546.
[36] Milborrow B V, Burden R S, Taylor H F. The conversion of 2-cis-[14C] Xanthoxic acid into [14C] ABA., 1997, 45: 257–260.
[37] Muday G K, Rahman A, Binder B M. Auxin and ethylene: collaborators or competitors?, 2012, 17: 181–195.
[38] Shin R, Berg R H, Schachtman D P. Reactive oxygen species and root hairs inroot response to nitrogen, phosphorus and potassium deficiency., 2005, 46: 1350–1357.
附图1 亚麻低钾胁迫的信号转导和离子转运体调控通路[22]
Fig. S1 Signal transduction and ion transporter regulation in responses to low-K+stress in flax[22]
a: 低钾胁迫处理的植株表型; b: 对照的植株表型。植物能感知外界低K+胁迫, 并在植物细胞中产生低K+信号。信号(Ca2+、ROS等)可在细胞溶质中转导, 最终在转录和翻译后水平调节下游靶点(尤其是K+通道和转运体)。“P”表示磷酸化, “X”表示抑制作用。
a: plant phenotype of low-K+treatment; b: plant phenotype of control. Plants are able to perceive external low-K+stress and generate low-K+signals in plant cells. The signals (Ca2+, ROS, etc.) can be transducted in cytosol, and eventually regulate the downstream targets (particularly K+channels and transporters) at transcriptional and posttranslational levels. P represents phosphorylation and X indicates inhibition effect.
附图2 亚麻低钾下生长素、细胞分裂素和乙烯合成通路相关基因[23]
Fig. S2 Genes related to auxin, cytokinin and ethylene synthesis pathway under low-K+in flax[23]
a: 低钾胁迫处理的植株表型; b: 对照的植株表型。矩形框为参与这些途径的蛋白质。蛋白质-蛋白质相互作用用箭头表示。箭头表示激活, 短横线表示抑制, 交叉线表示解离, 直虚线箭头表示间接作用, +u表示泛素化。
a: plant phenotype of low-K+treatment; b: plant phenotype of control. Rectangular boxes indicate proteins be involved in these pathways. Protein-protein interactions are represented by arrows. Arrows represent an activation, bar-headed lines an inhibition, crossed lines a dissociation, dotted arrows an indirect effect, +u denotes ubiquitination.
附图3 亚麻低钾下纤维素合成通路相关基因[24]
Fig. S3 Genes related to cellulose synthesis pathway under low-K+in flax[24]
Transcriptome profiling of flax (L.) response to low potassium stress
HUANG Wen-Gong1, JIANG Wei-Dong1, YAO Yu-Bo1, SONG Xi-Xia1, LIU Yan1, CHEN Si1, ZHAO Dong-Sheng1, WU Guang-Wen1, YUAN Hong-Mei1, REN Chuan-Ying2, SUN Zhong-Yi3, WU Jian-Zhong4, and KANG Qing-Hua1,*
1Institute of Industrial Crops, Heilongjiang Academy of Agricultural Sciences, Harbin 150086, Heilongjiang, China;2Food Processing Institute, Heilongjiang Academy of Agricultural Sciences, Harbin 150086, Heilongjiang, China;3Institute of Animal Husbandry Research, Heilongjiang Academy of Agricultural Sciences, Harbin 150086, Heilongjiang, China;4Institute of Forage and Grassland Science, Heilongjiang Academy of Agricultural Sciences, Harbin 150086, Heilongjiang, China
Potassium (K) is an essential element for the growth and development in flax. Transcriptome sequencing and qRT-PCR were used to investigate the regulation of differential gene expression after 12 h and 96 h of low-K+treatment. The results showed that the leaf edge of flax treated with low-K+for 7 days turned yellow, and the plants were dwarfed compared with the control.(Lus K channel 1),(Lus STELAR K+outward rectifier) and(Lus high affinity K+transporter 5) were detected to respond to low-K+with response peak time of 12 h and 96 h. Compared with the control, 1154 differentially expressed genes (DEGs) (508 up-regulated and 646 down-regulated genes) were identified in low-K+treatment for 12 h. GO enrichment showed that DEGs were mainly concentrated on five categories: metabolic process, cellular process, single biological process, catalytic activity and binding function. KEGG pathway enrichment showed that DEGs involved in energy metabolism, carbohydrate metabolism, carbon metabolism, amino acid metabolism, terpenoid metabolism and plant hormone signal transduction pathways. Furthermore, 7 genes directly related to K (4K transporters, 2K channel proteins and 1 sodium-potassium-calcium exchanger protein), 13 genes related to hormone and 6 genes related to cellulose synthesis were screened. Among the 7 genes directly related to K, the relative expression of 2 genes were up-regulated by 1.75 and 2.64 times and 5 genes down-regulated by 1.21–9.57 times. In summary, DEGs preliminarily revealed the transcriptional regulation pathway involved in low-K+in flax, which laid a foundation for cloning and functional verification of flax low-K+tolerance related genes.
flax; low potassium; transcriptome profiling; differentially expressed genes
10.3724/SP.J.1006.2021.04133
本研究由国家重点研发计划项目(2018YFD0201100), 黑龙江省现代农业产业技术协同创新体系麻类药用资源遗传改良与创新利用协同创新岗(YYM19SQ-24), 国家现代农业产业技术体系建设专项(CARS-16-E04)和黑龙江省农业科学院科技创新工程专项(2019JCQN003, HNK2019CX08-05)资助。
This study was supported by the National Key Research and Development Program of China (2018YFD0201100), the Heilongjiang Province Modern Agricultural Industry Technology Collaborative Innovation System-Hemp (medicinal) Resources Genetic Improvement and Innovative Utilization Collaborative Innovation Post (YYM19SQ-24), the China Agriculture Research System (CARS-16-E04), and the Science and Technology Innovation Project of Heilongjiang Academy of Agricultural Sciences (2019JCQN003, HNK2019CX08-05).
康庆华, E-mail: qinghuakang111@163.com
E-mail: huangwengong1736@163.com
2020-06-22;
2020-11-13;
2020-12-15.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20201215.0849.006.html

