人KIBRA基因真核表达载体的构建及生物信息学分析
王 博,宋少苒,田碧霞,杨泽健,张 妙,高小倩,孙 伟,蒋依娜,刘培军
(1. 西安交通大学第一附属医院转化医学中心,陕西西安 710061;2. 陕西省肿瘤精准医学重点实验室,陕西西安 710061;3. 西安交通大学医学部,陕西西安 710061;4. 西安交通大学第一附属医院病理科,陕西西安 710061)
Hippo信号通路主要通过调节细胞内增殖和凋亡相关基因,进而控制细胞接触抑制、组织大小、肿瘤的发生发展过程[1]。该通路的核心机制是Lats1/2磷酸化下游效应分子YAP/TAZ使其被泛素化降解,从而抑制其入核调控下游靶基因的表达。目前,已有大量的研究表明,Hippo信号通路在人类恶性肿瘤中明显失调,且与患者较差的预后相关[2]。同时,本课题组前期的研究发现,Hippo信号通路上游膜蛋白CRB3可以通过稳定肾脏脑蛋白(human kidney and brain protein, KIBRA)参与乳腺癌的发生发展过程[3],但具体的相互作用机制和KIBRA在乳腺癌中的作用机制仍不是十分清楚。因此,KIBRA的克隆和表达对研究Hippo信号通路上游信号及在乳腺癌中的作用机制具有重要意义。
本研究从人乳腺癌细胞系MCF7的总RNA中特异性PCR扩增KIBRA编码区全长序列,并将其克隆到真核表达载体pCMV-Blank上,通过测序鉴定以及转染MCF7细胞验证该载体的表达情况,同时分析KIBRA的蛋白物理特性、翻译后修饰情况、二级结构和三级结构,为全面了解Hippo信号通路上游重要分子的特性提供基础,也为研究KIBRA在乳腺癌中的分子机制奠定良好的实验基础。
1 材料与方法
1.1 质粒、菌株和细胞系载体质粒pCMV-Blank(上海碧云天生物技术有限公司);宿主菌E.coliDH5α[天根生化科技(北京)有限公司];人乳腺癌细胞系(MCF7细胞)为实验室受赠保存细胞,使用含100 mL/L胎牛血清(fetal bovine serum, FBS)的高糖DMEM培养基,置于37 ℃、50 mL/L CO2培养箱中培养。
1.2 主要试剂高糖DMEM培养基(Hyclone公司),FBS(Gibco公司),Trizol(Invitrogen公司),cDNA反转录试剂盒、高保真PCR酶(Thermo公司),限制性内切酶EcoRⅤ和ApaⅠ、T4 DNA连接酶(NEB公司),DNA凝胶回收试剂盒、质粒小量提取试剂盒和无内毒素质粒中量提取试剂盒 [天根生化科技(北京)有限公司],转染试剂X-tremeGENE HP DNA Transfection Reagent(Roche公司),实时定量PCR试剂SYBR®Premix ExTaqTMⅡ(TaKaRa公司),NC膜(Pall公司),兔抗人KIBRA多克隆抗体(CST公司),HRP标记山羊抗兔IgG抗体(北京博奥森生物技术有限公司),ECL试剂盒(Pierce公司),相关引物由苏州金唯智生物科技有限公司合成。
1.3 KIBRA基因PCR扩增消化MCF7细胞,使用Trizol按照说明书提取总RNA,Nano Drop进行定量取5 μg总RNA,按照RevertAidTMfirst strand cDNA synthesis kit说明书将总RNA反转录成cDNA。根据GenBank中人的KIBRA(NM_001161661.2)CDs区序列,运用Oligo 7软件设计特异性PCR引物,并在上游引物引入EcoRⅤ酶切位点和保护碱基,5′-CCGGATATCATGCCCCGGCCGGAGCTGCC-3′;下游引物引入ApaⅠ酶切位点和保护碱基,5′-CGCGGGCCCTTAGACGTCATCTGCAGAGA-3′。以该cDNA作为PCR模板,通过上述引物克隆人KIBRA全长CDs区的序列。PCR反应体系:cDNA模板1 μL,上游和下游引物(10 μmol/L)各1 μL,2×Mix 25 μL,补ddH2O至50 μL。PCR反应条件:95 ℃ 5 min;95 ℃ 10 s,58 ℃ 15 s,72 ℃ 3.5 min,40个循环;72 ℃ 5 min。PCR反应产物长度3 378 bp,通过8 g/L琼脂糖凝胶电泳进行鉴定分析。
1.4 构建KIBRA真核表达载体用DNA凝胶回收试剂盒将大小正确的PCR产物进行胶回收,再用限制性内切酶EcoRⅤ和ApaⅠ双酶切回收的PCR产物和真核表达载体pCMV-Blank,37 ℃双酶切2 h。胶回收经8 g/L琼脂糖凝胶电泳后的双酶切产物,按照KIBRA与载体pCMV-Blank摩尔比3∶1的量,用T4 DNA连接酶将KIBRA连接到真核表达载体pCMV-Blank多克隆位点上。将连接产物转化E.coliDH5α感受态细胞,并涂布于含有卡那霉素(Kan)的LB平板上。次日挑取单克隆菌落,接种于液体LB(KanR)培养基中,置于37 ℃摇床中振荡培养。待培养基浑浊后进行菌液PCR,对克隆的KIBRA基因进行鉴定;将鉴定阳性的单克隆菌液提取质粒,再通过限制性内切酶EcoRⅤ和ApaⅠ双酶切验证。随后将菌液PCR和双酶切鉴定成功的阳性KIBRA克隆送至苏州金唯智生物科技有限公司进行测序。对测序结果进行BLAST比对分析,并将序列正确的阳性克隆载体命名为pCMV-KIBRA。
1.5 转染质粒pCMV-KIBRA使用无内毒素质粒中量提取试剂盒提取无内毒素质粒pCMV-Blank和pCMV-KIBRA。将对数生长期的乳腺癌MCF7细胞按照7×105/孔的密度接种6孔板,培养箱中培养过夜。使用转染试剂X-tremeGENE HP将无内毒素质粒pCMV-Blank和pCMV-KIBRA分别转染MCF7细胞,同时设置对照组,即未加质粒转染细胞。
1.6 分析载体pCMV-KIBRA表达情况将转染48 h的细胞经预冷PBS冲洗后,分别加入Trizol提取总RNA及RIPA蛋白裂解液(含蛋白酶抑制剂)提取总蛋白。将总RNA反转录成为cDNA,使用SYBR®Premix ExTaqTMⅡ进行实时定量PCR,其中引物KIBRA:上游5′-AGCTCCAAGTATGACCCTGAG-3′,下游 5′-AAAGCCACGCTCTTTGAACTG-3′,产物大小123 bp;GAPDH:上游5′-ACAACTTTGGTATCGTGGAAGG-3′,下游5′-GCCATCACGCCACAGTTTC-3′,产物大小101 bp。qPCR反应体系:cDNA模板0.5 μL,上游和下游引物(10 μmol/L)各1 μL,2×Mix 12.5 L,ddH2O 10 μL。qPCR反应条件:95 ℃ 30 s,95 ℃ 5 s,58 ℃ 15 s,72 ℃ 30 s,40个循环。将总蛋白进行定量,再按照每孔50 μg的上样量进行8% SDS-PAGE电泳,湿转至NC膜,50 g/L脱脂牛奶室温封闭2 h,加入一抗兔抗人KIBRA多克隆抗体(1∶1 000),4 ℃孵育过夜。TBST洗膜后,加入二抗HRP标记山羊抗兔IgG抗体(1∶5 000),室温孵育1 h,TBST洗膜,ECL化学发光并使用成像系统进行照相和分析。
1.7 KIBRA蛋白特性分析使用在线的ProtParam tool软件(https://web.expasy.org/protparam/)对KIBRA蛋白的理化性质进行分析。使用PhosphoSitePlus软件(https://www.phosphosite.org//homeAction.action)对KIBRA蛋白的磷酸化、乙酰化和泛素化位点进行分析。使用SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)对KIBRA蛋白的二级结构进行分析[4]。使用SWISS-MODEL软件(https://www.swissmodel.expasy.org/)对KIBRA蛋白的三维结构进行分析。
1.8 统计学分析采用SPSS 23统计学软件对数据进行统计学分析。计数资料以均数±标准差表示,组间均数比较采用t检验。P<0.05为差异具有统计学意义。
2 结 果
2.1 人KIBRA编码区的克隆以人MCF7细胞的cDNA为模板,PCR扩增KIBRA全长编码区序列,并将PCR产物通过8 g/L琼脂糖凝胶电泳分析,结果表明,PCR得到大小约3 400 bp的基因条带(图1),与预期的大小一致。

图1 人KIBRA编码区的PCR产物电泳图Fig.1 Electrophoretic diagram of PCR fragment with CDs of human KIBRAM:1 kb marker;1,2:人KIBRA编码区的PCR产物。
2.2 真核表达载体pCMV-KIBRA的构建将大小正确的PCR产物经胶回收和双酶切过程,连接到pCMV-Blank载体多克隆区,转化宿主细胞后挑起单克隆菌落进行如下鉴定。真核表达载体pCMV-KIBRA结构示意图(图2A)。将单克隆菌液进行菌液PCR鉴定,结果表明,菌液PCR得到大小约3 400 bp的基因条带(图2B),与KIBRA理论大小相同。将菌液PCR鉴定阳性的菌液提取质粒,并进行EcoRⅤ和ApaⅠ双酶切鉴定,结果显示,双酶切得到大小分别约3 400 bp和4 300 bp的两条条带(图2C),与预期KIBRA和载体pCMV-Blank大小一致。将双酶切鉴定阳性克隆送至基因检测公司进行测序,比对结果表明,pCMV-KIBRA中KIBRA序列与人KIBRA编码区序列完全相同。

图2 重组质粒pCMV-KIBRA结构示意图及菌液PCR和双酶切鉴定Fig.2 Structure schematic and identification of recombinant plasmid pCMV-KIBRA with colony PCR and restriction enzyme digestion
2.3 mRNA和蛋白水平检测KIBRA表达情况将对照质粒pCMV-Blank和重组质粒pCMV-KIBRA分别转染MCF7细胞后,继续培养48 h,提取细胞总RNA和总蛋白,通过时实定量PCR和Western blottting分别检测KIBRA表达情况。时实定量PCR检测结果表明,pCMV-KIBRA转染组相比未转染组,KIBRA的mRNA水平明显升高,且升高了13.83倍(图3A),差异具有统计学意义(P<0.01),而未转染组与pCMV-Blank转染组比较无统计学意义。Western blotting检测结果同样表明,pCMV-KIBRA转染组KIBRA的蛋白水平明显升高(图3B),且升高了4.57倍(图3C),差异具有统计学意义(P<0.001)。该部分结果充分说明,构建的真核表达载体pCMV-KIBRA可在细胞中瞬时过表达目的基因KIBRA。
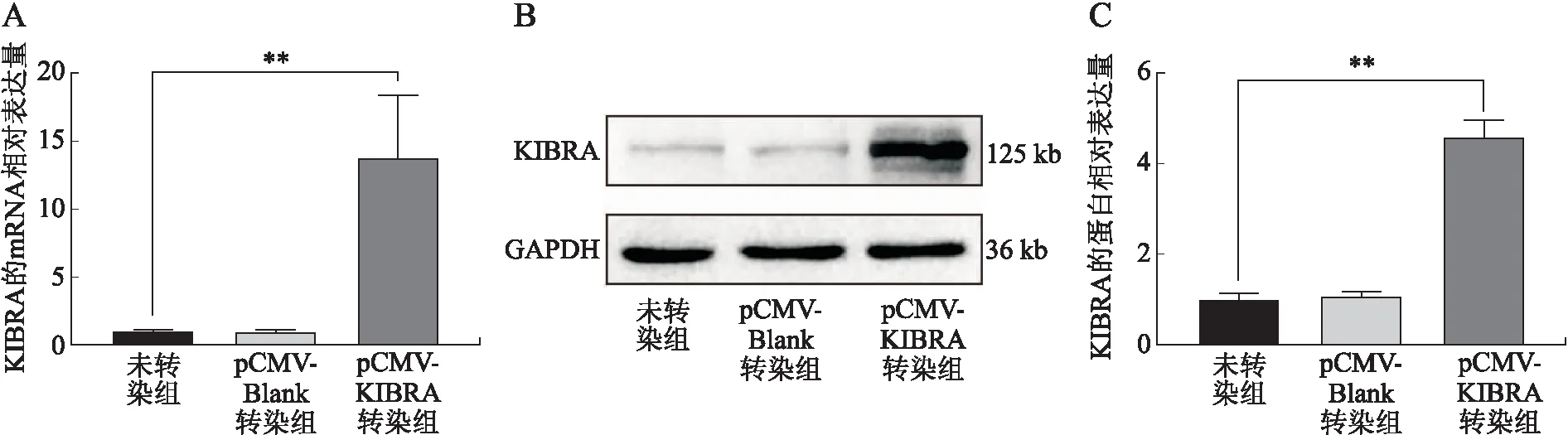
图3 转染后KIBRA的mRNA和蛋白表达水平检测Fig.3 Detection of mRNA and protein expressions of KIBRA after transfectionA:mRNA水平检测KIBRA表达情况;B:蛋白水平检测KIBRA表达情况;C:蛋白水平检测KIBRA表达情况的统计分析;未转染组、pCMV-Blank转染组分别与pCMV-KIBRA转染组相比,**P<0.01,***P<0.001。A:重组质粒pCMV-KIBRA结构示意图;B:重组质粒pCMV-KIBRA菌液PCR鉴定;C:重组质粒pCMV-KIBRA双酶切鉴定。M:1 kb marker;1~4:载体pCMV-KIBRA单克隆菌液PCR产物,其中2号和4号为阳性克隆;5、 6:2号和4号阳性克隆质粒的双酶切结果。
2.4 KIBRA蛋白特性及结构预测通过在线软件ProtParam tool对KIBRA蛋白的理化性质进行分析,结果表明,人KIBRA蛋白是由1 119个氨基酸组成,分子质量为125 904.55,理论的等电点为5.65,降解的半衰期为30 h,不稳定系数为58.64,脂肪族指数为76.64,平均亲水值为-0.682。通过在线软件PhosphoSitePlus对KIBRA蛋白的磷酸化、乙酰化和泛素化位点进行分析,结果表明,共有52个磷酸化位点,包括35个丝氨酸(S141、S144、S208、S241、S255、S427、S434、S438、S523、S525、S530、S533、S535、S539、S542、S548、S623、S626、S651、S815、S834、S841、S876、S899、S922、S927、S931、S945、S947、S962、S967、S974、S975、S978、S1081)、5个酪氨酸(Y128、Y781、Y786、Y788、Y938)和12个苏氨酸(T284、T527、T532、T837、T893、T895、T901、T912、T923、T929、T951、T1006)磷酸化位点,1个乙酰化位点(K154),5个泛素化位点(K117、K161、K324、K347、K379)(图4A)。通过在线软件SOPMA对KIBRA蛋白的二级结构进行分析,结果表明,α-螺旋530处、外延伸链104处、β-折叠49处和无规则卷曲436处,分别占其二级结构的47.36%、9.29%、4.38%和38.96%,故其二级结构以α-螺旋和无规则卷曲为主。同时KIBRA蛋白结构中包含两个WW结构域,分别位于氨基酸6~39位和53~86位,一个C2样结构域(C2-like domain)位于658~781位,三个卷曲螺旋结构域(coiled-coil motif),分别位于107~193位、293~431位和1 001~1 032位(图4B)。通过软件SWISS-MODEL对KIBRA蛋白的三维结构进行分析,2.2Å的X射线结果表明,KIBRA蛋白的三级结构主要为α-螺旋和无规则卷曲[5](图4C)。

图4 人KIBRA蛋白修饰化位点、结构域示意图和三级结构Fig.4 Modified sites, domain schematic diagram and 3D constitution of human KIBRA proteinA:人KIBRA蛋白修饰化位点示意图;B:人KIBRA蛋白结构域示意图;C:人KIBRA蛋白三级结构图。
3 讨 论
KIBRA作为支架蛋白参与多种重要的细胞功能和进程,如细胞骨架的调控、MAPK(mitogen-activated protein kinase)的活化以及细胞移动方向的调节等[6-7]。2003年首次在脑组织中发现KIBRA,其主要表达于细胞质,并在细胞核周围有明显聚集[8]。根据蛋白质结构分析,KIBRA属于进化保守的WWC家族蛋白,由2个WW结构域、卷曲螺旋结构域、C2样结构域、谷氨酸富集区(glutamic acid stretch)以及PDZ结合结构域组成[6]。在肿瘤研究中,乳腺上皮细胞中KIBRA与DDR1(discoidin domain receptor 1)结合,促进胶原诱导的MARK信号通路活化[9]。乳腺上皮细胞系MCF10A中CRB3调控KIBRA的蛋白稳定性,进而参与Hippo信号通路[3],其主要是通过WW结构域和NF2、Lats2相互结合影响Hippo信号通路[8]。同时敲低KIBRA的表达,可下调磷酸化Lats和YAP导致EMT的发生[10]。MDA-MB-231乳腺癌细胞系中,Notch3通过上调KIBRA蛋白水平抑制EMT过程[11]。KIBRA的卷曲螺旋结构域可结合Aurora A/B促使 KIBRA磷酸化以及结合PP1促使KIBRA去磷酸化[12],但目前肿瘤中KIBRA磷酸化的机制研究较少。C2样结构域是KIBRA二聚化重要结构域[13]。肿瘤组织中低表达CDC14A,在CDC14A敲除细胞系中过表达KIBRA可恢复细胞运动的表型[14],其原因可能是KIBRA的PDZ结合结构域可与极性蛋白PATJ结合,被招募到细胞膜边缘参与细胞的运动[15]。因此,KIBRA可被认为是抑癌基因,但在肿瘤中的具体分子机制尚不清楚,还值得深入探究。
本研究成功构建了真核表达载体pCMV-KIBRA,并建立了在细胞水平瞬时过表达KIBRA的方法,在mRNA和蛋白水平进行了验证。同时通过生物信息学网站分析了KIBRA蛋白的一级、二级和三级结构,并总结了KIBRA的磷酸化、乙酰化和泛素化位点。KIBRA克隆和真核表达载体的构建将为细胞水平研究KIBRA生物学功能提供基础,翻译后修饰的分析将为其在乳腺癌中的分子机制研究,特别是磷酸化KIBRA的功能研究提供了保障。

