定点突变鉴定Myxococcus sp.V11麦芽糖基海藻糖水解酶MTHase的催化中心
陈允妲,李雪亮,吴映函,赵晓艳,吴晓玉,王 飞*,陈金印
(1.江西省农业微生物资源开发与利用工程实验室,江西农业大学 生物科学与工程学院,江西 南昌 330045;2.江西省果蔬保鲜与质量安全创新中心,江西 南昌 330045)
【研究意义】海藻糖(Trehalose),又名蕈糖,是由2 个吡喃环葡萄糖分子以a,a-1,1 键连接而成的双糖,化学名为a-D-吡喃葡糖基a-D-吡喃葡糖苷[1-2]。海藻糖作为一种天然糖类,它在自然界的动植物和微生物中广泛存在,对生物有抗脱水、抗冷冻保护和抗高渗保护等多种作用。【前人研究进展】利用淀粉为底物直接生产海藻糖涉及2种酶,麦芽寡糖基海藻糖合成酶(maltooligosyltrehalose synthase,MTSase,由TreY 基因编码)和麦芽寡糖基海藻糖水解酶(maltooligosyltrehalose trehalohydrolase,MTHase,由TreZ 基因编码)[3],作用途径(TreY-TreZ)如下:麦芽寡糖在MTSase作用下通过转糖苷作用,将起始的2个葡萄糖之间的a,a-1,4 键扭转成为a,a-1,1 键,形成麦芽寡糖基海藻糖,继而在MTHase 水解作用下,将起始的海藻糖从麦芽寡糖基上水解下来,形成麦芽寡糖(比初始底物少2 个葡萄糖基)和1 分子的海藻糖[4]。α-淀粉酶(EC 3.2.1.1)是一种水解淀粉α-1,4-葡萄糖苷键,生成低聚麦芽糖和葡萄糖的糖苷水解酶类,在食品发酵、制药等领域广泛利用。在根据蛋白质的序列同源性分类的CAZy数据库中,GH13家族被认为是最主要的α-淀粉酶家族,具有4-7个保守的序列区,包括了Asp-Glu-Asp 的催化三联体和重要的底物结合位点[5],而这些特征在MTHase中也同时具有[6]。【本研究切入点】实验室分离筛选到的一株粘细菌Myxo⁃coccussp.V11在子实体形成阶段,细胞内可积累10倍于营养细胞阶段的海藻糖,通过基因组扫描发现存在着一个TreY-TreZ 合成途径,分别由orf6342 和orf3024 编码,将orf3024(TreZ,mthase)异源表达后发现不能以麦芽寡糖基海藻糖水解为底物,而是具有淀粉水解活性。【拟解决的关键问题】本文通过对推测的几个活性中心位点进行突变,来考察其功能。
1 材料与方法
1.1 材料
菌株:E.coliDH5α(pET-29a-mthase)克隆菌株(含麦芽糖基海藻糖水解酶mthase基因),E.coliBL21(pET-29a-mthase)表达菌株,克隆菌株E.coliDH5α,表达宿主菌株E.coliBL21(DE3)均为本实验室保藏。
CaCl2,NaOH,Tris,HCl,EDTA,SDS,胰蛋白胨,酵母提取物,NaCl,均购于国药集团化学试剂有限公司。琼脂糖凝胶、考马斯亮蓝G250,牛血清蛋白,卡那霉素(Km)购于上海生工。质粒提取试剂盒,凝胶回收试剂盒购自Solarbio 公司;定点突变试剂盒购自TransGen Biotech 公司;质粒提取回收试剂盒购自北京派泰克生物技术有限公司。引物合成及核酸测序委托湖南擎科生物科技有限公司。
LB培养基(g/L):酵母粉5.0,胰蛋白胨10.0,NaCl 10.0,pH 7.0~7.2。
1.2 仪器
PCR 扩增仪(Biometre T1)、Beckman台式高速冷冻离心机(Allegra X-22R)、DYCP-31DN水平核酸电泳仪、Bio-rad Mini-PROTEAN Tetra 垂直电泳仪等。
1.3 引物
定点突变引物序列见表1。
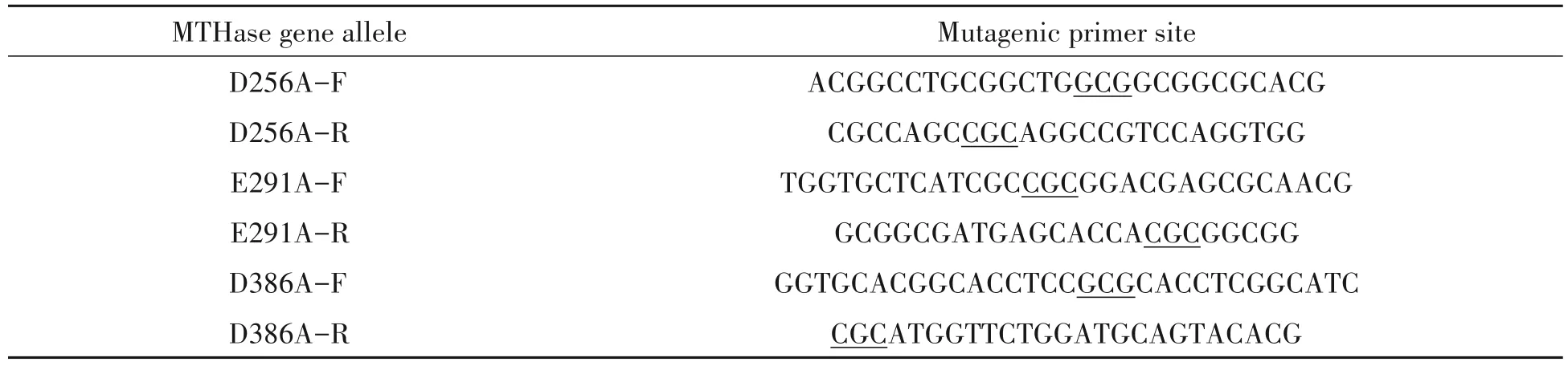
表1 MTHase定点突变PCR扩增所用引物Tab.1 Primers used for PCR amplification of MTHase directed mutation
1.4 氨基酸序列分析及突变位点的选择
将菌株V11 所产MTHase 氨基酸序列提交至NCBI 数据库,与已报道的MTHase 进行比对,下载相似度高的序列。利用MEGA 6.0软件构建系统发育树,BioEdit 7.0进行序列比对,分析保守氨基酸序列和活性中心氨基酸残基。以Swiss-Model进行三维结构在线建模。
1.5 PCR介导的定点突变
以提取的质粒为模板,进行重叠延伸PCR 扩增,PCR 体系为2× TransStart FastPfu PCR SuperMix 12.5µL,正向引物1.0µL,反向引物1.0µL,质粒DNA 1.0µL,ddH2O 9.5µL。
PCR 程序为95 ℃预变性2~5 min,95 ℃变性,55 ℃退火,72 ℃10 min,30 个循环,然后用0.75%琼脂糖凝胶电泳检测。
RCR 产物经DMT 酶(改良型的DpnI 限制性核酸内切酶)消化非突变型质粒模板后,转化至E.coliDH5α感受态细胞[7-9]。阳性克隆经电泳检测,大小无误后,送样品至祥音生物科技有限公司测序,测序正确的质粒转化至E.coliBL21(DE3)感受态细胞,构建突变表达菌株。
1.6 重组酶表达纯化
野生型和突变型的表达菌株接种至400 mL LB液体培养基(含有80 mg/L Kan),37 ℃,180 r/min培养至OD600约为0.6时,加入80µL 0.5 mmol/L IPTG,16 ℃诱导24 h。
将培养好的菌液12 000 r/min 离心5 min 收集菌体,用20 mL Tris-HCl 缓冲液(pH7.0,20 mmol/L)重悬洗涤菌体。12 000 r/min 离心5 min 后再以10 mL Tris-HCl 缓冲液重悬,超声破碎。12 000 r/min 4 ℃离心15 min,上清即为粗酶液。Ni2+-NTA 亲和层析柱预处理后将粗酶液上样,冰浴30 min。20 mL Tris-HCl 缓冲液(pH7.0,20 mmol/L)穿过,以含200 mmol/L 咪唑的20 mL Tris-HCl 缓冲液(pH7.0,20 mmol/L)洗脱,SDS-PAGE 电泳检测表达情况[10]。洗脱液在4 ℃下以截留分子量10 ku 的透析袋在20 mmol/L Tris-HCl(pH7.0)缓冲液中透析过夜去除咪唑。蛋白含量测定采用Bradford法[11]。
1.7 酶活测定方法
在含有底物玉米淀粉(0.03%,m/v)的Tris-HCl 缓冲液(20 mmol/L,pH6.5)中加入适量纯酶,40 ℃水浴反应3 h,用DNS 法测量,计算酶活。比较野生型(WT)和突变型(D256A,E291A,D386A 的单位酶活力)。酶活力定义:在40 ℃反应体系下,每分钟催化生成1µmoL 麦芽糖所需要的蛋白质含量(mg)1个酶活力单位。
2 结果与分析
2.1 MTHase的突变位点的选择
将菌株V11 所产MTHase 氨基酸序列提交至NCBI 数据库,与已报道的MTHase 进行比对,下载相似度高的序列,利用MEGA 6.0软件,邻接法构建系统进化树,结果见图1。

图1 Myxococcus sp.V11 MTHase系统发育树Fig.1 Phylogenetic tree of MTHaseof Myxococcus sp.V11
系统进化树结果表明Myxococcussp.V11 所产MTHase 与已报道的Deinococcus radiodurans所产麦芽寡糖基海藻糖水解酶(Maltooligosyltrehalose Trehalohydrolase,DR0464)相似度最高,但仅为49.23%,与已报道的淀粉酶相似度则有较大差异。
将Myxococcussp.V11 所产MTHase 与已解析的麦芽寡糖基海藻糖水解酶:来源于Deinococcus radio⁃durans(2BHU)[12],来源于Saccharolobus solfataricus(1EH9)[13],来源于Salmonella enterica(3M07)[13]氨基酸序列进行比较,结果见图2。麦芽寡糖基海藻糖水解酶具有几个严格保守的区域:GTFTPEG、WGYDG、ED、GLXVXLDXVXNHXGPXGNY、DGXRXDA、IQNHDQ。三联体活性中心(Asp256-Glu291-Asp386)[14]均位于保守区域之内。因此,拟将活性中心的3个氨基酸统一突变成没有侧链的Ala,通过观测其催化活性的变化来进行验证。
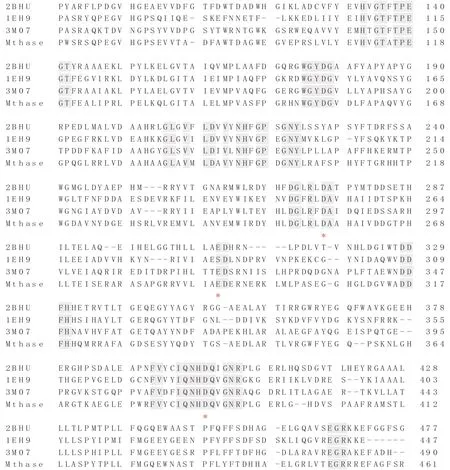
图2 MTHase与其它TreZ氨基酸序列的比较Fig.2 Comparison of amino acid sequences of different Maltooligosyltrehalose Trehalohydrolases
2.2 MTHase基因的定点突变
以表达载体pET-29a-mthase为模板,按表1 所设计的D256A、E291A、D386A 引物,按定点突变试剂盒说明进行PCR 扩增突变质粒,重叠延伸PCR 产物凝胶回收后与DMT 在37 ℃下孵育1 h,热激转化至E.coliDH5α,阳性克隆培养后提取质粒。以pET29a 空载和pET29a-mthase(WT)为对照,0.75%琼脂糖凝胶电泳检测(图3)。重组质粒大小为7 kb 左右,其超螺旋大小位于5 kb 左右。将大小无误的质粒送至湖南擎科生物科技有限公司测序,测序正确的突变质粒热激转化至E.coliBL21(DE3),构建表达菌株。
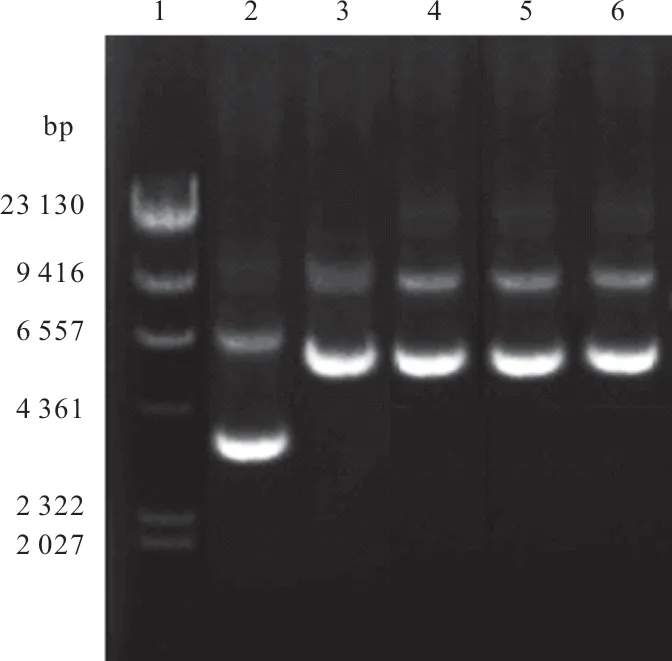
图3 重组质粒电泳图Fig.3 Agarose gel electrophoresis of site-directed mutagenesis mthase by overlapping PCR.
2.3 重组酶的诱导表达和纯化
将野生型和突变型MTHase进行诱导表达,收集菌体后,超声波破碎,12 000 r/min离心20 min 后,上清液以Ni2+-NTA 进行纯化(图4)。
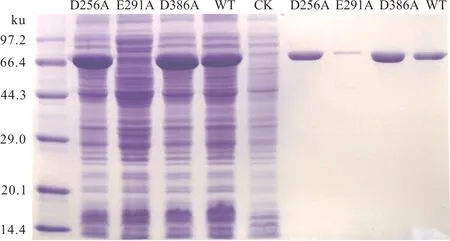
图4 突变型和野生型MTHase的SDS-PAGE凝胶电泳检测Fig.4 Analysis of the expression of the mutant enzymes on SDS-PAGE
SDS-PAGE 结果显示野生型海藻糖水解酶的蛋白质量约为68 ku,野生型WT和突变型D256A、D386A 都能够可溶性大量表达,但突变型E291A 表达量却非常低,并且也没有大量形成包涵体,这一氨基酸位点的改变对表达的影响机制尚不清楚。
2.4 突变酶的催化活性
对纯化所得重组酶洗脱液进行蛋白定量后,取1.11 mg重组酶,加入终浓度为0.03%底物淀粉后,在pH6.5、40 ℃水浴中反应3 h,以DNS 显色,测定酶活。结果显示突变型D256A、D386A 和E291A的活性均丧失,只有野生型能够测出酶活(图5)。
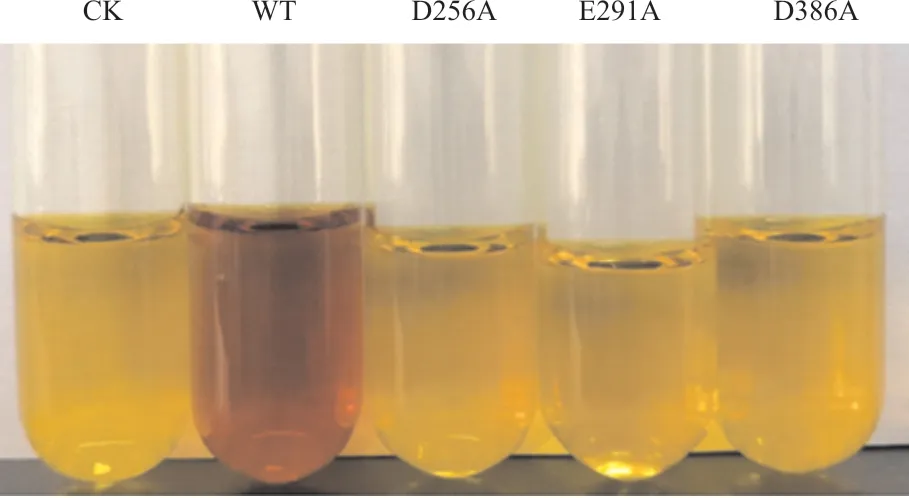
图5 野生型与突变体酶活对比Fig.5 Compared with activity of mutant recombinant MTHase and wild-type
2.5 MTHase同源建模
菌株Myxococcussp.V11 所产MTHase 与2BHU 相似度达49%,可以通过同源建模的方式来推测其三维结构。以2BHU为模板,在Swiss-Model进行三维结构预测,所得结果见图6。
MTHase 分子呈椭圆形,由3 个结构域组成(图6a),催化结构域Domain A(Ala95-Asp316,Gln372-Ser512)形成一个典型的(α/β)8桶状结构(图6b),这一结构是糖苷水解酶GH13家族的典型特征[15]。这一家族的糖苷酶包括α-淀粉酶、支链淀粉酶、环麦芽糊精酶、6-磷酸海藻糖水解酶、α-葡萄糖苷酶、海藻糖合酶、淀粉和蔗糖磷酸化酶等[16]。Domain A 的中间形成一个裂口,活性中心位于口袋之内;N 端结构域(Domain B)主要为β-折叠结构(图6b),Domain C 的差异性则被认为与底物特异性相关。催化三联体Asp256-Glu291-Asp386 位于靠近Domain C 的位置(图6c),其中Glu291 提供质子用于水解糖苷键,Asp256 为亲核试剂,Asp386 起着稳定构象的作用。图6c 显示MTHase 的Asp386(红色)与2BHU 的Asp400(蓝色)构象略有差异,这一改变是否对活性有影响尚待验证。
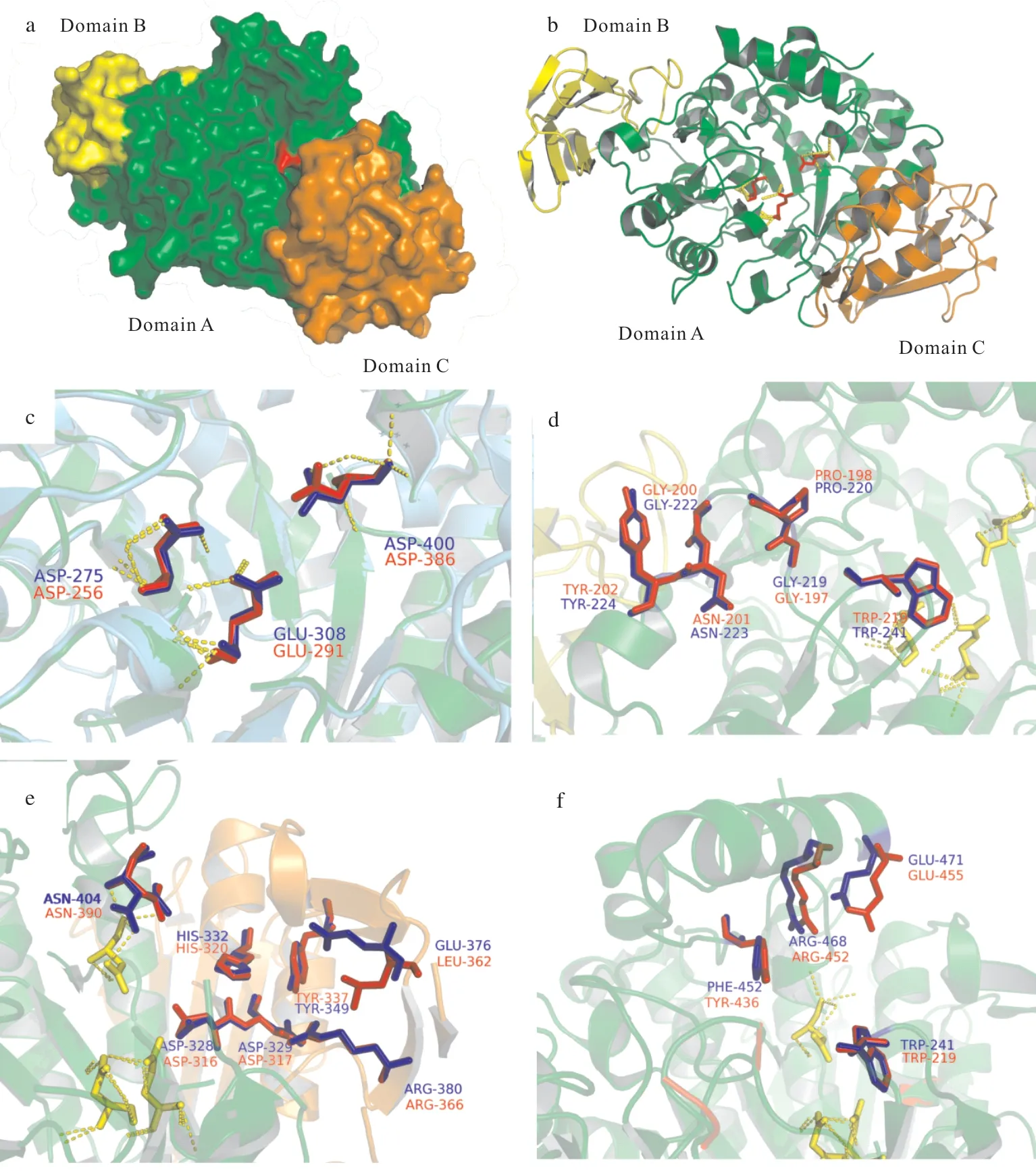
图6 MTHase的三维结构示意图Fig.6 3D structure of MTHase and 2BHU
3 结论与讨论
麦芽寡糖基海藻糖水解酶MTHase和淀粉酶amylase同属于GH13糖苷酶家族,后者随机切割淀粉分子内部的α-1,4 糖苷键,前者则对存在于海藻糖分子之后的α-1,4 糖苷键有更强作用。通常情况下,MTHase 也具有淀粉水解功能,双功能催化中心可能共用,但有些糖苷酶如海藻糖合酶的活性中心由不同的结构域组成。
本文通过定点突变的方法对预测的菌株Myxococcussp.V11所产MTHase活性中心的3个氨基酸残基Asp256-Glu291-Asp386进行了验证,结果证实这3个位点氨基酸的改变直接导致淀粉酶活力的丧失,突变重组酶以底物海藻糖-麦芽糖基进行验证时,同样也没有测到酶活。表明MTHase的催化中心正是由Asp256-Glu291-Asp386所构成。
学术界普遍认为GH13 家族糖苷酶对不同底物的适应性主要与底物结合的氨基酸位点有关[17-21],MTHase 与底物分子麦芽糖基海藻糖的第1 个葡萄糖苷连接的氨基酸分别为Gly197,Pro198,Gly200,Asn201,Tyr202,Trp210 均与2BHU 一致(图6d);第2 个葡萄糖苷连接的氨基酸Asp316,Asp317,His320,Tyr337,Arg366,Asn390 也与2BHU 一致,唯一的区别是2BHU 里的Glu376 在MTHase 中被替换成了Leu362(图6e);与第3个葡萄糖苷连接的氨基酸Trp219,Arg452,Glu455也与2BHU一致,不同的是2BHU里的Phe452 在MTHase 中被Tyr436 所替换(图6f)。导致MTHase 的活性表现为淀粉酶活性的原因是否由于Glu362Leu和Phe436Tyr所致尚待进一步研究。

