应用Illumina MiSeq测序技术比较传统发酵乳、肉食品中细菌多样性
杜 瑞,王柏辉,罗玉龙,王 宇,田建军,沙如拉,靳 烨*
(1 内蒙古农业大学 呼和浩特010018 2 内蒙古乌拉特中旗畜牧工作站 内蒙古巴彦淖尔015300)
我国内蒙古地区草原资源丰富,畜牧养殖业历史悠久,形成了这一地区丰富的乳、肉发酵制品,并拥有大量的益生菌资源。传统发酵乳制品是以品质较好的牛乳、羊乳及马乳为原料,经过乳酸菌、双歧杆菌和酵母菌等发酵成的一类具有独特风味,并可以调节肠道菌群和提高蛋白质和维生素代谢作用的一类产品,代表产品有酸奶、奶豆腐及干酪[1-4]。发酵肉制品是以新鲜牛肉、羊肉、马肉为原料,吊挂于阴凉处,利用微生物或酶的发酵作用而形成的风味独特的一类肉制品[5]。
在发酵食品中的微生物包括乳酸菌、微球菌和酵母等,它们在其制品风味的形成和安全性方面都发挥了各自独特的作用。乳酸菌可分解碳水化合物产生乙酸、双乙酰,酵母菌可分解产生乙醇等物质。受地理环境、人为因素等影响,不同地区、同种发酵食品和同一地区、不同工艺发酵食品的品质特性存在较大差异,这归因于不同的微生物群落。张敏等[5]利用16SrDNA 高通量测序技术分析新疆西北部地区乳品中的微生物,结果原奶样品中的菌群主要以变形菌门(Proteobacteria)为主,其中牛奶的优势菌属为假单胞菌属(Pseudomonas),驼奶为埃希菌属-志贺菌属(Escherichia-Shigella),马奶为明串珠菌属(Leuconostoc),羊奶则为乳球菌属(Lactococcus);而酸奶样品主要以厚壁菌门(Firmicutes)为主,酸牛奶、酸驼奶和酸马奶的优势菌属均为乳杆菌属(Lactobacillus)[6]。通过Illumina 测序发现天然发酵香肠及冷鲜滩羊肉中厚壁菌门和变形菌门为优势菌门,链球菌属(Streptococcus)、克吕沃尔氏菌属(Kluyvera)、弧菌属(Vibrio)、乳球菌属(Lactococcus)、明串珠菌属(Leuconostoc)为发酵香肠的优势菌属,假单胞菌属及不动杆菌属(Acinetobacter)为羊肉腐败变质的主要优势菌属[7-8]。
目前,发酵乳、肉制品中微生物的多样性研究越来越受到人们的关注,早期研究仅依赖培养的方法对微生物进行分离鉴定,而高通量测序技术不仅可以检测到普通的可培养物种,还能检测到那些难培养、低丰度以及难以分离的微生物[3]。本研究通过研究传统发酵乳、肉制品中微生物群落的组成和结构,深入了解不同传统发酵食品中微生物多样,挖掘我国特色发酵食品的微生物资源。
1 材料与方法
1.1 材料与试剂
DNA 快速提取试剂盒,德国Qiagen 公司;PCR 扩增的全套试剂及扩增引物,日本TaKaRa公司;5×TBE 电泳缓冲液(Tris 碱54g,Na2EDTA-2H2O 3.72 g,硼酸27.5 g,定容1 000 mL,pH 8.0);1%琼脂糖凝胶(1.0 g 琼脂糖溶于100 mL 0.5×TBE 电泳缓冲液),天津基准化学试剂公司;保护液,日本TaKaRa 公司。
1.2 仪器与设备
紫外-可见分光光度计(TU-1810),北京普析通用仪器有限责任公司;凝胶成像系统,美国Biorad;稳压稳流电泳仪(BG-power 3500 型),北京百晶生物技术有限公司;核酸蛋白分析仪(Nano-Drop2000),美国Thermo Fisher Scientific 公司;Veriti96 Well Thermal Cycler PCR 仪,美国Applied Biosystems 公司;高速台式冷冻型离心机(SIGMA3-18K),德国Sartorius 公司;旋涡振荡器(UVS-1),北京优晟联合科技有限公司。
1.3 方法
1.3.1 样品采集 本试验样品随机采自内蒙古阿拉善牧区的不同牧民家庭,包括自然发酵香肠(Sausage)、风干牛肉(Beef_jerky)、风干羊肉(Mutton_jerky)、饼状奶酪(Cheese1)、棒状奶酪(Cheese2)以及奶豆腐(Mike_cake)等6 种传统发酵制品。具体采样过程:用无菌采样勺采集20 g样品,装入无菌、无酶离心管后立即加入15 mL 保护液,密闭后低温运回实验室,于-80 ℃保存,用于后续试验分析。
1.3.2 样品总DNA 提取和检测 准确称取混匀的样品0.3 g,依照DNA 快速提取试剂盒说明书提取样品中细菌宏基因组DNA,之后用1%琼脂糖凝胶电泳和微量紫外分光光度计检测DNA 完整度、纯度及浓度,保证其满足后续试验要求。将DNA 样品暂存于-20 ℃冰箱保存,备用。
1.3.3 细菌16S rDNA 序列扩增和MiSeq 测序将纯化的DNA 作为模板,以515F(5’-GTGCCAGCMGCCGC GG-3’)和926R(5’-CCGTCAATTCMTTTGAGTTT-3’)为引物,对细菌的16S rDNA V3~V4 可变区进行PCR 扩增。扩增体系(20 μL):4 μL 5×TaqFastfu Buffer,2 μL 2.5 mmol/L dNTPs,0.4 μL 引物,0.5 μL DNA 模板(50 ng),用ddH2O 补至20 μL。扩增程序为:95 ℃预变性3 min,27 个循环(95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s),最后72 ℃延伸10 min。将Illumina 平台测序所需接头、引物、标签序列添加到目的片段两端。全部PCR 产物采用AxyPrepDNA 凝胶回收试剂盒回收,用PCR 仪进行荧光定量,均一化混匀后完成文库构建,在Illumina MiSeq 平台上完成测序。
1.4 数据处理
使用QIIME 分析平台开展序列的生物信息学分析[9-10]。用PyNAST 校准排齐序列[11],以100%相似性进行UCLUST 归并,从而建立无重复的926R→515F 序列集。采用两步UCLUST 归并,在100%相似性归并的基础上进行97%相似性的归并,建立分类操作单元(Operational Taxonomic Units,OTUs)。通过Chimera Slayer 检测去除属于嵌合体的OTU。将OTU 代表性序列通过RDP[12]和Greengenes(Release13.8)数据库进行同源性比对,整合两个数据库的比对结果,确定每个OTU 最终的分类学地位。通过Alpha 多样性分析反映样品菌群构成的丰度和多样性。同时使用稀疏曲线(rarefaction curve)和香农指数曲线(Shannon index map)评估每个样本测序的多样性以及在不同的OTUs 划分水平下的多样性,以评价测序量是否能够代表原始群落的多样性。对样品进行的PCoA 分析并基于16S rRNA 基因扩增子测序结果,对微生物基因进行16S 功能预测分析[13]。采用Origin 8.0 和Excel 2010 软件作图。
2 结果与分析
2.1 基因序列质量评估
通过细菌的16S rDNA 基因V4~V5 区测序,6 种发酵食品共产生194 568 条有效序列,其中3种发酵肉制品共得到94 158 条高质量序列,3 种发酵乳制品总共得到100 410 条高质量序列(见表1)。将高质量的序列在相似度98%的水平上归类,发现6 份样品可在种属水平上分类,共聚成237 个OTU。其中,发酵肉制品中共143 个OTU,发酵乳制品中共94 个OTU。为了评估此次测序深度是否达到分析要求,绘制样品细菌稀疏曲线(Rarefaction Curves)和香农曲线(Shannon index),如图1 和图2所示。在当前的测序量下,每个样品的细菌稀疏曲线都接近平台期。与此同时,每个样品的细菌香农曲线多样性曲线已饱和,表明测序量增加新的种系型可能被发现,然而细菌多样性不再随之发生变化,说明现有测序量可反映样品中绝大多数的细菌物种信息。本研究测序量满足后续生物信息学分析要求。
2.2 传统发酵制品菌群多样性分析
由表1所示,所有样品的覆盖率均大于99%,说明样品文库的覆盖率很大,可反映本测序结果代表样本的真实情况。在发酵制品中,Shannon 指数大,Simpson 指数小,表明样本菌群多样性指数高[14]。发酵肉制品平均Shannon 和Simpson 指数分别为3.0 和0.2,发酵乳制品平均Shannon 和Simpson 指数分别1.6 和0.4。试验中发酵肉制品与发酵乳制品相比,Shannon 指数大,Simpson 指数小。通过Chao1 指数对样品中微生物的丰富度进行评估,Chao1 指数越大样品中菌群丰富度越高。发酵肉制品中Chao1 指数平均值为157,发酵乳制品为109。由此可知,肉制品中细菌多样性和丰富度均高于发酵乳制品。

图1 稀疏曲线Fig.1 Rarefaction curves
图2 香农指数图Fig.2 Shannon index
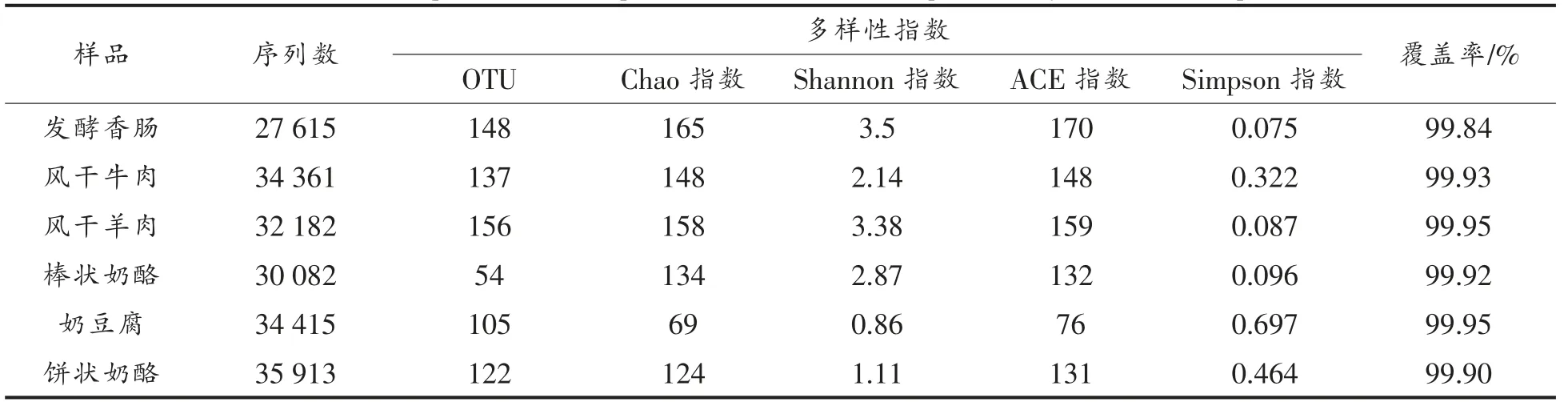
表1 不同样品的代表性序列及微生物多样性Table 1 Representative sequences and numbers sequenced by different samples
2.3 样品菌群主成分分析
基于样本OTU,采用PCA 主成分分析乳、肉样品中的群落结构差异。如图3所示,第1 主成分(PC1)和第2 主成分(PC2)的贡献率分别为74.43%和15.45%,可代表绝大部分的变量信息。通过菌群主成分分析,乳制品和肉制品的菌落能够被明显区分,因其制作原料和工艺不同,故乳、肉制品的微生物菌落也呈现一定的聚类趋势,说明二者有一定的同源性。
2.4 发酵制品菌群结构分析
2.4.1 菌群门水平分布 采用SILVER 数据库同源性序列比对并结合聚类的方法对序列进行鉴定,在发酵食品中鉴共定出14 个门。如图4所示,发酵乳制品中共有5 个菌门含量高于1.0%,含量最多的细菌门为厚壁菌门,其次为变形菌门,这与Aldrete-tapia 等[15]的研究结果一致。奶豆腐和饼状奶酪中厚壁菌门(95.48%)比例明显大于变形菌门(3.84%),而在棒状奶酪中相差不大。此外,在发酵食品中还检测到栖热菌门(Deinococcus-Thermus)、蓝藻菌门(Cyanobacteria)、拟杆菌门(Bacteroidetes),所占比例较低。
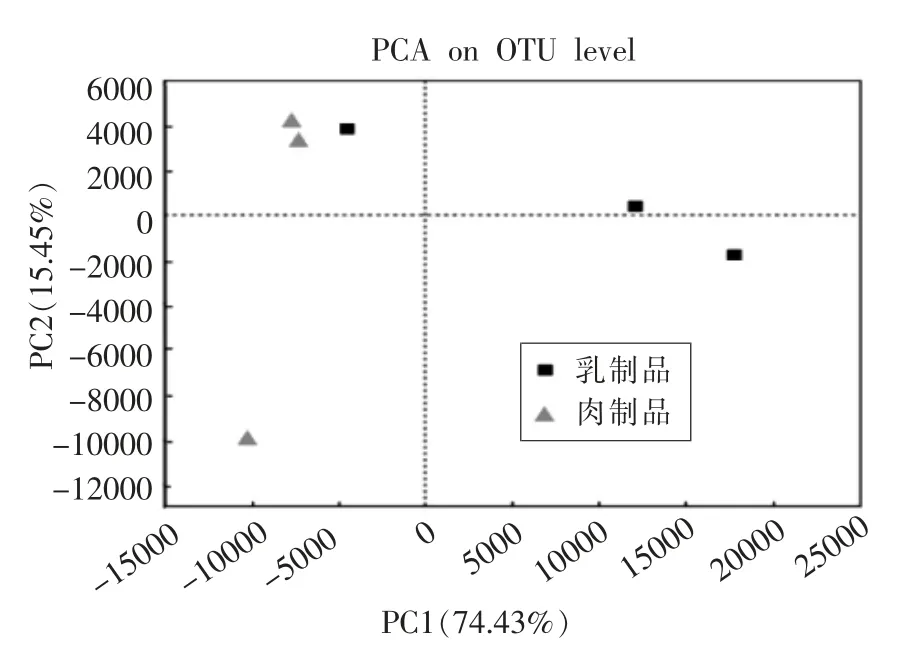
图3 细菌群落结构的主坐标分析Fig.3 Principal co-ordinates analysis of bacterial community
如图5所示,在发酵肉制品中发现门水平上菌群相对丰度高于1.0%的微生物共有8 个,其中厚壁菌门和变形菌门含量占到测序总数80%左右,是主要的优势菌;而栖热菌门(Deinococcus-Thermus)、蓝藻菌门、拟杆菌门、螺旋菌门(Spirochaetae)、互养菌门(Synergistetes)、放线菌门(Actinobacteria)所占比例较低,这与郭明亮[16]研究结果一致。风干牛肉中厚壁菌门(66.15%)为优势细菌门,变形菌门(21.53%)为次优势细菌门,而风干羊肉厚壁菌门(22.92%,变形菌门(57.67%)与之相反,发酵香肠中厚壁菌门(36.40%)和变形菌门(44.26%)差距不大。对比风干牛肉、风干羊肉和发酵香肠中菌群种类,发现不同肉类产品,其菌群结构和相对丰度有较大差异,而菌群结构的不同可能会引起发酵肉中醇类、酯类和酸类等挥发性香气成分的差异[17]。风干羊肉中细菌的丰度和多样性高于风干牛肉,说明在风干羊肉中更易筛选出优良菌种。

图4 门水平上发酵乳制品中菌群含量(%)Fig.4 Bacteria abundance in fermented dairy products(%)at phylum level
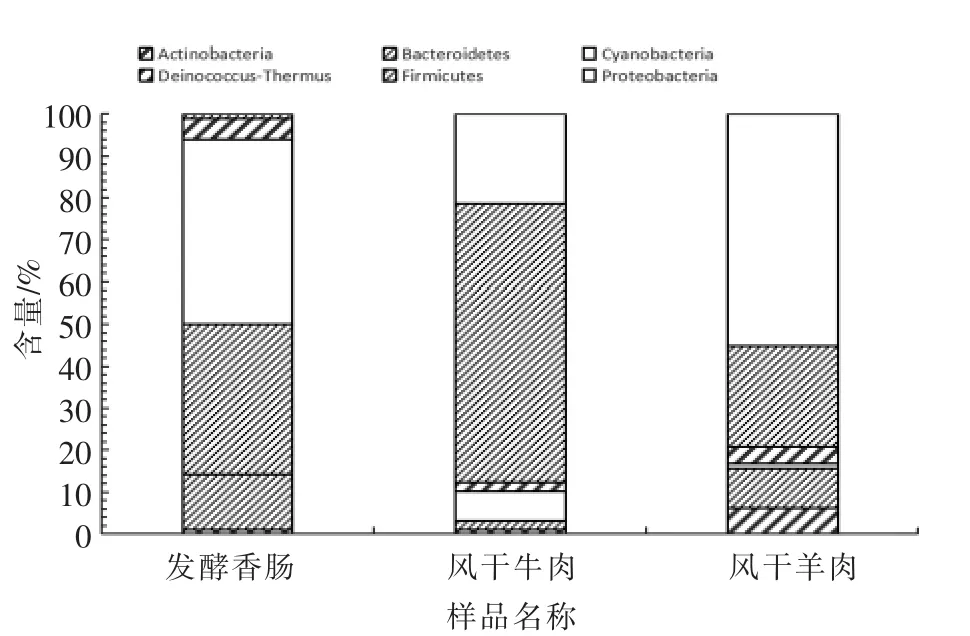
图5 门水平上发酵肉制品中菌群含量(%)Fig.5 Bacteria abundance in fermented meat products at phylum level(%)
2.4.2 菌群属水平分布 经序列比对,在传统发酵食品中共鉴定出81 个属。如图6所示,发酵乳制品中有15 个菌属含量高于1.0%,以乳杆菌属(Lactobacillus)、链球菌属(Streptococcus)、不动杆菌属(Acinetobacter)等细菌菌属为主,其中乳杆菌属为发酵乳制品优势菌属。乳酸菌将干酪中残留的乳糖转化为乳酸,在一定程度上抑制酸敏感性致病菌和腐败微生物的生长,并能提高凝乳酶的凝乳性,可用作发酵乳的菌种[18]。在饼状奶酪中含有较高的链球菌属(30.71%),其中棒状奶酪的乳球菌属(33.79%)、醋杆菌属(Acetobacter,1.70%)和葡糖杆菌属(Gluconobacter,0.28%)总丰度较低,原因可能是棒状奶酪在发酵过程时产酸,凝乳过程较缓慢。棒状奶酪中不动杆菌属(9.53%)、栖热菌属(5.93%)、链球菌属(7.91%)丰度高,这与干酪制作工艺中耐高温且产品黏度较高有关[19]。栖热菌是耐高温细菌,干酪加工温度达50℃时才能生长,推测该菌属可作为工业微生物资源应用于高温条件下乳糖的发酵或干酪加工。而不动杆菌属可以通过产生果聚糖(荚膜多糖)来提高牛奶黏度。Gennari 等[20]发现不动杆菌属存在于凝乳和软奶酪表面。
如图7所示,发酵肉制品在属水平上主要以乳杆菌属、假单胞菌属、不动杆菌属、链球菌属、明串珠菌属(Leuconostoc)、肉食杆菌属(Carnobacterium)、密螺旋体(Treponema)为主,这与郭明亮[16]的研究结果基本一致。发酵肉制品中菌群含量高于1.0%(选出有代表性的菌属)的共有21 个菌属,其中假单胞菌属为优势菌属,乳杆菌属为次优势细菌属,乳杆菌属在风干牛肉中所占比例较大(58.52%),在肉制品发酵过程中占绝对优势。乳酸菌能利用肉中碳水化合物产生乳酸及少量乙酸、甲酸、琥珀酸等,不仅能抑制病原菌和腐败菌的生长,还可赋予肉制品独特的风味。Ercolini 等[21]发现在好氧条件下假单胞菌属对肉制品腐败起主要作用。由图7 可知,发酵香肠中的肉杆菌属(Carnobacterium,7.94%)和葡萄球菌属(Staphylococcus,6.47%)的含量明显高于风干肉。Quijada等[22]利用高通量测序技术研究了干发酵香肠中的微生物群落,乳杆菌属可占65.1%~92.0%;Fontana等[23]利用高通量测序技术研究了骆驼肉发酵香肠中的乳酸菌群落,发现优势菌是乳杆菌属、明串珠菌属和假单胞菌属等,和本结果较为一致。
2.4.3 菌群种水平分布 经序列比对法,在传统发酵食品中共鉴定出33 个种。如表2 和表3所示,因未检测到的菌群含量高达80%,故在发酵乳制品和发酵肉制品中分别列出丰度排名前6 的菌种。如表2所示,发酵乳制品中丰度排名前6 的菌种含量均高于0.3%,棒状干酪中有较高数量的清酒乳杆菌(Lactobacillus sakei)和栖热菌(uncultured Thermus sp),饼状干酪中菌群结构与棒状干酪相似,较棒状干酪中菌种整体数量低。奶豆腐中清酒乳杆菌、栖热菌属和食窦魏斯氏菌(Weissella cibaria)含量较少。棒状和饼状干酪中含少量的棉子糖乳球菌(Lactococcus raffinolactis)。

图6 属水平上发酵乳制品中菌群含量(%)Fig.6 Bacteria abundance in fermented dairy products at genus level(%)
发酵肉制品中丰度排名前6 的菌种含量均高于3.0%,包括清酒乳杆菌、栖热菌属等。清酒乳杆菌在风干牛肉中的含量最高(55.43%)。发酵香肠中未分类的细菌高于风干肉,表明发酵香肠中发现新的菌种的可能性高。
2.5 16S 功能预测分析
COG 丰度越高,微生物代谢功能越强。通过COG 功能分类统计(表4)可知,传统发酵食品中的微生物代谢功能非常丰富,主要的功能有氨基酸转运与代谢、碳水化合物转运与代谢、核苷酸转运与代谢、能量生产与转换等。

图7 属水平上发酵肉制品中菌群含量(%)Fig7 Bacteria abundance in fermented meat products at genus level(%)

表2 种水平上发酵乳制品中菌群含量(%)Table 2 Bacteria abundance in fermented dairy products at species level(%)
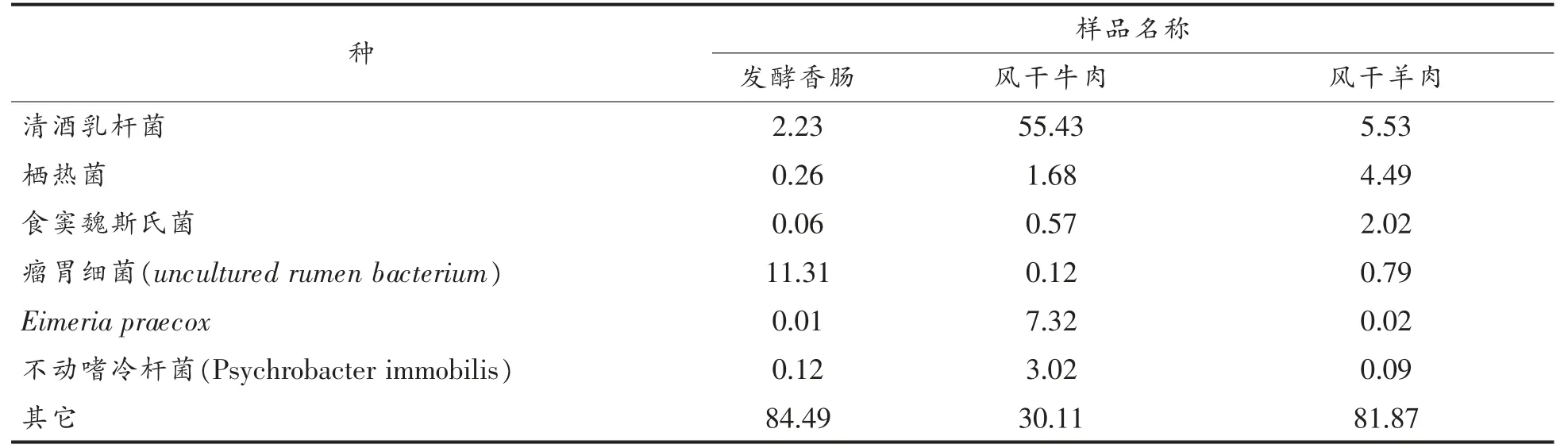
表3 基于种水平上发酵肉制品中菌群含量(%)Table 3 Bacteria abundancein fermented meat products at species level(%)

表4 基于COG 功能分类统计Table 4 Classified statistics based on COG Function
3 讨论
通过高通量测序技术全面比较分析内蒙古不同发酵食品中细菌群体结构及多样性,在两类发酵食品中,发酵肉制品具有较高的多样性。分析发酵乳制品的OTU 值发现,不同的发酵乳制品中细菌多样性和丰度的相差不大,这与张冬蕾[24]的研究结果一致。
本研究发现在门水平,发酵乳制品中的优势菌群为厚壁菌门和变形菌门,饼状干酪、奶豆腐与棒状干酪的细菌结构有差异,这可能是环境因素,如温度和湿度不同使得奶酪在发酵过程中菌落结构改变,进而改变了奶酪的风味[25]。在发酵肉制品中,厚壁菌门和变形菌门比例不同,这说明肉的种类影响菌群的比例。米瑞芳等[17]通过高通量测序技术发现4 种传统发酵酸肉的细菌群落中,厚壁菌门占绝对优势,占总细菌群落的83.73%~98.92%,其次为变形菌门和放线菌门。Wang 等[26]研究干腌肠与烟熏肠的菌群,厚壁菌门和变形菌门为优势菌门,其次是拟杆菌门和放线菌门,这与本试验结果一致。
在属水平上,乳杆菌属为发酵乳制品的优势菌属。Shangpliang 等[27]发现醋杆菌属、葡糖杆菌属是自然发酵乳制品中主要的产酸菌。在发酵肉制品中有近20%的菌种不能鉴定到属水平,而发酵乳制品中不能鉴定菌种相对较少,说明在发酵肉制品中还有发现新的菌属的可能性。假单胞菌属为发酵肉制品优势菌属,乳杆菌属为次优势细菌属。Wang 等[26]研究干腌香肠发现,葡萄球菌属是干腌香肠的优势菌属,食窦魏斯氏菌为次优势菌属,与本试验结果不一致,原因可能是制作工艺不同影响菌群的构成。在试验中发酵香肠的肉杆菌属和葡萄球菌属的含量明显高于风干肉,肉杆菌属有生物保护剂的用途,它能产生抑制李斯特菌(Listeria monocytogenes)生长的细菌素[28];而葡萄球菌属与微球菌属(Micrococcus)都具有硝酸盐还原酶活性,使肉制品在发酵过程中呈鲜艳的红色,起到稳定肉色的作用[16]。
在种水平上,由于未检测到的菌群含量较高,所以不能确定乳肉制品优势菌种。在可检测的菌群中,发酵食品中清酒乳杆菌为优势菌种。研究表明,清酒乳杆菌普遍存在于风干香肠、发酵香肠等发酵肉制品中,它不仅能缩短香肠的成熟期,而且能提高了产品的风味与质量,是肉制品发酵过程中的主要发酵微生物[29]。发酵乳制品中还发现少量的棉子糖乳球菌,其可发酵α-半乳糖苷,然而水解酪蛋白能力低,因此未在乳品行业广泛应用[30-33]。
通过16s 预测功能分析发现,传统发酵食品中绝大部分细菌与转运代谢功能有关。破坏正常菌群的结构可能对这些代谢通路产生阻碍。在菌群中还有一大部分功能未知,可能与传统发酵食品中特定的生物学特性有关,如蛋白修饰等功能,这是未来的一个研究方向。
4 结论
通过Illumina 高通量测序技术,在传统发酵食品中得到大量的高质量的细菌16S rDNA 基因序列,具有丰富的细菌多样性,其中厚壁菌门是干酪发酵过程中的优势菌门,乳杆菌属为优势菌属。厚壁菌门和变形菌门是发酵肉制品的优势菌门,假单胞菌属为优势菌属,乳杆菌属为次优势菌属。在种水平上,由于未检测到的菌群含量较高,因此不能确定乳肉制品优势菌种。清酒乳杆菌是发酵乳肉制品中可监测到的菌群含量最高的。通过16s预测功能分析,发现传统发酵食品中的绝大部分细菌与转运代谢功能有关。由此说明在内蒙古特色发酵食品中蕴含着丰富的微生物资源,且同一地区不同发酵产品中微生物的多样性及菌落结构也不同。本研究成果为传统发酵食品细菌群落结构研究和筛选优良菌种提供一定参考价值。

