免疫基因和代谢基因在胃癌患者预后分析中的比较研究
周 霞,江绍萍,叶明亮,喻文霞,钟 琦
(云南民族大学 数学与计算机科学学院, 云南 昆明 650500)
在恶性肿瘤中, 胃癌发病率与死亡率分别排行第4、第3[1], 其诊治和预后评估一直是专家学者和临床医生关注的重点之一. 胃癌起源于胃黏膜上皮, 其癌变的发展由多种因素, 多个阶段造成, 是一种目前还不能完全治愈的疾病, 且患者多数为50岁以上. 我国胃癌发病率极高, 胃癌患者约占世界胃癌患者一半, 纵然近些年来胃癌的诊治取得了一些良好的进展, 但胃癌患者的预后效果普遍较差[2].
现在越来越多的证据表明, 免疫基因在胃癌的发生和发展中起着关键作用[3], 如章婧文[2]运用CIBERSORT算法解析胃癌中22种免疫细胞的构成, 并筛选免疫细胞去建立免疫风险评分模型和个体化列线图模型以预测胃癌患者的死亡风险. 王尧等[4]采用ESTIMATE及CIBERSORT分别评估胃癌样本中免疫微环境及免疫细胞的组成, 总结出胃癌免疫分型有助于判断患者的预后, 且高免疫组患者具有较高的免疫原性及抗肿瘤免疫活性. 张静等[5]通过生物信息学方法分析发现胃癌Her-2基因高表达可能会对免疫系统产生一定的抑制作用, 从而影响免疫系统发挥抗肿瘤的作用. 这些都表明了免疫基因能够对胃癌患者产生直接或间接的影响.
胃癌的发生发展也与代谢基因的变化密切相关. 陶柱萍等[6]基于代谢基因发现了多种胃癌的内源性代谢生物标志物, 总结归纳出代谢组学技术在胃癌早期诊断及预后方面的研究进展,为胃癌的研究提供了1个参考. 向丽娟等[7]根据转录组测序, 筛选出胃癌与相邻非癌样本之间的所有基因表达量, 进行富集分析, 认为脂代谢中的某些基因与胃癌的相关通路有关联, 得到胃癌脂代谢中关键基因的异常表达可为寻找胃癌的诊断和预后指标提供线索. 赵丽沙等[8]从数据处理方式、取得的差异代谢物及其调节的代谢通路等方面研究了代谢组学技术在胃癌早期诊断与预后判断的临床应用, 为代谢基因应用于胃癌早期诊断及预后判断提供了参考的依据. 这些都说明了代谢基因能够对胃癌患者的诊断和预后分析提供1个参考价值.
综上可以看出免疫基因和代谢基因都可能影响胃癌患者的预后分析. 因而本研究将基于LASSO回归构建预后风险模型[9], 并根据特异性与灵敏度之和最大的点来划分患者的风险, 并从生存曲线, 生存风险分布等图表分别对免疫基因和代谢基因进行比较, 进一步了解哪个基因能更好的对胃癌患者进行预后及生存状况分析.
1 数据来源与预处理
在癌症基因组图谱(the cancer genome atlas, TCGA)数据库中获得胃癌完整的信使RNA表达数据和临床信息. 本次研究包含375个胃癌样本和32个相邻的非癌性样本. 研究中先从这些数据提取相应的临床信息, 包括患者的基本资料, 肿瘤分期, 肿瘤分级, TNM分类(见表1). 然后从Immport数据库中下载免疫基因相关信息. 使用R语言的limma包提取差异表达的免疫相关基因, 于是在胃癌的 56 753 个基因中找到 1 651 个免疫相关基因, 并提取所有免疫基因的表达量. 最后, 利用基因表达汇编(gene expression omnibus, GEO)数据库获得具有详细生存信息的433个胃癌样本及提取免疫基因表达量. 把从TCGA数据库和GEO数据库中获得的免疫基因矩阵取交集, 得到 23 355 组免疫基因对.

表1 部分临床样本数据表
同样的, 我们从下载的信使RNA表达数据和临床信息中获取代谢相关的基因, 最终在胃癌的 56 753 个基因中找到944个代谢相关基因, 并且把代谢基因的表达量提取出来. 最后把胃癌患者的生存信息与免疫基因和代谢基因合并.
2 实验设计
2.1 方法
用单因素Cox分析筛选出与胃癌患者总体生存率显著相关的免疫基因和代谢基因, 并计算出每个基因的风险比(hazard ratio, HR). 基于套索算法(least absolute shrinkage and selection operator, LASSO)构建预后模型. 依据风险值=∑(基因系数×基因表达量)计算出每个患者的风险评分, 与获得的最佳截断值(cutoff)进行比较, 绘出接收者操作特征曲线(receiver operating characteristic curve, ROC), 将患者区分为高低风险两组. 最后, 通过乘积极限法(kaplan-meier survival estimate, K-M)对胃癌患者进一步分析, 并采用对数秩检验. 绘制相关的风险曲线去分析预测胃癌患者的总体生存有效性, 计算ROC曲线下面积(area under curve, AUC), 用于分析模型的准确率.
2.2 实验环境
所有的统计分析均使用R4.0.1(www.r-project.org)和Strawberry Perl (www.perl.org/)进行,P值小于0.05为差异显著且具有统计意义.
2.3 预后基因筛选
通过单因素Cox分析方法筛选出与375例胃癌患者总体生存率显著相关的免疫基因和代谢基因, 得到与预后相关的37对免疫基因和16对代谢基因以及它们每个基因的HR值(见表2, 表3).
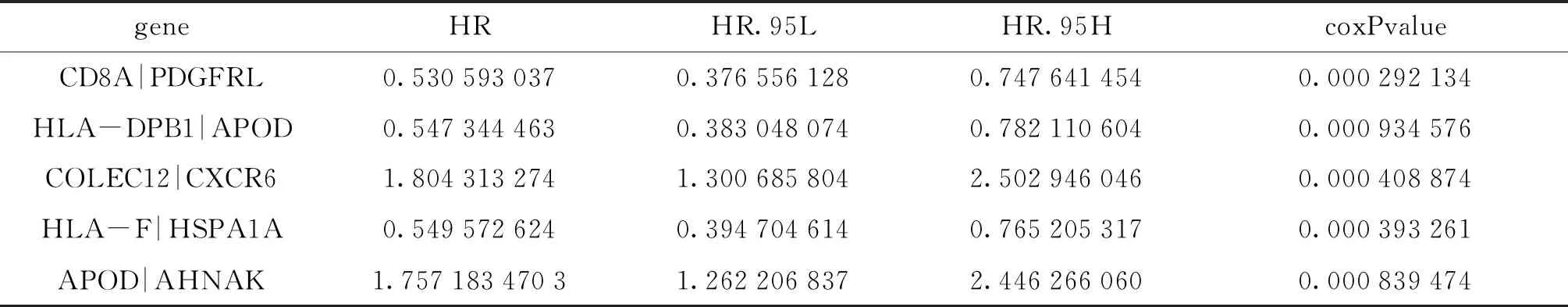
表2 部分免疫基因的HR值
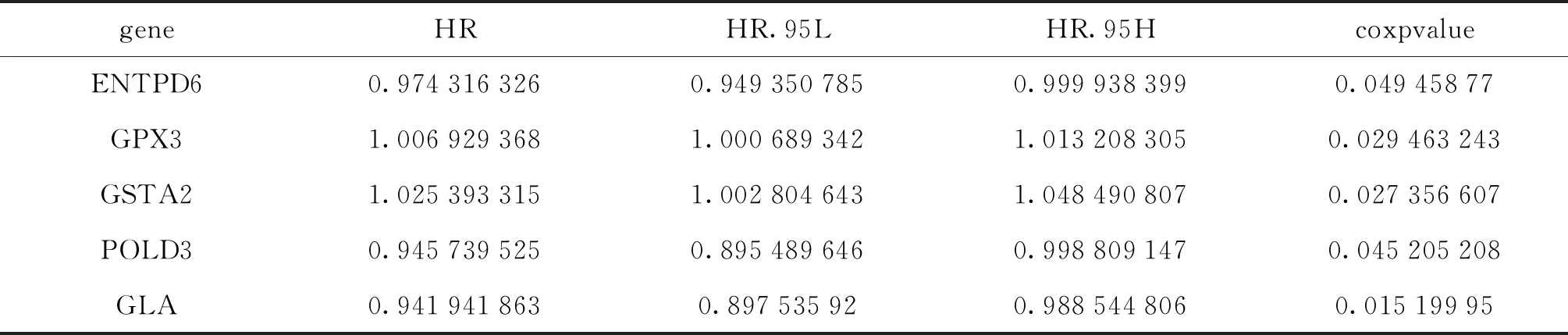
表3 部分代谢基因的HR值
表3中, 若HR值大于1表示该基因参与预后的风险较高, HR值小于1表示它参与预后的风险较低; 若coxPvalue值大于0.05说明这个基因与生存不相关, coxPvalue值小于0.05说明该基因与生存相关, 可以作为预后分析基因.
2.4 基于LASSO回归的预后模型建立与验证
2.4.1 LASSO回归模型
构建风险评分模型的最佳方法是LASSO回归, 它经过构造惩罚参数, 将变量的系数实行压缩, 使某些系数变为0, 从而取得一个更为精炼的模型. 同时为了避免出现过度拟合现象, 运用同一体系的参数来控制该模型的复杂性. LASSO回归可通过普通线性模型增加L1惩罚项得到. 普通线性模型为:
Y=Xβ+ε,
(1)
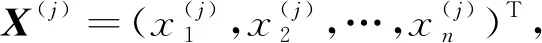
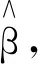
(2)
当矩阵X不满足列满秩时, 回归系数将不能使用OLS去求解, 在这种情况下, 引入惩罚方法, 即将最小惩罚似然函数的值作为回归系数的估计值, 从而可以获得LASSO回归的模型:
(3)
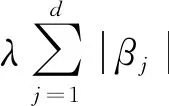
2.4.2 基于免疫基因和代谢基因的胃癌预后分析的LASSO回归模型
对预后相关的37对免疫基因使用LASSO回归, 最终选择了19对免疫基因用于构建模型(见表4): HLA-DQB1|CXCR4, HLA-F|HSPA1A, MICB|ESM1, MICB|NR2F1, MICB|NRP1, CXCL13|CD79A, S100A9|AGT, APOD|AHNAK, APOD|DMBT1, APOD|BMP4, ITGAV|MAPK3, TLR1|CTLA4, ARRB1|STC1, TNFSF4|CXCR6, VAV3|SCG2, PTPN6|PLAUR, EDN3|DKK1, SBDS|PLXNB2, FAM3C|ESRRA.
同样的,对预后相关的16个代谢基因使用LASSO回归, 最终选择了13个代谢基因用于构建模型(见表5): GSTA2, POLD3, GLA, GGT5, DCK, CKMT2, ASAH1, OPLAH, ME1, ACYP1, NNMT, POLR1A, RDH12.
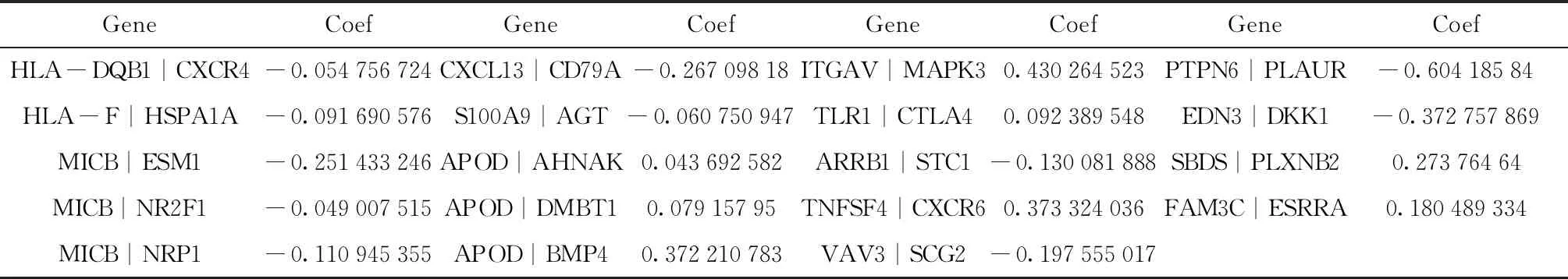
表4 免疫基因对及相应系数
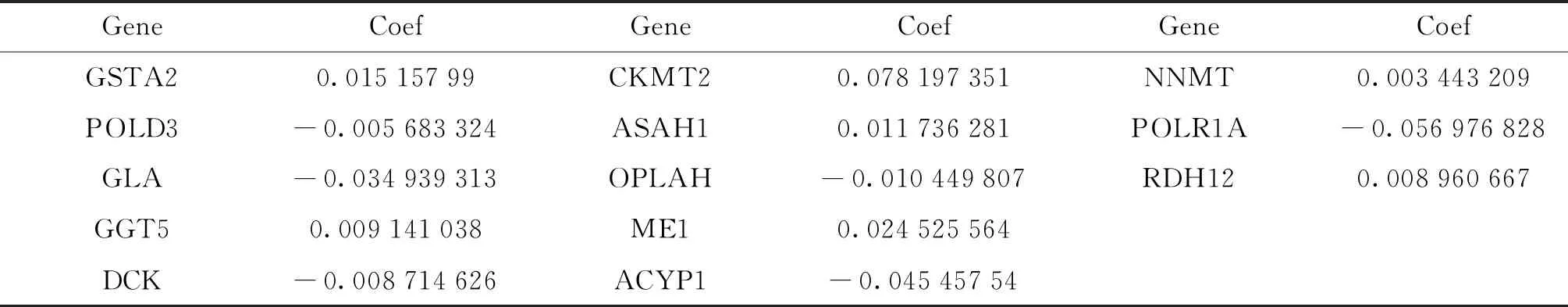
表5 代谢基因及相应系数
使用随时间变化的ROC曲线进行分析, 分别得到了1, 3, 5年的最佳截断值(cutoff),即特异性与灵敏度之和最大的点(见图1). 它们以假阳性率(1-特异性)为横轴, 真阳性率即灵敏度为纵轴. 特异性(Specificity)和灵敏度(Sensitivity)的公式[10]为:
(4)
(5)
其中TP为真阳性,Fp为假阳性,TN为真阴性,FN为假阴性.
按照风险值=∑(基因系数×基因表达量), 分别计算每个患者的风险值, 再让其与cutoff值进行比较, 将患者划分为高危组和低危组患者. 由图可以直观的看出, 免疫基因下1年的ROC曲线图最平缓, 最接近左上角的坐标轴, 这表明它有很高的诊断准确性, 同时对胃癌患者的预后研究具备较高的价值.
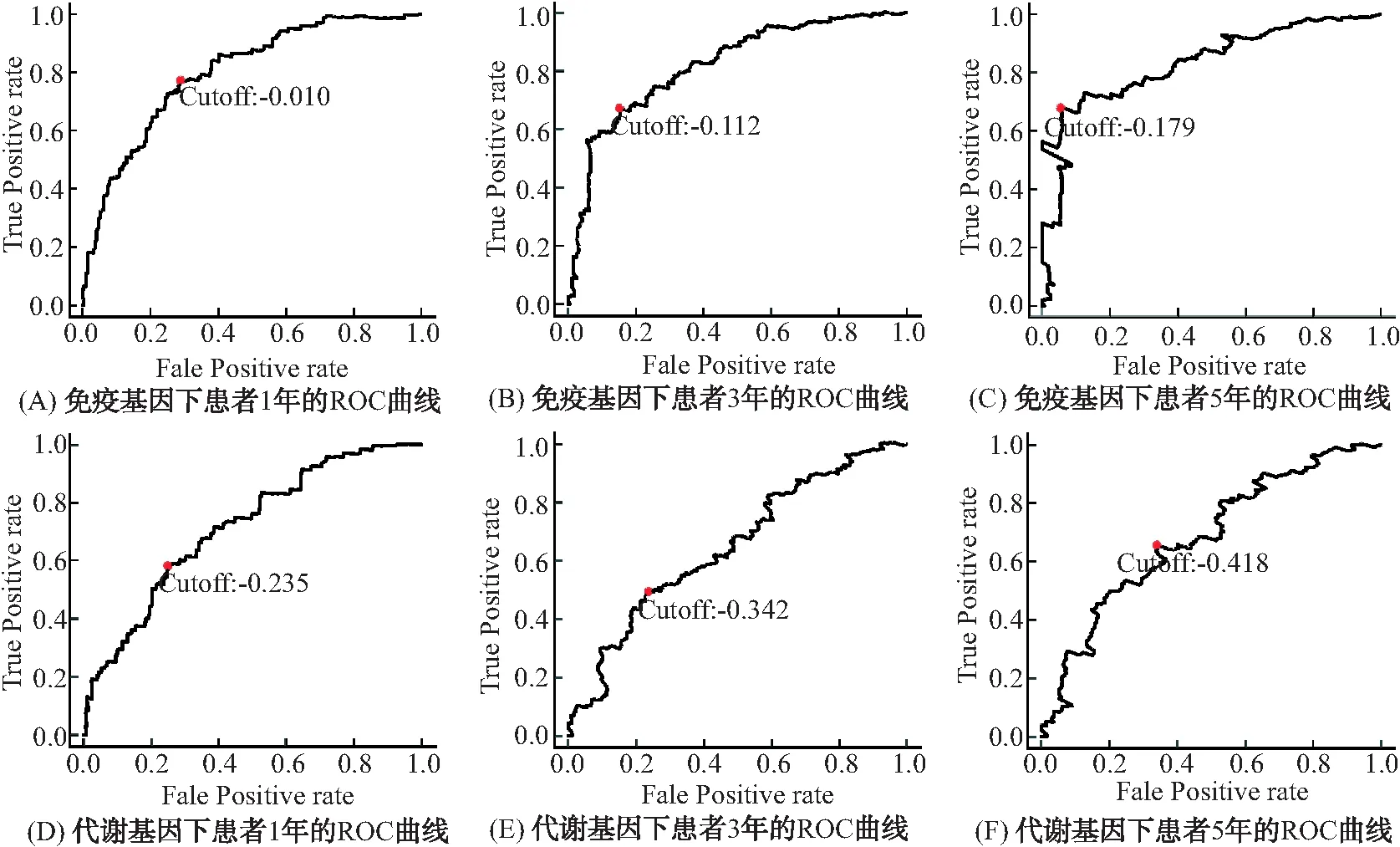
图1 免疫基因和代谢基因下患者在不同年份的ROC曲线
2.4.3 LASSO回归模型的准确性验证
计算出在免疫基因和代谢基因下患者的ROC曲线在不同时间下的AUC值(如表6). 用AUC值来验证模型的准确性, 其值越接近1表示准确性越高.发现在相同的年份里, 免疫基因的AUC值都比代谢基因的AUC值高, 且免疫基因的AUC值随着年份的增加而不断增加, 但是代谢基因的AUC值却随着年份的增加而减少, 说明利用免疫基因来构建LASSO回归模型具有较高的准确性.
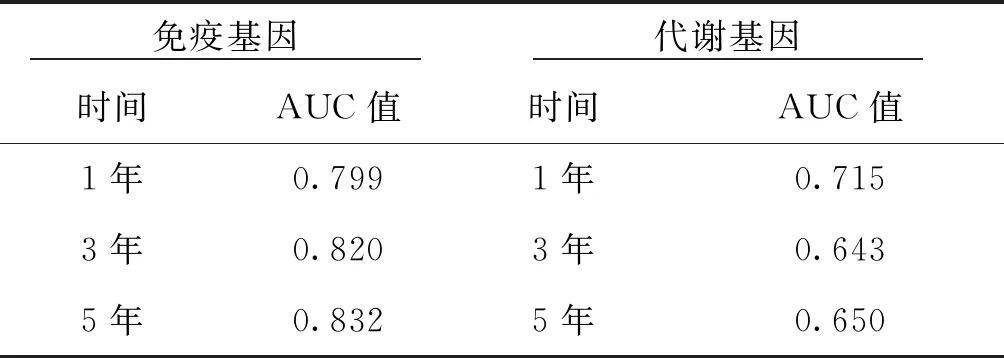
表6 免疫基因和代谢基因在不同时间下的AUC值
3 结果及分析
本文采用K-M法对已经分好的胃癌高低风险患者进行生存分析[11]. K-M法是1种非参数方法, 可以通过观察胃癌患者的生存时间来对患者的生存概率进行估计. 生存概率是指某段时间开始时存活的样本至该时间结束时仍然存活的可能性大小[12]. 生存概率可以表示为:
(6)
其中,S表示生存概率,U表示活过某段时间的人数,V表示该时段初期观察人数.
通过生存概率能够得到高低风险组随着时间的变化而变化的生存曲线. 在生存曲线上半部分,横轴为时间(年), 纵轴为生存率. 下半部分为在每个时间点上高低风险组患者存活的数目(见图2和图3).
由图可知,随着随访时间的增加, 两种基因的高低风险患者生存率都有所下降. 在免疫基因中, 高低风险组曲线之间存在的差异较大, 高危患者的生存率逐渐趋于0, 低危患者的生存率大概到达55%趋于稳定, 且随着时间的增加生存曲线比较平缓; 在代谢基因中, 高低风险组曲线之间存在交叉, 即差异较小, 生存率分别到达13%和35%就趋于稳定. 5年后, 免疫基因中高低风险患者存活的数目分别为1和6; 代谢基因中高低风险患者存活的数目分别为1和2.
免疫基因的生存曲线P值为0, 代谢基因的生存曲线P值为4.927e-06, 它们都小于0.05, 经过Log-rank检验, 它们的整体生存曲线间有明显的差异, 具有统计学意义.
同时我们还考察了患者的风险值分布和生存状况图(见图4). 把患者按照风险值从左到右由低到高的顺序进行排序, 并把这些患者作为横轴, G和I以风险值为纵轴. 随着胃癌患者往右增加, 它们的风险值也增加. 由于免疫基因的高低风险组之间差异较大, 风险值分布图呈现逐步上升趋势. 而代谢基因的风险值分布图上升较缓慢. H和J以生存时间为纵轴. 随着胃癌患者风险值的增加, 死亡的患者越来越多. 从免疫基因的生存状况分布图看出,表示死亡的红点由少变多较为明显, 能更好呈现出胃癌患者的生存时间.
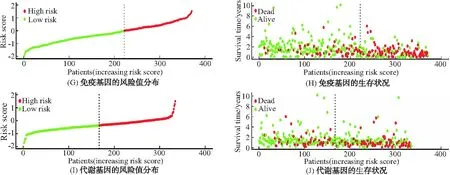
图4 免疫基因和代谢基因的风险分布图和生存状况图
总结出利用免疫基因能更好的分析胃癌患者的生存状态. 且随着时间的增加, 高低风险组之间的差异越来越明显. 患者风险值增加, 其风险评分增加, 死亡的病人也逐渐增多, 这与预期的结果一致.
4 结语
本研究通过单因素Cox分析筛选出与预后相关的免疫基因和代谢基因, 并利用LASSO回归构建预后风险评分模型, 找到cutoff值进行比较, 将胃癌患者划分为高低风险组, 进行生存分析的一系列图表对比研究, 结果显示利用免疫基因能更好的分析胃癌患者的生存状况, 即依据免疫基因在时间依赖的ROC曲线中找到了最佳截断点, 它能更好的将胃癌患者分为高危组和低危组, 且准确性较高. 由免疫基因计算得到的生存曲线显示, 高低风险组患者生存时间有明显的差异, 具有统计学意义. 综上所述, 免疫基因或许可成为预测胃癌患者生存时间的一个新指标, 为进一步研究胃癌的预后和临床辅助治疗提供一个参考价值.


