苹果SSR-PCR反应体系的建立与优化
刘 琳,孙晓帅,李 乾,范书臣,时鸿迪,马 钧,刘丹丹
(1云南大学农学院,昆明 650091;2西藏大学理学院,拉萨 850000;3云南省农业科学院园艺作物所,昆明 650091)
0 引言
苹果是蔷薇科苹果亚科苹果属(Malus)植物,为多年生落叶乔木,是国内主要的落叶果树之一,苹果产业在农业产业结构调整和农民增收中具有重要地位[1]。国内的主产区主要分布在四川、河北、河南、辽宁、山东、山西、陕西、甘肃和新疆等地[2],不同区域内苹果的品种和栽培生产方式差异较为明显,形成了有地域特色的品种优势,如新疆‘冰糖心富士’、山东‘红富士’、山西‘秦冠’、河北‘王林’等[3]。中国具有悠久的苹果栽培历史,是世界上苹果种植面积最大、总产量最高的国家[4],但良种苗木繁育体系不健全是影响苹果产量的重要原因之一[5]。中国已经成为世界上最大的苹果生产国和消费国[6],但自主培育具有地域特色的主栽品种较少,野生和地方特优种质资源的创新与利用日益突出,因此应充分挖掘利用国内资源,扩大优异基因来源,为选育具有自主知识产权的品种提供依据。
国内苹果种质资源丰富、品种类型多样,包括大量的野生种、半野生种及栽培品种,在果树栽培、生产和育种上具有较高的经济价值[7-9]。目前鉴定植物资源的方法主要有文献考证、野外考察、标本采集、品种的分类学鉴定以及DNA分子标记等方法[10]。而DNA分子标记是近年来使用最为广泛的一种遗传标记形式,也是继形态学标记、生化标记和细胞标记之后的一种较为理想的标记方法,同时也是一种鉴定种质资源较为有用的技术。它是以蛋白质、核酸分子的突变为基础,通过检测生物个体在基因或者基因型上所产生的变异来反映生物个体之间的差异[11]。DNA分子标记包含限制性片段长度多态性技术(RFLP)、序列标签位点技术(STS)、随机扩增多态性DNA技术(RAPD)、扩增片段长度多态性技术(AFLP)、简单序列重复技术(SSR)及单核苷酸多态性技术(SNP)等[12],其中SSR分子标记应用较为广泛。SSR分子标记即微卫星DNA,其序列串联重复,并且主要分布于植物基因组的编码区和非编码区中,在分类水平上具有很好的保守性,并且具有突变速率快、多态性高、重复性好、能区分纯合子和杂合子等特点,是一种理想的分子标记[13]。苹果SSR标记主要基于二核苷酸重复序列进行开发[14],SSR标记通过基因组扫描技术(GSA)及谱系基因分型技术可以很好地覆盖苹果基因组[15],前者主要用于绘制基因的抗性基因[16],后者主要用于等位基因的挖掘以及对性状关联的评估[17]。由于不同物种的最佳SSR-PCR反应体系不同,有必要对苹果属植物的SSR-PCR反应体系进行优化确定。
笔者以21种苹果属植物作为实验材料,采用L16正交实验对模板浓度、引物浓度、Mg2+浓度、dNTPs浓度以及Top Taq酶浓度等5个因素进行优化,并通过退火温度梯度实验对SSR-PCR扩增体系进行优化改良,旨在为利用SSR分子标记鉴定蔷薇科苹果属植物种质资源提供实验技术支撑。
1 材料与方法
1.1 材料
实验于2018—2019年在云南省云南大学农学院进行,所有实验均重复3次。实验所采用的21种苹果属材料均采自云南省昭通市水果推广站实验基地,详见表1。利用随机抽取的‘粉红女士’品种的DNA作为SSR-PCR反应优化体系模板。引物设计参照Liebhard等[18]的研究,由擎科生物工程股份有限公司合成,详见表2。PCR反应所用的dNTPs、Top Taq酶、DNAmarker,10×Top Taq buffer(Mg2+)以及6× DNA上样缓冲液购自云南金邦(普麦)生物科技有限公司(AP151-11)。
表1 21种苹果属植物
1.2 仪器设备
高速离心机(HC-2518),供试PCR仪(Eppendorf AG 22331),琼脂糖凝胶电泳仪(DYCP-31DN),聚丙烯酰胺凝胶电泳仪(MGV-202-33),切胶仪(天根生物科技有限公司),超微量分光光度计(tGem Spectrophotometer Plus)。

表2 引物序列
1.3 实验方法
1.3.1 DNA提取及质量检测
(1)DNA提取。方法参照Easy plant Genomic DNA kit(云科生物技术有限公司,code#E11101)。21种苹果属材料分别取0.1 g的嫩叶组织,液氮研磨后,参照DNA提取试剂盒说明书提取,提取的DNA用65℃预热的100 μL ddH2O溶解,-20℃保存。
(2)浓度检测。通过超微量分光光度计检测DNA浓度。1 μL ddH2O进行调零,检测1 μL DNA待测液的浓度及OD260/OD280值,取OD值于1.8~2.0范围内的DNA用于后续实验。
1.3.2 SSR-PCR反应体系正交设计实验 随机挑选‘粉红女士’作为优化体系的DNA模板,CH01h021作为引物,对表3中的引物浓度、Top Taq buffer(Mg2+)、dNTPs、Top Taq酶以及引物浓度等5个因素的4个水平进行正交实验,L16(45)16个组合体系正交表结果如表4。反应体系总体积为25µL,实验3次重复。
1.3.3 PCR扩增与检测
(1)PCR扩增程序。94℃预变性5 min,94℃变性30 s,49~58℃退火 30 s,72℃延伸 30 s,共 35个循环,72℃后延伸10 min,PCR产物4℃保存。取1µLPCR扩增产物加入0.2µL DNA上样缓冲液,分别用1.2%的琼脂糖凝胶电泳及8%的聚丙烯酰胺凝胶电泳进行检测。
(2)产物检测。根据琼脂糖凝胶电泳的条带清晰度以及条带的数量判断模板浓度、引物、Mg2+、dNTPs以及Top Taq酶浓度是否适合反应体系;根据聚丙烯酰胺凝胶电泳条带的清晰度来筛选引物。
1.3.4 最适SSR-PCR反应温度的筛选 参照何正文等[19]的实验方法,对聚丙烯酰胺凝胶电泳(PAGE)条带进行判断和筛选,其中特异性强、弥散度弱、条带清晰的条带视为正确扩增条带。根据扩增正确条带数判断SSR-PCR反应最适组合。
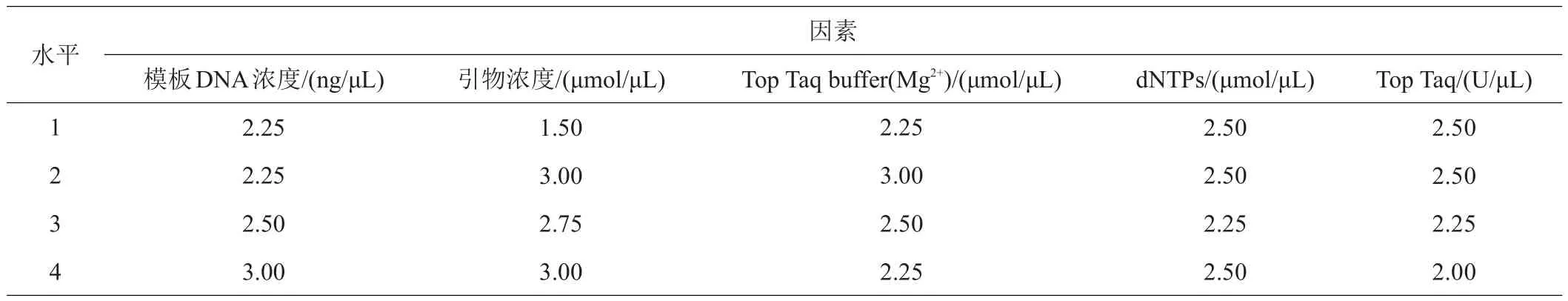
表3 SSR-PCR反应体系因素及水平

表4 苹果属植物SSR-PCR反应的正交实验设计[L16(45)]
运用最佳的SSR-PCR反应体系进行PCR实验,设置PCR反应程序不变,将退火温度设置49、52、55、58℃4个温度梯度进行实验(根据引物的退火温度设定),寻找最适退火温度。
2 结果与分析
2.1 蔷薇科苹果属品种DNA提取及浓度测定
对21个品种的DNA进行提取,琼脂糖凝胶电泳检测发现,所有样品的谱带清晰明亮,无明显杂质,DNA完整性较好(图1)。随后通过DNA浓度和纯度的测定,筛选出OD260/OD280值在1.8~2.0之间DNA样品(表5)。
2.2 苹果属植物SSR-PCR反应体系的优化
随机选取CH01h021作为引物(表2),根据表3中16个不同浓度梯度水平的组合进行PCR扩增,将反应产物进行聚丙烯酰胺凝胶电泳实验(PAGE)。由SSR-PCR正交实验结果可以看出,不同组合的扩增结果差异明显,组合6、11、14下扩增出的条带数量多且条带清晰明亮。经过多次重复实验发现组合11、14所扩增出的条带清晰度较高,实验结果较为稳定(图2)。经过多次实验证明,组合14可作为最优体系,即30~60 ng DNA模板、1.2µL正反向引物(5µmol/L)、0.5µL dNTPs(2.5 mmol/L)、0.5 µL Top Taq酶(2.5 U/µL)以及2.5 µL 10×Top Taq buffer(含Mg2+),用ddH2O补至25µL。
为进一步证明初步结论的准确性,对正交实验的各因素及水平进行分析(表6),其中K值代表在同一水平下所产生的扩增条带的平均值,R值能够反映某因素的极差,R值越大影响越显著。通过对模板DNA浓度、引物浓度、Mg2+浓度、dNTPs浓度、Top Taq酶浓度5个因素在4个水平内的综合分析发现,对结果影响从大到小依次为引物浓度、Top Taq buffer(Mg2+)、模板DNA浓度、Top Taq酶及dNTPs。经验证,上述实验中所获得的SSR-PCR反应体系为适合本实验的最适体系。

表5 21种苹果属植物品种的DNA浓度及OD值测定结果

图1 蔷薇科苹果属DNA质量检测电泳结果

图2 正交设计SSR-PCR反应体系扩增(聚丙烯酰胺凝胶电泳检测)结果
2.3 最适退火温度的筛选
引物的退火温度对PCR结果的影响较大,为减少误差,在以上反应体系的基础上对引物的退火温度进行了筛选。结果发现,当退火温度为52℃时,PCR扩增出的条带数目清晰(图3)。
2.4 最佳反应体系的稳定性检验
为了检测SSR-PCR反应的稳定性,随机选用另外一对引物CH03d11进行验证。以21种蔷薇科苹果属品种DNA为模板,利用优化后的SSR-PCR反应体系进行扩增,结果发现,21个品种的扩增结果条带清晰,无拖带并且大部分都具有2个或者3个等位基因,大小在100~200 bp左右(图4),说明该扩增体系比较稳定,可应用于蔷薇科苹果属植物SSR扩增反应。
3 结论与讨论
SSR是基于特异性引物进行PCR的分子标记技术[20],凡是影响PCR扩增效果的因素,如Mg2+、DNA聚合酶、引物、模板DNA、dNTPs等,均会影响到SSR-PCR的结果。不同物种的SSR-PCR分析报道虽多,但大多是采取单因素梯度实验,过程复杂且不能兼顾各因素之间的相互影响[21-22]。本试验采用正交设计实验能够分析不同因素之间的交互作用[23],同时也克服了单因素实验规模巨大、顾此失彼的问题[24],此种方法效率更高、稳定性更好,能够较为全面地找出各因素之间的规律[25]。因而本试验以此来获得最优反应体系,减少SSR技术的误差。

表6 SSR-PCR反应体系的正交实验直观分析

图3 SSR-PCR反应体系最佳退火温度

图4 SSR-PCR最佳反应体系检验结果
本试验研究前期参考Liebhard等[18]的研究,共选取了108对引物,通过琼脂糖凝胶电泳试验筛选出32对引物,本试验随机挑选了其中的2对引物进行后续的体系优化。研究发现,琼脂糖凝胶电泳的条带极易发生弥散拖尾现象,当模板浓度过低时产物容易弥散;模板浓度过高时引物与dNTPs过早耗尽,结果不稳定;引物浓度过高时会产生非特异性产物,极易形成引物二聚体[26-27]。PCR产物拖尾主要是由于退火温度过低,当退火温度较低时容易出现条带拖尾。通过正交设计实验发现,在SSR-PCR反应过程中,引物浓度的影响最为明显,但模板浓度、dNTPs、Mg2+以及退火温度也起到了较为关键的作用,需根据实验的具体要求灵活安排以提高结果的准确性,将人为因素降低到最小。在整个实验过程中,确定每个引物的最适退火温度实验过程比较繁琐,本研究只列举了引物CH03d11的退火温度。当采用多个引物同时扩增时,反应程序可采用降落PCR方法[28],降落PCR能够有效降低各组分浓度及退火温度引起的非特异性产物产生,大大缩短实验时间。通过本实验,最终确定适用于蔷薇科苹果属植物SSR-PCR的最适反应体系为:30~60 ng DNA模板,1.2µL正反向引物(5µmol/L),0.5µL dNTPs(2.5 mmol/L),0.5 µL Top Taq酶(2.5 U/µL)以及2.5µL10×TopTaqbuffer(含Mg2+),用ddH2O补至25µL。该体系经过验证可用于后续蔷薇科苹果属植物变异遗传稳定及分子身份证的构建研究。

