核内RNA对真核细胞基因组三维高级结构的影响
金加,沈文龙,李平,张彦,陈昭烈,赵志虎
军事医学研究院 生物工程研究所,北京 100071
细胞核内染色质在三维空间上的精确折叠包装和选择性表达决定了细胞的命运,也为各种染色体活动(如转录和复制[1-3])的正常进行提供了保障。一维线性染色质通过螺旋缠绕和高度折叠最终形成更高级的拓扑相关结构域(topologically associating domains,TAD)和A/B区室。A区室主要富集H3K4ac、H3K4me1等活跃的组蛋白修饰标志,存在高密度的基因分布和复制起始位点;相反B区室富含H3K27me3等沉默的组蛋白修饰标志,分布的基因较少,另外还有晚期复制区域[2-4]。然而这些结构并不是一成不变的,在分化或疾病发生时也可能会发生动态变化[5-6]。
Isoda等研究发现核内RNA参与了区室和环(loop)的形成[7-8],并且Erdel发现核内RNA可以通过“液相分离”[9]来创建局部细胞核环境,从而促进细胞核内无膜细胞器的组装,进而形成更高度有序的核结构,比如核糖体RNA介导的核结构的形成[10-11]。据报道,核内RNA不仅可以参与形成完整的区室[12-15],还参与形成其他核结构域,如剪接因子富集斑点、核仁等[16-19]。如果将核内RNA去除,通过电子显微镜进行显色观察发现高度有序的核结构发生混乱[14,20],所以核内RNA在核结构的形成中扮演重要的角色。
为了探究核内RNA对染色体三维高级结构的影响,我们用全基因组染色质构象捕获(highthroughput/resolution chromosome conformation capture,Hi-C)技术分析了野生型GM12878细胞和经RNase处理的GM12878细胞染色质的相互作用,以及A/B区室和拓扑相关结构域的变化。
1 材料与方法
1.1 材料
女性B淋巴细胞GM12878由本室保存。GM12878细胞存在特殊的X染色体失活现象,是研究核内RNA对染色体三维结构影响的很好的实验模型。GM12878细胞已经参与了许多组学研究,可为后续实验提供参考且便于培养。
RPMI培养基(Hyclone公司);多聚甲醛(国药集团化学试剂有限公司);RNaseA(50 U/mL)、RNaseT1(20 000 U/mL)、Pro Flex PCR System(Life Technologies公司);RNaseH(5000 U/mL)、HindⅢ-HF(100 U/μL)(NEB公司);T4DNA连接酶(5 Weiss U/μL)(Thermo Scientific公司);超声破碎仪(VCX-750 SONICS)。
1.2 GM12878细胞的Hi-C实验
采用Hi-C技术,分别对野生型GM12878细胞和经RNase处理的GM12878细胞进行实验。首先将约6×107GM12878细胞于4℃、500 r/min离心5 min,用2 mL 1×PBS洗涤1次,用4 mL 1×PBS重悬细胞沉淀,将细胞混匀后移入50 mL离心管,在其中加入40 μL 10%甲醛溶液,使其终浓度为1%,在摇床上交联10 min,加入终浓度为0.125 mol/L的甘氨酸,混匀后在摇床上终止交联5 min,4℃、500 r/min离心5 min,弃上清,用裂解液重悬细胞沉淀后在冰上静置5 min,再将裂解液移入Dounce管,轻轻上下转动管内的磨砂玻璃棒20次,然后将野生GM12878细胞Dounce管中的裂解液直接用5 μLHindⅢ酶切,RNase处理组Dounce管中的裂解液在5 μLHindⅢ酶切的同时加入 5.2 μL RNaseH、13 μL RNaseA/RNaseT1,4℃、500 r/min离心5 min,弃上清,用2 μL KLenow、2.4 μL 10mmol/L dA/T/GTP、2.4 μL 5 mmol/L生物素标记的dCTP、5 μL缓冲液2和39 μL ddH2O补平粘性末端,吹打混匀,在旋转混匀仪上于23℃混匀4 h,补平体系,500 r/min离心3 min,弃上清,加入215 μL ddH2O、25 μL T4DNA连接酶缓冲液洗涤1次,用10 μL T4DNA连接酶于16℃连接8 h,然后用蛋白酶K解交联,超声打断后,进行建库二代测序。
1.3 数据处理与分析
首先将得到的Hi-C数据进行质控,分别进行文库过滤统计、文库比对数据统计和文库有效数据过滤统计,在此基础上进一步处理实验数据,分别绘制全基因组相互作用热图、全基因组相互作用衰减指数(interaction decay exponents,IDE)比较分析、差异热图比较分析、统计A、B区室差异并采用GO注释进行功能分析。此外,对染色体之间的相互作用(秩和检验P<0.05)和TAD边界强度进行比较分析(Vilcox检验P<0.05)。
2 结果
2.1 Hi-C数据的质量控制
在文库过滤统计中(图1A),野生型GM12878末端完全配对片段占比97.92%,经RNase处理的GM12878末端完全配对片段占比98.43%,2组文库的完全配对片段占比较高。文库比对数据统计(图1B),与参考文库相比,野生型GM12878文库惟一配对片段占比74.65%,经RNase处理的GM12878文库惟一配对片段占比73.52%,没有配对的其他类型片段占比较低,2组文库样本质量较好。2组文库有效数据过滤统计(图1C),野生型GM12878文库相互作用片段占比95.45%,其中有效相互作用片段占比82.09%,在RNase处理的GM12878文库中,相互作用占比95.09%,其中有效相互作用74.1%,表明Hi-C实验质量较高。
2.2 核内RNA对染色体相互作用的影响
野生型GM12878染色体内相互作用热图(图2A)和RNase处理后的GM12878染色体相互作用热图(图2B)中的横坐标和纵坐标代表同一染色体的不同碱基位置,图中的红点代表了染色体两位置之间存在较强的相互作用,红色越强表示相互作用的强度越大。野生型GM12878和RNase处理的GM12878染色体内部的相互作用呈现出一样的相互作用状态,都是区室内部的相互作用大于区室之间的相互作用。
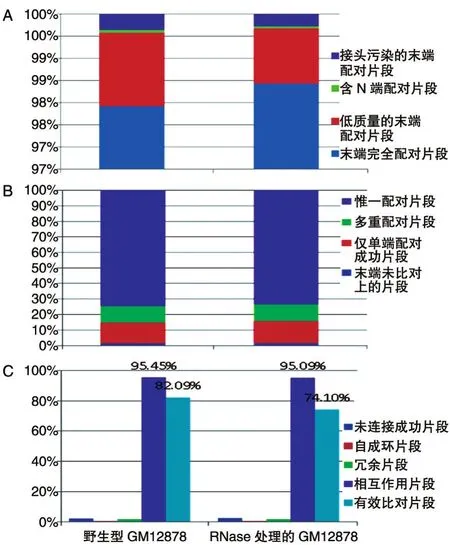
图1 Hi-C数据的质量控制
为了比较野生型GM12878和RNase处理的GM12878染色体内部的相互作用差异,将野生型GM12878染色体内相互作用减扣RNase处理的GM12878染色体内相互作用。图3横纵坐标分别代表同一染色体上的不同位置,图内红点代表野生型GM12878相互作用更强,蓝点代表RNase处理的GM12878相互作用更强。相比于近距离的染色质内相互作用,染色体内远距离的相互作用有更多的改变。这也表明核内RNA在远距离的相互作用上起到了更重要的作用。
我们绘制了全基因组IDE曲线(图4),随着基因距离的不断增大,野生型GM12878和RNase处理的GM12878细胞的相互作用频率由10-6下降到10-8,其中RNase处理组在基因距离108碱基对处的相互作用频率略高于野生型GM12878,说明相比于野生型GM12878细胞,经RNase处理后的GM12878细胞的染色体远距离相互作用有所增强,染色体的结构变得松散。
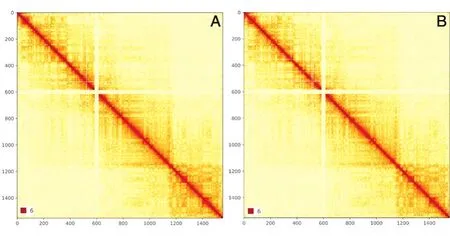
图2 X染色质相互作用热图

图3 染色质内相互作用的减扣
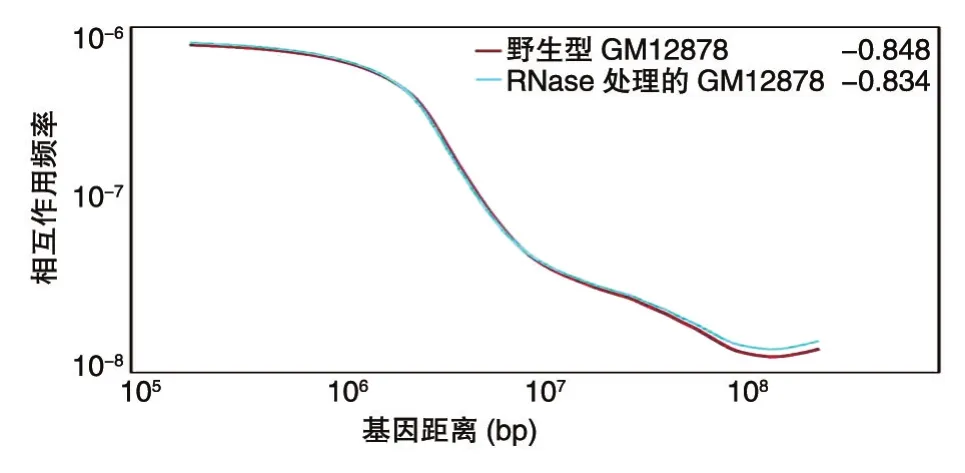
图4 染色质相互作用衰减指数
除此之外,我们还比较了2组细胞的染色体之间的相互作用(图5),RNase处理的GM12878细胞染色体间相互作用分数高于野生型GM12878细胞(秩和检验P<0.05)。这也说明经过RNase处理的GM12878细胞的染色质结构变得松散。
2.3 核内RNA对A/B区室的影响
为了进一步分析核内RNA对区室的影响,我们统计了2组GM1287细胞在A/B区室上的变化。所有染色体A/B区室转变比例饼图(图6A)中,A区室转变为B区室占比0.51%,B区室转变为A区室占比0.62%,未发生变化的A区室占比48.38%,未发生改变的B区室占比50.48%。经比较发现,B区室转变为A区室所占比例高于A区室转变为B区室。将发生转变区室上的基因进行GO注释功能分析,发现在转变区块中,A区室转变成B区室上的基因在功能上主要和脱氢酶相关,B区室转变成A区室上的基因在功能上主要和细胞外基质结构成分相关。在X染色体A/B区室转变比例饼图(图6B)中,A区室转变为B区室占比1.45%,B区室转变为A区室占比1.45%,未发生变化的A区室占比47.98%,未发生改变的B区室占比49.12%,所以相比于所有染色体的变化,X染色体上发生变化的A/B区室的比例更高。由于GM12878是女性淋巴细胞,含有2条X染色体,其中一条由于核内非编码RNA Xist的紧密缠绕而处于压缩状态,我们认为RNase的处理导致紧密的X染色体变得松散,所以相比于所有染色体A/B区室的变化,X染色体的A/B区室变化更大。对编码Xist基因的74M左右区域进行染色体相互作用分析(图7),野生型GM12878细胞Xist基因位置的相互作用减扣RNase处理的GM12878细胞Xist位置的相互作用显示蓝色增多,说明相比于野生型GM12878细胞,RNase处理后GM12878细胞的相互作用更弱。
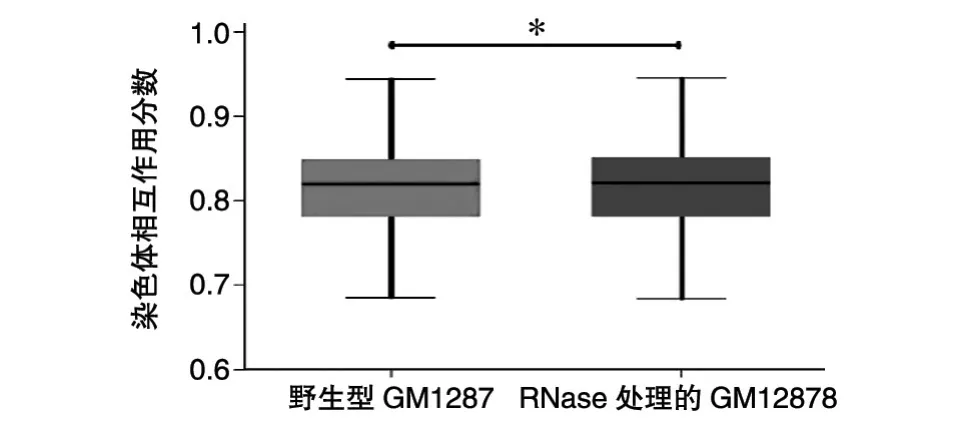
图5 染色质之间的相互作用比较(*P<0.05)

图6 A/B区室转变比例
2.4 核内RNA对TAD边界强度的影响
为了观察RNase处理后TAD边界强弱的变化,将野生型GM12878和RNase处理的GM12878细胞的TAD边界的强弱进行比较(图8),发现相比于野生型GM12878细胞,经RNase处理的GM12878细胞的TAD边界强度较小(Vilcox校验P<0.05)。这是因为由于核内RNA参与了loop形成,经RNase处理,导致环结构改变,进而影响TAD结构变得松散,从而减弱了TAD边界强度。
3 结论
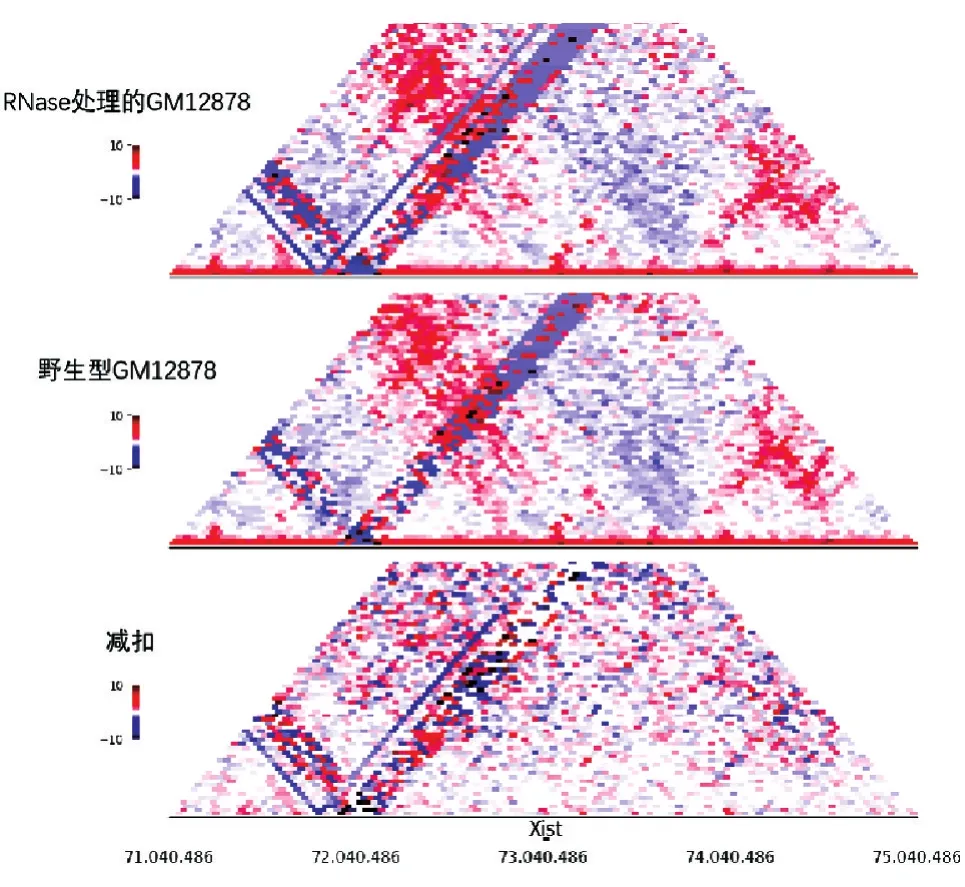
图7 X染色体74M左右区域相互作用热图

图8 TAD边界强弱变化
我们证实了核内RNA对真核细胞染色体三维高级结构是有影响的,RNA的去除导致真核细胞染色体三维高级结构在整体上变得松散,A/B区室发生改变,目前已经发现RNA可以通过结合RNA结合蛋白对染色质进行修饰,进而调控染色质环的形成,核内RNA还参与了TAD边界的形成和建立,而RNase处理导致了环结构的破坏,TAD边界强度减弱。基因组染色体三维结构的稳定性是表观遗传的关键,受到许多物质的调控,如核内RNA、蛋白质、激素等。如果基因组的三维结构或者调控出现了偏差,有可能导致疾病。我们认为在未来会研发出一些更好的分子生物实验,特别是研究核内RNA对染色质三维高级结构和染色质修饰物影响的实验,这对于揭示基因组三维高级结构形成的潜在原理至关重要。

