分离白细胞法、低渗红细胞法、全血直提法提取的冻存血RNA质量及其反转录后基因扩增效果
刘欣,彭贺含,唐晓慧,杨四梅,赵商岐,郑佳,周晓涛
1 新疆医科大学研究生院,新疆乌鲁木齐830011;2新疆昌吉职业技术学院;3 新疆中医学院;4新疆军区总医院;5 新疆医科大学基础医学院
RNA是基因表达过程中非常重要的分子,从细胞或组织中分离出RNA能更好的进行基因功能研究,其分离提取方法也是分子生物学最常用的技术。在临床工作中,外周血采集简单,对患者创伤小,是理想的RNA来源。目前常用的RNA提取方法有试剂盒法、SDS法、TRIzol法[1-3]等,这些方法在外周血RNA提取中得到了广泛应用。但在实际工作中,患者标本的采集情况非常复杂,许多血液标本在采集后需立即冻存于-80 ℃或-20 ℃环境中,待完全收集齐后才开展实验,一些年代较早的临床标本基本都是以这种方式保存的。由于长期冻存对血液标本成分的影响以及广泛存在的核糖核酸酶(RNase),使得从冻存血中提取数量和质量都能满足研究的RNA变得比较困难。有学者[4]用TRIzol法比较新鲜的马血和冻存马血提取RNA的差异,结果显示新鲜血液中提取的RNA在浓度和纯度均好于冻存血。基于科研的实际需求,特别是一些经过多年收集积累的冻存血标本,急需寻找一种有效的冻存血RNA提取方法。有研究[5]利用分离白细胞法从系统性红斑狼疮患者在-80 ℃中冻存1年的全血中提取了数量和质量都能满足要求的RNA。还有研究[6]利用低渗红细胞法和全血直提法也从-80 ℃的冻存血中提取到了RNA。2019年6月1日~2020年1月20日,本研究观察了分离白细胞法、低渗红细胞法、全血直提法提取的冻存血RNA质量及其反转录后基因扩增效果,现将结果报告如下。
1 材料与方法
1.1 RNA提取方法及分组 正常全血标本12例份,每例份血量约2.5 mL左右,于-80 ℃条件下冻存半年后,又置于-20 ℃条件下冻存半年。将冻存血标本12例份随机分为3组,分别采用分离白细胞法、低渗红细胞法、全血直提法提取RNA,提取出的RNA溶液分别记为A、B、C组。
1.1.1 分离白细胞法提取RNA 血液标本从-20 ℃冰箱取出后放置于室温环境,待标本血浆层融化后向其中加入2.5 mL Hanks液,用加样枪慢慢吹打混匀直至细胞层完全融化;取15 mL离心管,先加入5 mL淋巴细胞分离液待用,再将5 mL血液标本沿离心管壁缓慢地加到淋巴细胞分离液界面,20 ℃条件下2 000 r/min离心20 min;离心后离心管的界面分为3层,弃上层和中间层,向底层沉淀再加入5 mL的Hanks液,轻轻吹打混匀,20 ℃条件下2 000 r/min再离心10 min,弃上清液,留底层。将底层沉淀物转入新的EP管中,加入1 mL TRIzol充分混匀,室温静置5 min,加入0.2 mL氯仿,用力振荡15 s,静置2 min,4 ℃条件下12 000 r/min离心15 min。离心后样品分3层,RNA主要在上层水相中。取上清加入0.5 mL异丙醇,将管中液体轻轻混匀,室温静置10 min,4 ℃条件下12 000 r/min离心10 min,弃上清,加入1 mL 75%乙醇,轻轻洗涤沉淀,4 ℃条件下7 500 r/min离心5 min,弃上清,晾干,加入20 μL的DEPC水溶解,即为提取的RNA溶液,记为A组。
1.1.2 低渗红细胞法提取RNA 血液标本从-20 ℃冰箱取出后,直接与DEPC水按照1∶1比例进行稀释,静置5 min,然后用加样枪慢慢吹打混匀,直到标本完全融化,转入新的EP管中,4 ℃条件下12 000 r/min离心10 min,弃上清,留底物。底物中加入1 mL Trizol充分混匀,室温静置5 min,加入0.2 mL氯仿,用力振荡15 s,静置2 min,4 ℃条件下12 000 r/min离心15 min。离心后样品分3层,RNA主要在上层水相中。取上清加入0.5 mL异丙醇,将管中液体轻轻混匀,室温静置10 min,4 ℃条件下12 000 r/min离心10 min,弃上清,加入1 mL 75%乙醇,轻轻洗涤沉淀,4 ℃条件下7 500 r/min离心5 min,弃上清,晾干,加入20 μL的DEPC水溶解,即为提取的RNA溶液,记为B组。
1.1.3 全血直提法提取RNA 血液标本从-20 ℃冰箱取出后,直接加入1 mL TRIzol于冻存管中,出现白色絮状物后,静置10 min,用加样器吹打混匀,此时底部的血细胞会聚集成胶黏的团块状,黏附在加样枪上,较难混匀。先将加样枪深入底部沿着一个方向转10-15下,保证冻存管底部没有细胞沉淀,然后加样枪再来回上下运动2~3 min,充分混匀后,将细胞裂解液移至EP管中,4 ℃条件下16 000 r/min离心1 min,吸出管底絮状沉淀,直接加入0.2 mL氯仿,用力振荡15 s,静置2 min,4 ℃条件下12 000 r/min离心15 min。离心后样品分3层,RNA主要在上层水相中。取上清加入0.5 mL异丙醇,将管中液体轻轻混匀,室温静置10 min,4 ℃条件下12 000 r/min离心10 min,弃上清,加入1 mL 75%乙醇,轻轻洗涤沉淀,4 ℃条件下7 500 r/min离心5 min,弃上清,晾干,加入20 μL的DEPC水溶解,即为提取的RNA溶液,记为C组。
1.2 各组RNA浓度、RNA纯度观察 打开NANODROP 1000分光光度计取样臂,先用2 μL DEPC水校准归零,再吸取2 μL各组RNA溶液放置在测量基座的表面接收光纤的末端上,检测各组RNA浓度、RNA在230、260、280 nm处的吸光度值,计算OD(A260/A280)值、OD(A260/A230)值,以OD(A260/A280)值、OD(A260/A230)值表示RNA纯度。
1.3 各组RNA完整性观察 称取0.4 g琼脂糖加入烧瓶,加入1×TBE溶液20 mL摇匀,微波炉煮沸溶解30 s、3次,待琼脂糖溶胶稍冷却,加入3滴溴化乙锭(EB),轻轻混匀,配成2%琼脂糖凝胶。取合适孔径点样梳垂直放入胶板一端,再将凝胶轻轻倒入,防止产生气泡,待凝胶凝固后,取出点样梳。电泳槽中加入1×TBE电泳缓冲液,胶板放入电泳槽中,点样孔靠近负极。取4 μL各组RNA溶液与1 μL溴酚蓝Loading Buffer在塑料薄膜上混匀,依次加入上样孔,电泳设定电压110 V,电流200 mA,时间40 min,当指示剂跑过胶板的2/3时,用凝胶成像系统采集图像,观察电泳条带情况。
1.4 各组RNA反转录后基因扩增效果观察 ①采用PCR法。取各组RNA溶液1 μg,加入1 μL Random、1 μL 10 nmdNTP和DEPC ddH2O配成10 μL体系,65 ℃孵育5 min,置于冰上孵育1 min,再加入2 μL 10×RT buffer、4 μL 25 μm MgCl2、2 μL 0.1 MDTT、0.5 μL RNase Out、0.5 μL SSⅢRT、1 μL ddH2O,配成20 μL反应体系,利用PCR仪反转录得到cDNA。反转录条件:25 ℃ 10 min,50 ℃ 50 min,85 ℃ 5 min,4 ℃ 终止。将20 μL cDNA产物用ddH2O稀释至100 μL分装保存待用。在25 μL PCR反应体系里,分别加入cDNA 2 μL,上下引物(10 μm/L)各1 μL,2×Taq PCR Master Mix 12.5 μL,再加入RNase-free water至25 μL,以94 ℃、4 min预变性;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 30 s,扩增35个循环,72 ℃延伸8 min,4 ℃ 终止。反应结束后,取5 μL反应产物进行琼脂糖凝胶电泳检测,观察RNA反转录后的扩增产物电泳条带情况。②采用qPCR法。取各组RNA溶液,反转录成cDNA后,进行qPCR反应。qPCR反应体系20 μL:cDNA 2 μL,上下引物各0.8 μL,2×SYBR Green PCR Master Mix 10 μL,RNase-free water 至20 μL。qPCR反应条件:95 ℃ 2 min,95 ℃ 2 s,60 ℃ 10 s,扩增40个循环,最后在65~95 ℃条件下制备溶解曲线,计算循环阈值(Ct值)。Ct值是每个反应管内的荧光信号到达设定阈值时所经历的循环数,Ct值越小,表示RNA反转录后基因扩增效果越好。

2 结果
2.1 各组RNA浓度、RNA纯度比较 各组RNA浓度、OD(A260/A280)值、OD(A260/A230)值比较见表1。由表1可知,A组RNA浓度、OD(A260/A280)值、OD(A260/A230)值均高于B、C组(P均<0.05),且B组和C组OD(A260/A230)值均<2,说明B组和C组RNA提取过程中受到盐溶液污染或者有裂解液残留,纯度不好。

表1 各组总RNA浓度、OD(A260/A280)值、OD(A260/A230)值比较
2.2 各组RNA完整性比较 各组RNA琼脂糖凝胶电泳结果见图1。由图1可知,A组没有出现任何条带,B组条带严重弥散,C组有条带存在,说明C组RNA完整性较好。

图1 各组RNA琼脂糖凝胶电泳结果
2.3 各组RNA反转录后基因扩增效果比较 ①PCR实验结果显示,A、B、C组RNA均能在反转录成cDNA后,用于基因扩增,其中C组得到的扩增产物条带最为明亮,说明C组RNA反转录后基因扩增效果较好,见图2。②qPCR实验结果显示,A、B、C组Ct值分别为28.27±1.01、27.49±0.22、24.20±0.25,其中C组Ct值与A、B组相比,P均<0.05,说明C组RNA反转录后基因扩增效果较好。
3 讨论
本研究通过检索文献[7-9],找到了三种从冻存血中提取RNA的方法:分离白细胞法、低渗红细胞法及全血直提法。其中,分离白细胞法通过淋巴细胞分离液对室温融化的冻存血进行分离,在底层物质中加入TRIzol后进行RNA的提取。低渗红细胞法是在血液标本冻存的状态下,直接加入DEPC水,利用DEPC水去除RNA酶,同时通过低渗的方法将红细胞全部破坏,最后利用TRIzol进行抽提。全血直提法则是将TRIzol直接加入冻存血中进行RNA抽提。这些方法的共同之处,都是为了避免RNA酶的干扰。
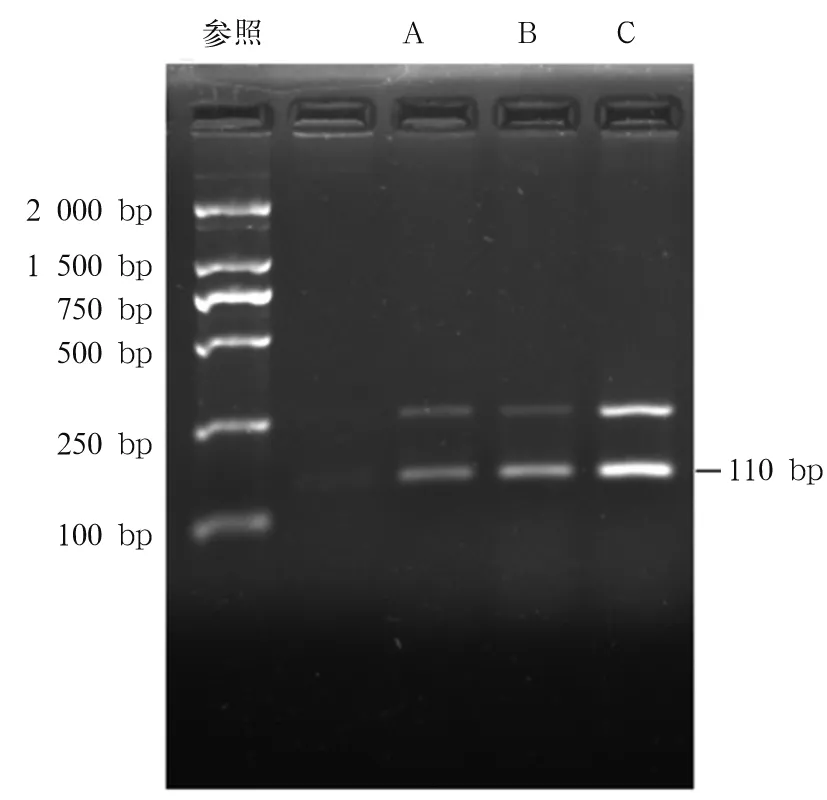
图2 各组RNA反转录后PCR扩增产物琼脂糖凝胶电泳结果
A260是RNA的吸收峰,A280是蛋白质的吸收峰,A230是其他碳源物质的吸收峰,因此可以根据OD(A260/A280)值和OD(A260/A230)值来判断RNA纯度。若OD(A260/A280)值在1.8-2.1之间,则提取的RNA纯度较理想;若OD(A260/A280)值<1.8,则存在蛋白质污染;若OD(A260/A280)值>2.2,说明RNA已降解成单核苷酸。OD(A260/A230)值一般要大于2.0,比值较小,说明有裂解液的残留或盐溶液的污染,提取的RNA纯度不好。在对RNA浓度检测的结果中,可以看出A组提取的RNA纯度跟浓度都是最好的,其OD(A260/A280)值为1.89±0.18,OD(A260/A230)值为2.94±2.42,浓度为(424.13±102.24)ng/μL。B组提取的RNA中,OD(A260/A230)值为0.79±0.23,推测有裂解液残留或盐溶液污染;C组提取的RNA中,OD(A260/A280)值为1.77±0.07,有蛋白质污染。
但在对RNA完整性检测的琼脂糖电泳中,可以看到A组无条带,可能是由于提取的RNA已经降解,呈片段化,碱基暴露,所以虽然可以检测出RNA浓度,但降解后的片段太小,EB染色无法显示条带。B组条带严重弥散,可能存在RNA降解、DNA污染或外源性RNase污染。C组的结果相对较好,但也存在RNA降解。
为了进一步验证RNA反转录后基因扩增效果,以等量RNA反转录得到的cDNA为模板,利用PCR扩增β-actin,三种方法均在110 bp处出现了特异性条带,但C组的条带更为明亮,说明C组RNA反转录后基因扩增效果较好。在qPCR实验中,Ct值又被称为循环阈值,其与模板起始拷贝数存在负相关的线性关系,即起始拷贝数越多,Ct值越低。C组的Ct值明显低于其他两种方法,进一步也说明其RNA反转录后基因扩增效果较好。
综上所述,本研究通过分离白细胞法、低渗红细胞法、全血直提法三种方法对冻存血RNA进行提取,检测3种方法所得的RNA浓度、RNA纯度、RNA完整性,后将各组RNA反转录制备cDNA进行PCR和qPCR扩增β-actin内参基因,观察三种方法提取的RNA反转录后基因扩增效果,结果显示全血直提法在冻存血中提取的RNA完整性高,其反转录后基因扩增效果较好。但是,全血直提法在标本的前处理过程中较为麻烦,冻存血中直接加入TRIzol后,吹打混匀过程中血细胞聚集成胶黏的团块状,黏附在容器壁上,需要用枪头裹挟着胶黏的团块在容器壁上反复旋转保证血液全部溶解,但容器壁上仍旧会有少量血细胞残留,影响RNA的提出量。

