引入养殖俄罗斯红参线粒体全序列结构与分析*
赵 强 杨 爽 刘心田 孙国华 冯艳微 王卫军 杨建敏
(1. 烟台市海洋经济研究院 烟台 264003;2. 鲁东大学农学院 烟台 264025;3. 威海市渔业技术推广站 威海 264200;4. 上海海洋大学水产与生命学院 上海 201306)
近年来,随着市场需求的增加,刺参养殖业发展迅速,但养殖效率不高、良种匮乏、生长性状稳定性较差等一直影响刺参产业的发展(曲洪霞等, 2016),因此,不同品种和品系的刺参逐步进入养殖和消费市场。俄罗斯红参(Apostichopus japonicus)简称“红参”,原产于俄罗斯远东近海,是本地刺参的地理远缘种,由于环境及摄食等因素体表呈红棕色。海参崴附近纬度高,海水温度较低,食物较少,俄罗斯红参生长缓慢,代谢及生命力较为旺盛,且体积略小,被认为其体内可以积蓄更多营养(关崇新等, 2015)。目前,俄罗斯红参已引入我国养殖。
线粒体基因组(mtDNA)为闭合的环状双链结构,大部分动物的mtDNA 由37 个基因与1 段长度可变的控制区组成,基因排列较为保守,结构相对紧凑。mtDNA 遗传方式为母性遗传方式,不存在双亲遗传的遗传差异,很少发生重组,无组织特异性,易于取材与测序分析(康立森, 2018)。相比核基因组,mtDNA的分子量相对较小,仅占细胞的1%,易于分离纯化。具有选择压力更小、结构相对稳定、编码速率更快等特点(赵娜娜, 2013)。线粒体DNA 包含更全面的信息,利用其进行系统进化分析的可信度更高(Cui et al,2009)。目前,针对水产动物mtDNA 已有大量研究,但海参mtDNA 的研究较少。俄罗斯红参与普通养殖刺参在生长及营养含量方面稍有差异,为了解俄罗斯红参在遗传上的特点及其与其他海参间的差异,本研究对俄罗斯红参线粒体全基因组进行测序,分析其线粒体基因组的构成及其系统发育关系,为海参线粒体基因组的研究及后续俄罗斯红参的种质创新应用提供遗传数据基础。
1 材料与方法
1.1 样品采集
实验所用俄罗斯红参取自山东华春渔业有限公司,2010 年引入,养殖于黄河三角洲滩涂区刺参池塘,持续进行生长性状的养殖选育。2017 年11 月取样,数量为20 个,参龄约为1 年半,平均体质量为(48.96±7.56) g。清洗、解剖并取肌肉部分,将所取组织送至上海天昊生物公司进行测序。
1.2 实验方法
1.2.1 基因组DNA 提取及质量检测 本实验取约30 mg 肌肉组织,按常规酚–氯仿法获得俄罗斯红参总DNA。使用NanoDrop 检测样品的浓度和纯度;琼脂糖凝胶电泳检测样品的完整度。
1.2.2 随机文库构建及PCR扩增DNA文库 采用转座酶建库试剂盒构建随机测序文库。利用转座酶随机打断DNA,同时在片段两端加接头,并使用高保真聚合酶扩增原始文库,利用PCR(扩增循环数控制在12~15之间)在文库两端引入特异性标签(Index)和测序接头。
1.2.3 文库片段筛选及质量检测 使用Agencourt SPRIselect 核酸片段筛选试剂盒纯化放大的文库,同时筛选片段的大小。采用两步法筛选(Double size selection),先用SPRI 磁珠去掉目标区域左侧小片段(Left-side sizes election),再除去位于目标片段区域右侧的大片段(Right-side size selection),最终筛选片段峰值在300 bp 的测序文库。使用Qubit 准确定量文库浓度,均匀混合样品,确保每个样本的数据量合适均衡。并用Agilent 2100 Bioanalyzer 确定文库片段大小分布,评估是否适合上机。
1.2.4 混样并进行上机测序 将质检合格的样品稀释,以等摩尔数对多个样品混样并上机。使用Illumina Hiseq 平台,以2×150 原始双端测序策略来测序文库。
1.3 数据处理
所用序列经串联后,使用BioEdit 7.0.9(Hall, 1999)软件中的ClustalW 进行序列比对,利用软件Gblocks 0.91b (Castresana 2000)去除不可信区间。利用Mega 4.0 软件统计碱基组成、密码子使用等。使用tRNAscanSE 在线网站(http://lowelab.ucsc.edu/tRNA scan-SE/)预测tRNA 的二级结构。
1.4 系统发生分析
本研究使用俄罗斯红参、半索动物门及棘皮动物门共28 个物种的线粒体序列构建系统发生树,线粒体序列为13 个蛋白编码基因串联的氨基酸序列与核苷酸序列,线粒体序列均从NCBI 获得(表1)。以半索动物门的囊舌虫(Saccoglossus kowalevskii)与肉花柱头虫(Balanoglossus carnosus)为外类群,将处理后的核苷酸序列使用jModelTest2.1.5 (2014)软件AIC 标准选择出最佳替代模型为GTR+F+R5,氨基酸序列使用ProtTest 2.4 (Abascal et al, 2005)软件AIC 标准选择出最佳替代模型为CpREV+I+G+F,根据最佳模型使用应用最大似然法 PhyML3.0 (http://www.atgcmontpellier.fr/phyml/?tdsourcetag=s_pcqq_aiomsg)构建系统发生树。最大似然法节点的支持度通过1000 次重抽样获得。
2 结果与分析
2.1 DNA 提取结果
提取的俄罗斯红参DNA 浓度≥20 ng/µl,总量≥100 ng,OD260nm/OD280nm=1.8~2.2;基因组DNA 主带清晰可见,无明显降解弥散存在,可用于下一步实验。
2.2 测序质量
测序获得序列根据比对结果(表2),Q20=94.42%,Q30=88.13%测序错误率在正常范围内,测序结果可进行后续分析。
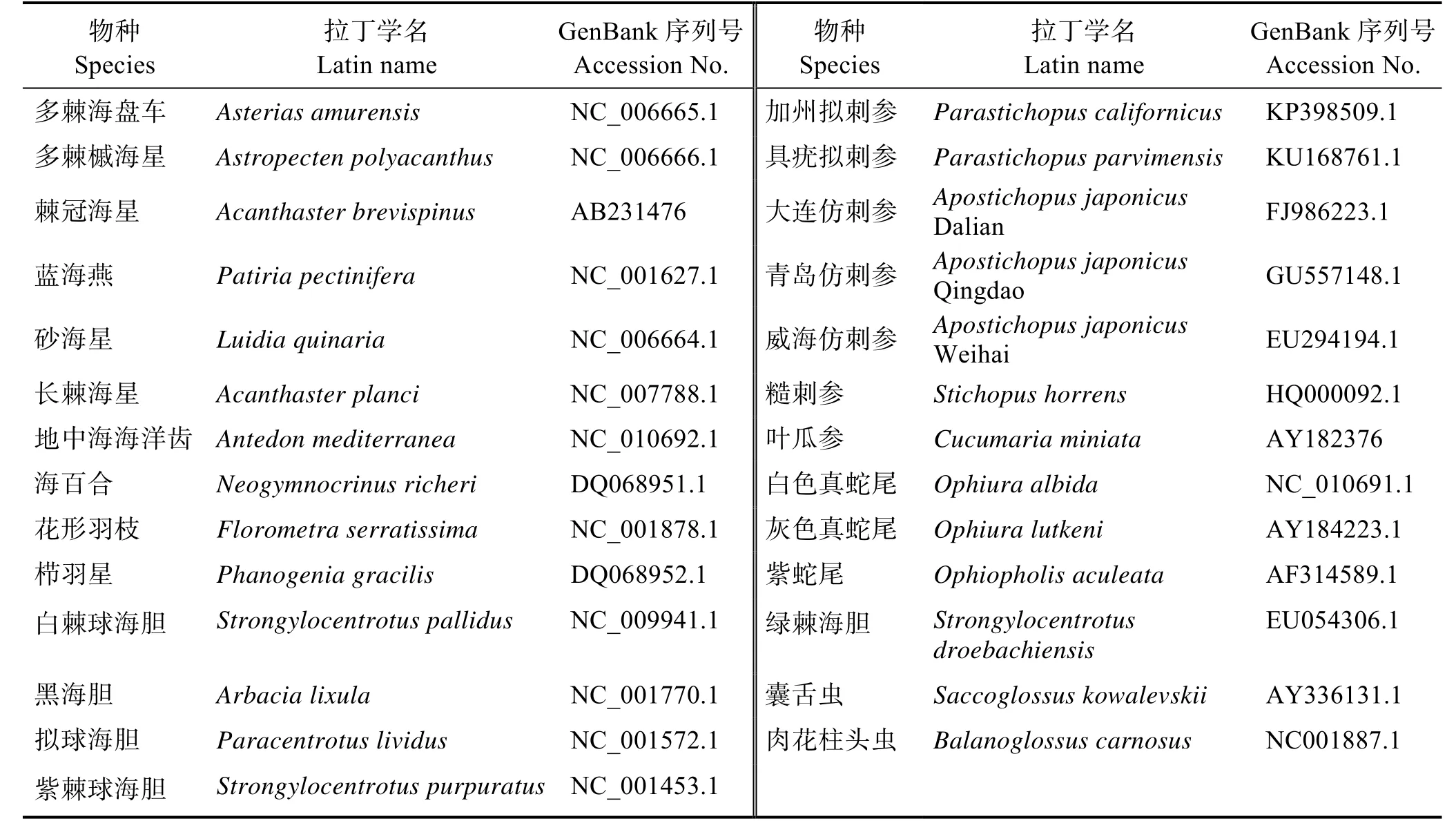
表1 构建系统发生树所用物种及其GenBank 号Tab.1 The GenBank number of the species used to construct the phylogenetic tree

表2 比对结果统计Tab.2 Statistics of comparison results
2.3 线粒体基因组结构组成
2.3.1 基因结构 本研究测得俄罗斯红参线粒体基因组全长为16111 bp,共含有37 个基因(图1),为环状DNA 结构,与目前已知的棘皮动物相似。37 个基因(表3)包括13 个蛋白编码基因、22 个tRNA 基因、2 个核糖体RNA 基因。13 个蛋白编码基因由11 个电子传递系统的基因(复合体Ⅰ、复合体Ⅳ、Cytb 基因)与2 个氧化磷酸化基因(ATP6、ATP8)组成,其中包括7 个还原酶复合体的亚单位复合体Ⅰ(ND4L、ND1、ND2、ND3、ND4、ND5 和ND6)、3 个氧化酶的亚单位复合体Ⅳ基因(COX1、COX2 和COX3)、1 个细胞色素b 基因(Cytb)、2 个ATP 合成酶基因(ATP6 和ATP8)。除tRNA-Gln、tRNA-Ala、tRNA-Val、tRNA-Asp和tRNA-Ser 与ND6 位于L 链外,其他基因均位于H链上。
2.3.2 基因间隔区与重叠区 俄罗斯红参线粒体的基因间隔较多,共存在 24 处基因间隔(表 3),tRNA-Thr 与tRNA-Pro 间隔最大为460 bp,占全长序列的2.86%,其次为ND6 与ND5 基因间隔,为189 bp,以及ND4 与ND3 间隔为181 bp。全序列共存在5 处基因重叠区,在tRNA-Pro 与tRNA-Gln 之间存在4 bp的重叠,l-rRNA 与COX1 之间存在6 bp 的重叠,COX3与tRNA-Ser 存在3 bp 的重叠。ND4 与tRNA-His 间存在10 bp 的重叠,tRNA-His 与tRNA-Ser2 间存在1 bp 的重叠。
2.3.3 碱基含量 线粒体基因全序列碱基组成中,A、G、C 和T 分别为31.79%、17.87%、20.17%和30.17%。A+T 和C+G 含量分别为61.96%和38.04%,A+G 含量高于G+C,表现为较强A、T 偏倚。蛋白编码基因碱基组成中,A、T、C 和G 含量分别为30.03%、31.34%、20.89%和17.74%,A+T 和C+G 的含量分别为61.37%和38.63%,蛋白编码基因的碱基使用与全序列相似,均表现出A、T 偏倚。
2.4 蛋白编码基因的密码子使用

图1 俄罗斯红参线粒体基因组结构Fig.1 Gene organization of Russian red sea cucumber mitochondrial genome
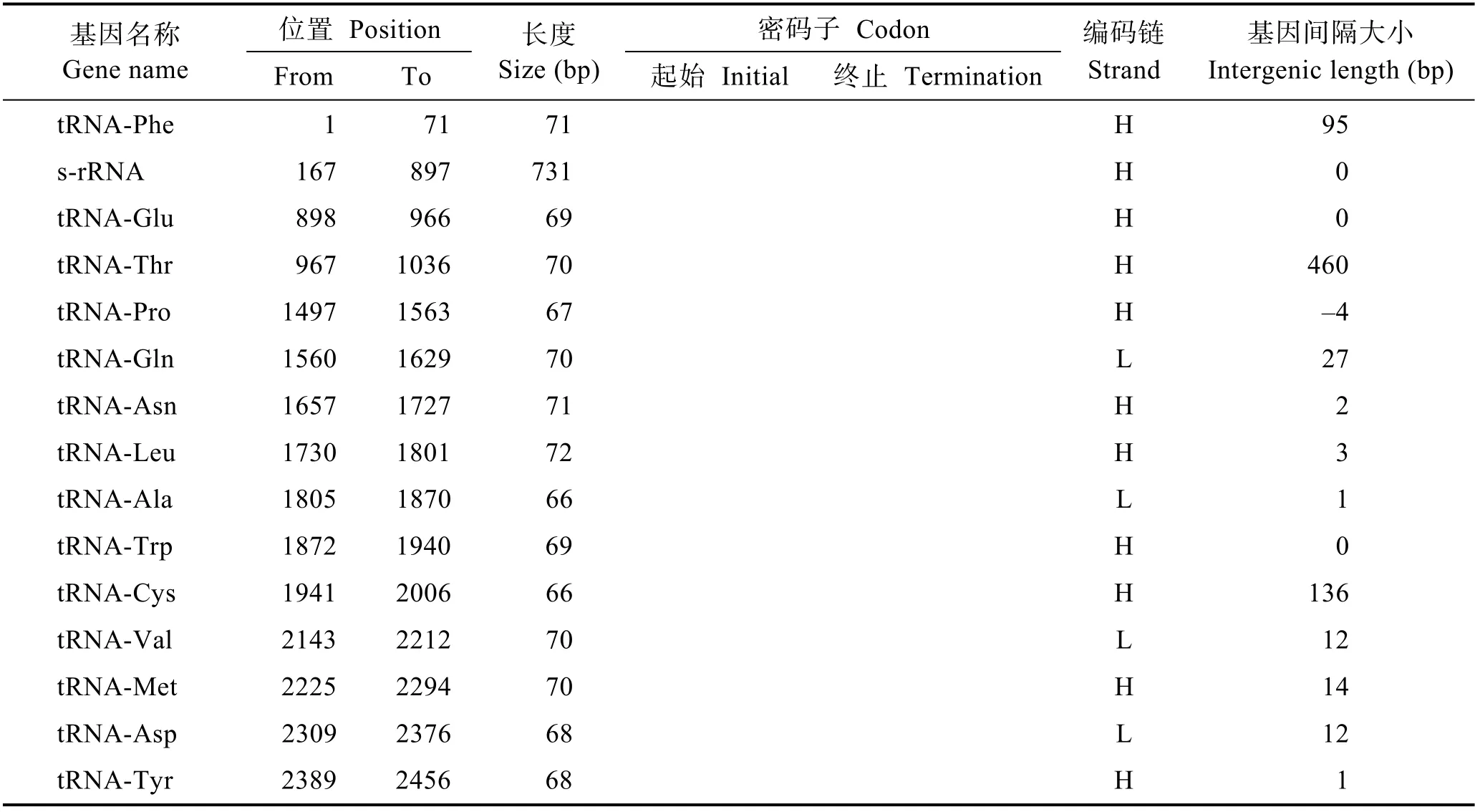
表3 俄罗斯红参线粒体基因组中基因位置及特征Tab.3 Localization and features of genes in the mitochondrial genome of Russian red sea cucumber

续表3

表4 俄罗斯红参线粒体全基因组编码蛋白基因密码子使用情况Tab.4 Codon usage of the protein-coding genes in Russian red sea cucumber mitogenome
13 个编码蛋白基因全长为10868 bp,占全序列的67.5%,共编码3186 个氨基酸(除终止密码子外)。氨基酸编码频率最高为丝氨酸Ser(S),其次为亮氨酸Leu(L)。使用Mega 软件统计密码子使用情况(表4),13 个编码蛋白基因的密码子完整,且存在30 个偏好密码子,其中第3 位点对A 或T(U)的使用频率较高。除ND1 以GTG 为起始密码子,其他均以ATN 为起始密码子。ND2、ND4L、COX2,ATP8、COX3、ND3、ND5 和Cytb 起始密码子为ATG,COX1、ATP6 和ND6 以ATT 为起始密码子,而ND4 以ATA 为起始密码子。终止密码子都是完整密码子,除ND2 与ND4的终止密码子分别为TTG 与TAG 外,其他终止密码子以TAA 为主。
2.5 tRNA
俄罗斯红参mtDNA 序列包含22个tRNA,长度范围为66~72 bp,tRNA-Gln、tRNA-Ala、tRNA-Val、tRNA-Asp 和tRNA-Ser 位于轻链,其他tRNA 位于重链。tRNA-Ser 与tRNA-Leu 各重复1次,最小的为tRNA-Cys,最大的为tRNA-Leu 和tRNA-Ser。除1个tRNA-Ser2 缺少D 环,无法形成三叶草结构,其他tRNA 使用tRNAscanSE 在线软件预测tRNA 的二级结构均为三叶草结构(图2),包括受体臂、TΨC 臂、反密码子臂,D 臂。受体臂:tRNA-Met、tRNA-Gly和tRNA-Ala 发生1 对碱基错配,tRNA-Tyr 插入1 个碱基,tRNA-Arg 发生2 对碱基错配。TΨC 臂碱基组成变化不大,tRNA-Cys、tRNA-His 在TΨC 臂上存在4 对碱基,其他为5 对。D 臂:tRNA-Ile 只有1 对碱基,tRNA-Gly、tRNA-Leu、tRNA-Tyr、tRNA-Leu2、tRNA-Thr、tRNA-Gln 和tRNA-Val 为3 对碱基,其他均为4 对。反密码子臂:tRNA-Pro 存在1 对碱基错配,tRNA-Lys、tRNA-Asp、tRNA-Ser、tRNA-Tyr 的反密码子臂存在4 对碱基,其他tRNA 为5 对。
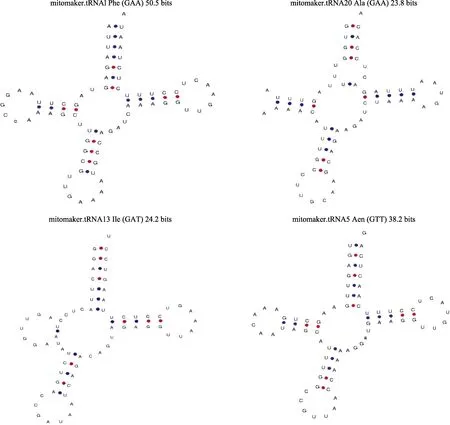



图2 俄罗斯红参的tRNA 的二级结构Fig.2 The secondary structure of tRNA genes from Russian red sea cucumber
2.6 rRNA
俄罗斯红参线粒体全基因组中只存在 2 个rRNA,分别是位于tRNA-Phe 与tRNA-Glu 之间的s-rRNA 和位于tRNA-Ile 与COX1 之间的l-rRNA。s-rRNA 位于H 链,长度为731 bp,碱基A、G、C和T 的含量分别为37.76%、18.74%、18.74%和23.12%,A+T 为60.88%。l-rRNA 位于H 链上,长度为1516 bp。碱基A、G、C 和T 的含量分别为37.86%、19.92%、18.14%、24.08%,A+T 的含量为61.94%。2 个基因的长度和位置不相同,但对碱基的使用相似。
2.7 基因排列比较
从NCBI 下载的动物线粒体编码蛋白基因顺序与所测俄罗斯红参序列对比(图3),发现这些物种间基因排列顺序有一定相似性。俄罗斯红参与海参纲(Holothuroidea)及海胆纲(Echinoidea)基因排列顺序相同,海星纲(Asteroidea)与海百合纲(Crinoidea)中的花 形 羽 枝(Florometra serratissima) 、 栉 羽 星(Phanogenia gracilis)及蛇尾纲(Ophiuroidea)中的紫蛇尾(Ophiopholis aculeata)基因排列顺序相同,白色真蛇尾(Ophiura albida)与灰色真蛇尾(Ophiura lutkeni)基因排列顺序相同。俄罗斯红参、海参纲、海胆纲与海星纲等相同基因排序的物种共享COX1-ND4L-COX2-ATP8-ATP6-COX3-ND3-ND4-ND5-ND6-CYTB 基因模块,与白色真蛇尾和灰色真蛇尾共享COX1-ND4LCOX2-ATP8-ATP6-COX3-ND3-ND4-ND5-ND6 基因模块。几乎每个物种都与其他物种间共享某些特定模块,最多为海百合(Neogymnocrinus richeri)与肉花柱头虫,存在ND1-COX1-COX2-ATP8-ATP6、COX3-ND3和ND4-ND5共3个基因模块,并与海星纲等相同基因排序物种共享ND1-COX1、COX2-ATP8-ATP6、COX3-ND3-ND4-ND5-ND6-CYTB-ND2共3个基因模块,剩余物种都同其他物种存在共享特定模块。

图3 基因排列对比Fig.3 Gene arrangement contrast
2.8 遗传距离
根据不同种类海参及不同地理分布刺参的线粒体核苷酸序列,利用Mega 软件计算遗传距离,使用Kimura-2-parameter 计算其遗传距离(表5)。结果显示,俄罗斯红参与所选海参的遗传距离范围为 0.017~0.385,相较其他海参的遗传距离,俄罗斯红参与青岛仿刺参、威海仿刺参和大连仿刺参间的遗传距离更为接近,分别为0.017、0.019 和0.021。
2.9 BLAST 分析
将俄罗斯红参线粒体全序列与NCBI 上的物种比对(表6),结果显示,其与同名物种的相似率达98%以上,与同源性最高的仿刺参(GenBank 序列号:AB525437.1)的相似性为99.59%,存在24 处碱基的插入与缺失(图4),突变的基因存在转换与颠换,其中,保守位点16024 个,变异位点81 个,变异位点约占分析位点的0.5%。

表5 不同海参核苷酸序列的遗传距离Tab.5 Genetic distances of nucleotide sequences of different sea cucumbers

表6 俄罗斯红参BLAST 分析表Tab.6 Analysis of on Russian red sea cucumber BLAST
2.10 系统发生分析
基于表1 及俄罗斯红参的蛋白编码基因的氨基酸序列,利用最大似然法构建系统发生树(图5),结果显示,海百合纲聚为一枝。俄罗斯红参与具疣拟刺参(P.parvimensis)聚为一枝,并与海参纲其他物种以及多棘槭海星(Astropecten polyacanthus)、蓝海燕(Patiria pectinifera)、灰色真蛇尾、紫蛇尾聚为一大枝。海星纲的长棘海星(Acanthaster planci)、棘冠海星(Acanthaster brevispinus)、砂海星(Luidia quinaria)、多棘海盘车(Asterias amurensis)与海胆科(Temnopleuridae)聚为一大枝。基于表1 及俄罗斯红参的蛋白编码基因核苷酸序列,利用最大似然法构建系统发生树(图6),结果显示,俄罗斯红参与3 个海域的仿刺参聚为一枝,这4 种海参再与海参纲的其他物种聚为一大枝,并与海百合纲4 个物种构成姊妹群。海星纲与海胆纲构成姊妹群。蛇尾纲聚为一枝。

图4 BLAST 比对碱基的缺失与插入Fig.4 Deletion and insertion of alignment bases by BLAST
3 讨论

图5 氨基酸序列基于最大似然法构建的系统发生树Fig.5 Phylogenetic tree of amino acid sequences based on maximum likelihood method

图6 核苷酸序列基于最大似然法构建系统发生树Fig.6 Phylogenetic tree of nucleotide sequences based on maximum likelihood method
本研究通过高通量测序技术对俄罗斯红参的线粒体基因组进行随机测序并分析,利用线粒体序列进行系统发生分析,结果发现,俄罗斯红参与棘皮动物门下的其他动物存在较高的相似性,均存在37 个基因(13 个蛋白编码基因、2 个rRNA 以及22 个tRNA基因),31 个基因编码在H 链上,5 个tRNA (RNA-Gln、tRNA-Ala、tRNA-Val、tRNA-Asp 和 tRNA-Ser)和1 个蛋白编码基因(ND6)编码在L 链上。基因的组成与分布情况均与孙秀俊(2013)所研究的刺参完全相同。全序列与蛋白编码基因的A、T 碱基含量同大部分后生动物一样存在A、T 偏向性(付婉莹, 2015),分别为61.96%与61.37%,且同脊椎及无脊椎动物的mtDNA 蛋白编码基因的第3 位点也存在明显的A、T偏向性,但碱基的含量存在差异,A、T 含量因物种的差异略有不同。
俄罗斯红参的13 个蛋白编码基因的密码子使用分析表明,除ND1 以GTG 为起始密码子外,其他均以ATN 为起始密码子。终止密码子与大连仿刺参(李云峰等, 2012)相同,均为完整密码子,大多以TAA为主。基因重叠区与基因间隔区同糙刺参(Stichopus horrens)(范嗣刚等, 2011)及某些水产动物线粒体相似(李亚莉, 2012; 袁亮等, 2015)。22 个 tRNA 中除tRNA-Ser 外,其余的tRNA 均能预测其三叶草结构,这同魏峦峦等(2011)研究的瘤背石磺(Onchidium struma) tRNA 一样,都是由于缺少D 臂而无法预测其结构,Cheng 等(2012)证明了缺失D 臂并不会影响其进入核糖体及转运氨基酸等功能;相比细胞质中tRNA,线粒体中的tRNA 存在差异,在受体臂与反密码子臂等部分均有碱基错配的情况。此外,俄罗斯红参的 tRNA(tRNA-Cys、tRNA-Trp、tRNA-Ala、tRNA-Leu 和tRNA-Asn)同某些后生动物一样,均出现簇集现象,即几个tRNA 集中在一起的现象。tRNA的重复与闫锦锦(2014) 所研究的光唇鱼(Acrossocheilus fasciatus)相同,皆存在tRNA-Ser 与tRNA-Leu 基因重复1 次。俄罗斯红参的2 个rRNAs(s-rRNA 和l-rRNA)及位置均与申欣等(2011)研究的威海仿刺参和范嗣刚等(2011)研究的糙刺参相同。线粒体基因组排列顺序在研究系统进化关系等方面起着重要作用(李石磊等, 2015),俄罗斯红参线粒体编码基因的排列与大部分棘皮动物具有一定相似性,且与其他物种共享基因模块。
线粒体基因组数据广泛应用于系统进化生物学、保护生物学及种质鉴定等领域的研究,如利用线粒体基因组中的COⅠ及16S rRNA 进行分子标记对海参品种及产品进行鉴别(Wen et al, 2013)。An 等(2013)对韩国红海参野生种群与养殖种群进行微卫星位点多重分析,发现两种群间存在高水平的多态性及遗传异质性。本研究通过对所选海参的核苷酸序列进行遗传距离分析,发现俄罗斯红参与3 个海域的仿刺参较为接近,与青岛仿刺参遗传距离最近,为0.017。将俄罗斯红参的线粒体全序列进行BLAST 分析,与仿刺参(GenBank 序列号:AB525437.1)的相似度为99.59%。基于28 个物种的编码蛋白基因的氨基酸及核苷酸序列,利用最大似然法分别构建系统发生树,结果发现,2 种方法所建的发生树有一定相似性,但在俄罗斯红参与其他海参聚簇上存在差异。在利用核苷酸构建的系统发生树中,俄罗斯刺参与3 个海域的仿刺参聚支的节点的自展值(bootstrap 值)都在95%以上,具有较高可信性;利用氨基酸构建系统发生树在较远距离的系统进化分析中更具有优势和可信性。通常考虑这2 种序列的进化信息,才可能得到更自然的系统发生关系,本研究中核苷酸序列建树结果与传统分类学更为相似。
目前,利用物种线粒体全基因组序列的测定与分析已成为研究物种进化必不可少的手段,广泛应用于各个领域。通过对线粒体基因组的比对和分析,为种质鉴定、地理种群划分和亲缘关系鉴定等提供基础信息。本研究对引入养殖的俄罗斯红参线粒体全基因组进行随机测序,分析发现,其基因结构、碱基含量、密码子、tRNA、rRNA、基因间隔与重叠、组成排列等皆与棘皮动物相似;遗传距离及BLAST 分析发现,俄罗斯红参与仿刺参较为接近。本研究中,俄罗斯红参为引入养殖,作为刺参的地理远缘种,其遗传特点、结构等方面与仿刺参差异较小,具有较高相似性。本研究可为引入养殖俄罗斯红参进一步的遗传系统进化和分类等研究提供资料,同时,对后续俄罗斯红参养殖的推广发展和种质创新与应用提供参考。

