多鳞鱚phds基因家族序列特征及其在低氧胁迫后表达变化
林星桦,叶明慧,SEATAN Wanida,潘炎杨,陈芳圆,黄 洋,朱春华,李广丽,田昌绪
多鳞鱚基因家族序列特征及其在低氧胁迫后表达变化
林星桦,叶明慧,SEATAN Wanida,潘炎杨,陈芳圆,黄 洋,朱春华,李广丽,田昌绪
(广东海洋大学水产学院 // 广东省名特优鱼类生殖调控与繁育工程技术研究中心 // 南方海洋科学与工程广东省实验室(湛江),广东 湛江 524088)
【】分析多鳞鱚 () 脯氨酸羟化酶(prolyl hydroxylases, Phds)基因家族序列特征,探讨其在低氧响应过程的作用。通过硬骨鱼类基因,在多鳞鱚转录组和基因组数据中比对筛选多鳞鱚基因。对多鳞鱚Phds蛋白序列进行结构域预测、系统进化和共线性分析等。采用实时荧光定量PCR检测基因在低氧胁迫和复氧后的表达变化。在多鳞鱚基因组中共鉴定到三个基因,分别是、和,开放阅读框 (ORF) 全长分别为1 635、1 092、723 bp,分别编码544、363、240个氨基酸。结构域预测表明,多鳞鱚Phd1、Phd2、Phd3蛋白均含有P4Hc保守结构域。系统进化分析发现,多鳞鱚Phds均与大黄鱼() 亲缘关系最近。共线性分析表明,多鳞鱚与尼罗罗非鱼()上下游基因高度一致。在低氧处理4 h鳃组织中mRNA表达显著高于常氧组(< 0.05),但在心脏中无显著性差异(> 0.05);在低氧处理4 h的鳃组织中显著低于常氧组(< 0.05),而在心脏中显著高于常氧组(< 0.05)。多鳞鱚存在、和基因,并在不同组织的低氧响应中起重要作用。
多鳞鱚;基因;基因家族;低氧胁迫;基因表达
缺氧影响大多数有氧动物的代谢和存活(如影响鱼类生长、繁殖以及行为活动),已成为阻碍水产养殖业发展的限制性因子之一[1-2]。低氧诱导因子 (hypoxia inducible factor, HIF) 介导的低氧信号通路是细胞产生低氧应答的核心信号通路之一[3-5]。在常氧时,脯氨酸羟化酶 (prolyl hydroxylases, Phds) 会利用氧分子将HIF-1α蛋白上脯氨酸羟基化,从而被泛素连接酶复合体识别并被蛋白酶降解。在低氧时,因无充足氧分子,Phds蛋白无法完成羟基化,细胞内HIF-1α积累并与HIF-1β结合,激活下游基因的表达[3-4],因此,Phds在低氧信号通路中起重要作用。Phds利用氧分子、α-酮戊二酸、二价铁离子和维生素C起羟化酶作用[6]。在人类中发现三种基因,分别是(又名Egl Nine Homolog 2,EGLN2)、(又名Egl Nine Homolog 1,EGLN1)和(又名Egl Nine Homolog 3,EGLN3)。三种基因序列有所不同,在C端有高度同源的序列,Phd2在N端有一个特有的锌指结构域[7]。Phds蛋白序列的差异导致其对人HIF-1α的脯氨酸残基的羟基化能力不同,Phd1和Phd2可羟基化两个保守的脯氨酸残基Pro402和Pro564,但羟化能力有所不同,而Phd3只能羟基化Pro564[8]。在Phd1和Phd3蛋白失活时,HIF-1α无法正常积累,当Phd2蛋白失活时,HIF-1α正常积累并启动下游通路[9],因此,Phd2也被认为是最重要的细胞内氧感受器。在鱼类中,共鉴定到四种基因,分别是、、和。但仅斑马鱼()、白斑狗鱼()、电鳗() 和遮目鱼() 等少数鱼类存在基因。在团头鲂()研究中发现,、和基因均参与低氧应答调控,且有组织特异性[10]。团头鲂Phds蛋白均有羟基化的作用[10],和基因编码时由于可变剪切而形成两种不同的蛋白产物,Phd1的两蛋白均有羟基化能力,而Phd3蛋白羟基化能力还需进一步研究。但是,目前对鱼类Phds蛋白功能的研究较少,研究鱼类基因家族在低氧响应中的功能有重要意义。
多鳞鱚()又名沙锥鱼,隶属鲈形目(Perciformes)鱚科(Sillaginidae)鱚属 (),主要分布印度洋-西太平洋热带浅海,是一种广温、广盐性鱼类[11-12]。多鳞鱚肉质鲜美,营养价值高,深受消费者喜爱,是我国重要的经济鱼类之一[13]。然而,近年来由于过度捕捞,多鳞鱚种群数量减少,产量降低,急需通过人工养殖增加多鳞鱚产量。但由于该物种耐低氧能力差,应激反应强[14],尚未实现大规模工厂化养殖,严重影响了其养殖业的发展。目前,已有多鳞鱚基因组大小、杂合度[15],低氧胁迫下鳃组织转录组[16]等相关分析,但其低氧响应机制尚不明确,亦未见其基因功能的研究报道。本研究通过基因组数据鉴定多鳞鱚基因家族,并对其基因结构,保守功能结构域、motif预测,进化关系,低氧胁迫后表达变化进行分析,为进一步揭示多鳞鱚对低氧的适应机制提供参考。
1 材料与方法
1.1 多鳞鱚phds基因鉴定
从NCBI(https://www.ncbi.nlm.nih.gov/)中检索尼罗罗非鱼()、月光鱼()、青鳉()、红鳍东方鲀()、大黄鱼()、斑马鱼()、鸡()、热带爪蟾()、小鼠()和人类()等10个脊椎动物基因序列(表1)。基于大黄鱼、罗非鱼和斑马鱼核苷酸序列,在多鳞鱚转录组数据(登录号:SRR9673344 ̶ SRR9673355)中检索基因,值为10-5。再将多鳞鱚氨基酸序列基于TBLASTN在多鳞鱚基因组数据(数据未公布,基因组大小521.63 Mb,Contig N50 13.54 Mb,Scaffold N50 21.46 Mb,预测到23 959个基因)中检索基因,值为10-5。两次比对结果中均有完整开放阅读框的基因认定为多鳞鱚基因家族成员。

表1 10种脊椎动物phds基因登录号
1.2 多鳞鱚phds基因序列分析
通过ExPASy (http://web.expasy.org/protparam/)预测多鳞鱚氨基酸序列的蛋白质分子质量和理论等电点。使用SMART (http://smart.embl.de/) 预测多鳞鱚序列结构域。通过多鳞鱚基因组数据,获得多鳞鱚基因染色体位置和外显子、内含子结构。
1.3 phds基因系统进化树构建与结构分析
将上述10种脊椎动物和多鳞鱚基因家族成员氨基酸序列通过MEGA X软件中MUSCLE程序进行比对,并通过邻接法构建系统进化树。将多鳞鱚序列通过Gsds2.0 (http://gsds.cbi.pku.edu. cn/)进行可视化基因结构分析。将11种脊椎动物Phds蛋白序列通过MEME v5.1.1 (http://meme-suite. org/tools/meme) 鉴定基因保守motif。
1.4 phds基因共线性分析
在Genomicus 97.01(https://www.genomicus.biologie.ens.fr/genomicus-97.01/cgi-bin/search.pl) 中查找人、斑点雀鳝() 和尼罗罗非鱼基因家族成员在染色体中的位置及上下游基因。通过多鳞鱚基因组数据查找多鳞鱚基因家族成员在染色体中的位置及上下游基因。将人、斑点雀鳝、尼罗罗非鱼与多鳞鱚基因及上下游基因进行共线性比对分析。
1.5 实验动物和低氧胁迫处理
实验用1龄多鳞鱚取自广东海洋大学东海岛海洋生物研究基地,体长(13.40 ± 1.05)cm,体质量(14.57 ± 3.17)g。将体型相近多鳞鱚随机分配到4个200 L鱼缸容器中(编号为1、2、3、4),每缸120尾,暂养一周后进行低氧胁迫处理。通过调节氮气与空气的充气速度控制溶解氧 (DO) 水平,实时监测DO以符合实验需求。实验前每缸水体溶解氧为8.0 mg/L。1、2、3号缸水体同时持续通入氮气,使DO至1.5 mg/L,1、2号缸分别低氧处理(1.5 mg/L) 1、4 h,3号缸低氧处理4 h后复氧处理(8 mg/L)4 h,4号缸(对照组)保持正常氧气浓度(8 mg/L)。每个缸分别取多鳞鱚10尾,麻醉,剖取各组多鳞鱚的鳃、心脏组织,放入液氮中速冻,转移到-80℃保存。
1.6 RNA提取和逆转录
用Trizol法提取总RNA,以琼脂糖凝胶电泳检测RNA完整性,参照Prime Script RT reagent Kit with gDNA Eraser (TaKaRa,大连) 试剂盒说明书将总RNA反转录为cDNA,置- 20 ℃下保存备用。
1.7 实时荧光定量PCR(qPCR)检测
将对照组和不同低氧处理的多鳞鱚鳃、心脏组织cDNA稀释5倍作为模板,多鳞鱚为内参基因,引物信息见表2。
使用LightCycler 96 (Roche,USA) 实时荧光定量分析仪进行qRT-PCR分析。反应体系为Power Green qPCR Mix (with ROX) (东盛生物科技有限公司)10 μL,正反向引物各0.8 μL,cDNA模板2 μL,双蒸水6.4 μL。反应程序:95 ℃ 10 min;95 ℃ 10 s、60 ℃ 30 s、72 ℃ 15 s,共40个循环。每个样品有3个生物学重复,2个技术重复。以相对ΔCT法(2–ΔΔCT法) 计算相对表达量,并通过SPSS 17.0软件进行单因素方差分析和多重比较。
2 结果分析
2.1 多鳞鱚phds基因的鉴定和序列分析
根据硬骨鱼类基因的比对结果,共鉴定出3个基因,分别为、和GenBank登录号分别为MT518875、MT518876和MT518877。表3显示多鳞鱚基因结构、编码蛋白基因信息。多鳞鱚,和基因mRNA长度分别为3 770、2 431、1 882 bp,开放阅读框(ORF)全长分别为1 635、1 092、723 bp,分别编码544、363、240个氨基酸。多鳞鱚基因分子质量为26.93 ~ 57.57 ku,等电点为7.85 ~ 8.93。

表3 多鳞鱚中phds基因特征
图1为3个多鳞鱚Phds蛋白结构域预测结果。多鳞鱚Phds蛋白均含有一个保守结构域,为脯氨酸-4-羟化酶(Prolyl 4-hydroxylase alpha subunit homologues, P4Hc) 结构域。多鳞鱚Phd1和Phd2中还含有部分低复杂度区域 (low complexity regions)。此外,Phd2中存在一个MYND型锌指结构域 (MYND-type zinc finger domains, zf-MYND)。
2.2 phds基因系统进化分析
基于多鳞鱚和其他脊椎动物的氨基酸序列构建系统进化树(图2)。结果显示,各物种、、各聚为一大支,且和的同源性更高。、、分支中鱼类聚为一支,哺乳类聚为一支。多鳞鱚、、分别于大黄鱼()、、亲缘关系最近。
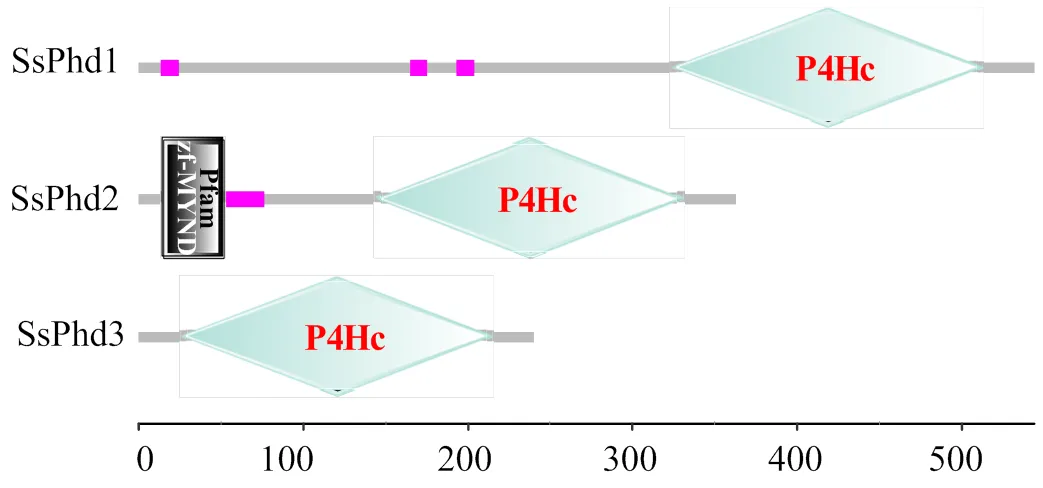
图1 多鳞鱚Phds蛋白保守结构域

11物种的Phds蛋白分别是:多鳞鱚SsPhd,大黄鱼LcPhd,尼罗罗非鱼OnPhd,月光鱼XmPhd,青鳉OlPhd,红鳍东方鲀TrPhd,斑马鱼DrPhd,鸡GgPhd,热带爪蟾XtPhd,人类HsPhd和小鼠MmPhd
2.3 phds基因结构分析
对多鳞鱚、、基因结构可视化分析结果(图3)显示,多鳞鱚、、基因分别包含6、5、5个外显子。使用MEME鉴定Phds蛋白的保守motif,共发现7个不同的保守motif(图4)。这些保守motif长度为15 ~ 50个氨基酸。在11种脊椎动物Phds蛋白中均发现motif1、motif2、motif3、motif4、motif6和motif7。基于SMART分析,motif1、motif2、motif3、motif4、motif6和motif7是高度保守的P4Hc结构域的6个片段。Phd2存在一个特有的motif5,预测motif5与zf-MYND结构域相关。
2.4 phds基因共线性分析
、和基因共线性分析结果(图5) 显示,除多鳞鱚在基因下游的(Neuronal PAS Domain Protein 3) 基因发生串联重复事件,产生一个(Neuronal PAS Domain Protein 3-like) 基因外,、和基因在多鳞鱚和尼罗罗非鱼的上下游基因表现出极高的一致性。
2.5 低氧胁迫处理后多鳞鱚phds基因表达变化
基因在多鳞鱚低氧胁迫和复氧后mRNA表达变化见图6。在鳃组织中,与常氧组相比,和基因在低氧处理4 h时mRNA表达显著上升 (<0.05),基因在低氧处理组间(低氧1 h和低氧4 h)mRNA表达均显著下降(<0.05),、和基因在复氧4 h后 mRNA表达均显著下降(<0.05)。在心脏组织中,与常氧组多鳞鱚相比,在低氧处理4 h时mRNA表达显著上升 (<0.05)而在各处理组间(低氧1 h、低氧4 h和恢复氧气4 h)mRNA表达无显著差异 (>0.05),相对表达量在低氧4 h、恢复氧气4 h时均显著上升(<0.05)。

图3 多鳞鱚中phds基因结构
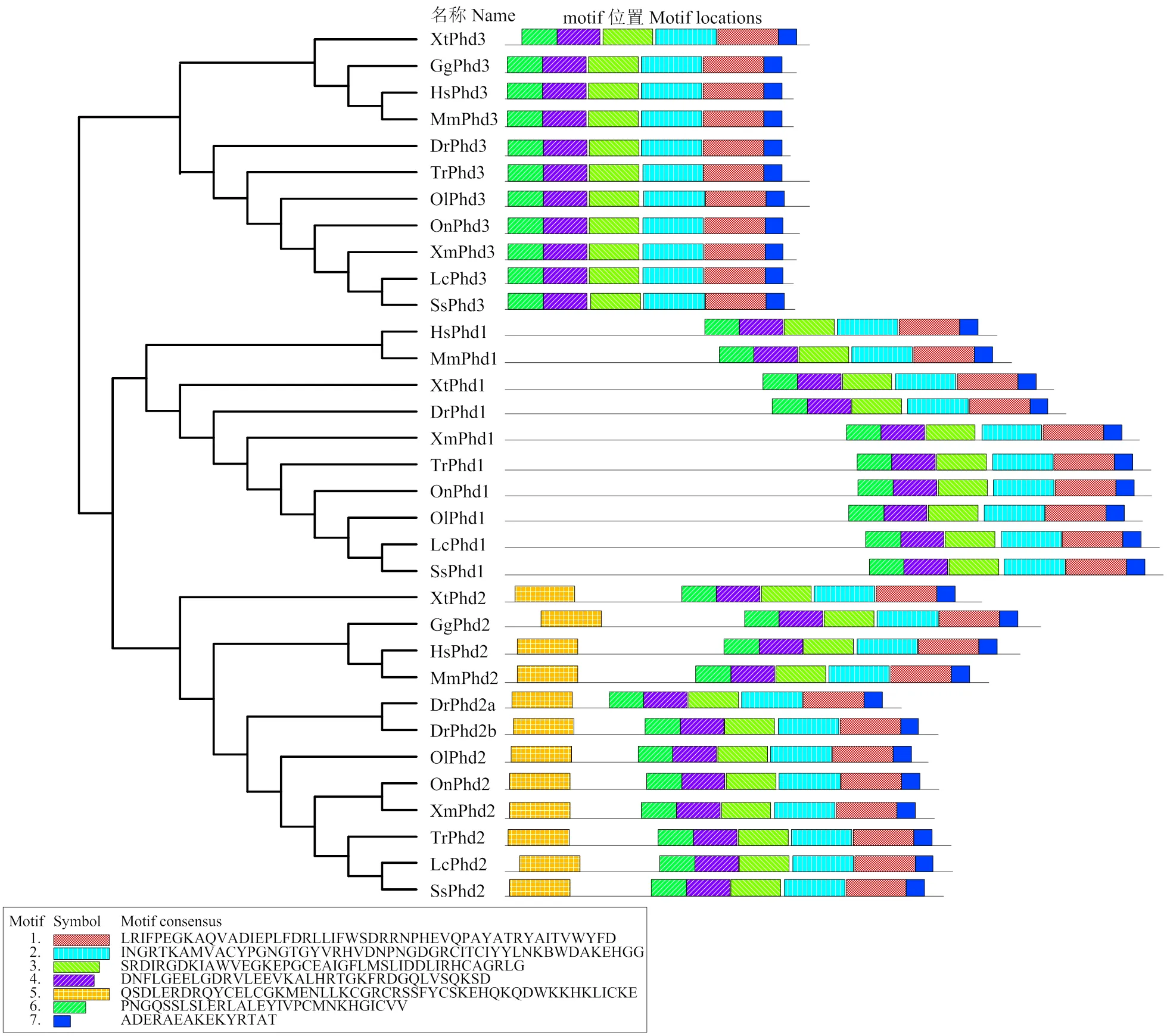
图4 Phds蛋白的保守motif分析

图5 人、斑点雀鳝、多鳞鱚与尼罗罗非鱼phds基因共线性分析
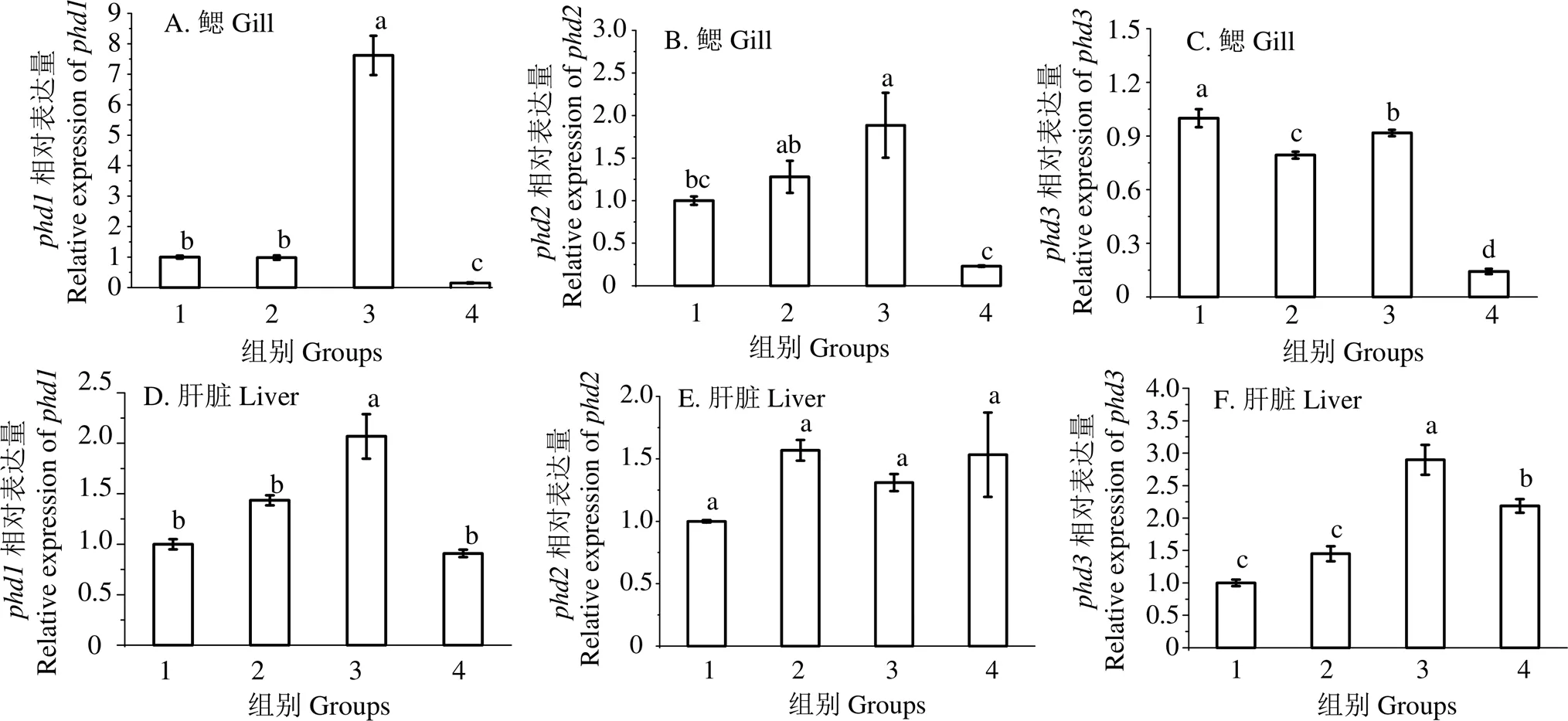
凡含一个相同字母表示组间差异无统计学意义(P > 0.05);1. 常氧对照;2. 低氧处理1 h;3. 低氧处理4 h;4. 复氧4 h
3 讨论
在鱼类低氧相关研究中,Phd是低氧信号通路的重要调节因子之一,开展鱼类Phds基因鉴定及低氧相关功能研究有重要意义。本研究在多鳞鱚中鉴定出等3个基因。系统进化分析表明,各聚为一大支,各大支中鱼类均聚为一支,说明基因在进化过程中比较保守。多鳞鱚分别与大黄鱼的亲缘关系最近。
基因家族成员C端均有高度同源的序列,但Phd2的N端有一特有的锌指结构域[7]。本研究中,多鳞鱚基因家族成员的C端均有一保守结构域P4Hc。P4Hc负责催化胶原蛋白和其他蛋白质-xaa-pro-gly-序列中4-羟脯氨酸[17],是基因羟基化功能的结构域。多鳞鱚基因N端有一个zf-MYND结构域,MYND由半胱氨酸和组氨酸残基以固定间隔排列形成潜在的锌结合motif,可能参与蛋白质间的相互作用[18]。基因对HIF-1α亚基有较强羟基化能力[19-20],可能与具有独特的zf-MYND结构域密切相关。
基因结构多样化和motif组成在许多基因家族的进化和功能中有重要作用。外显子数量在同一基因家族的不同基因中有所不同。多鳞鱚和基因外显子数量分别为6、5、5,与ensembl数据库(http://asia.ensembl.org) 中与斑马鱼外显子数量一致,但与大黄鱼有所不同(3基因外显子数量均为5个);同一基因中motif的类型和数量相同,表明基因结构较为保守;在中均有6个相同的motif,这6个motif与基因C端的P4Hc结构域相关[17],而基因中有一个独特的motif,与N端的zf-MYND结构域相关。基因结构决定其功能,多鳞鱚基因家族中基因的独特结构域与motif,可能与对HIF-1α亚基有较强羟基化功能相关[19-20]。共线性分析表明,人和基因上下游与鱼类相比有较大差异,而人基因与鱼类上下游一致性较高,说明基因在进化过程中较为保守。在鱼类基因中,和基因在鱼类中较为保守,而斑点雀鳝基因与尼罗罗非鱼和多鳞鱚上下游基因位置发生了互换。多鳞鱚与尼罗罗非鱼基因家族在染色体区段的上下游基因有保守性。多鳞鱚基因下游基因发生复制,而基因编码与多种人类精神病和神经发育障碍相关的转录因子[21],其在多鳞鱚中的功能尚需进一步研究。
本研究中,与对照组相比,多鳞鱚鳃组织基因在低氧处理4 h时mRNA表达显著上升 (<0.05),团头鲂在低氧处理后该2种基因亦有相同表达规律[22-23],说明和在不同鱼类低氧胁迫转录响应中发挥了作用。在低氧处理1、4 h后表达量均显著下降(<0.05),而团头鲂中表达显著上升[24],可能由低氧处理时间不同或低氧条件下表达模式存在物种差异所致。多鳞鱚在复氧4 h后表达量均显著下降,该结果与多鳞鱚转录组数据相符[16],可能是低氧胁迫引起多鳞鱚鳃组织受到损伤,导致基因无法恢复到正常表达水平。在多鳞鱚心脏组织中,与对照组相比,在低氧处理4 h时mRNA表达显著上升 (<0.05)而在各处理组间(低氧1 h、低氧4 h和恢复氧气4 h)mRNA表达无显著差异(>0.05),而在团头鲂中和表达量均显著下降[22-23]。在低氧4 h时,多鳞鱚均显著上升(<0.05),而团头鲂表达量也显著上升,表明多鳞鱚受到低氧调控。综上,多鳞鱚均受到低氧调控。在低氧条件下,和在鳃中发挥作用,而则在鳃和心脏中均发挥作用,说明基因在低氧调控时存在组织特异性。
[1] POLLOCK M S, CLARKE L M J, DUBÉ M G. The effects of hypoxia on fishes: from ecological relevance to physiological effects[J]. Environmental Reviews, 2007, 15(NA): 1-14.
[2] ROBERTS J J, BRANDT S B, FANSLOW D, et al. Effects of hypoxia on consumption, growth, and RNA: DNA ratios of young yellow perch[J]. Transactions of the American Fisheries Society, 2011, 140(6): 1574-1586.
[3] 肖武汉. 低氧信号传导途径与鱼类低氧适应[J]. 中国科学:生命科学,2014,44(12):1227-1235.
[4] DUNWOODIE S L. The role of hypoxia in development of the mammalian embryo[J]. Developmental Cell, 2009, 17(6): 755-773.
[5] 李福祥,全燕,夏前明. 低氧诱导因子及其低氧调节机制[J]. 中国临床康复,2004,8(30): 6744-6746.
[6] SINGH L, ALDOSARY S, SAEEDAN A S, et al. Prolyl hydroxylase 2: a promising target to inhibit hypoxia-induced cellular metabolism in cancer cells[J]. Drug Discovery Today, 2018, 23(11): 1873-1882.
[7] RYTKÖNEN K T, WILLIAMS T A, RENSHAW G M, et al. Molecular evolution of the metazoan PHD-HIF oxygen-sensing system[J]. Molecular Biology and Evolution, 2011, 28(6): 1913-1926.
[8] LANDÁZURI M O, VARA-VEGA A, VITÓN M, et al. Analysis of HIF-prolyl hydroxylases binding to substrates[J]. Biochemical and Biophysical Research Communications, 2006, 351(2): 313-320.
[9] BERRA E, BENIZRI E, GINOUVÈS A, et al. HIF prolyl-hydroxylase 2 is the key oxygen sensor setting low steady-state levels of HIF-1alpha in normoxia[J]. The EMBO Journal, 2003, 22(16): 4082-4090.
[10] 陈楠. 团头鲂基因家族参与低氧应答的分子机理解析[D]. 武汉: 华中农业大学,2017.
[11] 薛泰强. 鱚科几种鱼类的形态学及遗传学研究[D]. 青岛: 中国海洋大学,2010.
[12] 黄洋,杜涛,黄海立. 多鳞鱚Forskál人工繁殖研究[J]. 广东海洋大学学报,2013,33(01):15-21.
[13] 杜涛,黄洋. 多鳞鱚生物学特性及室内养殖试验[J]. 水产养殖,2009,30(3):1-3.
[14] 杜涛,黄洋,曹剑香. 多鳞鱚采捕暂养的初步研究[J]. 养殖与饲料,2009(10):15-17.
[15] LI Z Y, TIAN C X, HUANG Y, et al. A first insight into a draft genome of silver() via genome survey sequencing[J]. Animal, 2019, 9(10):756.
[16] SAETAN W, TIAN C X, YU J W, et al. Comparative transcriptome analysis of gill tissue in response to hypoxia in silver()[J]. Animals, 2020, 10(4): 628.
[17] ARAVIND L, KOONIN E V. The DNA-repair protein AlkB, EGL-9, and leprecan define new families of 2-oxoglutarate- and iron-dependent dioxygenases[J]. Genome Biology, 2001, 2(3): 1-8.
[18] GROSS C T, MCGINNIS W. DEAF-1, a novel protein that binds an essential region in a Deformed response element[J]. The EMBO Journal, 1996, 15(8): 1961-1970.
[19] HIRSILÄ M, KOIVUNEN P, GÜNZLER V, et al. Characterization of the human prolyl 4-hydroxylases that modify the hypoxia-inducible factor[J]. The Journal of Biological Chemistry, 2003, 278(33): 30772-30780.
[20] APPELHOFF R J, TIAN Y M, RAVAL R R, et al. Differential function of the prolyl hydroxylases PHD1, PHD2, and PHD3 in the regulation of hypoxia-inducible factor[J]. The Journal of Biological Chemistry, 2004, 279(37): 38458-38465.
[21] LUOMA L M, BERRY F B. Molecular analysis of NPAS3 functional domains and variants[J]. BMC Molecular Biology, 2018, 19(1): 1-19.
[22] CHEN N, HUANG C X, HUANG C H, et al. The molecular characterization, expression pattern and alternative initiation ofHif prolyl hydroxylase Phd1[J]. Gene, 2018, 678: 219-225.
[23] WANG H J, HUANG C X, CHEN N, et al. Molecular characterization and mRNA expression of HIF-prolyl hydroxylase-2 (phd2) in hypoxia-sensing pathways from[J]. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology, 2015, 186: 28-35.
[24] CHEN N, HUANG C H, CHEN B X, et al. Alternative splicing transcription ofHIF prolyl hydroxylase PHD3 and up-regulation of PHD3 by HIF-1α[J]. Biochemical & Biophysical Research Communications, 2016, 469(3): 737-742.
Sequence Characteristics and mRNA Expression Analysis of theGene Family in Silver Sillago () under Hypoxia Stress
LIN Xing-hua, YE Ming-hui, SEATAN Wanida, PAN Yan-yang, CHEN Fang-yuan, HUANG Yang, ZHU Chun-hua, LI Guang-li, TIAN Chang-xu
(////(),524088,)
To analyze the information of(prolyl hydroxylases) gene family in, and explore the role of the gene family in mediating the hypoxia response.Thegenes were screened in the transcriptome and genomic dataset by BLAST. The Phds protein were analyzed by domain prediction, phylogenetic analysis and collinearity analysis. The expression ofin gill and heart after hypoxia stress and reoxygenation treatment were studied by real-time fluorescence quantitative PCR.Threegenes were identified and the open read frame (ORF) of,andwas 1635, 1092 and 723 bp, encoding for proteins of 544, 363, and 240 amino acids respectively. The domain prediction results have shown that the Phds proteins consist of conserved P4Hc domains. Phylogenetic analysis revealed that the relationship betweenandwas the highest. The collinear analysis indicated that thegenes ofandconsisted of the upstream and downstream genes in chromosome. In the gill,andmRNA expression were significantly increased in hypoxia group in normoxia group after 4 hours, but there was no significant difference in the heart tissue. Thewas significantly decreased in hypoxia group than normoxic group after 4 hours of hypoxia treatment in the gill and was significantly increased than the normoxia group in the heart.,, andgenes have been identified in, and play an important role in the hypoxia response of different tissues.
; prolyl hydroxylases gene; gene family; hypoxia stress; gene expression
Q78;Q959.483.3
A
1673-9159(2020)06-0001-08
10.3969/j.issn.1673-9159.2020.06.001
林星桦,叶明慧,SEATAN Wanida,等. 多鳞鱚基因家族序列特征及其在低氧胁迫后表达变化[J]. 广东海洋大学学报,2020,40(6): 1-8.
2020-05-29
广东省普通高校特色创新类项目(2019KTSCX060);广东省普通高校青年创新人才类项目(2018KQCX111);广东省科技计划项目(2014A020208117, 2015A020209163);广东省大学生创新创业计划项目(CXXL2018048, CXXL2019138);广东海洋大学科研启动经费项目(R19026)
林星桦(1997―),男,硕士研究生,研究方向为鱼类分子遗传育种与功能基因组学。E-mail: linxinghua97@163.com
田昌绪(1985―),男,讲师,博士,研究方向为鱼类分子遗传育种与功能基因组学。E-mail: tiancx@gdou.edu.cn
(责任编辑:刘庆颖)

