基于分子对接技术从虾夷扇贝中筛选抗SARS-CoV-2活性肽研究
步 营, 何 玮, 王 飞,2, 朱文慧,*, 李学鹏,仪淑敏, 徐永霞, 励建荣,*
(1.渤海大学 食品科学与工程学院/生鲜农产品贮藏加工及安全控制技术国家地方联合工程研究中心/国家鱼糜及鱼糜制品加工技术研发分中心, 辽宁 锦州 121013;2.大连工业大学 海洋食品精深加工关键技术省部共建协同创新中心, 辽宁 大连 116034)
2019新型冠状病毒(SARS-CoV-2)感染的肺炎(COVID-19)严重威胁着全球公共卫生安全,截至2020年7月1日,全球累计COVID-19确诊病例已超1 059万,累计死亡病例超过51万[1]。我国海洋生物活性物质资源丰富,利用计算机模拟从水产原料中筛选具有抗SARS-CoV-2能力的生物活性的肽段,可为研发相应的海洋功能食品提供新的发展方向。
SARS-CoV-2属于β型冠状病毒,与人类严重急性呼吸综合征相关冠状病毒(SARS-CoV)具有79%的序列相似性,序列同源性较高[2];与其他冠状病毒相似,表现为包膜包被的单股正链RNA病毒。SARS-CoV-2由刺突蛋白(S)、包膜蛋白(E)、膜蛋白(M)和核衣壳蛋白(N)四种主要结构蛋白构成[3]。构成病毒刺突的S蛋白,在病毒侵袭宿主细胞的过程中发挥着至关重要的作用,S蛋白在结构上分为S1和S2两个亚基,其中S1亚基中具有的受体集合域(RBD)核心部位,与SARS-CoV的RBD核心部位高度一致[2,4]。不仅如此,对SARS-CoV-2的构象研究表明,许多单克隆抗体[5-7]也能够如同针对SARS-CoV一样以S蛋白的RBD作为关键靶标,并与血管紧张素转化酶2(ACE2)相互作用进入呼吸道上皮细胞[8],达到抑制病毒侵染的目的。另外,经过测量,SARS-CoV-2的RBD不仅可以与人ACE2发生强力结合,其亲和力和SARS-CoV的S蛋白与人ACE2之间的亲和力相当[9],而且SARS-CoV-2/ACE2结合蛋白与SARS-CoV/ACE2的原结构构象相比并未发生太大改变[10]。因此推测,SARS-CoV-2的感染和传播所依靠的受体和机制与SARS-CoV相差无几。除此之外,主蛋白酶(Mpro)作为冠状病毒在宿主细胞内通过转录翻译复制病毒时是必不可少的关键酶[11],以Mpro为靶标进行模拟筛选,也为研发抗SARS-CoV-2的功能性食品提供了新的思路。
分子模拟作为强大的科学研究工具,可通过模拟、计算和分析分子系统及其相互作用来研究实验现象或指导新实验的发展。分子对接的广泛利用可以清晰地了解活性肽和受体蛋白的对接位点及二者之间存在的相互作用力,从而分辨出肽段的生物活性和有效性。国内外研究团队在利用分子模拟进行SARS-CoV-2的防治药物研究方面做出颇多成果[12-16],但多数是在已知药物成分中进行筛选,未见有从食品原料中提取活性肽进行模拟研究的报道。虾夷扇贝(Mizuhopectenyessoensis)是一种重要的海洋经济贝类,同时具有较大的药用和保健价值。据报道,虾夷扇贝肌球蛋白的酶解肽段具有较好的潜在生物活性,部分还表现出优良的抗血管紧张素转化酶活性[17]。经蛋白基因库查询可知,虾夷扇贝肌球蛋白的蛋白序列明确且易得可供我们进行虚拟筛选。
本研究拟通过计算机模拟,从水产动物虾夷扇贝肌球蛋白中筛选出具有抗SARS-CoV-2潜在活性的小分子功能肽段,希望为抗SARS-CoV-2功能食品的开发与应用提供一定的理论参考。
1 实验方法
1.1 模拟水解虾夷扇贝肌球蛋白
利用ExPASy PeptideCutter(http:∥web.expasy.org/peptide_cutter)软件对虾夷扇贝的肌球蛋白序列(GenBank: BAB40711.1)进行模拟酶解,预测肌球蛋白被蛋白酶或化学物质裂解的潜在裂解位点,并在其上映射可能的切割位点和切割位点位置表。采用胃蛋白酶(pH值1.3)、低特异性胰凝乳蛋白酶(C端指[FYWML],而非P之前)和胰蛋白酶[17]对肌球蛋白进行模拟水解,整理水解所得肽段,以备后续研究。
1.2 肽段的活性和毒性预测
采用Peptide Ranker(http:∥distilldeep.ucd.ie/PeptideRanker/)服务器预测生物活性肽的生物学活性并对肽进行分类,当预测的肽段分数超过0.5,则表明肽段具有潜在生物活性。在蛋白数据库BIOPEP-UWM(http:∥www.uwm.edu.pl/biochemia/index.php/pl/biopep)中搜索预测分数超过0.5,即具有潜在活性的肽段序列,根据搜索结果选择蛋白库内未知的肽段进行毒性预测,采用ToxinPred(http:∥www.imtech.res.in/raghava/toxinpred/)程序预测所选肽段毒性。
1.3 受体蛋白晶体的选择
选择的受体蛋白晶体为SARS-CoV-S/ACE2复合蛋白[18-19]和COVID-19 Mpro 水解酶[20]的高分辨率晶体结构,可从RCSB PDB数据库(http:∥www.rcsb.org)中获得,PDB蛋白库编号分别为2AJF和6LU7。
1.4 分子对接
采用Discovery Studio(v17.2.016349)软件进行分子对接。首先使用DS /Macromoleculars根据肽段氨基酸序列构建肽段的三维结构,在构建好选定肽段的初始结构后,对肽段结构进行最小化优化,随后进行受体准备。小分子肽段处理结束后,将SARS-CoV-S/ACE2复合蛋白的三维结构导入软件中,选择SARS-CoV-S/ACE2复合蛋白晶体结构中的A链和E链作为受体,二者分别代表着ACE2和SARS-CoV-S蛋白结构。去除蛋白晶体结构周围存在的水分子和配体集团,做“Prepare Protein”处理,COVID-19 Mpro 水解酶蛋白晶体的三维结构与SARS-CoV-S/ACE2复合蛋白晶体处理方式相同。参考沈亮亮等[21]的研究,选择SARS-CoV-S/ACE2复合蛋白晶体结构中A链上的ASP38、GLU42、GLN325和GLU329以及E链上的TYR436和TYR491共6个关键氨基酸定义为SARS-CoV-S/ACE2复合蛋白分子对接的活性口袋,选择COVID-19 Mpro 水解酶蛋白晶体上的THR24、THR25、THR26、LEU27、ASN28和ASN119 共6个关键氨基酸定义为COVID-19 Mpro水解酶蛋白分子对接的活性口袋。将小分子肽段分别与SARS-CoV-S/ACE2复合蛋白上确定的活性结合口袋、COVID-19 Mpro水解酶蛋白上确定的活性结合口袋进行LibDock半柔性分子对接,选择构象参数为“BEST”,其余参数仍为默认值。对接成功的肽段结果按照LibDock Score分值高低降序排列,分析肽段与两种蛋白活性口袋中关键氨基酸形成的相互作用力,从而筛选出具有潜在COVID-19/ACE2结合阻断能力和COVID-19 Mpro水解酶抑制能力的肽段。
2 结果与讨论
2.1 虾夷扇贝肌球蛋白模拟酶解及酶解肽段潜在活性、毒性预测
经模拟酶解后1 945个氨基酸的肌球蛋白序列被酶解为415条肽段,将所有肽段进行Peptide Ranker预测,选择预测分数大于0.5的共28条肽段序列,序列位置及评分如表1。对表中28条具有潜在生物活性的肽段进行BIOPEP-UWM蛋白库搜索,肽段已知生物活性如表2 。表2中,肽段GM、GR、GY、GI、NCW和VAF具有不同程度的ACE抑制活性,NW、QW、GY、IM、NM和GI具有DPP IV抑制活性;除此之外,三肽ADF具有抗氧化活性,四肽QRPR能够进行免疫调节,其余肽段并未在活性蛋白库中搜索到明确的生物活性。在此基础上,对28条肽段序列进行毒性预测。表3预测结果表明,28条肽段均无毒性,可以用于后续抑制剂研究。
2.2 SARS-CoV-S/ACE2 蛋白受体与肽段的分子对接
以SARS-CoV-S/ACE2蛋白为对接受体,从而选择能够与复合蛋白构象关键氨基酸结合形成氢键的肽段,达到筛选COVID-19/ACE2结合阻断肽的目的。
图1为SARS-CoV-S/ACE2复合蛋白A链和E链的三维结构图,根据已知关键氨基酸所选择的结合口袋空间坐标为11.825 486、-2.174 257、70.748 771,半径13.051 520。28条肽段与SARS-CoV-S/ACE2复合蛋白对接结果汇总如表4。表4中,28条肽段均能进入复合蛋白结合口袋并与受体上的氨基酸结合,其中17条肽段能够与蛋白受体上的关键氨基酸形成氢键。图2为活性肽段与SARS-CoV-S/ACE2复合蛋白对接2D示意图。由图2(a)、图2(b)可见,肽段AM和QPGL除了能与关键氨基酸TYR491形成氢键外,还能以疏水相互作用力与之结合,LibDock Score 打分为103.22、86.64;而肽段CSNAIPEL可以与A链上的GLN42和E链上的GLU329两个关键氨基酸形成氢键(见图2 (c)),肽段LibDock Score打分136.03;肽段LPIY既能与受体蛋白E链上的TYR491作用形成氢键,也能够以静电作用与A链上的ASP38产生相互作用(见图2 (d)),LibDock Score为142.85。与其他能够与关键氨基酸形成氢键的肽段相比,CSNAIPEL和LPIY肽段能够与更多关键氨基酸发生相互作用,因此可以预测二者与SARS-CoV-S/ACE2复合蛋白结合更加紧密,具有更大阻断COVID-19/ACE2 蛋白受体结合的潜力。除此之外,虽然肽段HM、IM、NM、GM也可以与两个关键氨基酸以静电作用、疏水作用等方式发生相互作用,但它们并未与关键氨基酸形成氢键,结合强度较弱,因此阻断结合潜力相对较弱,不适合进行进一步研究。

表1 具有潜在生物活性的模拟酶解肽段Tab.1 Peptides with potential biological activity generated from Mizuhopecten yessoensis myosin digestedin silico
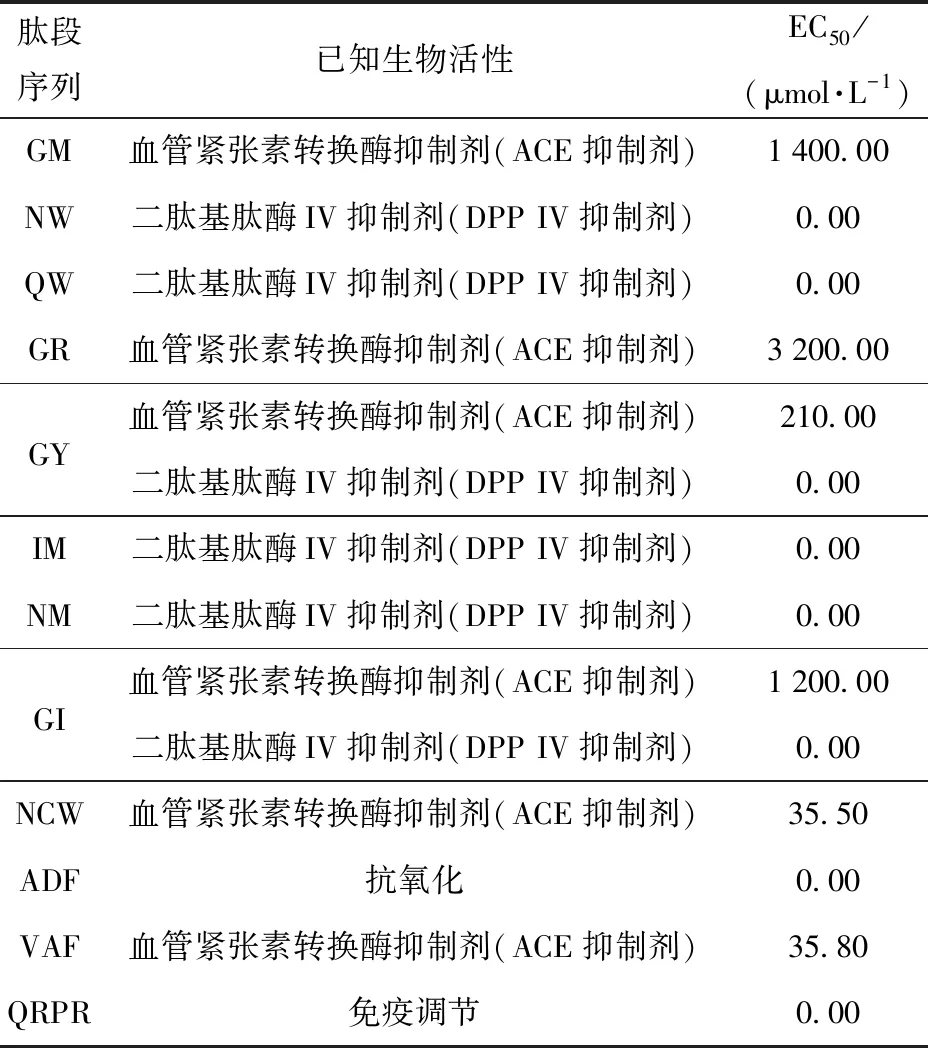
表2 BIOPEP-UWM蛋白库已知生物活性肽Tab.2 Known bioactive peptides in BIOPEP-UWM protein database

表3 选择肽段的模拟毒性预测Tab.3 Simulated toxicity prediction of selected peptides in silico
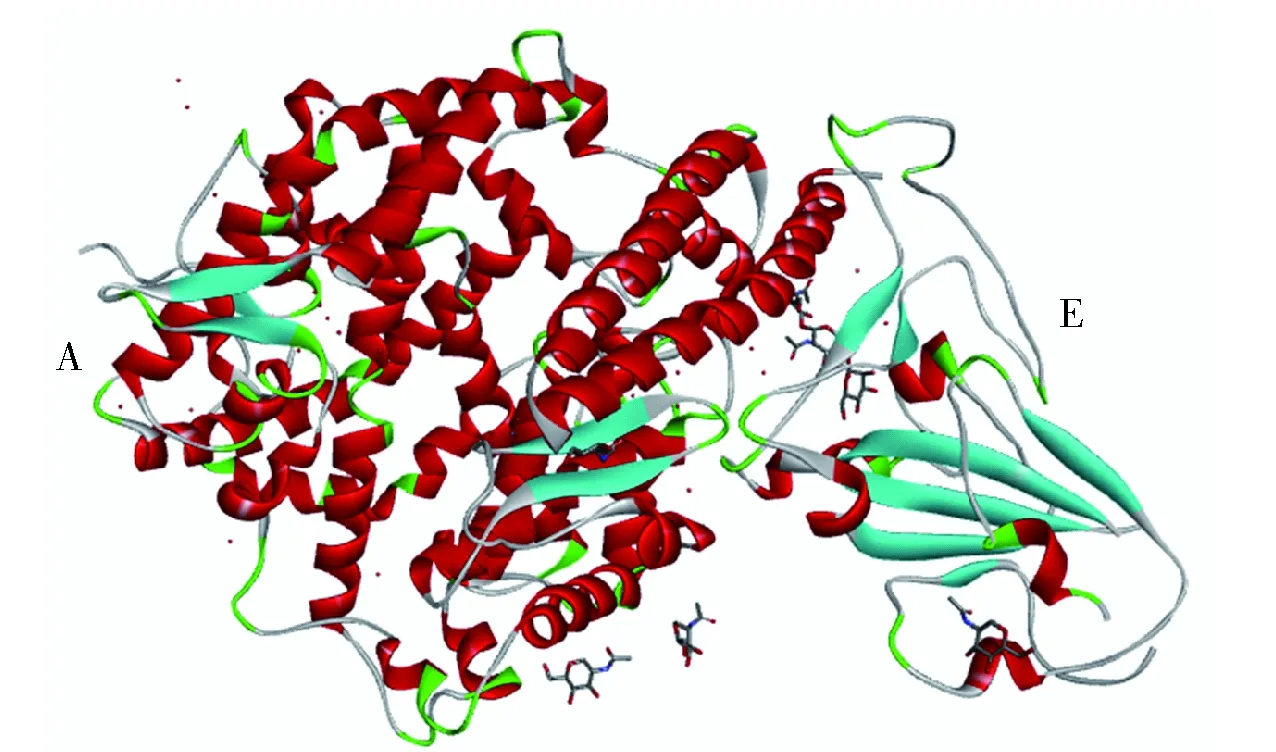
图1 SARS-CoV-S/ACE2复合蛋白A链和E链的 三维结构Fig.1 3D structure of SARS-CoV-S/ACE2 complex protein A and E chain
2.3 COVID-19 Mpro 水解酶蛋白受体与肽段的分子对接
选用的COVID-19 Mpro 水解酶高分辨率晶体结构如图3,根据已知关键氨基酸定义的结合口袋空间坐标为-9.063 889、24.552 533、65.154 444,半径11.788 826,对接结果汇总如表5。表5显示,28条肽段虽均能进入结合口袋并与受体上的氨基酸结合,但仅有10条肽段能够与蛋白受体上的关键氨基酸形成氢键。肽段QRPR、QPGL、LPIY和SITGW都能与关键氨基酸THR24、THR25和THR26形成氢键(见图4),LibDock Score打分为154.93、131.36、168.04和150.46;与此同时除SITGW之外的3条肽段还能与LEU27产生相互作用,与受体蛋白产生强烈的结合,表现出较强的Mpro水解酶抑制活性;肽段QRPR能够与关键氨基酸形成最多的氢键,与COVID-19 Mpro构象结合能力最紧密,从而预测出其为最有潜力的COVID-19 Mpro的水解酶抑制肽。
3 结 论
本研究以SARS-CoV-2和SARS-CoV所具有的相似作用机制为基础,通过计算机模拟和分子对接筛选虾夷扇贝肌球蛋白中的小分子抗新冠病毒活性肽段。通过模拟酶解虾夷扇贝肌球蛋白,从中筛选出具有潜在活性的28条肽段进行肽段和受体蛋白Y能够通过与SARS-CoV-S/ACE2复合蛋白中的关键氨基酸形成最多相互作用而表现出较好的病毒结合阻断作用;同时肽段QRPR、QPEL、LPIY和SITGW能够起到较好的COVID-19 Mpro水解酶抑制作用,其中QRPR与COVID-19 Mpro水解酶的关键氨基酸形成的相互作用数最多,结合能力最为稳定,相应的对水解酶的抑制能力最为理想。值得一提的是,肽段LPIY除了具有SARS-CoV-S/ACE2阻断能力,也显示出不俗的COVID-19 Mpro水解酶抑制能力,这也为后期研发出有助于提高人体抗感染能力和免疫能力双重作用的抗SARS-CoV-2病毒功能食品提供了可能性。
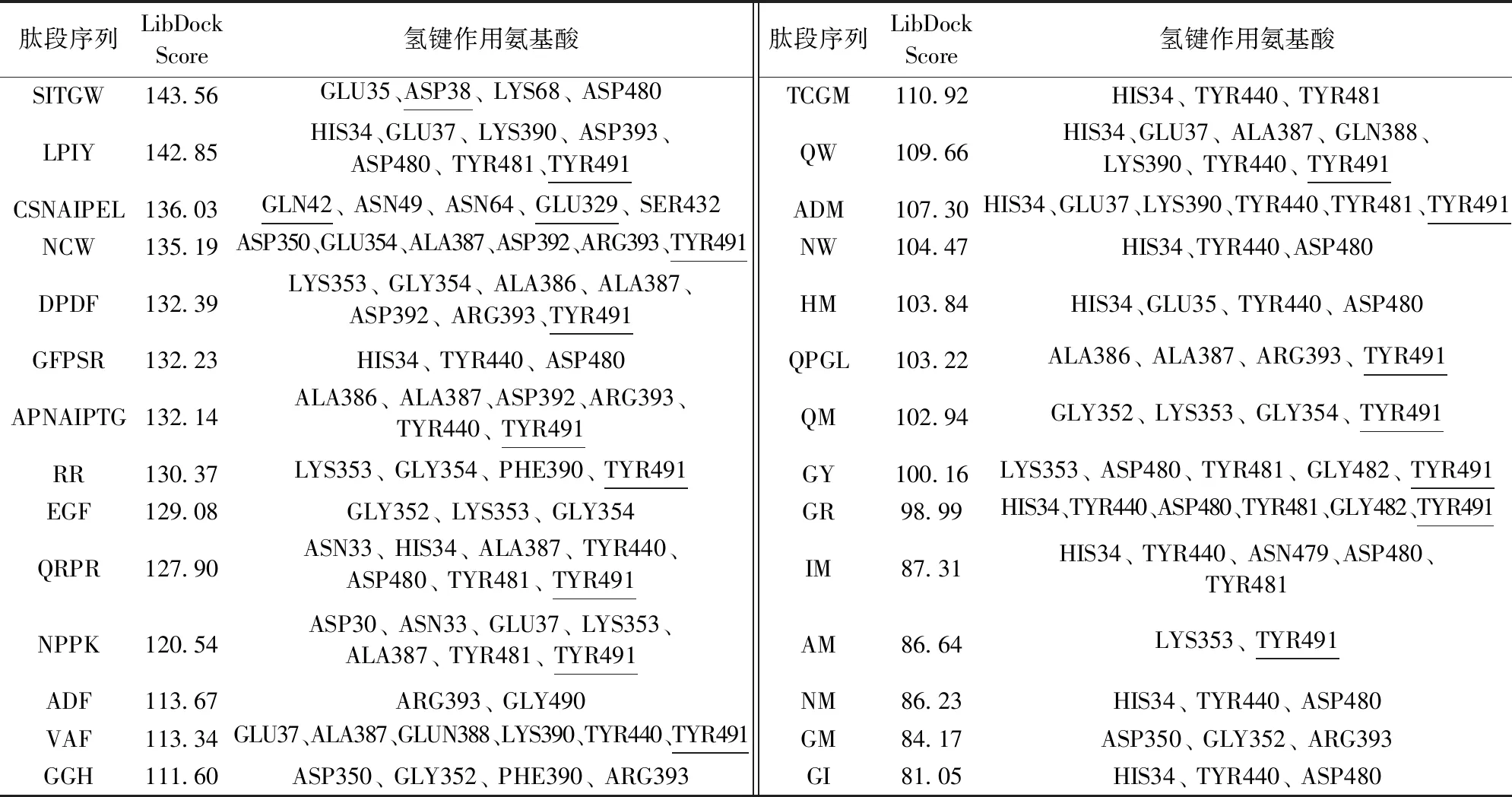
表4 28条肽段与SARS-CoV-S/ACE2复合蛋白对接结果评分与氢键作用氨基酸Tab.4 LibDock Score of docking results and hydrogen bonding amino acids of 28 peptides and SARS-CoV-S/ACE2 complex protein
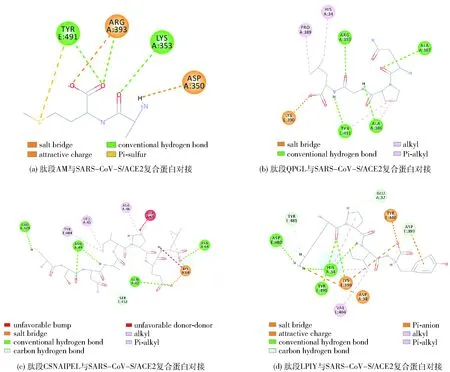
图2 活性肽段与SARS-CoV-S/ACE2复合蛋白对接2D示意图Fig.2 2D diagrams of interaction between bioactive peptides with SARS-CoV-S/ACE2 complex protein

图3 COVID-19 Mpro水解酶晶体三维结构Fig.3 3D structure of COVID-19 Mpro hydrolase

表5 28条肽段与COVID-19 Mpro 水解酶对接结果评分与氢键作用氨基酸Tab.5 LibDock Score of docking results and hydrogen bonding amino acids of 28 peptides and COVID-19 Mpro hydrolase
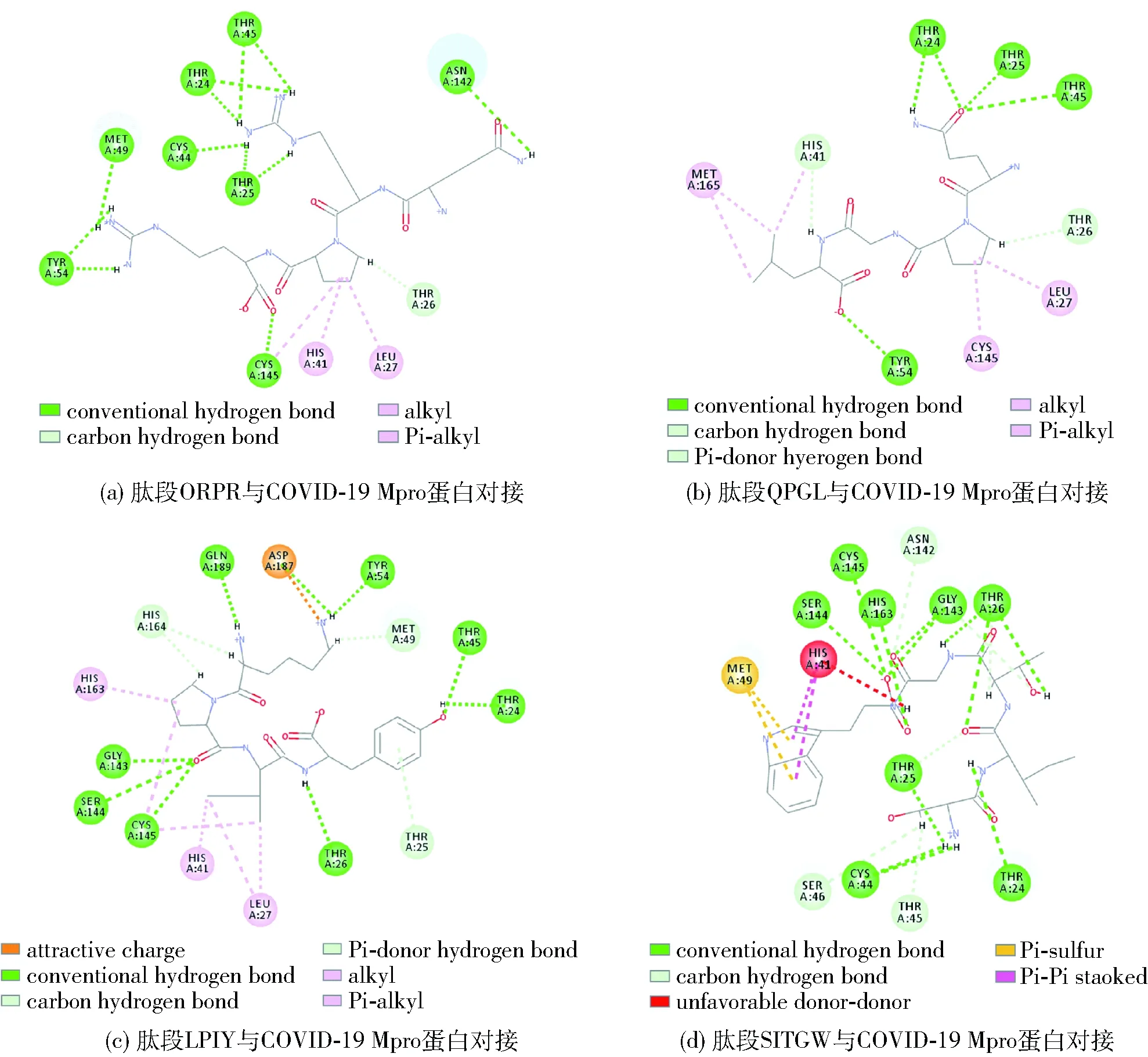
图4 活性肽段与COVID-19 Mpro蛋白对接2D示意图Fig.4 2D diagrams of interaction between bioactive peptides with COVID-19 Mpro protein

