40 株尖镰孢菌SRAP 遗传多样性分析
李新凤,姜晓东,郝晓娟,张祖维,冯铸,王建明*
(1.山西农业大学 植物保护学院,山西 太谷030801;2.山西农业大学 动物科技学院,山西 太谷030801;3.山西省农业种子总站,山西 太原030006)
尖镰孢菌(Fusarium oxysporum)属有有丝分裂孢子真菌,侵染多种农作物造成系统性病害发生,严重影响作物的产量和品质。该病原菌种内生理分化十分明显、致病性变异快,从分子水平研究病原尖镰孢菌的分类鉴定、遗传分化、群体遗传结构等,对深入开展病原镰孢菌的致病机制、抗病品种选育及尖镰孢菌病害的综合防治具有重要的理论与实践意义。
相关序列扩增多态性(Sequence-related am⁃plified polymorphism,SRAP)是由Li 和Quiros 基于PCR 技术开发的一种新型分子标记技术[1]。上游引物特异扩增目的基因序列的外显子区域,下游引物特异扩增目的基因序列的启动子和内含子区域。由于不同个体间内含子、间隔区长度的不同,扩增产物表现出多态性。该分子标记技术具有简便、多态性高、可靠、引物通用、中等产率、成本低廉、显示大量共显性标记、易得到选择条带序列、阅读目标是可读框等优点[2],已广泛应用于植物遗传学研究[3],真菌SRAP 分子标记相关研究应用最广的是食用菌[4],有关植物病原真菌的SRAP分析还相对较少,Li Z、杨潇湘、陈碧云等用SRAP技术研究来自油菜、太阳花、莴苣等作物上的核盘菌的遗传多样性,揭示了核盘菌丰富的遗传多态性[5~7];雷雨等研究四川西南部小麦白粉菌群体毒性及遗传多样性,发现SRAP 标记解释的群体遗传多样性与地理距离、毒性多样性间的相关性未达到显著水平[8];白玲玲与Il̇knur P 及王辉等分别进行了黄瓜霜霉病菌与辣椒疫霉病菌的SRAP 遗传多样性分析[9~11];Pasquali M、Liping X、Dinolfo M I 等分别用SRAP 技术研究了锈菌与丝黑穗病菌及梨孢镰孢菌的遗传多样性[12~14];刘志恒等建立了水稻茄丝核菌SRAP 遗传多样性分析体系[15],这些研究结果表明,SRAP 分子标记技术可检测到供试病原菌丰富的遗传多样性,该技术适宜于供试植物病原真菌种内遗传遗传多样性分析。
有关尖镰孢菌SRAP 分子标记方面的研究还很少,国内刘丽芳与田叶韩等分别利用SRAP 技术研究了不同地区西瓜枯萎病菌和瓜类枯萎病菌的遗传多样性,结果表明,供试尖孢镰刀菌的遗传变异丰富,同一寄主的尖镰孢菌聚在同一SRAP类群分支上[16,17]。有关山西省不同寄主尖镰孢菌的SRAP 遗传多样性分析还未见相关报道。本研究采用SRAP 分子标记技术对山西省不同寄主、地理来源的的38 株与河北的2 株尖镰孢菌进行遗传多样性分析,以期明确各菌株间的遗传关系,为尖镰孢菌分类鉴定、病害早期监测、抗病育种及病害综合防治提供理论依据。
1 材料与方法
1.1 试验材料
1.1.1 供试菌株
山西省不同地区与河北保定不同寄主植物上采集,并分离鉴定40 株尖镰孢菌,所有菌株均为单孢分离物,且经致病性测定为致病菌。菌株的寄主和地理来源信息见表1。
1.2 试验方法
1.2.1 供试菌株培养与菌株DNA 提取
供试单孢菌株培养采用李新凤的方法[18],用SDS-CTAB 法抽提供试菌株的基因组DNA,具体步骤参考Lodhi M A 的方法[19]。
1.2.2 供试菌株的SRAP−PCR 扩增
从Li、Budak 和Ferriol 等的标准引物[1,20,21]中,随机选出上、下游引物各5 条,送至上海生工生物工程有限公司合成,引物序列见表2。随机选取5株供试菌株基因组DNA 为模板,利用上下游引物自由组合成的25 对引物对其进行PCR 扩增,筛选条带清晰,多态性丰富,且重复好的引物对,用于40 株供 试菌株DNA 的PCR 扩增。
SRAP-PCR 扩增反应体系:20 uL 扩增反应体系 包 含 上 下 游 引 物 各0.5 μmol·L-1,20 ng 模 板DNA,1.0 UTaqDNA 聚合酶,1.5 mmol·L-1MgCl,0.2 mmol·L-14×dNTP。
SRAP-PCR 扩增程序:PCR 扩增程序采用李新凤的方法[22]。
PCR 反应结束后,用2.0%的琼脂糖凝胶,在80 V 电压下,电泳分离PCR 扩增产物,并用凝胶成像分析系统采集记录分离图像。
1.3 数据分析
电泳结束后,检测、记录扩增结果。将在同一迁移率位置上,有条带记为1,无条带为记0,得到0/1 矩阵。利用POPGENE Version 1.32 软件计算各遗传多样性参数,对供试菌株进行遗传多样性分析。利用NTSYS-pc Version 2.10s 软件对供试菌株进行聚类分析,用Similarity SAHN 模块基于非加权法计算菌株间的遗传相似系数,生成聚类图,为验证聚类树能否有效反应供试菌株间相似 性,采 用UltrametricDis 方 法,用COPH 组 件 把目录树式数据转换成隐式距离数据,经Mantel 检测,计算协表征矩阵,用MXCOMP 模块对生成的遗传相似性矩阵和协表征矩阵进行相关性检验,最后生成Tree Plot 聚类图。然后对供试菌株进行主成分(PCA)分析,利用Output&transf Dcenter 模块进行中心化后,在Ordination 模块中利用Eigen计算特征向量和特征值,最后生成PCA 聚类图。
2 结果与分析
2.1 引物筛选与供试菌株的遗传多样性分析
用筛选出的12 对条带清晰,多态性丰富的引物,用于供试菌株的SRAP 遗传多样性分析,40 株尖镰孢菌共扩增125 个条带,引物扩增条带数分布在4~24 条之间,平均每对引物产生10.4 条条带,且均为多态性条带。其中,扩增条带数最多的引物为F1-R4,扩增的条带数最少的引物为F1-R5。所有扩增条带均分布于100~1500 bp 之间(见表3)。由图1 可以看出,引物F4-R5 的所有扩增条带均显示多态性,具有很好的扩增效果。12 个引物组合的多态性信息含量介于0.145~0.443 之间,平均为0.273,多态性信息含量最高的是引物组合F3-R5 与F4-R5,最低的是F5-R5。供试12 对引物中,有7 对引物PIC 值大于0.25,表现出较好的扩增多态性。

表1 供试菌株及其寄主与地理来源Table 1 Tested strains and their hosts and sampling locations in the study
遗传多样性分析结果表明,40 株尖镰孢菌的平均有效等位基因数(Ne)为1.280 3,介于1.051 2~2.000 之间。平均Nei' s 基因多样性指 数(H)为0.187 6,最大值为0.5,最小值为0.048 8。Shannon 信息指数(I)变幅为0.116 9~0.693 1,平均0.313 9。由此可以看出,供试尖镰孢菌遗传变异程度及遗传差异较大,遗传多样性丰富。
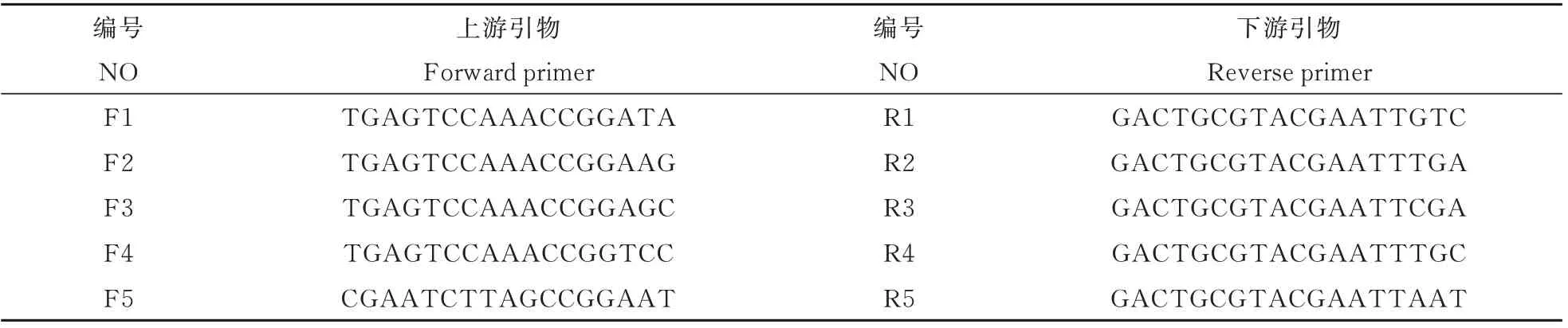
表2 引物序列Table 2 SRAP primers sequences
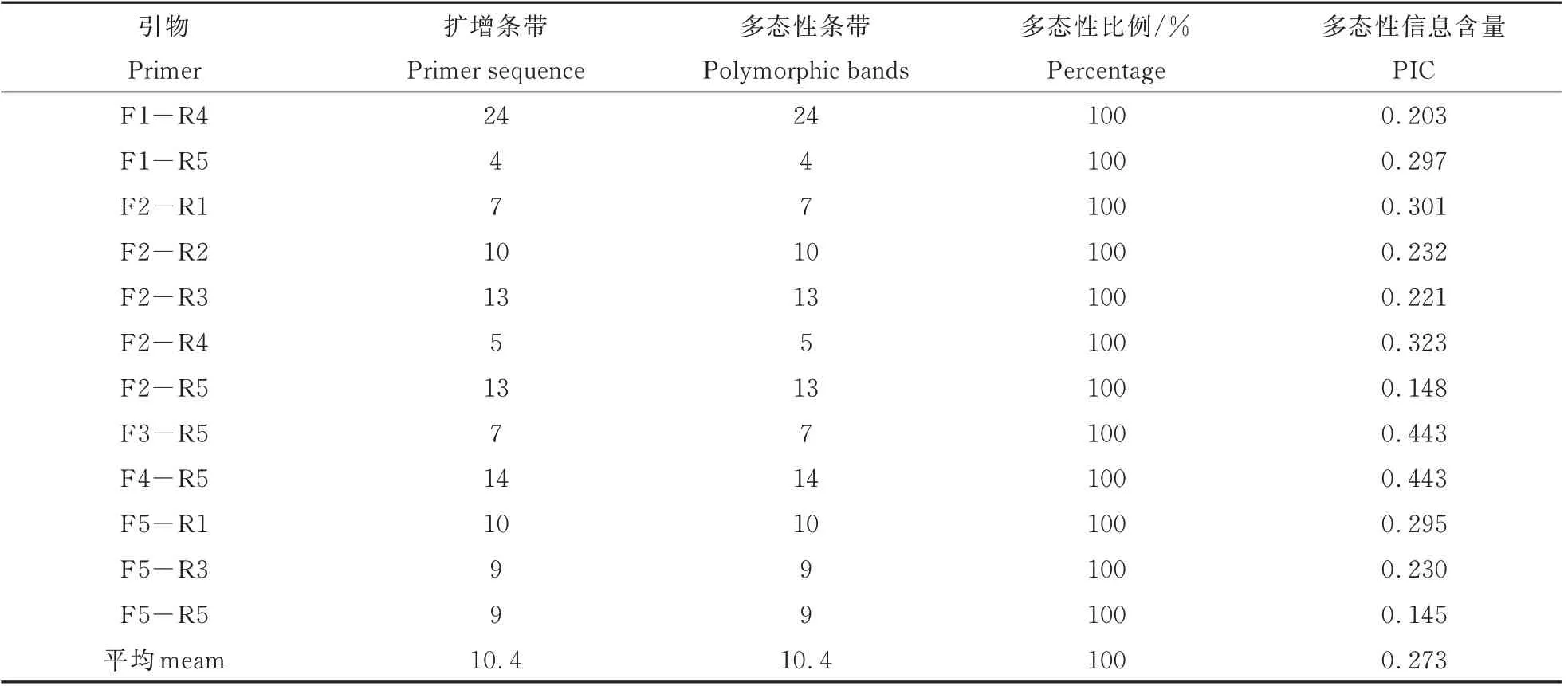
表3 12 对多态性引物对40 株尖镰孢菌的扩增结果Table 3 The amplification results of 12 SRAP primers from 40 strains of F.oxysporum
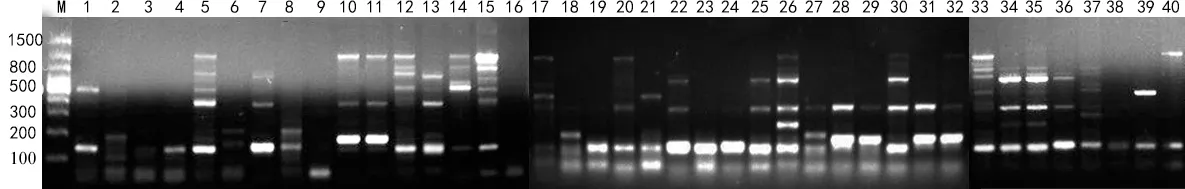
图1 引物F4-R5 对40 株尖镰孢菌的SRAP 扩增结果Fig.1 SRAP amplification results of 40 strains of F.oxysporum with primer F4-R5
2.2 不同地理来源尖镰孢菌遗传多样性分析
根据不同地理来源,将供试40 株菌划分为晋东南、晋中、晋北、河北四个地理居群。利用POP⁃GENE 1.32 软件对其进行遗传多样性分析,结果由表4 可知,4 个地理群体的多态位点数在11~106之间,平均为57.25;多态位点百分率在8.8%~84.8% 之间,平均为45.80%;有效等位基因数(Ne)变 幅 为1.283 2~1.088,平 均 为1.222 8;Nei's 基因多样性指数(H)分布在0.172 到0.044之间,平均为0.135 5;Shannon's 指数(I)在0.287 6到0.061 之间,平均为0.209。其中晋中地区菌株的Nei's 基因多样性指数和Shannon's 指数均最高,其遗传分化水平最高,多样性最丰富。晋北地区和晋东南地区菌株相差不大。

表4 4 个地理居群的遗传信息Table 4 Genetic information of four populations
各地理群体间的遗传分化参数分析显示,4 个地理居群总的遗传多样度(Ht)为0.1965,居群内遗传多样度(Hs)为0.135 4,说明地理居群内菌株间亲缘关系较近。群体间遗传分化系数(Gst)为0.310 6,基因流(Nm)为1.109 8,群体间的遗传变异占到31.06%,68.94%的遗传变异存在于地理居群内部,说明4 个尖镰孢菌地理居群间存在一定的遗传分化,但亲缘关系较近,不同群体的菌株间存在较大的基因流动,且群体内遗传多样性大于群体间遗传多样性。
2.3 不同地理来源尖镰孢菌的遗传一致度和遗传距离分析
利用Popgene Version 1.32 计算不同地理来源尖镰孢菌群体间的遗传距离和遗传相似系数(Nei’s,1978),从表5 可以看出,4 个地理居群间的遗传相似系数分布在0.8510 -0.9711 之间,其中晋东南群体与晋中群体间的遗传相似系数最高,为0.9711,遗传距离最近;而晋东南菌群和河北群体间的遗传相似系数最低,为0.8510,遗传距离最远。山西省内3 个居群间的遗传相似系数均高于0.94,明显高于其与省外居群间的相似性,省内晋东南、晋中、晋北居群与河北居群间的遗传相似系数 分 别 为0.8510、0.8695 和0.8596。4 个 地 理 居群的聚类图(图2)显示,山西省3 个群体聚在一个类群,河北群体另外分为一支,山西省3 个菌群中,晋东南菌株与晋中菌群先聚在一起,然后再和晋北菌群聚在一起。
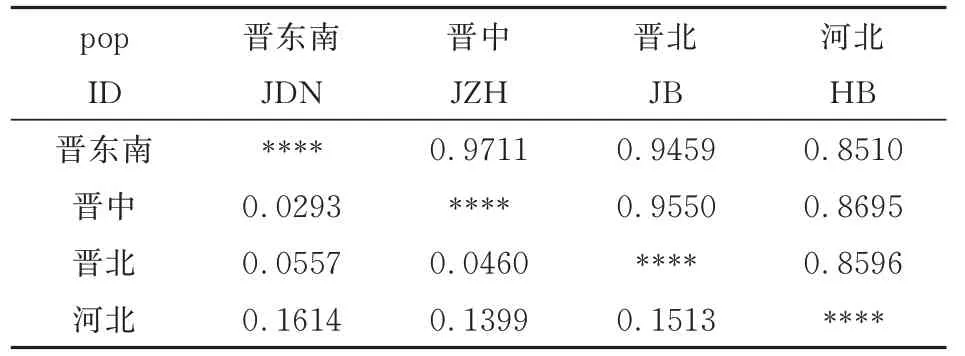
表5 尖镰孢菌不同地理居群的遗传距离和遗传相似系数Table5 Genetic distances and similarity of different geo⁃graphic populations of F.oxysporum

图2 基于Nei's(1972)遗传距离的尖镰孢菌地理居群聚类图Fig.2 Dendrogram of Fusarium oxysporum geographic populations Based on Nei's(1972)Genetic distance
2.4 晋中地区不同寄主来源尖镰孢菌遗传多样性分析
晋中地区4 个寄主群体的遗传多样性分析结果(表6)表明,群体间共有106 个多态性位点,多态性位点比例为84.80%。Nei's 基因多样性指数和Shannon's 指数均以葫芦科寄主群体最高,豆科寄主群体最低。4 个寄主群体的平均Nei's 基因多样性指数和Shannon's 指数分别为0.137 5 和0.207 2。四个寄主群体总基因多样度为0.190 1,种群内基因多样度为0.137 5,种群间遗传分化系数为0.277 0,基因流为1.305 3,说明遗传分化主要发生在不同寄主群体之间。
2.5 供试尖镰孢菌遗传相似性与聚类分析
Mantel 检测结果表明,供试菌株的聚类树矩阵和协方差特征矩阵相关,相关系数为0.855 74,说明该聚类树可以很好地代表供试菌株的原始距离矩阵。供试菌株两两之间的平均遗传相似系数为0.81,分离自晋中太谷黄瓜的14 号菌株与分离自晋北大同番茄的22 号菌株的遗传相似系数(0.64)最低,分离自晋中祁县绿豆的3 号菌株和晋中太谷6 号菌株间的遗传相似系数最高,为0.99。聚类分析结果如图2 所示,由图2 可知,当遗传相似系数为0.228 时,供试的40 个尖镰孢菌可以明显的划分为8 个SRAP 类群(SG)。SG1 包括2、3、6、9、8 共5 个菌株,均分离自晋中地区豆科作物;SG2 和SG3 类群各包含1 个菌株,分别为分离晋北和晋中茄子的37 号和16 号菌株;分离自晋中西瓜的18、19 号菌株和分离自晋北五寨茄子的25 号菌株构成SG4 类群;SG5 包含分离自晋中地区黄瓜和番茄的11 号和25 号菌株;类群SG6 只有分离自晋中地区黄瓜的14 号菌株;SG8 类群包含来自豆科作物的1、4、5、7 号菌株和来自葫芦科的10、11、12、13 及15 号菌株;剩余所有菌株均聚在SG7类群。在更高遗传相似系数水平上,除单个菌株构成的SG 类群外,其它类群又可以进一步划分出不同的亚群。当相似系数达到0.750 时,40 株供试尖镰孢菌被全部分开。
由聚类图可以看出,同一地理来源或同一寄主来源的菌株分散在不同SG 类群中;同一SG 类群中又包含不同地理来源、不同寄主的菌株。此外,来自同一地区且寄主相同的菌株也会分散在不同SG 类群中,如,7 号、8 号、12 号、21 号、38 号和39 号菌株均分离自晋中地区太谷县,其中7 号菌株(菜豆)和12 号菌株(黄瓜)聚在SG8 类群,而8 号菌株(菜豆)聚在SG1 类群。分离自番茄的39 号菌株与分离自西瓜的38 号菌株聚在SG7,同是分离自番茄的21 号菌株却分散在SG5 类群中。说明SRAP 类群的划分与菌株地理和寄主来源均没有明显相关性。
2.6 供试尖镰孢菌主成分分析(PCA)
根据供试菌株间的遗传相似系数,利用Nt⁃sys2.10s 软件进行主成分分析,结果表明,共有8个主成分特征根大于1,前3 个主成分的方差贡献率分别为81.380 7%、3.083 0%、和2.044 1%,累积贡献率为86.507 7%,因此,它们可以作为主成分反映各供试菌株的遗传多态性信息。由第一、二、三主成分生成的三维空间聚类图(图4)可以看出,菌株2、3、6、9 聚在PS1-1 群体,然后又和菌株8聚为群体PS1;PS2 包含18、19 和25 三个菌株;群体PS3 有17 和21 两个菌株;菌株5、7、10、11、12、13 聚在PS4-1,然后又和菌株1、4 聚为群体PS4;PS5 包 括 除 菌 株14、16、37 和29 之 外 的 剩 余18 个菌株。菌株14、16、37 和29 与其他菌株间的空间距离相对比较远,分散在五个PS 群体之间。这一结果与聚类分析结果基本一致,供试菌株间的空间位置越近表示亲缘关系越密切,越远表示其亲缘关系越疏远。
3 结论与讨论
本试验利用筛选出的12 对SRAP 多态性引物对40 株尖镰孢菌进行遗传多样性分析,共扩增125 条谱带,多态性比例达100%,平均每对引物扩增10.4 条。供试菌株平均Nei' s 基因多样性指数(H)为0.187 6,平均Shannon 信息指数为0.313 9。菌株间的遗传相似系数介于0.64~0.99 之间。说明供试菌株间遗传分化明显。刘丽芳、田叶韩等研究西瓜枯萎病菌及瓜类的SRAP 遗传多样性得出与本研究相同的结果[16,17]。说明尖镰孢菌种内SRAP 遗传多样性丰富,存在明显的遗传分化,SRAP 分子标记技术可以用于检测尖镰孢菌种内的遗传多样性。
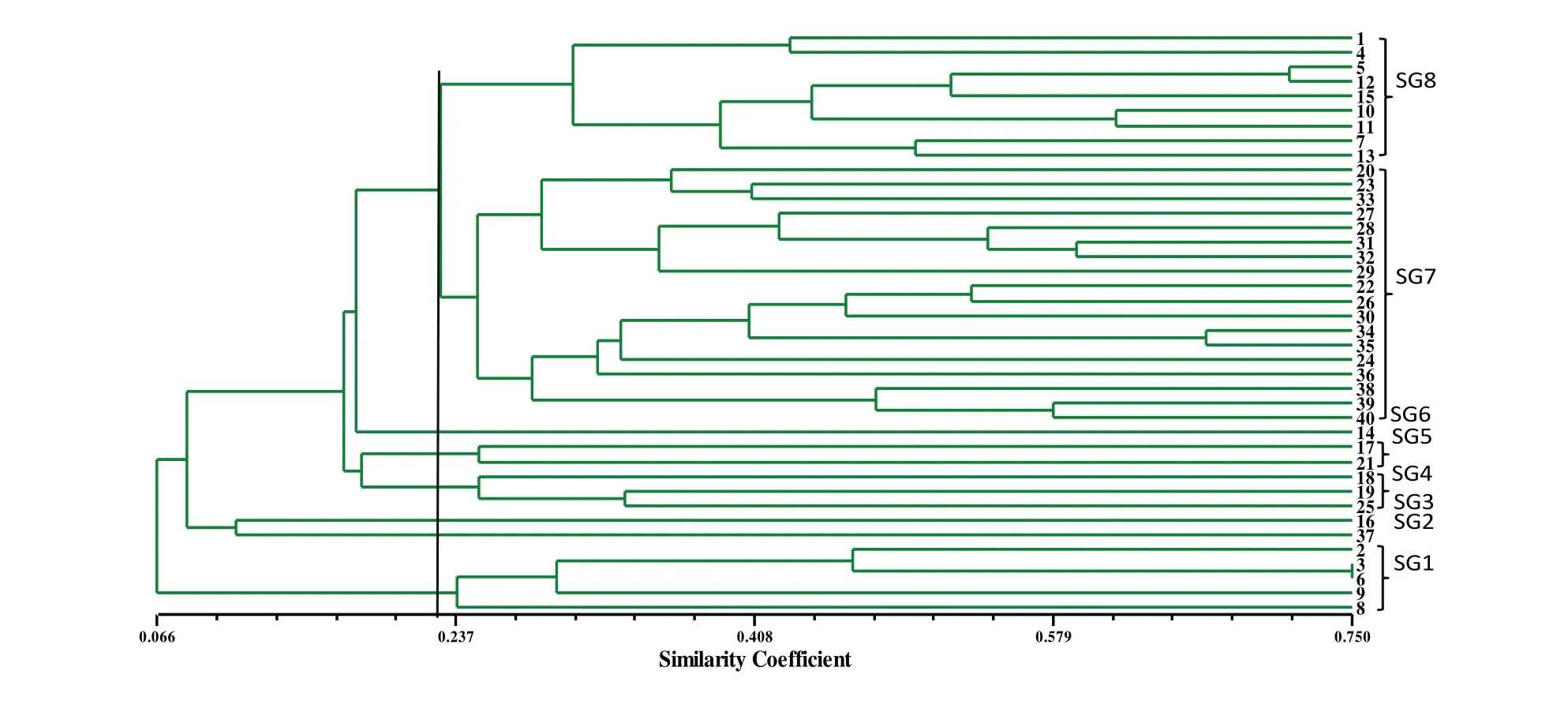
图3 40 株尖镰孢菌的SRAP 聚类图Fig.3 Dendrograms of 40 F.oxysporum strains with SRAP-PCR

图4 基于SRAP 的40 株尖镰孢菌主成分分析Fig.4 Principal component analysis of F.oxysporum strains based on SRAP
目前可用于尖镰孢菌遗传多样性分析的分子标记技术有多种,如AFLP、RAPD、ISSR、SSR等,每种分子标记技术所扩增菌种DNA 区域与位点不同,前期我们用18 对引物对除本试验中2 和3号菌株外的38 株尖镰孢菌中进行EST-SSR 扩增,共检测到75 条多态性条带,多态性比率达92.6%,平均每对引物可扩增4.2 条,遗传相似系数 介 于0.565 ~0.946 之 间,平 均 为0.721[18]。同时还利用ISSR 分子标记技术对本试验菌株中的33 株进行了遗传多样性分析,共扩增出105 条条带,其中86.7%的条带(91 条)呈现多态性,平均每条引物扩增8.3 条,供试菌株间的遗传相似系数为0.606~0.962,平均0.756[23]。与本试验中尖镰孢菌SRAP 遗传多样性分析结果相比,EST-SSR、ISSR 标记扩增多态性位点百分率及单对/条引物扩增多态性条带数均低于SRAP 标记;菌株间的遗传相似系数跨度以EST-SSR 最大,另外两种标记相差不大;检测得到的平均遗传相似系数也以SRAP 标记技术最大,其次是ISSR,EST-SSR 最小。说明SRAP 标记技术较其他两种能检测更多、更高比例的多态性位点,3 种标记技术中,SRAP 所反应菌株间亲缘关系也最近。这可能与该标记技术扩增尖镰孢菌DNA 序列开放阅读框,最终翻译成蛋白的基因核苷酸序列,能更多地反映供试菌株表型性状有关。此外,SRAP 标记综合了RAPD 标记的简便性和AFLP 标记的稳定性,及两者共有的高多态性于一身[20],可随机搭配上下游通用引物使用,可以极大地提高引物的利用效率。因此,SRAP 技术是一种进行真菌种内遗传多样性评价的有效工具。
地理居群间遗传距离与地理距离具有一定的相关性,省内3 个居群间的遗传距离较其与河北居群间的遗传距离近,遗传相似性也较高,一定程度上体现了土传病原真菌群体存在相对地理隔离的特性。各地理群体间遗传分化系数(Gst)为0.310 6,群体内遗传分化系数为0.689 4,4 个居群间存在一定的遗传分化,且群体内遗传多样性高于群体间的遗传多样性。基因流(Nm)反应基因在群体间的运动情况,Nm<1,表示群体被强烈分化,Nm>1 时,证明群体间存在一定基因流动,发挥匀质化用[24]。4 个地理群体基因流Nm = 1.1098,表明群体间存在一定程度基因交流,基因流是导致病菌群体间遗传结构分化的主要原因之一,因此加强山西省各地区病原尖镰孢菌群体遗传变异的监测,对选育和利用抗病品种具有重要的指导意义。
聚类分析与主成分分析结果表明,基于供试尖镰孢菌SRAP 区域DNA 序列扩增结果划分的类群(SRAP 类群)与菌株地理来源和寄主均没有明显相关性。刘丽芳等[16]认为30 株供试西瓜枯萎病菌的聚集与地理来源没有明显的相关性,和本试验所得结果一致,而田叶韩[17]对95 株瓜类枯萎病菌进行SRAP 遗传多样性分析得出结果与本研究结果不同。这可能是供试菌株DNA 序列的真实反映,也可能与本试验供试菌株数量有限,试验结果还存在一定的局限性。有关尖镰孢菌SRAP类群划分与地理和寄主来源的关系,还有待于扩大样本数进一步研究证实。此外,尖镰孢菌与寄主作物在长期互作进化过程中,存在遗传变异快,遗传分化明显的现象,但是否存在部分菌株,趋向于侵染多种寄主,对寄主选择专性较差的现象,还有待于进一步研究。4 个地理群体中,晋中地区菌株Nei's 基因多样性指数和Shannon 信息指数最高,该地区菌株遗传多样性最高,遗传分化最明显,也可能与本试验选取该地区菌株数相对较多有关,还有待于扩大各地区试验样本数进一步验证。

