水稻包穗突变体sui2的遗传分析和基因精细定位
孙 琦 赵志超 张瑾晖 张 锋 程治军,* 邹德堂
水稻包穗突变体的遗传分析和基因精细定位
孙 琦1,**赵志超2,**张瑾晖2张 锋2程治军2,*邹德堂1,*
1东北农业大学寒地粮食作物种质创新与生理生态教育部重点实验室, 黑龙江哈尔滨 150030;2中国农业科学院作物科学研究所, 北京 100081
水稻穗下倒一节间的伸长和发育对株型的形成起着重要作用, 在水稻不育系中常见的包穗现象是由于穗下倒一节间的伸长和发育受阻造成的, 深入研究水稻包穗分子机制能为水稻不育系株型的改良提供一定的理论意义。我们在粳稻Kitaake的组织培养后代中获得了1个包穗突变体, 其穗部被剑叶叶鞘包裹程度处于半包裹和全包裹之间, 倒一节间严重缩短细胞学形态分析表明, 倒一节间的缩短是由于节间薄壁细胞的伸长生长不足造成的。通过对与IRAT129杂交后代分析表明,为单基因显性突变。对该组合F2的608个正常单株遗传定位分析结果表明, 候选基因被精细定位在4号染色体长臂端, 由InDel标记S4-14.1和S4-14.2界定的110 kb的区域内。该区间内的编码基因在基因组序列水平上无差异, 而基因LOC_Os04g39430表达量在突变体中升高了264倍, 该基因编码1个参与油菜素内酯(brassinolide, BR)合成的细胞色素P450蛋白, 是的等位基因。qRT-PCR分析结果表明, 突变体中BR信号途径的基因表达量增高, 预示着BR信号途径的基因可能参与了穗下倒一节间的伸长和发育的调控。
水稻; 包穗; 精细定位;; BR信号途径的基因
杂交水稻不育系普遍存在包穗现象, 主要表现在穗颈节缩短、穗部约30%~60%颍花包裹在剑叶叶鞘内而无法伸出[1]。在大田生产实际中, 一般通过外部喷施一定浓度赤霉素(GA3)的方法来解除包穗现象[2]。但喷施GA3不仅在一定程度上增加了制种成本、降低了杂交种质量, 而且其喷施效果在很大程度上依赖喷施者的相关技术、天气情况及施用时期等[3-4]。因此利用包穗突变体, 克隆包穗相关基因, 研究包穗形成的分子生物学机制, 对于降低制种成本, 提高杂交种产量和质量具有重要的理论指导意义。
水稻倒一节间的缩短导致了包穗现象, 该性状不仅受主效基因的控制, 而且同时受微效基因的影响。至今12个控制倒一节间长度的QTL已被检测到, 并且通过自然变异、理化诱变、组织培养和转基因等技术获得了11种包穗突变体, 这些突变体包括、、、、、、、、、和[5-17]。根据穗部被包裹的程度可分为全包穗和部分包穗。、、、、、和为部分包穗的突变体,和为全包穗的突变体其中各节间均表现出不同程度的缩短, 而,和的节间缩短仅特异地发生在倒一节间。和均已经被克隆, 且为同一个基因[10,15],和分别定位在4号染色体上和2号染色体上[14,16]。除此之外, 有关水稻包穗突变体相关基因的研究报道极少。因此, 通过突变体挖掘新的包穗基因, 为深入探究水稻倒一节间生长发育的分子机制奠定一定的理论基础。
Zhu等[11]报道了2个突变体和, 其倒一节间发育受阻。Ma等[15]也发现了倒一节间缩短的突变体, 并命名为本实验的突变体材料表型和前人报道突变体表型相似, 因此我们将突变体命名为。突变体在表型上属于1个半包穗和全包穗之间的突变体, 表现出倒一节间缩短, 遗传上表现为显性。通过与正常抽穗品种IRAT129杂交, 构建了对该突变位点的遗传作图群体, 利用图位克隆的方法, 我们最终获得了目标基因。初步结果表明, 该基因表达量增加导致了包穗的表型, 该突变体为深入研究BR (brassinolide)信号通路基因控制水稻包穗表型提供了材料。
1 材料与方法
1.1 供试材料
水稻包穗突变体来源于日本粳稻品种Kitaake的组织培养后代。以粳稻品种IRAT129为父本, 以突变体为母本进行杂交, 杂交种种于中国农业科学院作物科学研究所顺义试验基地。同年, 将F2分离群体种植于海南省三亚实验基地, 常规水肥管理。成熟时, 田间随机选取野生型Kitaake和突变体各10株, 考察株高、分蘖数、倒一节间长度、粒长、粒宽、粒厚、每穗粒数、千粒重、结实率等农艺性状, 进行数据分析。
1.2 倒一节间细胞学观察
抽穗后取Kitaake和突变体的倒一节间, 徒手用刀片切3 mm左右的节间组织, 放置于50%甲醇和10%乙酸混合固定液中, 4℃固定12 h。固定后, 转移至80%乙醇中, 80℃孵育5 min, 再移至固定液固定1 h, 用水洗净后置于1%高碘酸中, 室温静置40 min。洗净后置于100 μg mL–1碘化丙啶希夫试剂中染色1~2 h, 再置于水合氯醛溶液过夜透明。用激光共聚焦显微镜(ZEISS Microsystems LSM 700)观察并照相。
1.3 包穗突变基因的精细定位与候选基因分析
在田间分别取样2个亲本IRAT129、及总共608个F2分离群体中隐性(正常表型)单株的叶片, 进行突变基因精细定位。叶片全基因组DNA的提取采用改进的CTAB法。首先, 随机选取取样的F2单株9株与2个亲本的DNA一起构建混池, 利用均匀分布于12条染色体上, 且在双亲间有多态性的186个分子标记进行基因连锁分析。随后, 再随机选取66个隐性单株验证连锁的分子标记, 进行初定位。最后, 在初定位的基础上, 用新开发的多态性InDel标记对全部608个隐性单株基因型进行精细定位。新标记开发参考网站Gramene (http://ensembl.gramene.org/genome_browser/index.html)上籼稻和粳稻日本晴的基因组序列, 在初定位区间内进行序列对比寻找插入/缺失位点, 用Premier3在线软件(http: //primer3.ut.ee/)设计InDel分子标记, 委托上海英潍捷基贸易有限公司合成(表1)。采用北京宝日医生物技术有限公司的Premix进行PCR扩增, 产物经8%非变性聚丙烯酰胺凝胶电泳分离, 染色显色后观察。利用网站(http://rice.plantbiology.msu.edu/cgi-bin/ gbrowse/rice/)确定区间内ORF, 利用南京诺唯赞生物技术有限公司的Phanta Max Super-Fidelity DNA Polymerase扩增, 委托北京博迈德基因技术有限公司测序。

表1 新开发的多态性InDel和SNP引物
1.4 候选基因及油菜素内酯相关基因表达量分析
成熟期分别取野生型和突变体的倒一节间基部组织。采用TIANGEN公司的RNA试剂盒提取植物组织总RNA, FastKing cDNA试剂盒反转录合成cDNA。采用北京宝日医生物技术有限公司的TB Green Premix ExII试剂盒进行实时荧光定量PCR, 在Applied Biosystems 7500 Real-time PCR仪器上进行扩增反应, 评价油菜素内酯合成及信号相关基因(引物表2)在野生型和突变体倒一节间基部组织中的表达情况, 内参基因为(NCBI Locus: XM_020816636), 每个样品3个重复。
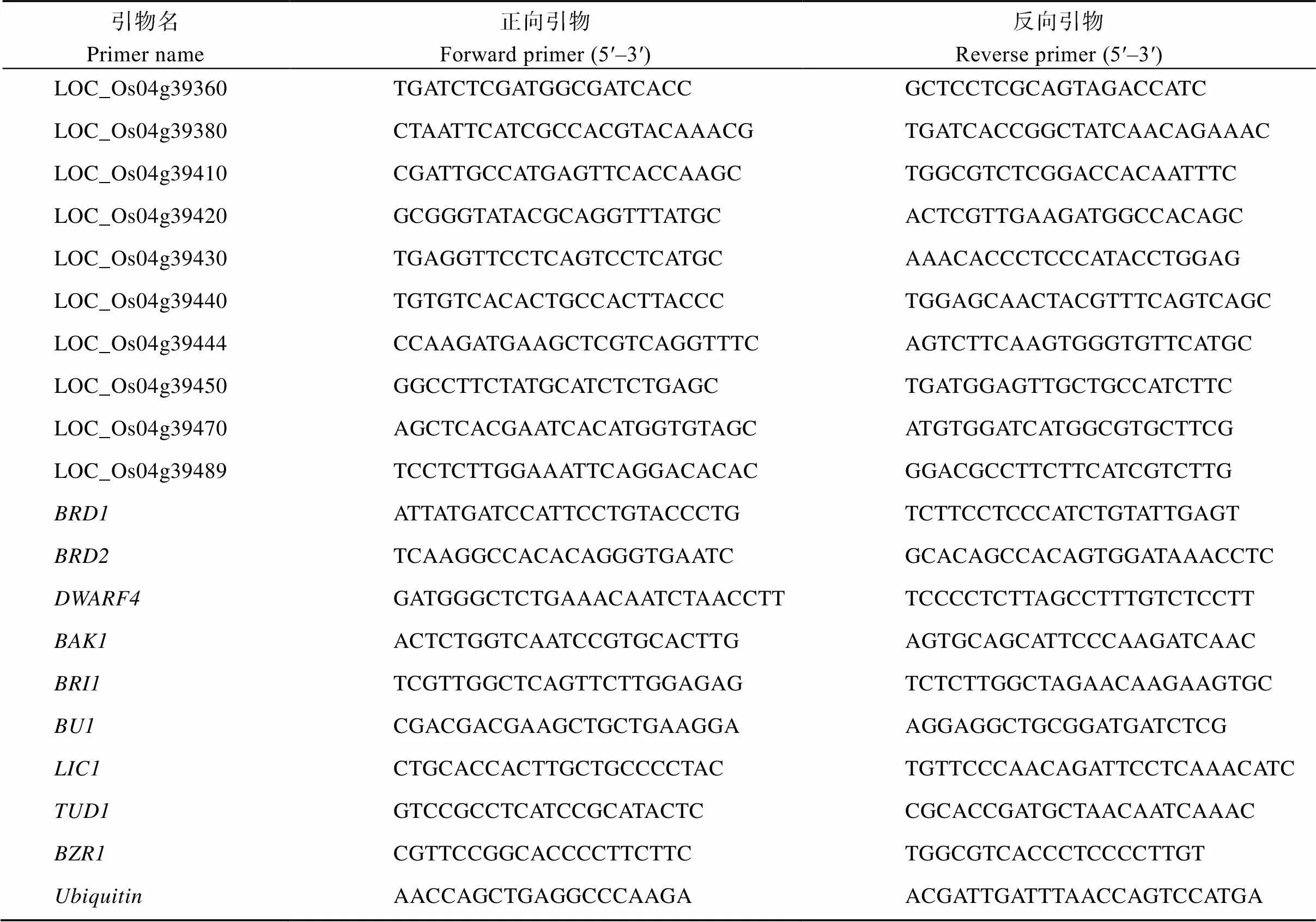
表2 qRT-PCR所用引物
2 结果与分析
2.1 包穗突变体sui2的表型分析
在成熟期, 突变体的穗部被剑叶叶鞘包裹, 其包裹的程度介于半包裹的和全包裹之间(图1-A, B)。的包穗表型是由于节间的伸长与叶鞘的伸长不同步造成的, 在突变体中, 除倒三、倒四节间没有表现出明显的差异外, 倒一、倒二节间长度都相应缩短, 尤其是倒一节间的长度仅为对照Kitaake的50% (图1-C, 表3)。此外, 突变体还表现植株矮化、分蘖数增加、每穗粒数减少、结实率下降、籽粒变大和剑叶变窄(表3), 尤为特别的是,的倒一和倒二叶的夹角显著增大(图1-G, H)。

图1 野生型与突变体sui2的表型比较
A: 野生型野生型(左)与突变体(右)成熟期表型; B: 成熟期野生型(左)与突变体(右)的1个典型的包穗的表型比较; C: 成熟期野生型(左)与突变体(右)的各节间表型比较, 自左至右为倒一、二、三、四节间; D, E: 野生型(D)和突变体(E)倒一节间横切面细胞学比较; F: 野生型和突变体倒一节间纵切面细胞学比较; G, H: 野生型(G)与突变体(H)苗期倒一和倒二叶叶片夹角。标尺: (A) 15 cm; (B) 5 cm; (C) 5 cm, (D, E) 200 μm; (F) 50 μm。
A: Phenotypes of WT (left) and(right) plants at rippenperiod; B: Phenotype comparison of the sheathed panicle of WT (left) and(right) at the maturationperiod; C: Comparison of internodes between WT (left) and(right), from left to right is the uppermost internode, penultimate internode, antepenult internode, fourth internode from top down; D, E: Transverse sections of the first internode of WT (D) and(E); F: Longitudinal sections of the uppermost internode of wild type and; G, H: The leaf angles between the last and penultimate leaf in the WT (G) and(H) at the seedlingstage. Scale bars: (A) 15 cm; (B) 5 cm; (C) 5 cm; (D, E) 200 μm; (F) 50 μm.

表3 野生型Kitaake和突变体sui2农艺性状比较
*表示在< 0.05水平差异显著;**表示在< 0.01水平差异显著。
*represents significantly different at< 0.05;**represents significantly different at< 0.01.
2.2 sui2节间形态分析
突变体株型矮化, 成熟期株高约43 cm, 只有野生型的80%左右。突变体倒一节间缩短严重, 其长度仅为野生型倒一节间的50% (表3)。由于节间细胞数量减少或细胞伸长受到抑制, 造成了矮秆突变体节间缩短的表型。为了深入研究细胞形态及数目对突变体倒一节间长度的影响, 我们通过PI染色处理成熟期野生型和突变体的倒一节间并进行显微观察。结果发现, 相对于野生型, 在横截面上, 突变体薄壁细胞数目没有明显增多(图2-B), 薄壁细胞的体积变小, 茎秆壁增厚, 空气髓腔明显减小, 维管束数目减少(图1-D, E); 在纵切面上, 突变体的细胞长度明显缩短, 突变体的细胞宽度也变窄(图1-F, 图2-A)。因此, 突变体倒一节间的薄壁细胞伸长出现了障碍, 突变体表现出的矮秆及其包穗表型是细胞伸长受阻所导致。

图2 野生型和突变体节间组织薄壁细胞体积和数目比较
A: 野生型和突变体倒一节间纵切面薄壁细胞长度及宽度; B: 野生型和突变体倒一节间横截面半径含有的细胞数目。* 表示在<0.05水平差异显著; ** 表示在< 0.01水平差异显著。
A: Comparisons of the length and width of stem parenchyma cells from longitudinal sections of the uppermost internode in wild type and; B: comparison of cell numbers of the radius in the transverse sections of the uppermost internode in wild type and. * represents significantly different at< 0.05; ** represents significantly different at< 0.01.
2.3 遗传分析
对与抽穗正常的IRAT129品种的杂交组合后代分析结果表明, F1植株全部包穗, 为突变体表型。F2群体出现明显的性状分离。随机调查927个单株, 217株表现为正常表型, 710株表现为包穗表型, 经卡方(χ2)测验(χ2= 1.25, χ20.05= 3.84), 该表型符合1对显性基因3 (显性)∶1 (隐性)的分离比。
2.4 基因定位与候选基因分析
从F2群体中随机选取9个隐性(正常抽穗)单株叶片构建DNA混池。选用实验室186对均匀分布在水稻12条染色体上, 且在双亲间具有多态性InDel或SSR引物, 对DNA混池进行连锁分析。结果发现, 位于4号染色体长臂端的2个标记S4-14和S4-15与混池DNA的单株连锁。之后进一步用66个F2取样单株对这两个标记进行验证, 并确定了物理距离为0.83 Mb初定位区间(图3-A)。利用Gramene网站(http://ensembl.gramene.org/genome_browser/index. html)的籼粳稻基因组序列对比信息, 在初定位区间新开发了2个分子标记S4-14.1和S4-14.4, 利用全部F2单株将基因定位在S4-14.1与S4-14.4之间, 物理距离为287 kb的范围。进一步开发2个SNP分子标记S4-14.2和S4-14.3, 将基因最终定位在了标记S4-14.1和S4-14.2之间物理距离为110 kb区间内(图3-B)。
根据水稻基因组注释信息数据库(http://rice. plantbiology.msu.edu/cgi-bin/gbrowse/rice/), 该区间内共包含14个基因和2个转座子(表4和图3-C)。对所有14个基因的基因组测序在野生型和突变体之间均没有发现差异, 说明的突变表型并不是由于DNA水平上的差异引起的。由于基因表达量的差异也会引起植株表型的变化, 我们进一步分析了区间内14个基因在成熟期的表达水平。如图4所示, 与野生型相比, 在中有4个基因明显上调, 1个基因明显下调, 而其他候选基因的表达量在两者之间无明显差异。鉴于该突变体为显性, 我们重点关注表达量差异较大的2个基因LOC_Os04g39420 (上调9倍)和LOC_ Os04g39430 (上调264倍)。经查阅文献, LOC_ Os04g39420编码了6-磷酸果糖激酶-2 (6-phosphofructokinase-2), 可能参与水稻籽粒淀粉的理化性质形成[17]; LOC_Os04g39430编码了1个细胞色素450蛋白, 为的1个等位基因, 该基因的过表达植株的表型除了有包穗外, 其他表型也与本实验的突变体相似[18]。因此, 我们认定LOC_Os04g39430是参与调控水稻包穗表型的候选基因。

表4 定位区间内基因的功能注释

图3 候选基因SUI2基因的图位克隆示意图
A:初步定位在4号染色体标记S4-14和S4-15之间; B:被精细定位在S4-14.1和S4-14.2之间的110 kb范围内; C:精细定位区间内包含的14个基因和2个转座子。横线的上方为定位所用的分子标记, 横线下方为交换单株数。
A: Rough mapping of the geneflanked by two markers S4-14 and S4-15; B:was fine mapped to a 110 kb interval delimited by two markers S4-14.1 and S4-14.2 region; C: 14 ORFs and 2 transposons within the fine-mapped fragment. The molecular markers used in linkage analysis were indicated above the bold lines, and the umbers beneath the bold lines represented the recombinants identified by the corresponding markers.
对不同组织中表达量检测结果表明,在突变体的幼穗、叶枕、茎秆中表达水平均显著提高,在幼穗中表达量为514倍, 在茎中表达量为369倍, 在叶枕表达量为214倍(图5)。

图4 候选基因的qRT-PCR检测
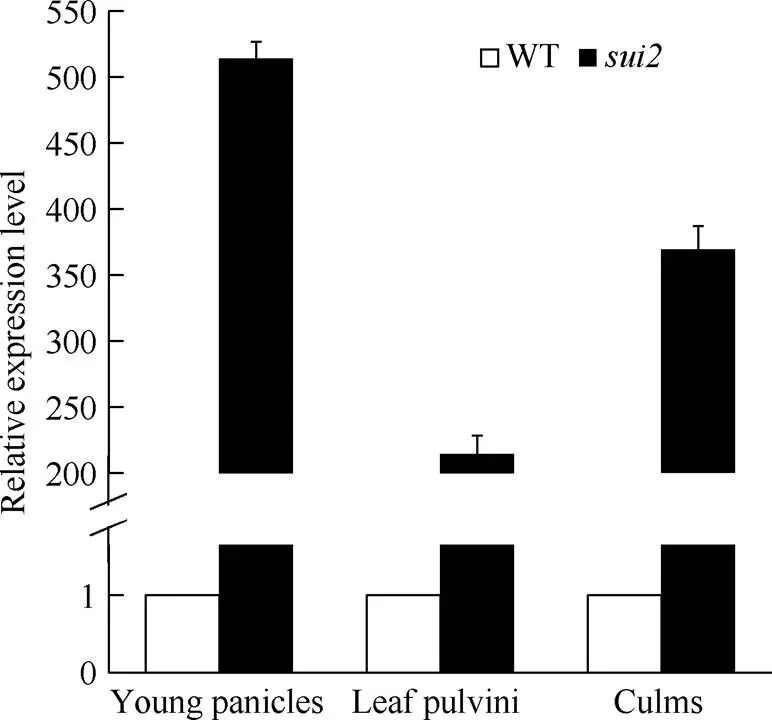
图5 SUI2在不同组织中的表达量变化情况
2.5 突变体油菜素内酯合成及信号途径相关基因表达量分析
LOC_Os04g39430编码了1个细胞色素P450酶, 它在油菜素内酯合成通路中发挥作用, 可能参与了油菜素内酯生物合成通路中6-DeoxoTY和TY的合成[18]。为了检测突变体中其他油菜素内酯合成及信号通路基因表达量的变化, 我们利用qRT-PCR分析了野生型和突变体中油菜素内酯合成相关基因[19][20][21]及信号通路相关基因[22][23][24][25][26]在茎秆中的表达情况。结果表明, 与野生型相比, 3个合成基因在突变体的表达量没有一致变化规律, 1个表达量上调()、1个下调()、1个没有明显变化()。而被检测的6个信号转导基因中, 有4个基因的表达量明显上调, 尤其是基因BU1的表达量增加了6倍多, 预示着可能一些BR信号转导途径的基因参与了穗颈节的伸长和包穗表型的调控。
3 讨论
水稻的穗外露主要由倒一节间的长度决定, 由于突变体倒一节间显著缩短, 导致穗颈节伸出度降低, 从而产生包穗。包穗突变体来自于粳稻品种Kitaake的组织培养后代, 突变体籽粒增大、单穗粒数减少、结实率下降明显。对的茎秆细胞生物学观察发现, 株高变矮和包穗产生的原因可能与细胞的纵向伸长不足有关。对该突变体的遗传分析结果表明, 它为1个显性突变体, 突变基因被定位在第4号染色体的标记S4-14.1和S4-14.2之间, 物理距离为110 kb的区间内。区间内的基因LOC_Os04g39430的表达量在突变体内发生了明显的上调, 高达230倍。由于LOC_Os04g39430是的等位基因, 加之该基因的过表达会导致包穗现象[27], 我们认为LOC_Os04g39430是引起包穗表型的关键基因。由于LOC_Os04g39430的基因组和启动子没有DNA序列水平上的差异, 该包穗表型可能不是由于DNA水平上的差异引起的, 我们推测可能是由于表观遗传修饰引起的基因表达量升高而造成包穗表型。表观遗传学修饰涉及到DNA甲基化、非编码RNA的调控、组蛋白修饰、染色质重塑等多种类型[28-29], 具体哪种现象导致突变体的表型差异, 尚不明确, 需要对该基因进行深入研究。
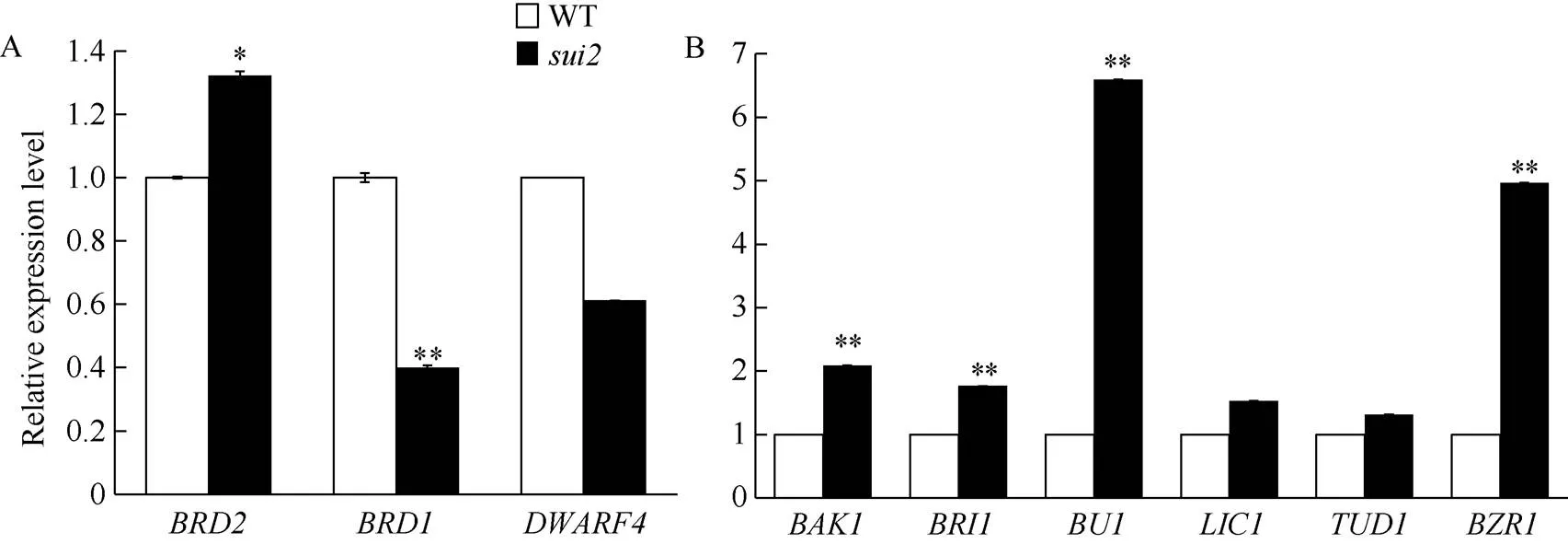
图6 油菜素内酯相关基因在野生型和突变体中的表达量变化情况
A: 野生型及的茎秆中油菜素内酯合成相关基因的表达水平; B: 野生型及的茎秆中油菜素内酯信号相关基因的表达水平。*表示在< 0.05水平差异显著;**表示在< 0.01水平差异显著。
A: Relative expression levels of the genes related to BR biosynthesis in WT andculms. B: Relative expression levels of the genes related to BR signaling in the young WT andyoung culms.*represents significantly different at< 0.05;**represents significantly different at< 0.01.
基因LOC_Os04g39430最初被Tanabe等[18]命名为基因, 它编码了1种细胞色素P450蛋白, 是BR生物合成过程中的1种关键酶。后来研究又陆续报道了几种的等位基因, Wu等[27]研究表明表现为株高降低、叶夹角变小、籽粒变小, 突变体中表达量上调了5倍; Shi等[30]研究表明,表现籽粒变小且为黄褐色, 植株高于野生型, 叶片直立, 该基因在突变体的叶中的表达量为野生型的2.5倍, 而在茎、根、叶鞘、小穗中该基因表现为下调; Guo等[31]报道突变体表现为半包穗、叶夹角变小、叶宽增加、籽粒发育不正常。以上3个突变体均为隐性突变体。而本实验中为显性突变体, 表现为株高降低, 第1节间缩短, 籽粒变长, 自苗期之后的全生育期叶夹角明显增大, 叶宽减少。这与之前报道的其他几个突变体明显不同。Tanabe等[18]又发现基因与BR生物合成有关, 本研究对野生型和突变体茎秆组织中的BR合成与信号通路相关基因进行了表达量分析, 结果表明与野生型相比,中BR合成途径中相关基因表达量无一致的变化趋势, 而信号转导途径相关基因表达量显著上调, 因此过表达该类基因可能导致了BR信号途径基因表达量增加。
GA是调节细胞伸长和最上节间长度的主要植物激素[32]。Zhu等[33]和Luo等[34]研究发现,与具有生物活性赤霉素GA4代谢有关, 同时发现穗下倒一节间伸长与赤霉素积累有关, 穗下倒一节间伸长突变体在节间部位积累了过量的赤霉素。Oikawa等[35]研究结果表明基因突变可以抑制植株体内赤霉素合成, 体内赤霉素含量降低, 最终导致细胞伸长受阻, 使植株矮化, 但喷施外源GA可恢复其表型。此外, 一些研究也指出磷脂酰丝氨酸合酶也参与了倒一节间伸长[15]。但是, 关于BR途径基因对穗下倒一节间伸长影响的报道极少, 它们具体作用的分子机制仍待探究。BR相关途径的基因表达水平分析表明, 有可能信号途径的基因参与了倒一节间伸长的表达调控,也为这方面深入研究提供了难得的材料。
4 结论
通过对来源于组织培养后代的包穗突变体研究发现, 突变体倒一节间长度缩短是包穗的主要原因。此外,还表现植株矮化、分蘖数增加、每穗粒数减少、结实率下降、籽粒变大、剑叶变窄、倒一和倒二叶的夹角显著增大等表型。该性状受1对显性基因控制,基因被定位在水稻4号染色体上标记S4-14.1和S4-14.2之间物理距离为110 kb区间内, 候选基因LOC_Os04g39430编码了1个细胞色素P450蛋白。本研究同时也证明了油菜素内酯信号途径的基因有可能参与了倒一节间伸长的表达调控, 这为进一步研究BR信号通路基因控制水稻包穗机制奠定了一定的理论基础。
[1] 邓华凤. 中国杂交粳稻研究现状与对策. 杂交水稻, 2006, 21(1): 1–6. Deng H F. Status and technical strategy on development ofhybrid rice in China., 2006, 21(1): 1–6 (in Chinese).
[2] 朱斌成. 施用“920”对杂交水稻制种父母本农艺性状的影响. 江西农业科技, 1988, (6): 4–6. Zhu B C. Effects of “920” on parental agronomic traits of hybrid rice seed production., 1988, (6): 4–6 (in Chinese).
[3] 李安祥, 李慈厚, 丁克信, 严国红. 杂交制种稻粒黑粉病的综合防治. 江苏农业科学, 1995, (4): 34–36. Li A X, Li C H, Ding K X, Yan G H. Comprehensive control of rice seed powdery mildew of hybrid seed production., 1995, (4): 34–36 (in Chinese).
[4] Li J, Yuan L P. Hybrid rice: genetics, breeding, and seed Production. In: Janick J ed. Plant Breeding Reviews. Springer, 2010. pp 15–158.
[5] Guan H Z, Duan Y L, Liu H Q, Chen Z W, Zhuo M, Zhuang L J, Qi W M, Pan R S, Mao D M, Zhou Y C. Genetic analysis and fine mapping of an enclosed panicle mutantin rice (L.)., 2011, 56: 1476–1480.
[6] Heu M H, Shrestha G. Genetic analysis of sheathed panicle in a Nepalese rice cultivar Gamadi. Manila: Rice Genet I: (In 2 Parts), 1986. pp 317–322.
[7] Kinoshita T. Report of committee on gene symbolization, nomenclature, and linkage groups., 1995, 12: 13–74.
[8] Maekawa M. Allelism test for the genes responsible for sheathed panicle., 1986, 3: 62–63.
[9] Shrestha G L, Heu M H. Gene location for “Gamadiness” in rice (L.)., 1984, 29: 128–135.
[10] Yin H F, Gao P, Liu C W, Yang J, Liu Z C, Luo D. SUI-family genes encode phosphatidylserine synthases and regulate stem development in rice., 2013, 237: 15–27.
[11] Zhu L, Hu J, Zhu K M, Fang Y X, Gao Z, He Y Z, Zhang G H, Guo L B, Zeng D L, Dong G J, Yan M X, Liu J, Qian Q. Identification and characterization of SHORTENED UPPERMOST INTERNODE 1, a gene negatively regulating uppermost internode elongation in rice., 2011, 77: 475.
[12] 刘庄, 罗丽娟. 水稻矮秆鞘包穗突变体茎的形态解剖学研究. 中国农学通报, 2006, 22(12): 409–412. Liu Z, Luo L J. Anatomical studies on the stem of rice of dwarf and sheathed panicle., 2006, 22(12): 409–412 (in Chinese with English abstract).
[13] 王伟平, 朱飞舟, 唐俐, 陈立云, 武小金. 一种水稻全包穗突变体的发现及初步分析. 中国农学通报, 2008, 24(6): 222–226. Wang W P, Zhu F Z, Tang L, Chen L Y, Wu X J. Discovery and preliminary analysis of a rice mutant with fully sheathed panicle., 2008, 24(6): 222–226 (in Chinese with English abstract).
[14] 朱克明. 水稻包穗基因 SHP6 的遗传与定位. 扬州大学硕士学位论文, 2006. Zhu K M. Genetic Analysis and Mapping ofGene in Rice. MS Thesis of Yangzhou University, Yangzhou, Jiangsu, China, 2006 (in Chinese with English abstract).
[15] Ma J, Cheng Z J, Chen J, Shen J B, Zhang B C, Ren Y L, Ding Y, Zhou Y H, Zhang H, Zhou K N, Wang J L, Lei C L, Zhang X, Guo X P, Gao H, Bao Y Q, Wan J M. Phosphatidylserine synthase controls cell elongation especially in the uppermost internode in rice by regulation of exocytosis., 2016, 11: e0153119.
[16] Maekawa M. Genes linked within rice., 1992, 42: 212–213.
[17] Lestari P, Koh H J. Development of new CAPS/dCAPS and SNAP markers for rice eating quality., 2013, 20: 15–23.
[18] Tanabe S, Ashikari M, Fujioka S, Takatsuto S, Yoshida S, Yano M, Yoshimura A, Kitano H, Matsuoka M, Fujisawa Y. A novel cytochrome P450 is implicated in brassinosteroid biosynthesis via the characterization of a rice dwarf mutant,, with reduced seed length., 2005, 17: 776–790.
[19] Makarevitch I, Thompson A, Muehlbauer G J, Springer N M.gene in maize encodes a brassinosteroid C-6 oxidase., 2012, 7: e30798.
[20] Hong Z, Ueguchi-Tanaka M, Fujioka S, Takatsuto S, Yoshida S, Hasegawa Y, Ashikari M, Kitano H, Matsuoka M. The ricemutant, defective in the rice homolog ofDIMINUTO/DWARF1, is rescued by the endogenously accumulated alternative bioactive brassinosteroid, dolichosterone., 2005, 17: 2243–2254.
[21] Choe S, Dilkes B P, Fujioka S, Takatsuto S, Sakurai A, Feldmann K A. Thegene ofencodes a cytochrome P450 that mediates multiple 22α-hydroxylation steps in brassinosteroid biosynthesis., 1998, 10: 231–243.
[22] Nam K H, Li J. BRI1/BAK1, a receptor kinase pair mediating brassinosteroid signaling., 2002, 110: 203–212.
[23] He J X, Gendron J M, Sun Y, Gampala S S, Gendron N, Sun C Q, Wang Z Y.is a transcriptional repressor with dual roles in brassinosteroid homeostasis and growth responses., 2005, 307: 1634–1638.
[24] Zhang C, Xu Y, Guo S, Zhu J Y, Qing H, Liu H H, Wang L, Luo G Z, Wang X J, Chong K. Dynamics of brassinosteroid response modulated by negative regulator LIC in rice., 2012, 8: e1002686.
[25] Tanaka A, Nakagawa H, Tomita C, Shimatani Z, Ohtake M, Nomura T, Jiang C J, Dubouzet J G, Kikuchi S, Sekimoto H, Yokota T, Asami T, Kamakura T, Mori M. BRASSINOSTEROID UPREGULATED1, encoding a helix-Loop-helix protein, is a novel gene involved in brassinosteroid signaling and controls bending of the lamina joint in rice., 151: 669–680.
[26] Hu X, Qian Q, Xu T, Zhang Y, Dong G J, Gao T, Xie Q, Xue Y B, Li J M. The U-box E3 ubiquitin ligasefunctions with a heterotrimeric G α subunit to regulate brassinosteroid-mediated growth in rice., 2013, 9: e1003391.
[27] Wu Y Z, Fu Y Z, Zhao S S, Gu P, Zhu Z F, Sun C Q, Tan L B. CLUSTERED PRIMARY BRANCH 1, a new allele of, controls panicle architecture and seed size in rice., 2016, 14: 377–386.
[28] Shi J, Dong A, Shen W H. Epigenetic regulation of rice flowering and reproduction., 2015, 5: 803.
[29] Heard E, Martienssen R A. Transgenerational epigenetic inheritance: myths and mechanisms., 2014, 157: 95–109.
[30] Shi Z Y, Rao Y C, Xu J, Hu S K, Fang Y C, Yu H P, Pan J J, Liu R F, Ren D Y, Wang X H, Zhu Y Z, Li Z, Dong G J, Zhang G H, Zeng D L, Guo L B, Hu Z, Qian Q. Characterization and cloning of SMALL GRAIN 4, a novelallele that affects brassinosteroid biosynthesis in rice., 2015, 60: 905–915.
[31] Guo M, Yang Y H, Liu M, Meng Q C, Zeng X H, Dong L X, Tang S Z, Gu M H, Yan C J. Clustered spikelets 4, encoding a putative cytochrome P450 protein CYP724B1, is essential for rice panicle development., 2014, 59: 4050–4059.
[32] Tong H, Chu C. Functional specificities of brassinosteroid and potential utilization for crop improvement., 2018, 23: 1016–1028.
[33] Zhu Y Y, Nomura T, Xu Y H, Zhang Y Y, Peng Y, Mao B Z, Hanada A, Zhou H C, Wang R X, Li P J. ELONGATED UPPERMOST INTERNODE encodes a cytochrome P450 monooxygenase that epoxidizes gibberellins in a novel deactivation reaction in rice., 2006, 18: 442–456.
[34] Luo A D, Qian Q, Yin H F, Liu X Q, Yin C X, Lan Y, Tang J Y, Tang Z S, Cao S Y, Wang X J., encoding a putative cytochrome P450 monooxygenase, regulates internode elongation by modulating gibberellin responses in rice., 2006, 47: 181–191.
[35] Oikawa T, Koshioka M, Kojima K, Yoshida H, Kawata M. A role of, encoding an isoform of gibberellin 20-oxidase, for regulation of plant stature in rice., 2004, 55: 687–700.
Genetic analysis and fine mapping of a sheathed panicle mutantin rice (L.)
SUN Qi1,**, ZHAO Zhi-Chao2,**, ZHANG Jin-Hui2, ZHANG Feng2, CHENG Zhi-Jun2,*, and ZOU De-Tang1,*
1Key Laboratory of Germplasm Enhancement, Physiology and Ecology of Food Crops in Cold Region, Ministry of Education, Northeast Agricultural University, Harbin 150030, Heilongjiang, China;2Institute of Crop Sciences, Chinese Academy of Agricultural Sciences, Beijing 100081, China
The elongation and development of rice uppermost internode plays an important role in plant architecture development. In general, the sheathed panicle phenomenon in rice sterility line is caused by the elongation and development hindrance of the uppermost internode. The study on molecular mechanisms underlying sheathed panicle would be helpful for improving plant architecture in sterility line. At the present study, we reported the study on a sheathed panicle mutant, named, originated from a tissue-culture progeny. Its uppermost internode severely shortened, resulted in its pancleen closed by flag leaf sheath, without significantly length alternation at other internodes. The cytological analysis demonstrated that the shorten of the uppermost internode is caused by insufficient elongation of the parenchyma cells. Genetic analysis of the progeny derived from the cross-combination ofand IRAT129 revealed thatis a single gene dominant mutant. Linkage analysis to 608 normal individuals from F2generation showed thatwas located in a 110 kb region delimited by InDel marks S4-14.1 and S4-14.2 on the end of chromosome 4 long arm. The annotated genes on this region did not display any difference in genomic sequence, while the expression level of, encoding a cytochrome P450 protein and an allele of, increased by 264 times expression amount in mutant. Analysis of qRT-PCR for several crucial genes on BR (brasssinolide) signaling pathway showed that in mutant the expression level of all these genes increased, indicating that genes in BR signaling pathway may be involved in the regulation to the elongation and development of uppermost internode.
rice; sheathed panicle; fine mapping;; BR signaling pathway
10.3724/SP.J.1006.2020.02009
本研究由国家自然科学基金项目(31871603)资助。
This study was supported by the National Natural Science Foundation of China (31871603).
邹德堂, E-mail: zoudtneau@126.com; 程治军, E-mail: chengzhijun@caas.cn
**同等贡献(Contributed equally to this work)
孙琦, E-mail: 517596634@qq.com; 赵志超, E-mail: zhaozhichao@caas.cn
2020-02-14;
2020-06-02;
2020-06-22.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20200622.1052.002.html

