异常寡核苷酸结合折叠域蛋白基因对肝细胞癌DNA复制起始的影响
李根亮,倪安妮,唐玉莲,冯 权,黄小程,阮文慧,李曙波,陆瑞群
0 引 言
癌症发生起始于正常体细胞突变成永生的癌细胞。在这一过程中,癌基因和抑癌基因突变,逆转录酶和生长因子异常激活[1-2]。这些变化使会走向死亡的体细胞去分化,从而开始新的不断分裂增殖的细胞周期。细胞增殖过程中,首先开始的是DNA复制,复制过程则起始于复制起始复合物的生成。微小染色体维持(mini chromosome maintenance,MCM)复合物在DNA复制起始中发挥着不可替代的作用[3-4]。当MCM复合物与复制起始点结合时,能解开结合部位的双链DNA,募集DNA聚合酶并启动DNA合成。MCM功能失调会导致染色体缺陷,因此可能参与肿瘤发生[5]。MCM复合体组成蛋白常含有寡核苷酸结合(oligonucleotide binding,OB)折叠域。该结构域能够识别端粒结构并招募端粒酶及其他相关蛋白构成端粒复合物,以维持端粒的稳定[6-7]。Zhu等[8]研究发现,在人肺癌细胞中,OB折叠域蛋白TPP1可通过抑制端粒酶向端粒的募集抑制细胞增殖,诱导细胞凋亡。OB折叠域蛋白基因(OB fold domain protein-encoding genes,OBGs)突变则会引起端粒的不稳定,从而诱发肿瘤的发生[9-10]。肝癌的影响因素很多[11-12],然而,OBGs异常在肝细胞癌中通过MCM复合物影响复制起始的相关作用报道较少。由于逆转录在癌症发生中具有独一无二的作用,因此,本研究旨在探讨逆转录相关基因(reverse transcription related genes,RTGs)在肝细胞癌中的作用及可变剪接和单核苷酸位点变异(single nucleotide variants,SNV)与基因表达异常的相关性。
1 材料与方法
1.1 动物模型构建及样本获取选取150只洁净级昆明小鼠,鼠龄6周,体重(30±5)g。采用随机数字表法取100只,双前肢腋下注射对数生长期肝细胞癌细胞株H22等渗盐水悬液作为H22组。细胞悬液密度105个细胞/mL,注射量0.2 mL;其余50只同样方法注射0.2 mL不含H22的等渗盐水作为对照组。小鼠正常饲养40 d。从H22组小鼠中选出成瘤小鼠,解剖取出瘤体;从对照组小鼠中取健康肝组织。
1.2 RNA提取和测序利用RNA提取试剂盒提取H22组和对照组各样本总RNA,同组内等量取各样本总RNA混合成总RNA样本。RNA-seq、质量检测、组装及高质量测序数据(clean reads)筛选按常规程序完成[13]。
1.3 差异表达基因分析以小鼠基因组为背景对高质量测序数据进行比对、注释和较正。以各基因匹配的clean reads为依据,统计分析每百万reads中来自于某基因每千碱基长度的reads数(reads per kilo bases per million reads,RPKM)的差异。以H22组和对照组同一基因之间的RPKM比值≥2或≤0.5,且错误发现率(false discovery rate,FDR)≤0.05的基因为差异表达基因(differentially expressed genes,DEGs)。
1.4 RDEGs的筛选以"Reverse transcript(逆转录)"为关键词,在NCBI中的Nucleotide(核苷酸)数据库进行检索,将检索结果通过DAVID在线转换成Ensembl基因组数据库的基因代码和正式基因符号。将转换结果与测序数据中的DEGs进行比较,筛选出在2类样本之间差异表达的逆转录相关DEGs(reverse-transcription-related DEGs,RDEGs)。
1.5 RDEGs的功能分析利用DAVID 6.8软件(https://david.ncifcrf.gov)对RDEGs进行GO和KEGG分析,并对富集到的生物学程序、细胞组分、分子功能、信号通路和功能部位等进行功能注释聚类,其中FDR≤0.05的为有统计学意义。
1.6 RDEGs编码蛋白的互作分析通过STRING 11.0版本(https://string-db.org)在线对RDEGs编码蛋白进行互作分析,以评价其在表达上的相关性及其中发挥功能的关键分子。
1.7 RDEGs多态性与基因表达的相关性分析利用Excel软件,对RDEGs中出现的可变剪接、SNV及插入-删除(insertion-deletion,INDEL)等基因多态性变异进行χ2检验,其中P<0.05且2样本间发生频率的倍数≥2的为差异有统计学意义。同时,对OBGs的可变剪接和SNV/INDEL进行位点分布分析,探讨基因的突变情况。
1.8 RT-qPCR验证RDEGs随机选择4个在2类样本中均表达的RDEGs,进行RT-qPCR分析,用以验证RNA-seq结果。PCR条件参照文献[13],引物利用Blast-primer软件设计,由上海生工进行合成。
2 结 果
2.1 测序数据H22组和对照组分别获得了5.01G base(35.28M clean reads)和5.02G base(35.32M clean reads)的数据。H22组和对照组所得clean reads对基因组的匹配率分别为93.65%和89.20%,且2组测序数据的Q20皆>99%。表明本研究测序数据的质量较高,能满足后续分析的需要。
2.2 RDEGs分析H22组和对照组总共有4533个DEGs。RTGs检索结果显示,在nucleotide数据库中共存在28123个与逆转录相关的核苷酸序列。DAVID6.8软件共成功注释到9264个基因,与DEGs匹配的RDEGs有193个,其中77个上调表达,116个下调表达。随机选择4个在H22组和对照组样本中均表达的RDEGs(Gclc、Gclm、Comt、Agt)进行RT-qPCR分析,内参基因为Rn18s,肝细胞癌中这4个基因的表达皆下调,该结果与RNA-seq结果一致,见图1。引物序列见表1。
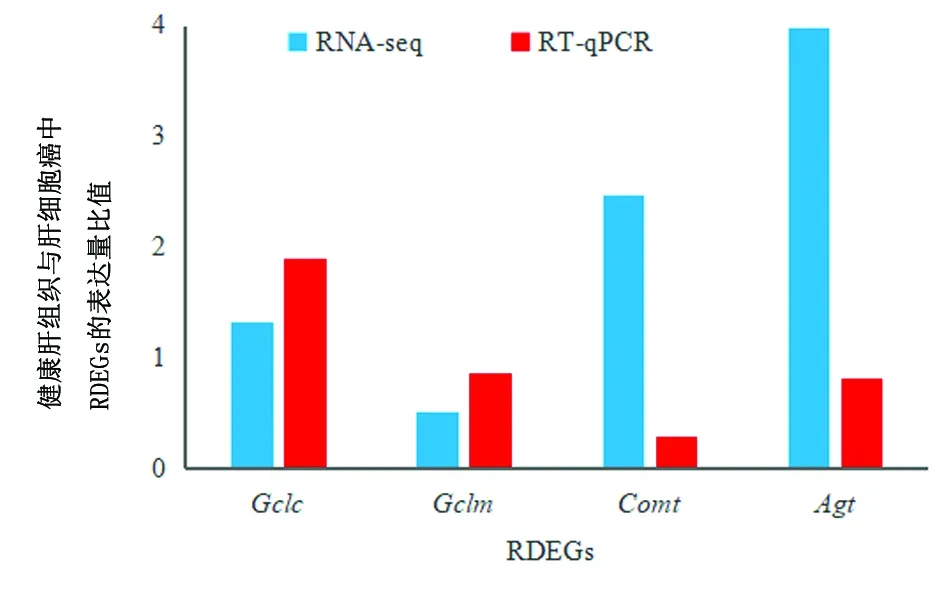
图1 RT-qPCR与RNA-seq分析肝细胞癌和健康肝组织中RDEGs的表达情况

表 1 RT-qPCR引物
2.3 RDEGs的功能分析功能富集分析结果显示,本研究所得193个RDEGs共富集到2个生物学程序、3个细胞组分、1个分子功能、3个信号通路和3个功能部位。其中,2个生物学程序为:DNA复制起始和凋亡过程的负调控;3个细胞组分为:核染色体端粒区、膜和MCM复合体;1个分子功能为:损伤DNA的结合;3个信号通路为:细胞周期、DNA复制和错配修复;3个功能部位为:核酸结合的OB-折叠、微型染色体维持的保守位点、微型染色体维持相关DNA依赖的ATP酶。其中,除了负调控凋亡程序及膜组分中存在下调表达基因外,参与其他10个功能的基因,如构建MCM复合体和复制起始复合体的RDEGs,皆上调表达。除了功能紊乱的细胞凋亡负调控和膜组分外,RDEGs的功能主要集中在生物学程序中的DNA复制起始、细胞组分中的MCM复合物和核染色体端粒区,并通过细胞周期中的DNA复制和错配修复的信号通路及损伤DNA结合的分子功能来实现相关功能。能与核酸结合的OBGs功能包括DNA复制起始、MCM复合物构建、通过保守位点和DNA依赖的ATP酶的MCM。见图2、图3。
红星表示该基因上调表达
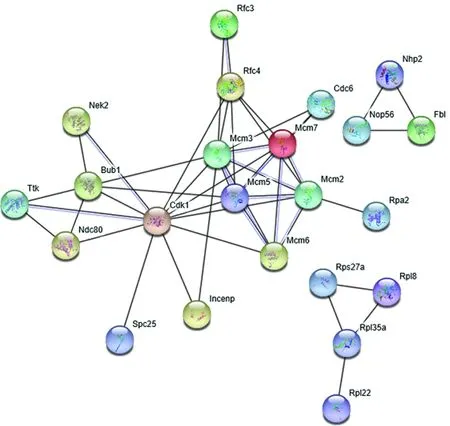
图 3 RDEGs编码蛋白的互作关系
2.4 肝细胞癌及健康肝组织中的可变剪接和SNV/INDEL可变剪接分析结果显示,4个RDEGs在基因的3个位点发生了差异表达的可变剪接,即第一个外显子可变剪切、最后一个外显子可变剪切、单内含子滞留(包括ON及OFF 2种),且可变剪接与相应基因的表达呈正相关,即Cd81和Agt的可变剪接和基因表达皆下调,Lsm3和Mycr的可变剪接和基因表达皆上调。结果表明,可变剪接在基因表达中可能起着正调控作用。
SNV和INDEL分析显示,肝细胞癌组织中共有157个RDEGs,发生了1541个SNV和78个INDEL位点的改变。上述基因位点的改变主要发生在基因的6个部位,即外显子区、内含子区、基因间区、基因下游区、基因上游区和拼接区。上述核苷酸位点发生改变的基因中,共有28个基因的SNV改变差异有统计学意义(P<0.05),其中26个基因的SNV改变与其基因的表达呈正相关。但是,INDEL的改变与相关基因的表达没有明显的相关性(P>0.05)。基因位点多态性分析结果显示,5个OBGs(Mcm2、Mcm3、Mcm5、Mcm6、Rpa2)在肝细胞癌中出现了SNV,而健康肝组织中则没有SNV,见表2。上述SNV全部发生在外显子区,且其中4个OBGs参与MCM复合物构建。
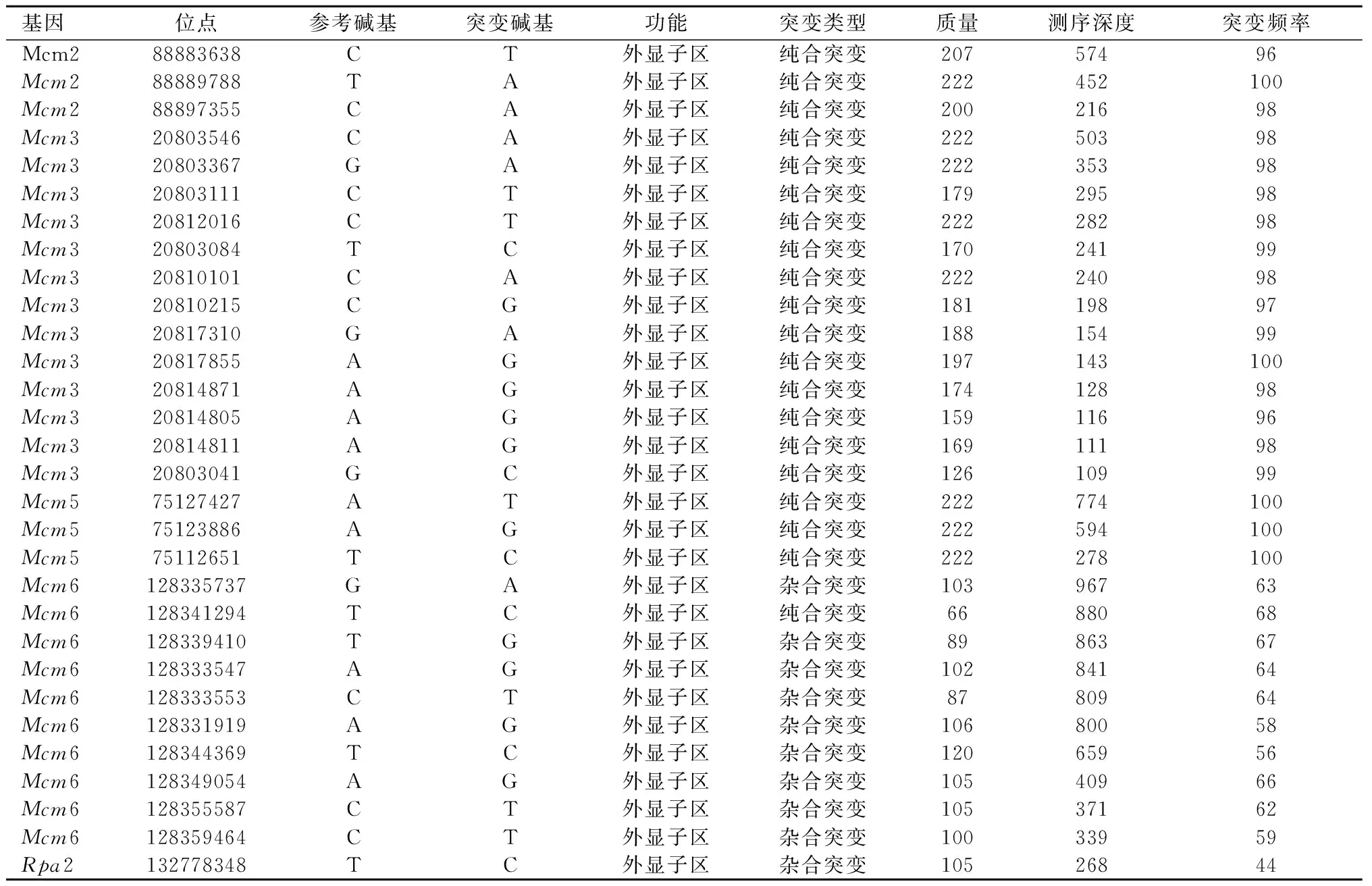
表 2 肝细胞癌中OB折叠域蛋白编码基因内 SNV的发生情况
3 讨 论
无论在生殖细胞发生,还是在体细胞癌变中,逆转录都是关键环节[1]。本研究发现,在肝细胞癌组织中的RDEGs参与DNA复制,尤其OBGs参与的MCM复合体及DNA复制起始。本研究结果显示,在MCM六聚复合体中有5个组分的基因在肝细胞癌中上调表达,包括Mcm2、Mcm3、Mcm5、Mcm6和Mcm7。MCM复合体是复制起始和复制调控所必需的。Patterson等[14]发现,MCM复合物参与恶性疟原虫非典型DNA复制周期中的复制前复杂组织形成。Hiraga等[15]发现,在MCM复合物控制DNA复制的过程中,Rif1通过指导蛋白磷酸酶1逆转Cdc7介导的磷酸化来实现。MCM复合体异常可能与癌症发生相关[5]。而且,上调表达的5个MCM复合体组分都具有OB折叠域。OB折叠域又称寡核苷酸/寡糖结合基序[16]。OB折叠域蛋白属于核酸结合蛋白,如ssDNA结合蛋白CDC13、DNA连接酶、DNA复制启始子、KREPA4等[16-18]。因此,OB折叠域在DNA复制、RNA转录、蛋白翻译中具有广泛的作用。Gao等[19]还发现,OB折叠复合物控制DNA双链断裂的修复途径。在肺癌细胞中,OB折叠域蛋白具有抑制细胞增殖和诱导细胞凋亡的功能[8],但其基因的突变可能引起端粒的不稳定从而起始肿瘤的发生或引起端粒复制和复制应激的异常[8,20-21]。
涉及复制起始的RDEGs包括Cdc6、Mcm7、Orc6、Mcm2、Mcm3、Mcm5和Mcm6,它们与另外5种RDEGs:Rpa2、Rfc3、Rfc4、Pcna和Rpa3,共同参与DNA的复制过程。这些基因中,Rpa2和Rpa3也属于OBGs。以上基因中的7种,即Rpa2、Mcm7、Pcna、Mcm2、Mcm3、Mcm5和Mcm6,及另外4个RDEGs:Cdk1、Thoc6、Orc1和Orc2,还参与核染色质中端粒区域的构建。端粒长度的维持是长寿及细胞干性的重要原因[2]。而细胞去分化、突变细胞的癌变、配子的发生都与逆转录酶的激活相关[1]。本研究发现与端粒相关的多种基因在肝细胞癌中上调表达,表明肝细胞癌细胞中参与逆转录及端粒维持的蛋白组分可能过量表达。由于大量的端粒复合体相关基因含有OB折叠域,因此OB折叠域可能与这些端粒复合体相关基因的功能相关。
本研究GO分析结果还表明,这些基因中的3种,即Rpa2、Pcna和Rpa3,与另外4种上调表达的RDEGs:Msh6、Polh、Xrcc1和Ogg1,共同参与损伤DNA的结合和修复,并协同调控癌细胞的细胞周期。KEGG分析同样表明,上调表达的RDEGs也参与以上GO功能相关的3个信号通路,即细胞周期、DNA复制和错配修复等。上述结果表明,在肝细胞癌中的RDEGs,尤其MCM复合体的异常在改变复制起始中发挥着极其重要的作用。而OB折叠域则在参与肝细胞癌中MCM复合体构建及复制相关其他功能如端粒复合体构建、DNA复制中的错配修复等中发挥着广泛的作用。本研究中RDEGs编码蛋白的互作分析同样说明了肝细胞癌中OBGs在这些功能上的重要性和相关性。
本研究中的可变剪接分析结果进一步表明,引起基因多态性的遗传因素可变剪接与基因表达呈正相关。另外,在肝细胞癌组织中,SNV的改变与其基因的表达呈正相关,而INDEL则与基因的表达无明显的相关性。也就是说,可变剪接和SNV引起的基因多态性在肝细胞癌中扮演着调控基因表达的重要功能。但有2个基因的SNV改变与相关基因的表达呈负相关,说明基因表达的调控是多层次的,还有一些调控方式可能改变了SNV对特定基因表达的正调控功能。这还有待进一步研究。本研究结果还显示,肝细胞癌中上调表达的OBGs多具有SNV现象。OB折叠域蛋白具有抑制癌细胞增殖和诱导癌细胞凋亡的功能,但突变后则会影响这些功能。基因多态性与基因编码蛋白的功能改变密切相关[9,22]。OBGs的多态性参与肿瘤发生和促进癌细胞生存[9-10,20-21]。因此,OBGs中的SNV可能在肝细胞癌发生中起着重要作用,如异常起始DNA的复制、招募端粒酶延长端粒等。
综上所述,RDEGs中的OBGs可能在肝细胞癌中通过自身的基因多态性突变引起MCM复合体及端粒复合体的改变,从而调控DNA复制起始及保护端粒的完整性,进而在癌细胞的增殖过程中发挥重要作用。而可变剪接及SNV则可能是一些基因上调表达的重要调控因素。

