细粒棘球绦虫肌动蛋白抗原表位预测※
李超群,张发斌,赵 婧,李凤辉,高瑞雪,樊海宁
(1.青海大学医学院;2.青海大学附属医院;3.青海省包虫病研究重点实验室)
目前,疫苗因具有安全、无残留、动物无休药期的优点在棘球蚴病的防治中得到广泛地研究、应用[1]。预测抗原表位有助于了解抗体抗原识别的免疫基础,有助于疫苗设计和药物研发[2]。
本研究通过相关课题设计,对细粒棘球绦虫肌动蛋白(Echinococcus granulosus actin Eg ACT)的结构和抗原表位进行分析,为筛选疫苗候选抗原提供依据。
1 研究方法
1.1 氨基酸序列获取
通过GenBank数据库查寻下载细粒棘球绦虫肌动蛋白的氨基酸序列,登录号为EUB56817.1。
1.2 蛋白质特性预测
利用Expasy线上分析系统(https://web.expasy.org/protparam/)的protparam数据库预测细粒棘球绦虫肌动蛋白的等电点、相对分子量、稳定性等理化性质,利用Expasy系统的protscale数据库预测亲/疏水区域。利用SignalP-5.0 Server(http://www.cbs.dtu.dk/services/SignalP/)和 TMHMM(http://www.cbs.dtu.dk/services/TMHMM/)软件确定蛋白质信号肽序列与跨膜结构区域,利用DNAstar Protean线下软件分析蛋白的亲水性、柔性区域、抗原指数及表面可及性。利用 SOPMA 和 SWISS-MODEL(http://swissmodel.expasy.org)在线软件预测空间结构中的二级结构与三级结构。
1.3 蛋白质抗原表位预测
用DNAstar Protean线下软件中的AMPHI法预测辅助性T淋巴细胞抗原表位、Rothbard-Taylor法预测T淋巴细胞抗原表位,综合两种方法分析获得优势T细胞表位;用IEDB软件和ABCpred软件综合预测细粒棘球绦虫肌动蛋白的优势B细胞表位。
2 结果
2.1 Eg ACT的理化性质
如表1所示,细粒棘球绦虫肌动蛋白相对分子质量为41.95 kDa;等电点为5.38;半衰期为30h(mammalian)>20 h(yeast)>10h(Escherichiacoli)。该蛋白经过稳定性分析显示,其不稳定指数为39.28,小于40,为稳定蛋白。
对蛋白质信号肽序列与跨膜结构区域进行分析,所得结果显示该蛋白质无信号肽序列、无跨膜螺旋区域。
2.2 空间结构特性
如图1所示,Eg ACT的二级结构中β转角占6.65%、α螺旋占38.83%、延伸链占21.54%、无规则卷曲占32.98%,以α螺旋和无规则卷曲为主。

图1 Eg ACT二级结构预测结果图Figure 1 Predictio.Results of Eg ACT secondary structure
如图2所示,Eg ACT的三级结构呈多折叠型。
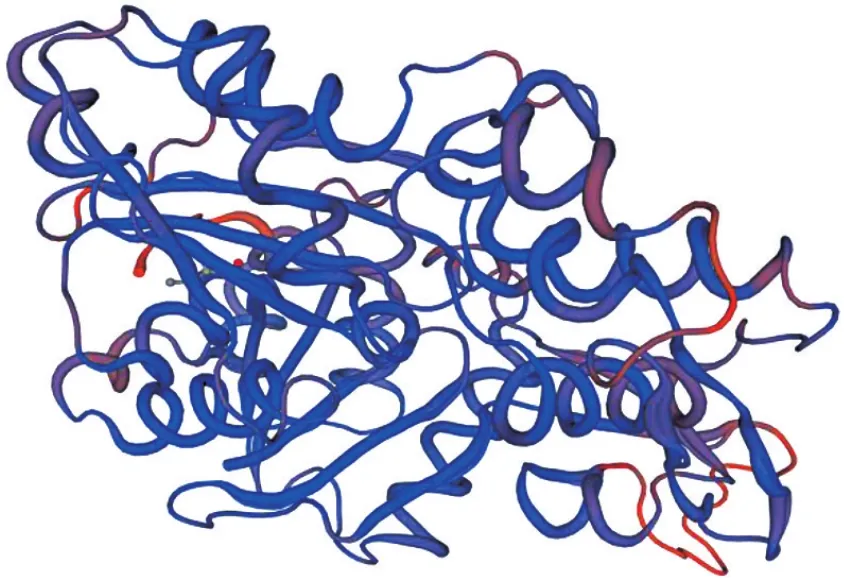
图2 Sw issmodel网站构建的Eg ACT三级结构图Figure 2 The three-level structure of Eg ACT built by the Sw issmodel website
2.3 亲水性、柔性区域、抗原指数及表面可及性
如图3所示,综合Expasy和DNAstar Protean软件分析结果,该蛋白可明显看到分布较多的亲水性区域,位于 21-30、50-67、70-92、95-106、109-121、191-216、222-246、283-295、311-321、331-340、355-346氨基酸区段,这些区域暴露于蛋白表面,易于形成抗原表位。

图3 Expasy与Protean软件预测的亲/疏水区Figure 3 The pro-hydrophobic region predicted by Expasy and Protean software
如图4所示,柔性区域呈散在分布,较明显的分布区域位于 12-30、37-42、57-63、79-85、107-118、195-208、232-240、282-293、312-319、360-368 氨基酸区段。表面可及性得分较高的区域主要位于58-63、78-86、109-119、231-244、286-294、312-318、359-365氨基酸区段。抗原性指数得分较高区域与柔性区域的分布一致。
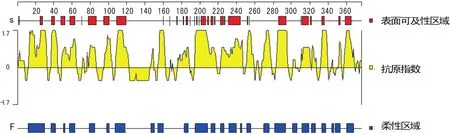
图4 Eg ACT柔性区域、抗原指数及表面可及性区域图Figure 4 Eg ACT proteinflexible region,antigen index and surface accessibility region
2.4 B细胞表位
ABCpred软件预测阈值>0.8的优势B细胞表位的氨基酸序列区域位于13-29、42-72、74-90、173-189、210-242、245-261、283-299、302-318、327-343氨基酸区段;如图5所示,IEDB软件预测长度大于6个氨基酸的区域位于14-31、36-42、52-61、77-82、97-105、108-118、155-165、199-208、233-244、285-290、319-325、361-369 氨基酸区段。 综合上述两种软件预测结果,可能形成的优势B细胞表位氨基酸序列位于 14-29、52-61、77-82、233-242、285-290氨基酸区段。
2.5 T细胞表位
如图6所示,AMPHI法预测可能形成辅助性T淋巴细胞抗原表位的区域位于29-36、75-91、185-193、208-217、220-228、273-285、309-321、346-355氨基酸区段;Rothbard-Taylor法预测可能的T淋巴细胞抗原表位位于 21-24、52-56、75-78、85-88、126-129、148-155、185-195、208-211、227-230、232-235、260-264、274-277、209-312、320-323、355-359氨基酸区段。综合上述两种软件预测结果,可能的优势T细胞表位区域位于75-78、85-88、185-193、208-211、274-277、309-312 氨基酸区段。
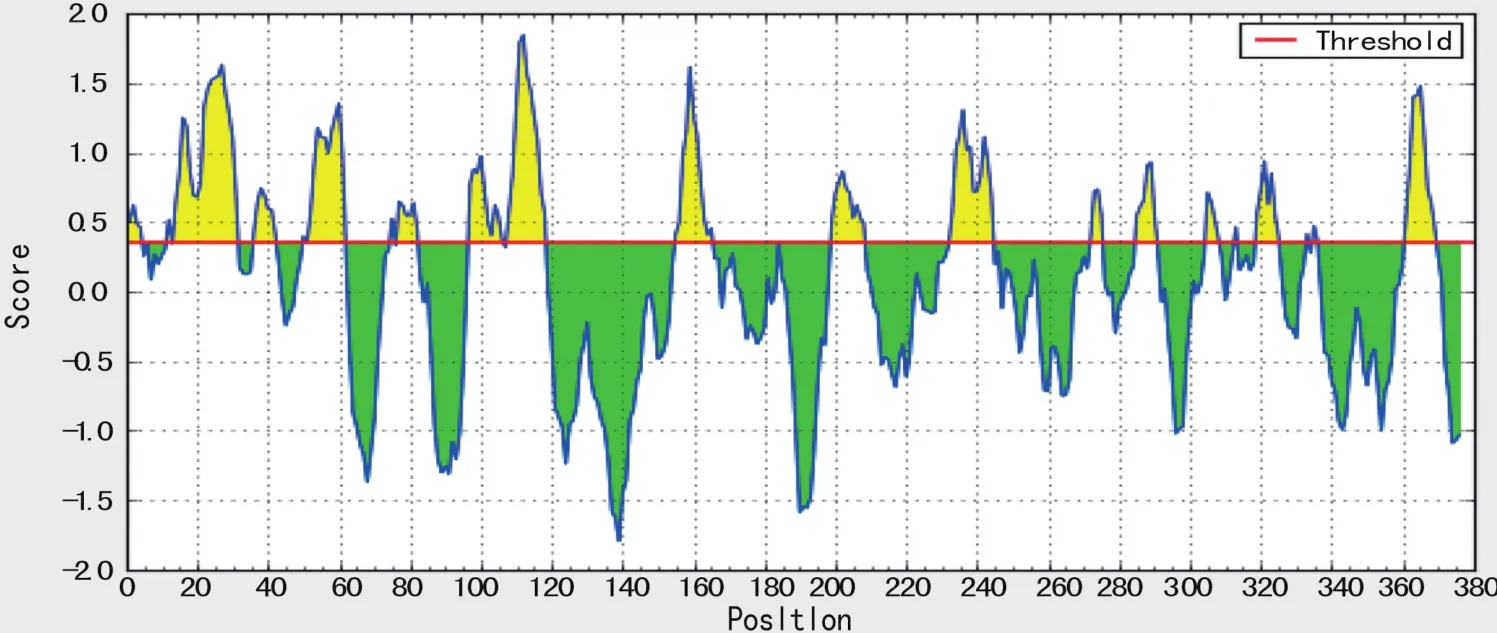
图5 IEDB软件预测的Eg ACT B细胞线性表位图Figure 5 Eg ACT B cells predicted by IEDB software

图6 DNAStar软件预测的T细胞表位图Figure 6 T-cell epitopes predicted by DNAStar software
3 讨论
本研究借助生物信息学的方式,对细粒棘球绦虫肌动蛋白的结构和抗原表位进行了分析,为筛选疫苗候选抗原提供了依据。
抗原表位为抗原分子表面一种特有的结构,可刺激机体产生能够被其识别的一个免疫活性区,该免疫活性的氨基酸基团又称为抗原决定簇。借助生物信息学的方式预测抗原表位的成本低、效率高[3]。
本研究通过蛋白分析软件对细粒棘球绦虫肌动蛋白的理化性质进行分析,确定该蛋白是否适宜作为研究对象。对蛋白质的信号肽和跨膜区域进行分析,确定该蛋白分布是否存在跨膜区域,是否为分泌蛋白并参与转运。对二级结构中的β转角、α螺旋、无规则卷曲等进行分析是为了解其与抗体结合的难易程度[4]。本研究发现,细粒棘球绦虫肌动蛋白的β转角、无规则卷曲所占二级结构的比例之和超过50%较易形成抗原表位。
分析亲水性、柔性区域、抗原性指数及表面可及性区域是为了解各个氨基酸残基的分布情况,亲水性区域一般位于蛋白质表面,细粒棘球绦虫肌动蛋白亲水性区域较多,易形成抗原表位;抗原性指数得分较高区域更易形成抗原表位;表面可及性区域反映蛋白质与其他分子的接触情况。
综合分析细粒棘球绦虫肌动蛋白的理化性质、结构特点和优势B、T细胞表位区域显示,该蛋白具有稳定性蛋白特征和易形成抗原表位的特点。

