基于复合物信息和亚细胞定位信息的关键蛋白质识别
毛伊敏,章宇盟,胡 健
(1.江西理工大学信息工程学院,赣州 341000;2.江西理工大学应用科学学院,赣州 341000)
关键蛋白质一般是指通过基因剔除式突变将其移除后会造成生物体相关功能缺失,并导致生物体生病或无法生存的蛋白质[1]。早期,在关键蛋白质研究方面,主要是通过生物实验方法进行预测,例如RNA干扰、单基因敲除、条件基因敲除。虽然该类方法的预测准确率高,但存在操作复杂、耗时成本代价高等缺陷。因此,研究人员逐渐转向基于生物计算的预测方法。随着高通量蛋白质组技术、计算机相关技术的迅猛发展以及蛋白质相互作用数据日益完善,这使得通过基于计算机的计算方法来识别关键蛋白质成为可能。
Jeong等[2]在2001年就指出,在蛋白质相互作用网络(PPI)中,存在着“中心-致死性”法则,即在PPI中邻居节点较大的蛋白质对细胞的生存起着非常重要的作用。Yu等[3]经过研究蛋白质相互作用(protein-protein interaction,PPI)网络相关数据发现,关键蛋白质的平均度值大约是非关键蛋白质的两倍。基于这些网络拓扑特征,众多基于拓扑中心性的关键蛋白质识别方法相继被提出,如度中心性(degree centrality,DC)[4]方法、接近度中心性(closeness centrality,CC)[5]方法、介数中心性(betweenness centrality,BC)[6]方法、子图中心性(subgraph centrality,SC)[7]方法、特征向量中心性(eigenvector centrality,EC)[8]、信息中心性(information centrality,IC)[9]方法、方法、邻居中心性方法(network centrality,NC)[10]和局部连通中心性方法(local average connectivity,LAC)[11]。由于在PPI网络中存在大量的假阳性和假阴性数据(噪声数据),使直接利用拓扑中心性特征来识别关键蛋白质的方法有缺陷;同时,这些中心性的方法也忽略了关键蛋白质本生固有的生物属性[12]。因此,为进一步提高关键蛋白质的预测精度,研究者将一种生物信息或多种生物信息融入PPI网络中。Li等[13]和Tang等[14]基于基因表达信息分别提出一种名为PeC(integrate ECC and person correlation)和WDC(weighted degree centrality)的关键蛋白质预测方法;Peng等[15-16]基于蛋白质同源信息和蛋白质域信息,将其与蛋白质在PPI网络中的拓扑特性相结合,提出UDoNC(united the domain features and the normalized ECC)和ION(the integration of the properties of orthologous and the features of neighbors)关键蛋白质预测方法;除此之外,研究者还提出基于蛋白质在复合物内的参与程度来衡量PPI网络的可靠性以及关键蛋白质与复合物之间紧密程度。胡赛等[17]通过计算两个蛋白质共享结构域的概率和共享复合物的概率以构建高可靠性加权PPI网络;Zhao等[18]利用PPI网络的模块化特性挖掘重叠的复合物,并通过计算蛋白质在复合物内的加权度来衡量蛋白质的关键性。Qin等[19]提出了基于局部密度、介数中心性(BC)和复合物度中心性(in-degree centrality of complex,IDC)的关键蛋白质识别算法LBCC。虽然基于复合物信息的关键蛋白质识别取得了一定的成效,但只考虑了蛋白质在复合物内的参与程度,忽略了蛋白质的关键性与复合物参与频率成正相关性这一特性。目前,基于拓扑特性的关键蛋白质识别算法虽然取得了一定的成效,但是通过高通量生物技术获得的大规模蛋白质相互作用网络数据中存在较高比例的噪声数据和不完备数据,以及基于复合物信息的关键蛋白质预测方法对复合物信息考虑不够全面导致关键关键蛋白质识别的准确性不高等缺陷,仍是亟待解决的问题。
针对以上问题,现提出一种名为基于复合物信息和亚细胞定位信息(united protein complexes and subcellular locallizations,PCSL)的关键蛋白质预测方法。主要开展以下3个方面工作:①融合PPI网络的拓扑属性、生物属性和空间属性3个方面构建加权网络,以降低原始PPI网络中噪声数据和不完整数据对关键蛋白质预测精度的负面影响,其中拓扑属性用边聚集系数计算,生物属性用生物功能相似性计算,空间信息用亚细胞定位信息计算;②基于复合物信息和亚细胞定位信息,综合考虑复合物参与频度和空间位置重要性,提出一种蛋白质关键性度量,以提高关键蛋白质预测精度;③基于加权PPI网络,利用改进的CPPK算法对PPI网络进行寻优操作,以提升关键蛋白质挖掘的效率。
1 PCSL方法
针对蛋白质PPI网络中存在大量噪声,现有大多关键蛋白识别方法对蛋白质关键性描述不全面以及关键蛋白质挖掘效率不高等问题,提出一种新的关键蛋白质预测方法PCSL。首先该方法基于边聚集系数、GO功能相似性和空间位置重要性,提出一种综合性边权值度量对PPI网络进行加权,从而构建加权网络;然后,综合考虑复合物参与度和复合物参与频率,提出一个名为复合物参与频度的度量,以更全面地描述关键蛋白质与复合物之间的紧密联系,然后结合复合物参与频度和亚细胞定位信息,提出一个新的衡量蛋白质关键性的公式;最后,基于应用于PPI网络的CPPK寻优算法,对其扩张策略进行改进,利用聚集度设计一个试探策略,避免CPPK算法陷入局部最优,以提高挖掘关键蛋白质的效率。
1.1 构建加权网络
由于高通量方法获得的蛋白质相互作用数据中存在假阳性、假阴性和不完整性(噪声),因此仅仅依靠PPI网络的拓扑特性来识别关键蛋白质较依赖于网路本身,限制了关键蛋白质识方法的性能。因此,基于PPI网路的拓扑特性和蛋白质生物功能相似性,结合PPI网络的空间属性(亚细胞定位信息),对原始PPI网络进行加权,以提升原始PPI网络的可靠性。
1.1.1 PPI网络的拓扑特性
边聚集系数[20]是网络拓扑特性中的重要一种,不仅考虑了边在网络中的重要程度,还能评估节点u、v邻居之间的紧密程度,且能较好地识别PPI网络中的关键蛋白质。边聚集系数的定义为
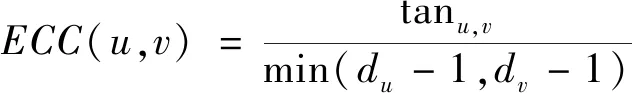
(1)
式(1)中:tanu、v表示节点u、v共同构成三角形的个数;du、dv分别表示节点u、v的度。
1.1.2 PPI网络的生物功能相似性
考虑到两个蛋白质之间的功能相似程度越高,它们之间相互作用就越可靠[21]。已有研究表明,若两个蛋白质的共享的GO功能注释越多,它们的生物功能相似程度越高。因此用GO语义相似度描述蛋白质之间的功能相似度,其计算公式为

(2)
式(2)中:|Gu|和|Gv|分别表示蛋白质u、v的GO功能注释集合的大小;|Gu∩Gv|表示蛋白质u、v之间的GO功能注释交集的大小。
1.1.3 PPI网络的空间属性
由于现有的大多数关键蛋白质预测方法都是从PPI网络的拓扑特性和生物属性两个方面去衡量蛋白质之间的可靠性,忽略了PPI网络的空间属性,对蛋白质相互作用关系考虑不够全面。文献[22]表明,如果两个蛋白质出现在同一细胞区域,它们之间的关系就越可靠。因此,用Lin描述两个蛋白质之间的可靠性,其计算公式为
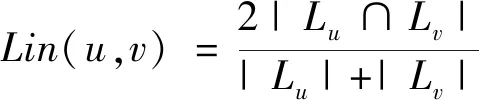
(3)
式(3)中:Lu和Lv分别表示蛋白质u、v的亚细胞定位信息集合;Lu∩Lv为蛋白质u、v之间的亚细胞定位信息交集。
为避免人为产生的假阴性的负面影响,综合考虑PPI网络的拓扑、生物特性和空间属性,提出一种综合性边权值度量公式:
ESL(u,v)=[ECC(u,v)+1][Sim(u,v)+
1][Lin(u,v)+1]
(4)
1.2 蛋白质关键性度量
1.2.1 复合物参与频度
由于基于复合物信息的关键蛋白质识别方法大多用蛋白质在复合物内的参与程度来衡量关键蛋白质与复合物之间的紧密联系,忽略了蛋白质的关键性与复合物参与频率(蛋白质出现在复合物的频率)之间的相关性[18],为更加准确描述蛋白质的模块化特性,综合考虑复合物参与度[19]和复合物参与频率两个方面,提出复合物参与频度来衡量蛋白质与复合物之间的紧密联系,其计算公式为
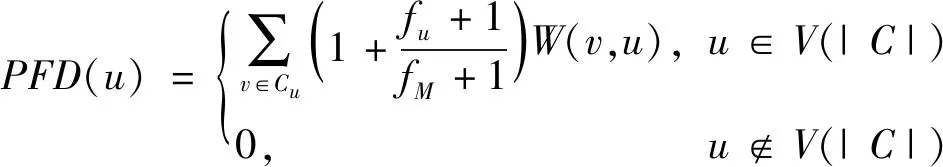
(5)
1.2.2 空间位置重要性
已有研究表明,蛋白质的关键性不仅仅与PPI网络的生物属性相关,还与蛋白质的空间位置相关。因此,充分利用亚细胞定位信息对关键蛋白质预测有重要意义。从PPI网络角度描述,蛋白质的关键性与细胞位置的置信水平相关[23];从蛋白质合成过程角度描述,蛋白质的关键性与细胞位置本身相关[24]。综合以上两点,提出一种利用空间信息衡量蛋白质关键性的度量公式:

(6)
式(5)中:li(v)表示蛋白质v所在的细胞区域;Sli(v)表示对应细胞区域中蛋白质集合;Smax表示所有细胞区域中蛋白质数量最大的集合;LI(li)表示相应细胞区域的关键指数[24]。
鉴于蛋白质的关键性与复合物、空间位置密切相关,为了提高关键蛋白质预测精度,将复合物参与频度、空间位置重要性用线性组合模型整合。整合之后的蛋白质关键性的度量式为

(7)
1.3 关键蛋白质识别
现有的大多数关键蛋白质识别方法都是首先根据蛋白质的某种重要性指标(如局部中心性LAC)排序,然后取前P个。虽然这样做可以识别关键蛋白质,但是这些方法都需要逐一计算顶点的某种指标并排序,无形中大大增加了计算量。为提高关键蛋白质挖掘的效率,引入CPPK寻优算法[25],并对其扩张策略进行改进。
1.3.1 CPPK算法的改进
由于CPPK算法本质上是一种贪心算法,为避免该算法容易陷入局部最优的缺陷,设计一种试探策略来实现跳出局部最优的目的。试探策略大致思想如下:在种子节点扩张之前,先计算种子的聚集度[26],如果聚集度大于或等于阈值∂,则向其邻居扩张,如果聚集度小于阈值∂,则从剩余的蛋白质集合中随机选取一个聚集度大于或等于阈值∂的蛋白质作为种子。
1.3.2 改进的CPPK算法描述
输入:加权PPI网络G,部分已知关键蛋白质集合K,需要预测的关键蛋白质数量n
输出:包含n个关键蛋白质的集合ES
ES=Φ,TES=Φ
For each node∈G
Compute C(node)
ES=K
WHILE(|ES|< n)
For each node∈K
TES=TES∪{u|max(PSLC(u)),u∈Nnode}
If(|ES|+|TES|<=n)
ES=ES∪TES
For each node∈TES
If(C(node)<∂)
Delete node from TES,select one in unprocessed proteins
K=TES
Else
Descend TES according to PSLC
ES=ES∪TESn-|ES|
Output ES
2 实验结果与分析
2.1 实验环境
实验所用的计算机配置为windows 7操作系统,Inter i5双核处理器,2.6 GHz主频和8 G内存。实验所用的程序代码用python编写,用IPython进行解释和交互。
2.2 标准实验数据集的选取
由于酵母PPI网络数据相对比较完善。因此以酵母蛋白质网络为研究对象,并展开相关实验。实验所需数据介绍如下。
(1)PPI数据集。酵母PPI网络数据从DIP数据库[27]中下载,经过数据预处理后得到5 093个蛋白质,24 743条相互作用关系边。
(2)亚细胞定位信息。酵母蛋白的亚细胞定位数据分为11类,从COMPARTMENTS[28]中下载获得。
(3)标准关键蛋白质集合。通过整合MIPS[29]、SGD[30]、DEG[31]和SGDP[32]4个数据库得到标准关键蛋白质数据,共有1 285个关键蛋白质(1 167个关键蛋白质出现在酵母PPI中)。
(4)酵母蛋白质GO注释信息[33]下载自基因本体数据库(2016年12月24日的版本),它主要包括3部分:生物过程、分子组件和分子功能。
(5)蛋白质复合物。蛋白质复合物集合从文献[34]中下载得到,其由CM270、CM425、CYC408和CYC428这4种蛋白质复合物集合整合。
2.3 标准参数α对关键蛋白识别的影响
在PCSL方法中,蛋白质的关键性评分由两部分组成:①蛋白质的复合物参与频度得分;②蛋白质的空间位置得分。由参数α调节两种不同得分重要性的比重,其中α的取值范围为[0,1],如表1所示。当α为1时,蛋白质的关键性仅取决于由复合物信息决定的复合物参与频度得分;当α为0时,蛋白质的关键性仅依靠蛋白质的空间位置。

表1 不同参数α对识别关键蛋白质数量的影响比较Table 1 The number of true essential proteins correctly identified by PCSL with different α
从表1可以看出,当α的取值范围为[0.3,0.5]时,PCSL方法的关键蛋白质的识别数目较多。特别是当α值为0.4时,PCSL方法识别的关键蛋白质最多。因此将α值设置为0.4。
2.4 关键蛋白质预测方法性能比较分析
2.4.1 不同比例关键蛋白质预测数量比较
为评估PCSL方法的关键蛋白质识别性能,首先,只与基于拓扑特征的中心性方法DC、BC、SC、EC、IC、LAC进行比较,验证PCSL方法中通过融合拓扑特性、生物功性能相似性和亚细胞定位信息而构建的加权PPI网络是否更加可靠,且有助于提高关键蛋白质识别准确率;其次,与基于拓扑数据和基因表达数据的预测方法PeC、WDC比较,以检验PCSL方法是否降低了对原始蛋白质相互作用网络本身的依赖;最后,与基于复合物信息的关键蛋白质预测方法LBCC比较,证明本文提出的蛋白质关键性度量是否能够识别更多的蛋白质。实验中,为得到较为精确的对比结果,首先将本文提出的PCSL方法应用于酵母PPI网络上,计算各个比例下关键蛋白质的个数;然后,利用以上提到的10种关键蛋白质预测方法,根据各自的节点重要性指标对候选蛋白质集合进行降序操作,得到一组根据各自节点重要性指标值降序排列的候选关键蛋白质集合;最后,选取每个候选关键蛋白质集合前1%、5%、10%、15%、20%、25%的候选蛋白质与一组标准关键蛋白质集合进行比较,获取各方法在不同规模下预测正确的关键蛋白质数量。图1所示为各方法识别候选集前1%~前25%中的关键蛋白质数量。
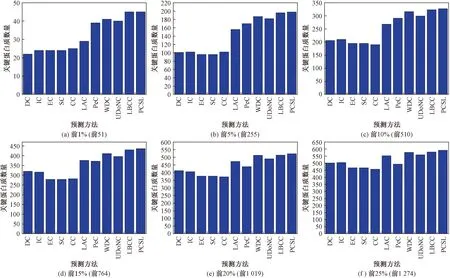
图1 PCSL与其他方法在不同规模样本中识别出的关键蛋白质数量Fig.1 The number of true essential proteins predicted by PCSL and other ten methods on DIP datase
从图1中可以明显看出,PCSL方法要优于其他10种预测方法,尤其是在前1%、5%、10%的候选关键蛋白质集合当中,预测关键蛋白质的准确率分别达到了86.3%、76.9%、63.5%。在仅基于PPI网络拓扑特征的中心方法中,表现最好的是方法LAC,PCSL方法与其相比,关键蛋白质预测精准率分别提高了29.4%、15.7%、11.0%、8.0%、4.5%、3.1%,其说明了PCSL方法构建的加权PPI网络拥有较高的可靠性;与融合基因表达信息的中心方法PeC相比,关键蛋白质预测精准率分别提高了9.8%、10.2%、6.5%、8.5%、8.2%、9.1%,并且在关键蛋白质候选集规模为1%、5%、15%的情况下,PCSL方法优势明显,PCSL方法降低了对原始PPI网络的依赖;与预测性能表现最佳的基于蛋白质复合物信息的预测方法LBCC相比,随着候选关键蛋白质规模的增加,PCSL方法的优势保持稳定。这说明PCSL方法在预测关键蛋白质的过程中,其不但能进一步提高预测方法的精度,还能提升挖掘关键蛋白质的效率。相比于其他10种关键蛋白质预测方法,PCSL方法之所以性能较好,是因为其不但能通过构建的加权网络减少PPI网络的噪声的负面影响,而且从复合物信息和亚细胞定位信息两个角度考虑蛋白质的关键性,同时有通过改进CPPK寻优算法提升挖掘关键蛋白质的效率。
2.4.2 统计指标分析
为进一步分析PCSL方法的性能,基于文献[21]中的敏感度(SN)、特异性(SP)、阳性预测值(PPV)、阴性预测值(NPV)、F-测度(F-measure)和准确率(ACC)这6个统计指标与其他10种预测方法进行比较实验。由于从DIP数据库下载的酵母PPI网络中仅有1 167个关键蛋白,因此选取排序后的前1 167个蛋白质作为候选关键蛋白质,并对比各方法在6个统计指标值,以深入分析PCSL方法的识别性能。由于现有方法预测准确率提升幅度不大,为尽可能表现出各指标的精度,避免各指标数据重合,对指标数据保留小数点后4位。PCSL方法与其他10种方法的比较结果如表2所示。
从表2可以看出,相比于其他方法,PCSL方法的6个指标均有所提升。与预测性能最差的中心方法CC相比,6项指标分别高出16.34%、4.96%、16.34%、4.96%、16.34%和7.65%,与最好的方法LBCC相比各项指标仍具有较好优势。PCSL方法预测性能比其他10种预测方法更好的原因主要有两点:一是本文构建的加权PPI网络不仅降低了对原始PPI网络的依赖和网络中噪声数据带来的负面影响,提高了网络的真实性和可靠性,而且从拓扑特性、生物特性和空间属性3个方面考虑蛋白质之间的紧密联系;二是本方法综合考虑复合物信息和亚细胞定位信息来衡量蛋白质的关键性。
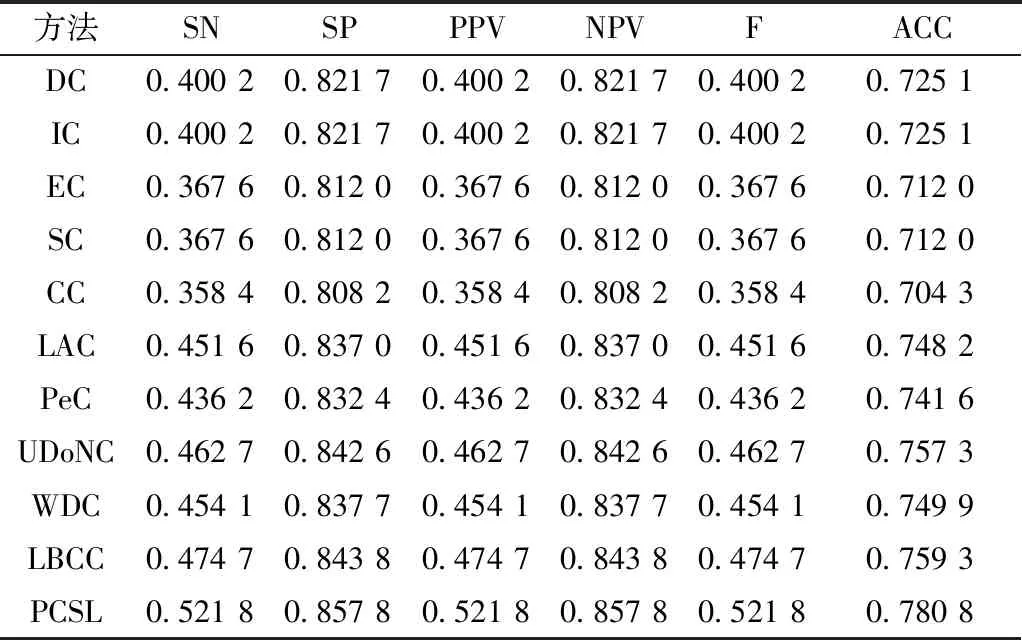
表2 PCSL方法与其他10种方法的统计指标比较Table 2 Comparison of the SN,SP,PPV,NPV,F and ACC between PCSL and other ten methods
3 结论
将关键蛋白质识别方法的主要挑战归纳为3个方面:①如何降低PPI网络中假阳性和假阴性数据的负面影响,从而构建更加真实可靠的PPI网络;②如何有效地整合多元生物和其他PPI网络相关信息,设计一个能够较好地衡量蛋白质关键性的度量方式;③如何选择合理的选择计算算法,以提升挖掘关键蛋白质的效率。
为改善关键蛋白质识别方法识别准确率不高的问题,首先利用PPI网络的拓扑特性、GO功能相似性和空间属性构建可靠的加权网络;其次,基于复合物信息和亚细胞定位信息设计一种衡量蛋白质关键性的度量;最后,设计一种应用于PPI网络寻优的试探策略,以提升挖掘关键蛋白质的效率。实验结果表明,利用DIP数据集进行关键蛋白质预测精度比较,PCSL方法的识别准确率高于被比较的10种预测方法。

