大白菜GID1家族基因鉴定及表达模式分析
宋晴晴 张慧敏 张烨 张一卉 李景娟 李化银 贺立龙 王凤德 高建伟


摘要:大白菜是我国分布最广、种植面积最大的蔬菜作物,其丰产对安定人民生活起着举足轻重的作用。GID1蛋白是赤霉素信号转导过程中的重要受体,在调控植物生长发育过程中发挥重要作用。本研究采用生物信息学分析方法对大白菜基因组中GID1家族基因进行鉴定,并对该基因结构、染色体分布、系统进化以及表达模式等进行分析。结果表明,大白菜基因组中含有5个GID1家族成员,分别位于4、5、6、7、9号染色体上,各家族成员都只含有1个内含子。系统进化结果显示,单子叶和双子叶植物的GID1蛋白明显处于两个不同分支,大白菜GID1家族成员与拟南芥GID1具有更近的亲缘关系。通过表达模式以及启动子顺式响应元件分析发现,大白菜GID1基因不仅参与生长发育调控还可能参与对不同环境因子的响应过程。
关键词:大白菜;GID1基因;生物信息学分析;表达模式分析
Abstract: Chinese cabbage is the vegetable with the widest distribution and the largest planting area in China. Its yield plays an important role in the stability of the people's life. GID1 protein is an important receptor in the process of gibberellin signal transduction and plays an important role in regulating plant growth and development. In this study, the GID1 family genes in the Chinese cabbage genome were identified by bioinformatics methods, and their gene structure, chromosome distribution, phylogenetics and expression patterns were analyzed. The results showed that the Chinese cabbage genome contained 5 GID1 gene members and was evenly distributed on chromosomes 4, 5, 6, 7 and 9. In addition, members of the Chinese cabbage GID1 family contain only one intron. In phylogenetic evolution, the GID1 family of proteins has distinct branches between dicotyledons and monocotyledons, while the GID1 family members of Chinese cabbage have a closer genetic relationship with Arabidopsis GID1. Through expression pattern and promoter cis-element analysis, it was found that the GID1 gene of Chinese cabbage not only participates in the regulation of growth and development but also may participate in the response process to different environmental factors. This study not only provides clues for us to understand the function of GID1 gene in Chinese cabbage, but also provides an important reference for further application in molecular breeding of Chinese cabbage.
Key words: Chinese cabbage; GID1 gene;Bioinformatics analysis; Expression pattern analysis
大白菜(Brassica rapa L. ssp. pekinensis)起源于中国,常年种植面积2×106 hm2左右,总产约1亿吨,是种植面积、产量最大的蔬菜作物,素有“国菜”之称。长期生产实践表明,大白菜产量与叶球大小直接相关并且受严格的遗传控制。因此,挖掘参与调控大白菜叶球大小的相关基因将不仅有助于阐明叶球发育的分子调控机制,还有助于通过分子辅助育种手段提高大白菜产量。
赤霉素(gibberellin, GA)作为一种重要的植物激素,参与调控植物生长和发育,如种子萌发、植株生长、花器官形成以及果实发育等[1,2]。GID1(GIBBERELLIN-INSENSITIVE DWARF1)蛋白是赤霉素信号转导过程中的重要受体,能特异性地结合活性GA,并进一步与DELLA蛋白结合形成复合体[3,4]。而这个复合体可以被特定的泛素E3连接酶复合体(SCFSLY1/GID2)多聚泛素化后被26S蛋白酶降解,从而产生GA效应[5]。2005年,人们在水稻中首次发现GID1蛋白,gid1-1突变体表现为植株矮化,对GA不敏感[6]。Nakajima等[7]研究发现拟南芥基因组存在GID1a、GID1b和GID1c三个成员,进一步研究[8]表明,三个成员中任何一个发生功能缺失突变对植株表型几乎没有影响,但同时发生功能缺失突变则可导致植株极其矮小,对GA完全不敏感。最近,Illouz-Eliaz等[2]发现另一种模式植物番茄基因組中也存在三个GID1成员,同样是在三者同时发生功能缺失时出现植株极其矮小的表型,从而说明GID1不同成员间存在功能冗余。然而,Cheng等[9]对桃树两个GID1成员的研究发现,一个基因(GID1c)发生突变即可导致植株矮小。因此,对于不同植物而言,GID1成员在调控生长发育的过程中存在一定分化,并且GID1基因在调控植物大小的发育过程中发挥作用。但目前尚未见关于大白菜GID1基因的研究报道。
基于上述分析,本研究首先在大白菜全基因组水平上对GID1家族基因进行鉴定,后对其在染色体上的分布、基因结构、系统进化以及表达模式等进行分析,以期了解GID1基因功能,并为其在大白菜分子育种中的进一步应用提供参考。
1材料与方法
1.1 大白菜GID1基因鉴定
本研究从拟南芥基因组网站(https://www.arabidopsis.org/)下载了拟南芥GID1家族成员的蛋白序列,并以其为探针分别在NCBI(https://www.ncbi.nlm.nih.gov)和大白菜基因组网站(http://brassicadb.org/brad/)上进行BLAST检索。提取最优比对结果,然后利用在线软件SMART (http://smart.embl-heidelberg.de)對候选基因进一步验证,确保其含有HGG和GXSXG蛋白保守结构域。
1.2 生物信息学分析
利用ExPASy Proteomics Server 工具(http://www.expasy.org/tools)中提供的ProtParam软件进行转录因子蛋白的分子量、理论等电点分析,GSDS软件(http://gsds.cbi.pku.edu.cn/)分析内显子/外含子结构,MEME软件(http://meme.nbcr.net/meme/)分析保守基序(Motif),SMART 软件(http://smart.embl-heidelberg.de)分析保守结构域,DNAMAN软件进行氨基酸序列的多重比对分析,MEGA6.0软件进行系统进化分析,MapChart软件进行染色体分布分析,PlantCARE软件(http://bioinform-atics.psb.ugent.be/webtools/pla-ntcare/html/)进行启动子区顺式作用元件分析。
番茄、水稻和玉米GID1家族成员的氨基酸序列下载自Phytozome基因组网站(https://phytozome.jgi.doe.gov/pz/portal.html)。
1.3 大白菜GID1基因表达模式分析
参照Tong等[10]的数据对BrGID1基因在大白菜根、茎、叶、花以及角果中的表达模式进行分析,RPKM值取对数值后利用TBtools软件绘制热图。
不同叶位叶片BrGID1基因表达模式分析所用大白菜品种为西白16(山东登海种业股份有限公司西由种子分公司产品)和夏绿50(香港惟勤企业有限公司)。其中,成熟期,西白16植株较大,整株重约7 kg,而夏绿50较小,重约1.5 kg。选取饱满一致的种子播于盛满营养土的方形塑料盆(7 cm×7 cm)中。待幼苗长至第2片真叶完全展开时,向叶片正反面喷洒100 μmol/L GA3,以喷水为对照(CK),并在处理后0、1、3 h采集第1片真叶进行表达模式分析。待长至5叶期时(第5片叶完全展开),按从下到上的顺序选取第1、3、5片叶作为样品进行表达模式分析。
本研究使用天根生化科技(北京)有限公司的植物总RNA提取试剂盒(目录号:DP432)提取RNA,宝生物工程(大连)有限公司的PrimeScript? RT reagent Kit with gDNA Eraser试剂盒(目录号:RR047A)合成cDNA、TB Green? Fast qPCR Mix试剂盒(目录号:RR430A)进行荧光实时定量PCR(RT-qPCR)。荧光实时定量PCR所用引物如表1所示,每个样品设3次生物学重复,利用2-△△CT法计算基因的相对表达水平,再使用TBtools软件绘制热图。
2结果与分析
2.1大白菜GID1基因家族成员鉴定
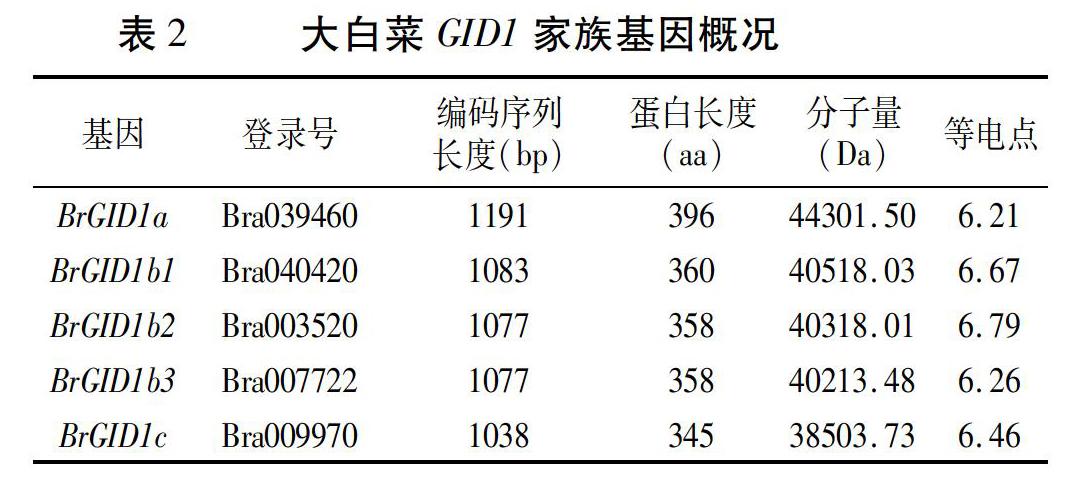
本研究利用相关软件对候选基因进一步验证发现,大白菜基因组中存在5个GID1家族基因。将这5个GID1基因与拟南芥GID1基因进行系统进化分析,发现Bra039460与AtGID1a基因在同一分支,Bra040420、Bra003520和Bra007722与AtGID1b基因在同一分支,而Bra009970则与AtGID1c基因位于同一分支(图1A)。因此,参照拟南芥GID1基因家族的命名方法并进一步根据5个基因在染色体上的相对位置(图1B),分别将其命名为BrGID1a、BrGID1b1、BrGID1b2、BrGID1b3和BrGID1c。利用GSDS软件对大白菜GID1基因外显子/内含子结构进行分析发现,5个成员均只含有一个内含子(图1C)。另外,对大白菜GID1家族基因不同成员进行分析发现,其CDS序列长度介于1038~1191 bp,预测编码蛋白长度在345~396aa之间,分子量在38503.73~44301.5 Da之间,等电点在6.21~6.79之间(表2)。
A:大白菜和拟南芥GID1基因系统进化分析;B:大白菜GID1基因在染色体上的位置分布;C:大白菜GID1基因外显子/内含子结构分析(标尺代表基因长度,绿色部分代表外显子区域,横线部分代表内含子)。
2.2大白菜GID1蛋白保守基序分析
利用在线分析软件MEME(http://meme.nbcr.net/meme/)对大白菜GID1家族蛋白的保守结构域进行预测,共得到10个Motif(保守基序)(图2)。其中Motif1、2、4、5、6、7、9同时存在于5个BrGID1蛋白,Motif10存在于BrGID1a和BrGID1c中,而Motif3、8则仅存在于BrGID1b1、BrGID1b2和BrGID1b3中。因此,本研究推测Motif1、2、4、5、6、7、9可能是维持BrGID1家族蛋白基本功能所必需的,而其它保守结构域在不同成员上的差异分布则可能是导致不同成员功能差异的原因。
此外,通过对大白菜GID1家族蛋白进行多重序列比对分析发现,它们的氨基酸序列一致性为73.67%,但均具有保守的激素敏感性脂肪酶活性的HGG和GXSXG结构域(图3)。Hirano等[11]研究表明,拟南芥GID1蛋白与GA或DELLA的蛋白结合区有13个结合功能域,从N端到C端依次是TWVLIS、LDR、FFHGGSF、HS、IYD、YRR、DGW、GDSSGGNI、GNI、MF、LDGKYF、WYW和GFY。而对大白菜BrGID1蛋白分析发现,BrGID1b3蛋白TWVLIS结构域中的L被F取代,BrGID1a蛋白GNI结构域中的I被V取代,其它区域则都是保守的。以上结果表明,大白菜BrGID1蛋白在GA信号转导中发挥重要功能的氨基酸残基与模式植物一样都是保守的,它们在GA作用机制中的受体功能可能是完整的。
[2]Illouz-Eliaz N, Ramon U, Shohat H, et al. Multiple gibberellin receptors contribute to phenotypic stability under changing environments[J]. Plant Cell, 2019, 31(7): 1506-1519.
[3]Hedden P. Gibberellins close the lid[J]. Nature, 2008, 456: 455-456.
[4]Sun T P. Gibberellin metabolism, perception and signaling pathways in Arabidopsis[J]. Arabidopsis Book, 2008, 6(1): 1-28.
[5]Sun T P. Gibberellin-GID1-DELLA: a pivotal regulatory module for plant growth and development[J]. Plant Physiology, 2010, 154(2): 567-570.
[6]Ueguchi-Tanaka M, Ashikari M, Nakajima M, et al. GIBBERELLIN INSENSITIVE DWARF1 encodes a soluble receptor for gibberellin[J]. Nature, 2005, 437: 693-698.
[7]Nakajima M, Shimada A, Takashi Y, et al. Identification and characterization of Arabidopsis gibberellin receptors[J]. Plant J., 2006, 46(5): 880-889.
[8]Griffiths J, Murase K, Rieu I, et al. Genetic characterization and functional analysis of the GID1 gibberellin receptors in Arabidopsis[J]. Plant Cell, 2006, 18(12): 3399-3414.
[9]Cheng J, Zhang M M, Tan B, et al. A single nucleotide mutation in GID1c disrupts its interaction with DELLA1 and causes a GA-insensitive dwarf phenotype in peach[J]. Plant Biotechnology Journal, 2019, 17(9): 1723-1735.
[10]Tong C, Wang X W, Yu J Y, et al. Comprehensive analysis of RNA-seq data reveals the complexity of the transcriptome in Brassica rapa[J]. BMC Genomics, 2013, 14: 689.
[11]Hirano K, Nakajima M, Asano K, et al. The GID1-mediated gibberellin perception mechanism is conserved in the lycophyteSelaginella moellendorffii but not in the bryophyte Physcomitrella patens[J]. Plant Cell, 2007, 19(10): 3058-3079.
[12]The Brassica rapa Genome Sequencing Project Consortium, Wang X, Wang HZ, et al. The genome of the mesopolyploid crop species Brassica rapa[J]. Nat. Genet., 2011, 43(10): 1035-1039.
[13]Cheng F, Mandáková T, Wu J, et al. Deciphering the diploid ancestral genome of the mesohexaploid Brassica rapa[J]. Plant Cell, 2013, 25(5): 1541-1554.
[14]Tanaka N, Matsuoka M, Kitano H, et al. gid1, a gibberellin-insensitive dwarf mutant, shows altered regulation of probenazole-inducible protein (PBZ1) in response to cold stress and pathogen attack[J]. Plant Cell and Environment, 2006, 29(4): 619-631.
[15]Chen L, Cao T, Zhang J, et al. Overexpression of OsGID1enhances the resistance of rice to the brown planthopper Nilaparvata lugens[J]. Int. J. Mol. Sci., 2018, 19(9):2744.
[16]Ariizumi T, Murase K, Sun T P, et al. Proteolysis-independent downregulation of DELLA repression in Arabidopsis by the gibberellin receptor GIBBERELLIN INSENSITIVE DWARF1[J]. Plant Cell, 2008, 20(9): 2447-2459.
[17]Powell A E, Lenhard M. Control of organ size in plants[J]. Current Biology, 2012, 22(9): R360-R367.

