SMA 小鼠中相关剪接因子表达与SMN2 外显子7 列入比率关系研究
杜利莉,马 钰,柏文清,吴刘成,邵义祥*
(1.南通大学神经再生重点实验室,江苏 南通 226001; 2.南通大学实验动物中心,江苏 南通 226001;3.南通大学杏林学院,江苏 南通 226001)
脊髓性肌萎缩症(spinal muscular atrophy,SMA)是由运动神经元生存1(survival of motor neuron1,SMN1)基因遗传缺陷引起,SMN1 基因编码SMN 蛋白,广泛表达于所有真核细胞,为运动神经元存活所必需。 较低水平的蛋白质导致脊髓前角运动神经元细胞功能丧失,随后全身骨骼肌萎缩,其他身体系统也可能受到影响,特别是早期发病的患者。SMA 是婴儿死亡最常见的遗传原因[1-2]。
SMA 的致病基因SMN1 是位于5q13 的基因,患者常因其外显子7 和8 或仅外显子7 同源缺失,从而导致不能表达有功能的全长SMN 蛋白[3]。 而人类的SMN1 基因在这个重复片段区域还存在唯一与之高度同源的SMN2 基因,两者的关键不同之处在于外显子7 第6 位核苷酸由SMN1 中的C 转变为SMN2 中的T,虽然没有造成翻译中氨基酸的改变,却严重影响了该外显子列入(exon inclusion)[4]。SMN2 的成熟转录产物约90%不包含外显子7,而没有外显子7 的蛋白(称作SMNΔ7)基本没有功能且极不稳定[5]。 是否能够通过增强SMN2 外显子7 的保留来获得全长(full-length,FL)的SMN,从而有效降低SMA 患者的临床症状? 这个问题为研究者所关注,有望使SMN2 成为治疗SMA 的理想靶点[6]。仅含有Smn 基因一个拷贝的小鼠,由于转入人的SMN2 基因能够挽救其Smn 纯合子敲除的胚胎致死性并延迟至出生后10 d 左右死亡,且症状与人类Ⅰ型的SMA 相似[7],从而使得SMA 小鼠成为目前国际相关领域公认的良好模型。 故在SMA 小鼠中,通过研究SMN2 剪接来获得全长SMN 蛋白,从而观察SMA 小鼠的病情及寿命是否有所改善,探讨脊髓性肌萎缩症有效的临床治疗方案,是当前国际SMA 研究关注的焦点。 本实验通过研究SMA Ⅰ型小鼠不同组织中SMN2 外显子7 列入与相关剪接因子的表达差异,筛选与SMN2 外显子7 列入具有密切关系的剪接因子,以期待发现可能参与SMA 相关基因SMN2 剪接的新调控因子,为SMA 疾病的治疗提供新思路。
1 材料和方法
1.1 实验动物
2 只雌性SMA Ⅲ型小鼠(基因型Smn-/-SMN22tg/2tg),1 只雄性杂合子Smn 敲除小鼠(基因型Smn+/-),6 周龄,体重18 ~22 g,用以交配繁殖。SMA Ⅰ型小鼠(基因型Smn-/-SMN22tg/0)、SMA Ⅰ型对照小鼠(基因型Smn+/-SMN22tg/0),4 d 龄,体重1~4 g,雌雄不限,各4 只,用以进行实验。 小鼠背景品系均为SPF 级FVB 近交系小鼠,均来源于南通大学实验动物中心[SCXK(苏)2014-0001]并在南通大学实验动物中心SPF 级屏障环境饲养繁殖。 动物实验在南通大学实验动物中心比较医学实验室进行[SYXK(苏)2017-0046]。 实验动物的使用坚持3R 原则,且所有动物实验均经南通大学实验动物伦理管理委员会审查(伦理审批号:20180822-002)。
1.2 主要试剂与仪器
小鼠基因型快速鉴定试剂盒(PD101-01)、TRIzol 总RNA 抽提试剂(R401-01)、反转录酶试剂盒(R021-01)、2×Taq master mix(P111),SYBR Green 荧光PCR 试剂盒(Q711-02)购自南京诺唯赞生物科技公司;Veriti 96-well PCR 扩增仪购自美国Applied Biosystems 公司;MICROCL 21R 台式冷冻离心机购自美国Thermo Fisher Scientific 公司;EPS-300 数显稳压温流电泳仪、HE-120 多功能水平电泳槽购自中国上海天能科技;Native-PAGE 垂直电泳系统购自美国Apogee 公司;BIO-RAD Gel Doc XR +凝胶成像分析系统购自美国Bio-Rad 公司;G:box Chemi XL 1.4 成像系统购自英国Syngene 公司;Light cycler 96 荧光定量PCR 仪购自瑞士ROCHE公司。
1.3 实验方法
1.3.1 小鼠繁殖与取材
雄性杂合子Smn 敲除小鼠和雌性SMA Ⅲ型小鼠交配,取同窝出生4 d 龄的后代,通过基因型鉴定,区分SMA Ⅰ型及对照小鼠,并分别取4 只SMAⅠ型小鼠(Smn-/-SMN22tg/0) 和4 只对照小鼠(Smn+/-SMN22tg/0)心脏、肝、脾、肺、肾、脑、脊髓、肌肉组织。
1.3.2 RT-PCR
按照TRIzol 试剂盒及反转录试剂盒提取总RNA 并合成cDNA,采用Cy5 标记的引物进行RTPCR,94°C 30 s,55°C 30 s,72°C 25 s,28 个循环,取8 μL 扩增样品,用2%琼脂糖凝胶电泳检验扩增结果,剩余样品用于非变性PAGE 定量分析,引物信息为:E6-F:5’-ATAATTCCCCCACCACCTCCC-3’,E8-467R:5’-TTGCCACATACGCCTCACATAC-3’。
1.3.3 非变性PAGE 及SMN2 外显子7 列入
6%非变性PAGE 胶110 V 电压下进行电泳,3 h后结束电泳。 G:BOX 系统显影,用Image J 分析条带灰度值, 并计算SMN2 基因外显子7 列入(Inc%),公式如下:
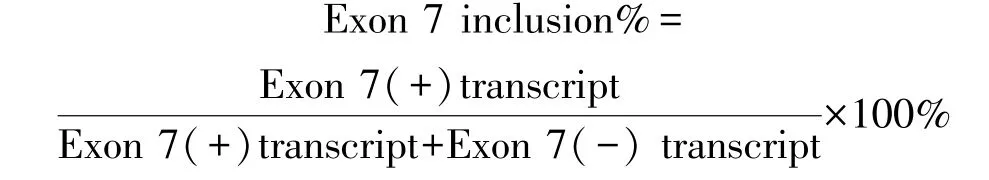
1.3.4 实时荧光定量PCR
反应体系:SYBR Green Master Mix 10 μL,上游引物1 μL,下游引物1 μL,cDNA 1 μL,补灭菌双蒸水7 μL 至20 μL 体系,每个样品设置3 个重复。 反应条件:95℃预变性4 min,94℃下变性15 s,60℃下退火20 s,72℃延伸20 s 并读取荧光值,45 次循环,循环后设置55℃~90℃,每隔0.3℃读荧光值生成熔解曲线。 所有设定保存后运行程序。 反应完成后,选择PCR base line subtracted 模式进行数据分析和修正。 在调整baseline cycles 和计算threshold value(阈值)后,得出cycle threshold(Ct 值),ΔΔCt法计算出样本的相对表达量(RQ 值)。 在gene bank中查找并下载相关基因的mRNA 序列及相关信息,实时荧光定量PCR 引物采用Primer premier 5.0 软件,严格按照相关原则设计。 引物信息见表1。
1.4 统计学方法
本实验数据采用STATA 7.0 和GraphPad Prism 5.0 进行数据处理和统计学分析。 计量数据用平均数±标准差(±s)表示,组间比较采用t 检验,P<0.05 表示两组间差异显著,P<0.01 表示两组间差异极显著。
2 结果
2.1 SMN2 外显子7 列入在脑和脊髓中与肝、肾和肌肉相比显著增加
通过RT-PCR 和非变性PAGE 凝胶电泳分析,在出生后第4 天(P4)的SMA Ⅰ型小鼠大脑和脊髓中,SMN2 表达全长的有功能RNA 比例显著高于其它三个非神经组织(图1)。 表明SMA Ⅰ型小鼠中,SMN2 mRNA 在神经与非神经组织中的剪接差异机制与其体内可能差异表达剪接因子密切相关。
2.2 HNRNP 剪接因子基因家族在SMA Ⅰ型和对照小鼠神经与非神经组织中的mRNA 水平
核内不均一核糖核蛋白(HNRNPs)是主要存在于细胞核中的由多个蛋白组成的复合体,其主要功能是结合RNA 参与转录后修饰,在新合成的RNA(pre-mRNA)过程中不可缺少,是成熟mRNA 形成的重要剪接因子[8]。 如图1 所示,本实验检测了出生后第4 天SMA Ⅰ型和对照小鼠的神经与非神经组织中HNRNP 家族中的Hnrnpk、Hnrnpl、Hnrnpll、Hnrnpu、 Hnrnpd、 Hnrnph3、 Hnrnpm、 Hnrnpf 以 及Hnrnph2 基因的mRNA 水平。 以心脏作为对照,这些剪接因子在心脏、肝及肌肉组织中表达相对较低,在脾、肺和肾中表达最高,在神经组织即大脑和脊髓中表达相对较高。

表1 引物序列Table 1 Primer sequences
2.3 SR 剪接因子基因家族在SMA Ⅰ型和对照小鼠神经与非神经组织中的mRNA 水平
富含丝氨酸和精氨酸剪接因子(serine/argininerich splicing factor,SRSF),常被简称为SR 蛋白,是一种涉及RNA 剪接的保守蛋白家族[9]。 如图3 所示,本实验检测了SR 家族中的Srsf10、Srsf1、Srpk1、Srsf3、Srsf6、Srsf2、Srsf4、Srsf5、Srsf7 以及Srsf9 基因在出生后第4 天的SMA Ⅰ型和对照小鼠神经与非神经组织中的mRNA 水平。 以心脏作为对照,这些剪接因子基因在心脏、肝及肌肉中表达相对较低,在脾、肺和肾中表达最高,在神经组织即大脑和脊髓中表达相对较高。
2.4 NOVA 剪接因子基因家族在SMA Ⅰ型和对照小鼠神经与非神经组织中的mRNA 水平
神经肿瘤腹侧抗原(neuro-oncological ventral antigen,NOVA)家族,有NOVA1 和NOVA2 两个成员,其作为神经元特异性剪接因子,在运动神经元中表达丰富[10]。 本实验检测了NOVA 家族中的Nova1 和Nova2 基因在出生后第4 天的SMA Ⅰ型和对照小鼠神经与非神经组织中的mRNA 水平。 以心脏作为对照,这些剪接因子基因在心脏、肝、脾、肾及肌肉中表达相对较低,在大脑和脊髓中表达最高,在肺中表达相对较高(见图4)。
2.5 HNRNP 剪接因子基因家族在SMA Ⅰ型和对照小鼠神经与非神经组织中的mRNA 水平差异化比较
通过比较HNRNP 剪接因子在出生后第4 天的SMA Ⅰ型和对照小鼠中的mRNA 差异水平,如图5可知,与对照小鼠相比,HNRNP 家族不同基因分别在SMA Ⅰ型小鼠不同组织中均有显著性差异。 其中,与对照小鼠相比,Hnrnph2 在SMA Ⅰ型小鼠脊髓中显著下调。
2.6 SR 剪接因子基因家族在SMA Ⅰ型和对照小鼠神经与非神经组织中mRNA 水平差异化比较
通过比较SR 剪接因子基因在出生后第4 天的SMA Ⅰ型和对照小鼠中的mRNA 差异水平,如图6可知,SMA Ⅰ型小鼠中,SR 家族不同基因分别在不同组织中与对照小鼠比较,均有显著性差异,其中,与对照小鼠相比,Srsf7 和Srsf9 在SMA Ⅰ型小鼠脊髓中显著上调,Srsf4 和Srsf10 在大脑中显著下调。
2.7 NOVA 剪接因子基因家族在SMA Ⅰ型和对照小鼠神经与非神经组织中mRNA 水平差异化比较
通过比较NOVA 剪接因子基因在出生后第4天的SMA Ⅰ型和对照小鼠中的mRNA 差异水平,如图7 可知,Nova1 和Nova2 基因分别在SMA 小鼠的不同组织中和对照小鼠比较,有显著性差异。 其中,与对照小鼠相比,在SMA Ⅰ型小鼠中,Nova2 在脊髓中显著上调。
3 讨论
脊髓性肌萎缩症是由于SMN1 基因功能丧失而引起的一种神经退行性疾病,表现为脊髓α 运动神经元坏死,导致进行性肌肉萎缩、瘫痪甚至死亡[11]。而与SMN1 序列主要有10 个碱基差异的高度同源的SMN2 基因,由于其可被调控的特殊选择性剪接即外显子7 跳跃模式,并能翻译出全长的SMN 蛋白,使得其成为治疗SMA 疾病的“靶基因”[12]。 尽管已经研发出有效治疗SMA 的药物并获批上市[13],但在病变累及多系统[14]的SMA 中,其仅针对特定组织且不能起到彻底治愈的效果,同时关于SMN2 外显子7 列入是否具有组织间差异及其是否是导致SMA 多系统病变的原因等相关问题是未知的。
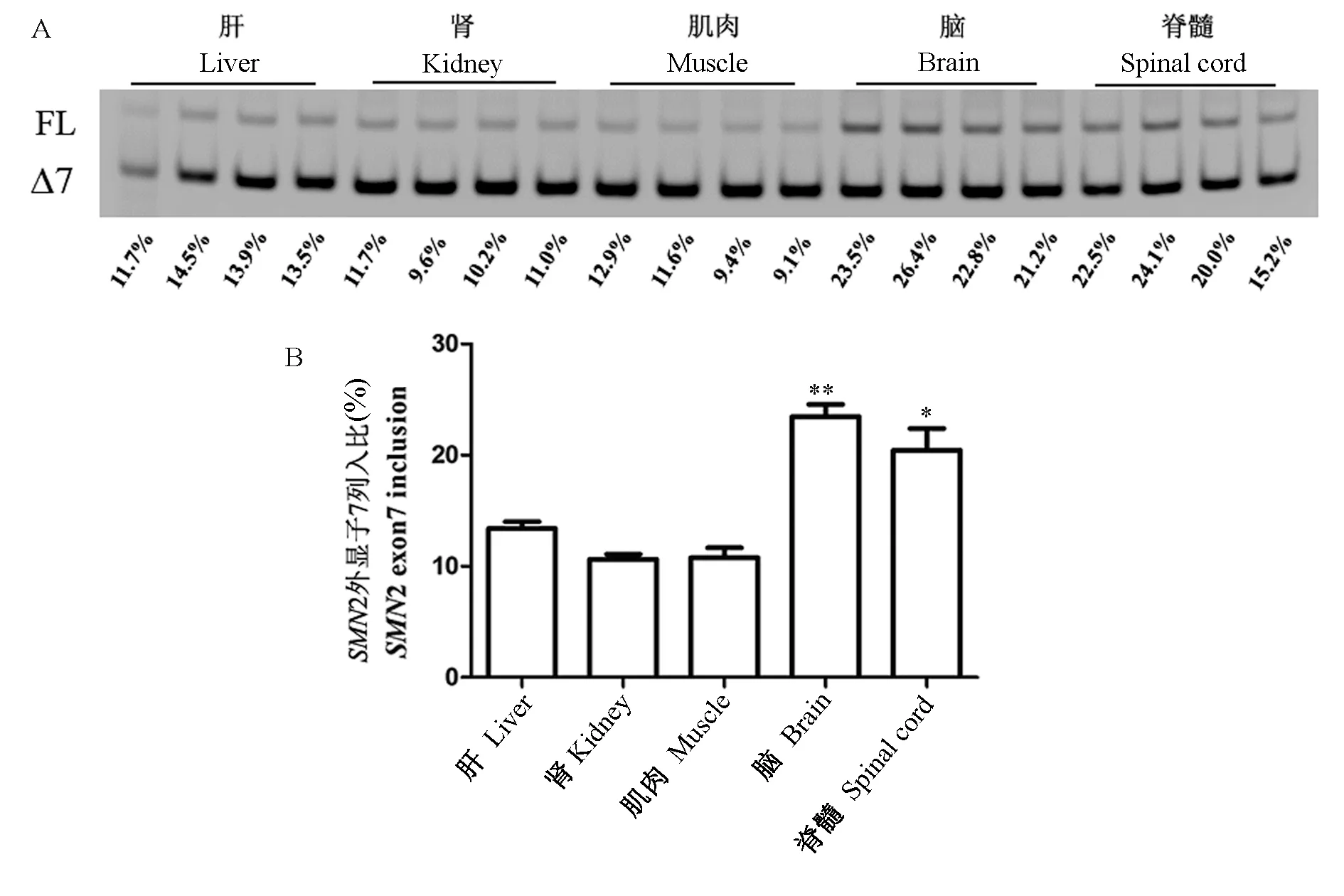
图1 SMN2 外显子7 在第4 天SMA Ⅰ型小鼠大脑和脊髓的列入比显著高于肝、肾和肌肉组织Note. A, Non-denaturing PAGE electrophoresis showing the inclusion ratio of SMN2 exon 7 in central and peripheral tissues. B,Bar chart showing the inclusion ratio of SMN2 exon 7 based on gray level statistics. Compared with the liver,*P <0.05,**P <0.01.Figure 1 The inclusion ratio of SMN2 exon 7 in brain and spinal cord tissues was significantly higher than that in liver, kidney, and muscle tissue from SMA type I mice at postnatal day 4

图2 HNRNP 剪接因子基因家族在出生后第4 天的SMA Ⅰ型和对照小鼠的心脏、肝、脾、肺、肾、肌肉、大脑和脊髓中的mRNA 水平Note. A, B, and C, QPCR analysis results of Hnrnpk, Hnrnpl, Hnrnpll, Hnrnpu, Hnrnpd, Hnrnph3, Hnrnpm, Hnrnpf, and Hnrnph2 gene expression in different tissues from SMA type I control mice at postnatal day 4. D, E, and F, QPCR analysis results of Hnrnpk, Hnrnpl, Hnrnpll,Hnrnpu, Hnrnpd, Hnrnph3, Hnrnpm, Hnrnpf, and Hnrnph2 gene expression in different tissues from SMA type I mice at postnatal day 4. Compared with the heart,*P <0.05,**P <0.01.The mRNA level of the control group was 1.Figure 2 mRNA expression levels of HNRNPs in the heart, liver, spleen, lung, kidney, muscle, brain,and spinal cord of type I and control SMA mice at postnatal day 4

图3 SR 剪接因子基因家族在出生后第4 天的SMA Ⅰ型和对照小鼠的心、肝、脾、肺、肾、肌肉、大脑和脊髓中的mRNA 水平Note. A, B, C, and D, QPCR analysis results of Srsf10, Srsf1, Srpk1, Srsf3, Srsf6, Srsf2, Srsf4, Srsf5, Srsf7, and Srsf9 gene expression in different tissues from SMA type I control mice at postnatal day 4. E, F, G, and H, QPCR analysis results of Srsf10, Srsf1, Srpk1, Srsf3, Srsf6,Srsf2, Srsf4, Srsf5, Srsf7, and Srsf9 gene expression in different tissues from SMA type I mice at postnatal day 4. Compared with the heart,*P <0.05,**P <0.01.The mRNA level of the control group was 1.Figure 3 mRNA expression levels of SRs in the heart, liver, spleen, lung, kidney, muscle, brain,and spinal cord from type I and control SMA mice at postnatal day 4

图4 NOVA 剪接因子基因家族在出生后第4 天的SMAⅠ型和对照小鼠的心脏、肝、脾、肺、肾、肌肉、大脑和脊髓中的mRNA 水平Note. A, QPCR analysis results of Nova1 and Nova2 gene expression in different tissues from SMA type I control mice at postnatal day 4. B, QPCR analysis results of Nova1 and Nova2 gene expression in different tissues from SMA type I mice at postnatal day 4. Compared with the heart,*P <0.05,**P <0.01.The mRNA level of the control group was 1.Figure 4 mRNA expression levels of NOVAs in the heart, liver, spleen, lung, kidney, muscle, brain,and spinal cord from type I and control SMA mice at postnatal day 4
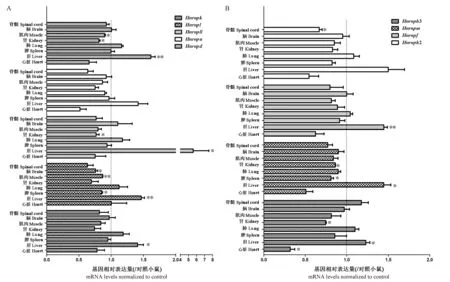
图5 HNRNP 剪接因子基因家族在出生后第4 天的SMA Ⅰ型和对照小鼠心脏、肝、脾、肺、肾、肌肉、大脑和脊髓中的mRNA 水平差异化比较Note. A, QPCR analysis results of the differences in Hnrnpk, Hnrnpl, Hnrnpll, Hnrnpu, and Hnrnpd gene expression in different tissues. B, QPCR analysis results of the difference between Hnrnph3, Hnrnpm, Hnrnpf, and Hnrnph2 gene expression in different tissues. Compared with the corresponding organization in control mice,*P <0.05,**P <0.01.The mRNA level of the control group was 1.Figure 5 Comparison of differences in HNRNP mRNA levels in the heart, liver, spleen, lung, kidney, muscle,brain, and spinal cord from type I and control SMA mice at postnatal day 4
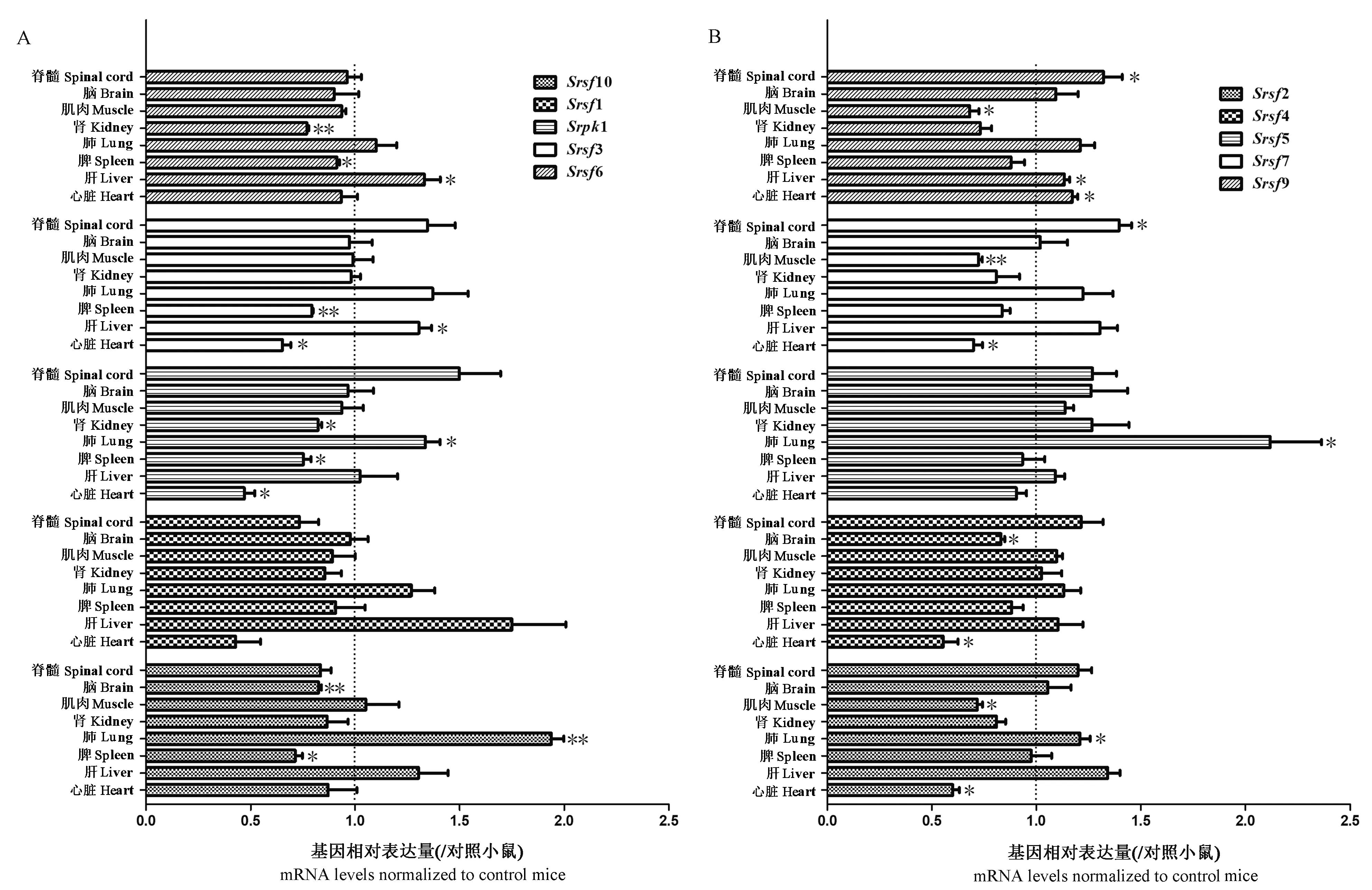
图6 SR 剪接因子基因家族在出生后第4 天的SMA Ⅰ型和对照小鼠心脏、肝、脾、肺、肾、肌肉、大脑和脊髓中的mRNA 水平差异化比较Note. A, QPCR analysis results of the differences in Srsf10, Srsf1, Srpk1, Srsf3, and Srsf6 gene expression in different tissues. B, QPCR analysis results of the differences in Srsf2,Srsf4, Srsf5,Srsf7,and Srsf9 gene expression in different tissues. Compared with the corresponding organization in control mice,*P <0.05,**P <0.01.The mRNA level of the control group was 1.Figure 6 Comparison of differences in SRs mRNA levels in the heart, liver, spleen, lung, kidney,muscle, brain, and spinal cord from type I and control SMA mice at postnatal day 4
本研究发现,在SMA Ⅰ型小鼠中,SMN2 外显子7 的列入具有组织间差异,且在神经组织中的列入显著高于非神经组织。 而外显子是否列入是由剪接因子所调控的选择性剪接来决定的,因此,有必要筛选在神经组织与非神经组织存在的差异表达剪接因子,寻找促进SMN2 外显子7 在神经组织中列入的剪接因子,为SMA 疾病提供新的治疗方法。 而本实验重点检测了SMA Ⅰ型小鼠8 个组织中HNRNP、SR 及NOVA 三类经典剪接因子的mRNA 水平,发现这21 个剪接因子的mRNA 水平均具有组织间差异,其中HNRNP 家族9 个成员及SR家族10 个成员均在非神经组织中表达最高,而NOVA 家族2 个成员均在神经组织大脑及脊髓中表达最高,与SMN2 外显子7 在神经组织中高列入呈正相关。
而在SMA Ⅰ型对照小鼠中,我们发现剪接因子表达与SMA Ⅰ型小鼠中检测到的表达变化相似,表明无论是在SMA Ⅰ型还是对照小鼠中,这些剪接因子基因水平表达均具有组织间差异,尤其是NOVA家族成员均在神经组织中高表达,再次证明了NOVA 是神经特异性表达的剪接因子[15]。 进一步对剪接因子的表达差异进行研究,发现与对照小鼠相比,SMA Ⅰ型小鼠脊髓组织中,Srsf7、Srsf9 及Nova2 显著上调,与SMN2 外显子7 在脊髓中高列入呈正相关,SRSF7、SRSF9 及NOVA2 极有可能参与SMN2 的剪接调控。 而在其它特定组织中,某些剪接因子显著变化,如心脏组织中HNRNPH3、SRSF3 显著下调,可能是引起SMA 疾病多系统病变的原因之一。

图7 NOVA 剪接因子基因家族在SMA Ⅰ型和对照小鼠出生后第4 天心、肝、脾、肺、肾、肌肉、大脑和脊髓中的mRNA 水平差异化比较Note. QPCR analysis results of the differences in Nova1 and Nova2 gene expression in different tissues. Compared with the corresponding organization in control mice,*P <0.05,**P <0.01.The mRNA level of the control group was 1.Figure 7 Comparison of differences in NOVAs mRNA levels in the heart, liver, spleen, lung, kidney,muscle, brain, and spinal cord from type I and control SMA mice at postnatal day 4
导致SMA 疾病的最直接原因是运动神经元存活蛋白(SMN)合成减少,功能丧失。 而SMN 是一种以其在SMN-Gemin 多蛋白复合物中的管家作用而闻名的蛋白,其参与了小核蛋白(SnRNP) 的组装[16],调控前体mRNA 剪接,同时还参与机体多种生理功能,包括应激反应、轴突运输、细胞骨架动力学、线粒体和生物能量途径以及泛素途径等[17]。Groen EJN 等[18]在严重型SMA 小鼠模型中发现SMN 蛋白的表达具有组织间差异,且在神经组织中表达最高,与本实验中SMN2 外显子7 列入的组织间差异相一致,SMN 可能是调控SMN2 外显子7 组织特异性列入的因子之一。 与此同时,HNRNP 家族成员HNRNPR,已被证明能够与SMN 蛋白在运动神经元中直接相互作用[19]。 同时,由RNA 识别基序(RRM)区和由丝氨酸和精氨酸的重复序列组成的RS 区两个结构域组成[8]的SR 蛋白,已发现其在不同组织中差异表达[20],且能够调控SMN 蛋白的表达[21]。 而本实验在研究时,发现Hnrnph2 显著下调,Srsf7、Srsf9 及Nova2 表达显著上调,这可能是因为受到SMN 蛋白的调控,从而表达发生上调或下调。
除此之外,神经特异性表达的剪接因子NOVA1或NOVA2 与多种神经系统疾病有密切关系[22],本研究发现SMA 小鼠中Nova1 及Nova2 表达与SMA相关基因SMN2 外显子7 列入呈正相关,表明NOVA 家族与SMA 疾病之间存在着某种联系。 同时,NOVA 与HNRNPK、MER1 等蛋白均具有KH 结构域[23],且具有调控剪接的作用,而其调控剪接的作用是与靶mRNA YCAY[24-25]元件结合来实现的,通过序列分析,发现在SMN2 外显子7 内有该序列,提示NOVA1 或NOVA2 可能结合该序列直接调控SMA 相关基因SMN2 剪接。 而有关SRSF7/9 和NOVA 剪接因子对SMN2 外显子7 列入的具体调控机制有待深入研究。
综上所述, SMA Ⅰ型小鼠中,SMN2 外显子7列入及相关剪接因子的表达变化具有组织间差异,且神经组织中,Srsf7/9 和Nova1/2 高表达与SMN2外显子7 列入显著提高呈正相关,剪接因子SRSF7/9 及NOVA1/2 可能参与SMN2 基因的剪接调控。

