广域照射拉曼光谱技术结合簇类独立软模式法快速鉴别原料肉及掺假肉
徐记各 韩莹 忻欣 史喜菊


摘 要:应用广域照射(wide area illumination,WAI)拉曼光谱技术与簇类独立软模式(soft independent modeling of class analogy,SIMCA)法,结合多元散射校正(multiplicative scatter correction,MSC)和光谱仪降噪和波长标定(spectrometer noise reduction and wavelength calibration,SNRWC)降噪技术,建立鸭、羊、猪3 种原料肉及掺假羊肉的定性识别模型。结果表明:经MSC与SNRWC处理后,鸭、羊、猪3 种原料肉之间及羊肉、掺假羊肉之间的主成分分析结果具有明显的聚类趋势,在此基础上建立SIMCA定性分类模型,对不同产地的37 个原料肉样品种属进行定性鉴别,识别正确率达100%;对4 个掺假羊肉和5 个未掺假羊肉样品识别正确率也为100%。因此,拉曼光谱分析技术结合有效的数据前处理方法及化学计量学方法可对鸭、羊、猪原料肉种属及掺假羊肉进行鉴别。与常规方法相比,该检测过程快速、方便,并且无需样品前处理。
关键词:广域照射;拉曼光谱;原料肉;快速鉴别;簇类独立软模式方法;主成分分析
Abstract: Qualitative recognition models were established by using wide area illumination (WAI) Raman scheme and soft independent modeling of class analogy (SIMCA) for rapid identification of duck, lamb, pork and adulterated meat. The spectra of all samples were pre-processed by multiplicative scatter correction (MSC) and spectrometer noise reduction and wavelength calibration (SNRWC) method and then principal component analysis was implemented to observe the clustering trend. It turned out that most of the duck, lamb and pork samples as well as most of the lamb samples and adulterations were well separated. Finally, the qualitative classification models were established by using SIMCA. All validation samples were identified by the SIMCA model with an accuracy of 100%, including 37 meat samples from different species and geographical origins, as well as four adulterated and five unadulterated lamb samples. Therefore, the WAI Raman scheme coupled with chemometrics could distinguish among lamp, duck, and pork and adulterated lamb, and it proved to be more fast, convenient, without the need for any sample pretreatment compared with the routine method.
Keywords: wide area illumination; Raman spectroscopy; raw meat; rapid discrimination; soft independent modeling of class analogy; principal component analysis
DOI:10.7506/rlyj1001-8123-20191211-303
中图分类号:TS251.7 文献标志码:A 文章编号:1001-8123(2020)05-0070-06
动物源性食品是人类饮食结构中的重要组成部分。肉类的營养价值、风味以及消费者习惯等的不同导致其价格差异较大,并且利益驱使导致市场上肉类掺假现象持续存在。据国家统计局数据显示,2015年起猪肉在饮食中的比重大幅下降,牛、羊肉的比重持续增长。这反映了人们的饮食结构由低价猪肉消费转变为高价牛、羊肉消费,导致市场上出现了以相对廉价的鸭肉冒充牛、羊肉的乱象。这不仅扰乱了正常的市场竞争秩序,还严重损害了消费者的利益。因此,开发肉类种属及掺假的快速鉴别技术成为肉类市场发展的迫切需求。动物源性食品种属鉴别通常依据外观和气味进行感官判别,但无法满足监管需要。实验室分析方法主要是以蛋白质为基础的酶联免疫吸附分析法、免疫层析试纸条法[1-4]和以核酸检测为基础的聚合酶链式反应[5-8]。近几年近红外光谱技术也被用于鉴别原料肉的掺假[9-10]。然而这些检测方法损坏样品、费时费力且易受水分的影响,不能满足口岸肉类种属和掺假鉴别的时效性和可靠性要求。
拉曼光谱技术是近几年发展起来的一种分子光谱分析技术,它可提供分子振动和转动的信息,属于分子指纹光谱,是分析物质组成和结构的理想工具,已在毒品检测、考古、石油化工、材料、医学、药物和环保等领域被广泛应用[11-18]。相关研究表明,拉曼光谱技术可应用于肉类鉴别,但尚缺乏广域照射(wide area illumination,WAI)拉曼光谱技术的报道[19-21]。WAI拉曼光谱技术是一种新型光谱技术,其激光光斑直径远大于传统激光光斑直径(<500 μm),极大地提高了信号收集的代表性和操作方便性[22]。Kim等[23]使用WAI拉曼系统(直径6 mm)和实时强度校正方法检测萘普生药片中活性成分的含量,方法的重现性和准确度均较好。采用WAI拉曼技术采集数据时无需传统拉曼光谱采集时的复杂调焦过程,这极大降低了对测试人员的专业要求。拉曼光谱为弱信号光谱,肉类样品尤其是瘦肉样品的拉曼效应较弱[24]。此外,采用拉曼光谱鉴别肉类种属时对肉类组织类型具有限制性。De Biasio等[25]使用显微拉曼光谱技术鉴别鸡肉、猪肉、火鸡、牛肉、马肉和羊肉的瘦肉组织;Beattie等[26]利用拉曼光谱技术对鸡肉、牛肉、羊肉和猪肉的脂肪组织进行分类。然而,同时使用原料肉中瘦肉和脂肪组织的拉曼光谱信息鉴别肉类种属鲜有报道。
本研究同时采集动物瘦肉和脂肪组织的拉曼光谱数据,采用光谱仪降噪和波长标定(spectrometer noise reduction and wavelength calibration,SNRWC)法对数据进行降噪预处理,结合多元散射校正(multiplicative scatter correction,MSC)和均值中心化方法进行数据前处理,采用簇类独立软模式(soft independent modeling of class analogy,SIMCA)法建立鸭肉、羊肉和猪肉原料肉的动物种属鉴别分析模型,同时建立真假羊肉的预测模型,以期实现对掺假羊肉的鉴别以及满足口岸检测任务的多种需求。
1 材料与方法
1.1 材料与试剂
本实验所用肉类样品均来自北京海关技术中心,样品包含不同种属、部位、产地的肉类,每种肉类样品种属均经北京海关技术中心疯牛病实验室检测确认。
1.2 仪器与设备
ExR810广域拉曼光谱仪 西派特(北京)科技有限公司;KMG-W905绞肉机 康佳集团股份有限公司。
1.3 方法
1.3.1 样品信息
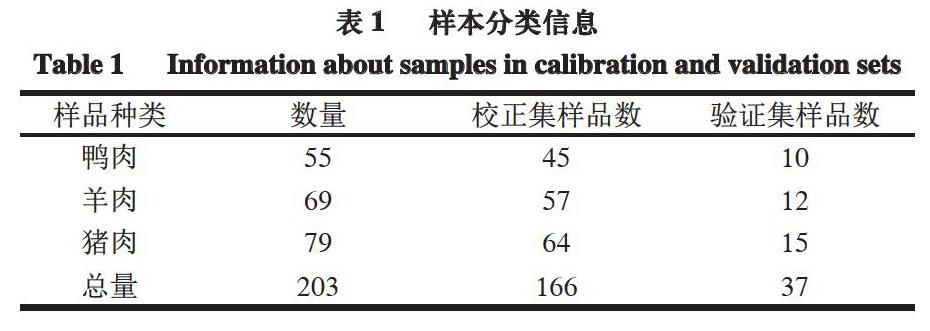
鸭肉、羊肉和猪肉光谱数据的数量、校正集及验证集数据信息如表1所示。
各取羊里脊、羊后腿、羊前腿、羊腩、去骨羊肉40 g绞成肉糜,制备未掺假羊肉。以羊肉质量计,分别添加20%、30%、40%、100%的鸭肉制备掺假羊肉。肉糜制作时,使用绞肉机(板孔直径5 mm)绞碎,每个样品绞碎时间约2 min,重复2 次。通过肉眼分辨肉糜的红白颜色差异,选取28 个未掺假羊肉样品;同样按照肉糜颜色差异,从各比例掺假羊肉中分别选取6 个肉糜样品,共24 个。未掺假羊肉和掺假羊肉样品数量、校正集及验证集数据信息如表2所示。
1.3.2 拉曼光谱数据采集
将样品置于样品台上,设定积分时间10 s,平均次数6 次,功率等级10 级,拉曼光谱扫描范围157~3 645 cm-1。
采集并保存每個样品的拉曼光谱数据。
由图1可知,不同种属动物脂肪组织的拉曼光谱数据差异较大。动物脂肪组织中脂肪含量在90%以上,水分含量约6%,蛋白质含量约2.2%,因此其拉曼光谱数据主要来源于脂肪。动物脂肪组织在800~1 800 cm-1范围内拉曼谱带的大致分布如表3所示。3 种原料肉的脂肪组织在部分峰型和峰强度上具有明显差异,如在1 266 cm-1
处的顺式烯烃H弯曲,羊肉峰强度相对较弱,猪肉和鸭肉峰强度较强;在1 126 cm-1处的C-C伸缩振动,鸭肉峰强度很弱,羊肉和猪肉峰强度相对较强;在920 cm-1处,由于具有多种振动模式,3 种原料肉的脂肪组织有较大不同;3 种原料肉脂肪组织在1 656 cm-1处的C=C伸缩谱带与1 742 cm-1处的C=O伸缩谱带或1 440 cm-1处的-CH2-剪切振动谱带的强度比值不同,常用此比值来定量分析脂肪的不饱和度[28]。
不同种属动物瘦肉组织的拉曼光谱数据如图2所示。动物瘦肉组织中蛋白质含量约20%,脂肪含量1%~15%,脂肪含量视瘦肉种类的不同而不同,无机盐1%,另有微量维生素和水分。肉色主要取决于肉中肌红蛋白和血红蛋白的含量及化学状态。与动物脂肪组织不同,动物瘦肉组织的拉曼光谱信号来源比较复杂,主要是蛋白质和脂肪拉曼光谱信号的叠加。因此,结合化学计量学中的多变量判别工具,可将拉曼光谱数据作为原料肉种属定性鉴别和掺假肉鉴定的依据。
1.3.3 数据预处理
拉曼光谱数据在采集过程中因受到荧光、样品平整度、环境中光线等因素的影响,光谱会出现不同程度的基线漂移;并且瘦肉样品拉曼光谱信号弱,从而使光谱信噪比降低,因此这些因素均会影响检测结果。本实验采用MSC、SNRWC与均值中心化法对光谱数据进行预处理,MSC法可以校正基线的平移和偏移现象[29]。SNRWC法通过调整横坐标将谱线频率调整至统一值f
(f为移动窗口尺寸),滤波后再将坐标恢复或标定至统一值,此方法可有效去除光谱数据的随机噪声,显著提高数据信噪比且不会使数据失真。均值中心化法是将样品光谱数据减去校正集的平均光谱数据,以增加样品光谱之间的差异,从而提高模型的稳健性和预测能力[30]。数据预处理方法程序均采用MATLAB(R2017b)软件编写。
1.3.4 SIMCA分类法
SIMCA分类法又称相似分析法,是建立在主成分分析(principle component analysis,PCA)基础上的一种模式识别法,即利用样品在主成分空间中与不同类别样品的类距离进行归类判别[31]。该方法包括2 个主要步骤,
第1步选定已知类别的训练集,并对训练集样本的原始光谱矩阵进行降维处理,建立每一类别的PCA模型;第2步是以未知样本逐一拟合各类SIMCA主成分模型,进行归类判别[32-33]。SIMCA算法程序采用MATLAB(R2017b)软件编写。定性识别准确率按下式计算。
2 结果与分析
2.1 拉曼光谱数据预处理结果
由图3可知,由于样本本身的荧光效应以及激光照射区域样品表面凹凸不平,样本拉曼光谱有明显的基线漂移和光谱散射现象。由于瘦肉样本中水分含量较高,脂肪和蛋白质含量较低,拉曼信号较弱,噪声较大。因此,需要采取合适的光谱预处理方法消除样品散射带来的基线漂移以及噪声对定性识别带来的影响。
利用MSC法和SNRWC法对鸭肉、羊肉、猪肉3 种原料肉的203 个光谱数据进行预处理。采用MSC法计算校正集样品的平均光谱值,以此平均光谱值作为标准光谱值。将每个样品的光谱值与标准光谱值进行一元线性回归分析,计算各样品光谱值相对于标准光谱值的线性和倾斜平移量,最后将每个样品的原始光谱值减去线性平移量并除以倾斜平移量,使每个样品光谱的基线平移和偏移都在标准光谱值的参考下得到修正。光谱降噪时将SNRWC法中的移动窗口尺寸设为27,以降低光谱的随机噪声和完好保留光谱信号。由图4可知,MSC法有效降低了光谱间的散射,而SNRWC法则明显降低了拉曼光谱的噪声。
2.2 PCA分析结果
为考察不同原料肉及真假肉样光谱的差异及各类样品的聚类趋势,首先用无监督识别模式的PCA法分别对3 种不同原料肉样的203 个原始光谱数据和真假羊肉样品的52 个原始光谱数据进行分析,3 种原料肉和真假羊肉样品的前3 个主成分的累计贡献率分别为98.67%、99.62%。由图5可知,3 种原料肉和真假羊肉样品均没有明显的聚类趋势。
利用MSC结合SNRWC法对3 种原料肉203 个样本和真假羊肉52 个样品数据进行前处理后再作PCA,得到3 种不同原料肉和真假羊肉样品的前3 个主成分累计贡献率分别为96.69%、97.10%。由图6可知,MSC结合SNRWC法对光谱数据处理后,样品之间有明显的聚类趋势,但仍有小部分样品存在一定程度的重叠。
2.3 SIMCA法定性分析结果
2.3.1 原料肉鉴别
SIMCA定性分析是基于PCA基础上的有监督模式識别方法。首先从3 种原料肉203 个样本中选择166 个建立训练集,其余37 个样本均用于验证定性模型的预测效果。数据经过前处理后,对训练集样本的光谱矩阵进行PCA,采用SIMCA法分别建立3 种原料肉的定性分析模型。为充分利用光谱中的有效信息并避免过拟合,需选择合适的主因子数。通过交叉验证计算预测残差平方和,确定鸭肉、羊肉和猪肉3 种原料肉的主因子数分别选取6、6、8。
建立不同种属肉类样品的SIMCA主成分模型,以Q与T2衡量样本与模型的拟合程度。Q为样本与PCA模型中主成分投影之间的残差,用于评价样本与模型拟合程度;T2用于描述样本在PCA模型内部的远离程度。经过有效的数据前处理以及结合SIMCA分类方法分别建立鸭肉、羊肉和猪肉的定性分析模型。由图7可知,3 种原料肉SIMCA主成分模型均可将3 种原料肉明显区分。这说明通过化学计量学方法可有效提取不同种属肉类样品的光谱信息,如脂肪酸不饱和度、蛋白质组成以及结构等方面的信息,实现对原料肉种属的鉴别。
为验证原料肉SIMCA主成分模型的预测效果,对验证集37 个样本的光谱数据进行预测分析。由表4可知,3 种原料肉的识别正确率均为100%,这表明所建立的SIMCA定性识别模型对3 种原料肉种属具有较好的识别能力。
2.3.2 未掺假羊肉与掺假羊肉样品的鉴别结果
采用SIMCA法分别建立未掺假与掺假羊肉的SIMCA主成分模型,2 种样品主因子数均选择3。由图8可知,数据经前处理后,未掺假与掺假羊肉的SIMCA主成分模型均可有效将未掺假和掺假羊肉分开。
为验证真假羊肉SIMCA模型的预测效果,采用定性模型对验证集中5 个未掺假羊肉和4 个掺假羊肉样品进行预测,由表5可知,识别正确率均为100%。
3 结 论
本研究应用WAI拉曼光谱技术对不同种属肉类样品进行分类判别。将采集的拉曼光谱数据经MSC法和SNRWC法处理后,利用PCA法提取样品光谱主成分以考察样品之间的聚类趋势。PCA分析结果显示大部分鸭肉、羊肉、猪肉样品都能得到较好分离,同时未掺假和掺假羊肉也有明显的聚类趋势,但未能完全区分。在前期数据前处理和PCA分析基础上,建立SIMCA定性分类模型,对37 个鸭肉、羊肉和猪肉样品种属进行定性鉴别,识别正确率均达100%。对4 个掺假羊肉和5 个未掺假羊肉样品鉴别正确率亦为100%。单个样品检测时间可控制在2 min以内,即放即测,操作简便。因此,采用WAI拉曼光谱技术和有效的数据前处理方法以及结合SIMCA算法可快速识别不同种属肉类样品和鉴别肉类掺假情况,有利于推进口岸检测任务前移。
参考文献:
[1] BALLIN N Z, VOGENSEN F K, KARLSSON A H. Species determination: can we detect and quantify meat adulteration?[J]. Meat Science, 2009, 83(2): 165-174. DOI:10.1016/j.meatsci.2009.06.003.
[2] KIM S H, HUANG T S, SEYMOUR A T, et al. Development of immunoassay for detection of meat and bone meal in animal feed[J]. Journal of Food Protection, 2005, 68(9): 1860-1865. DOI:10.1111/j.1745-4522.2005.00023.x.
[3] MULDOON M T, ONISK D V, BROWN M C, et al. Targets and methods for the detection of processed animal proteins in animal feedstuffs[J]. International Journal of Food Science and Technology, 2004, 39(8): 851-861. DOI:10.1111/j.1365-2621.2004.00858.x.
[4] KIM S H, HUANG T S, SEYMOUR T A, et al. Production of monoclonal antibody for the detection of meat and bone meal in animal feed[J]. Journal of Agricultural and Food Chemistry, 2004, 52(25): 7580-7585. DOI:10.1021/jf048789a.
[5] TARTAGLIA M, SAULLE E, PESTALOZZA S, et al. Detection of bovine mitochondrial DNA in ruminant feeds: a molecular approach to test for the presence of bovine-derived materials[J]. Journal of Food Protection, 1998, 61(5): 513-518. DOI:10.4315/0362-028x-61.5.513.
[6] 林彦星, 张彩虹, 阮周曦, 等. 实时荧光定量PCR检测畜禽肉制品中鸭源性成分[J]. 动物医学进展, 2016, 37(11): 48-53. DOI:10.16437/j.cnki.1007-5038.2016.11.009.
[7] AL-KAHTANI H A, ISMAIL E A, AHMED M A. Pork detection in binary meat mixtures and some commercial food products using conventional and real-time PCR techniques[J]. Food Chemistry, 2017, 219: 54-60. DOI:10.1016/j.foodchem.2016.09.108.
[8] MONTOWSKA M, FORNAL E. Label-free quantification of meat proteins for evaluation of species composition of processed meat products[J]. Food Chemistry, 2017, 237: 1092-1100. DOI:10.1016/ j.foodchem.2017.06.059.
[9] 杨志敏,丁武. 近红外光谱技术快速鉴别原料肉掺假的可行性研究[J]. 肉类研究, 2011, 25(2): 25-28. DOI:10.3969/j.issn.1001-8123.2011.02.007.
[10] 白京, 李家鹏, 邹昊, 等. 近红外光谱定性定量检测牛肉汉堡饼中猪肉掺假[J]. 食品科学, 2019, 40(8): 287-292. DOI:10.7506/spkx1002-6630-20180802-016.
[11] 耿莹莹, 李亚飞, 刘湘祁, 等. 便携式拉曼光谱仪在毒品和易制毒化学品快速检测中的应用[C]//第17届全国光散射学术会议摘要文集. 成都: 光散射学报编辑部, 2013: 260.
[12] 冯泽阳, 张卫红, 郑颖, 等. 2000年以来拉曼光谱在考古中的应用[J]. 光散射学报, 2016, 28(1): 27-41. DOI:10.13883/j.issn1004-5929.201601007.
[13] MATOUSEK P, PARKER A W. Bulk Raman analysis of pharmaceutical tablets[J]. Applied Spectroscopy, 2006, 60(12): 1353-1357. DOI:10.1366/000370206779321463.
[14] 田高友. 拉曼光譜技术在石油化工领域应用进展[J]. 现代科学仪器, 2009(2): 138-142.
[15] MISHRA P, TRIPATHI L N. Characterization of two-dimensional materials from Raman spectral data[J]. Journal of Raman Spectroscopy, 2020, 51(1): 37-45. DOI:10.1002/jrs.5744.
[16] SANCHIS-BONET A, MENOR GALVAN M A C, GARCIA RICO G E, et al. Application of Raman spectroscopy coupled with chemometrics to the early diagnostics and classification of prostate cancer[J]. European Urology Supplements, 2019, 18(11): e3511-e3512. DOI:10.1016/S1569-9056(19)34664-0
[17] AKIMOVA G A, GRIGORIEVSKY V I, SYRYKH P Y, et al. On a method for measuring methane concentration on extended atmospheric paths using a remote gas analyzer with a powerful Raman amplifier[J]. Journal of Communications Technology and Electronics, 2019, 64(11): 1251-1255. DOI:10.1134/S1064226919110019.
[18] 陳龙, 袁明洋, 明晶, 等. 基于改进siPLS法建立5 种含CaCO3中药的拉曼光谱定量模型[J]. 中国中药杂志, 2015, 40(18): 3608-3615. DOI:10.4268/cjcmm20151818.
[19] ZHAO Ming, DOWNEY G, ODONNELL C P. Dispersive Raman spectroscopy and multivariate data analysis to detect offal adulteration of thawed beef burgers[J]. Journal of Agricultural and Food Chemistry, 2015, 63(5): 1433-1441. DOI:10.1021/jf5041959.
[20] VELIO?LU H M, TEMIZ H T, BOYACI I H. Differentiation of fresh and frozen-thawed fish samples using Raman spectroscopy coupled with chemometric analysis[J]. Food Chemistry, 2015, 172: 283-290. DOI:10.1016/j.foodchem.2014.09.073.
[21] BOYAC S H, TEMIZ H T, UYSAL R S, et al. A novel method for discrimination of beef and horsemeat using Raman spectroscopy[J]. Food Chemistry, 2014, 148: 37-41. DOI:10.1016/ j.foodchem.2013.10.006.
[22] WIKSTR?M H, LEWIS I R, TAYLOR L S. Comparison of sampling techniques for in-line monitoring using Raman spectroscopy[J]. Applied Spectroscopy, 2005, 59(7): 934-941. DOI:10.1366/0003702054411553.
[23] KIM M, CHUNG H, WOO Y, et al. New reliable Raman collection system using the wide area illumination (WAI) scheme combined with the synchronous intensity correction standard for the analysis of pharmaceutical tablets[J]. Analytica Chimica Acta 2006, 579(2): 209-216. DOI:10.1016/j.aca.2006.07.036.
[24] BAKEEV K A. 过程分析技术: 针对化学和制药工业的光谱方法和实施策略[M]. 2版. 北京: 机械工业出版社, 2014: 174-176.
[25] DE BIASIO M, STAMPFER P, LEITNER R, et al. Micro-Raman spectroscopy for meat type detection[C]//Next-generation spectroscopic technologies Ⅷ. Baltimore: international society for optics and photonics. Baltimore, Maryland, USA, 2015. DOI:10.1117/12.2176321.
[26] BEATTIE J R, BELL S E J, BORGAARD C, et al. Classification of adipose tissue species using Raman spectroscopy[J]. Lipids, 2007, 42(7): 679-685. DOI:10.1007/s11745-007-3059-z.
[27] ODONNELL C P, FAGAN C, CULLEN P J. 食品工业中的过程分析技术[M]. 北京: 化学工业出版社, 2016: 100-112.
[28] 陈倩, 李沛军, 孔保华. 拉曼光谱技术在肉品科学研究中的应用[J]. 食品科学, 2012, 33(15): 315-321.
[29] 彭彦昆. 食用农产品品质拉曼光谱无损快速检测技术[M]. 北京: 科学出版社, 2019: 27-28.
[30] 褚小立. 化学计量学方法与分子光谱分析技术[M]. 北京: 化学工业出版社, 2011: 41-43.
[31] 陆婉珍. 现代近红外光谱分析技术[M]. 2版. 北京: 中国石化出版社, 2007: 73-78.
[32] 严衍录. 近红外光谱分析基础与应用[M]. 北京: 中国轻工业出版社, 2005: 225-231.
[33] 谢锦春, 袁洪福, 宋春风, 等. 基于近红外光谱与SIMCA快速识别乙烯醋酸乙烯酯共聚物树脂牌号的定性方法[J]. 现代化工, 2018, 38(9): 235-238; 240. DOI:10.16606/j.cnki.issn0253-4320.2018.09.052.

