分离缺陷蛋白6α在胶质瘤中的表达及其对临床病理和预后的意义
王帅,潘景臻,华实,张健,衡雪源
1.潍坊医学院临床医学院,山东 潍坊 261053;
2.临沂市人民医院微创与功能神经外科,山东 临沂 276002
胶质瘤是一种来源于神经外胚叶组织、由大脑或脊髓胶质细胞癌变导致的原发性颅脑肿瘤,年发病率为3~8 人/10 万人口[1],经综合治疗后中位生存期仍较短,死亡率极高,尽管接受了手术切除和放化疗,胶质母细胞瘤(GBM)的平均生存时间只有12~15个月[2]。研究表明,胶质瘤的发生与进展是多基因、多步骤共同作用的结果[3],因此从病因学研究脑胶质瘤的发病机制对脑胶质瘤患者的早期诊断和治疗以及提高脑胶质瘤患者的生命质量具有重要的意义。分离缺陷蛋白6 α(partitioning defective protein 6 homolog alpha,PARD6A)基因是PAR 家族的成员,该家族目前共发现了6 种PAR基因(PAR1-6),其中PAR6 有三个基因亚型,分别是PARD6A、PARD6B和PARD6G,均可编码PAR6蛋白,其分子量为37 000 kD,该细胞膜蛋白作为多蛋白复合物的成员参与不对称细胞分裂和细胞极化过程[4]。最新的研究表明,细胞的不对称分裂和细胞极化过程的异常均与肿瘤的发生和发展密切相关[5]。因此,对分离缺陷蛋白的研究将对脑胶质瘤的早期诊断、临床治疗及预后具有重要意义。
本研究利用癌症基因组图谱(TCGA)数据库、GTEx 数据库收集GBM、低级别胶质瘤(LGG)和正常脑组织(Normal)的mRNA 及临床信息资料,结合免疫组化实验,分析胶质瘤中PARD6A的差异性表达与临床病理学参数及预后的相关性。下载KEGG 数据库内的分子特征数据库(Molecular signatures database,MsigDB),利用基因富集分析(gene set enrichment analysis,GSEA)技术研究PARD6A 在胶质瘤中涉及的信号通路,研究PARD6A 在胶质瘤中可能的发病机制,探讨其作为潜在靶点的可行性,为胶质瘤的治疗提供新思路。
1 资料与方法
1.1 资料来源 本研究中的胶质瘤数据来源于TCGA 数据库中下载的胶质瘤GBM、LGG 数据集(leve3,mRNA-seq、Clinical);Normal 基因表达数据来源于GTEx 数据库(Genotype-Tissue Expression Project(GTEx)Brain-Cortex-GTEx mRNA-seq)。
1.2 资料筛选方法 下载GTEx数据库内正常脑组织中PARD6A 的mRNA-seq 表达数据114 例;下载TCGA 数据库中GBM 的mRNA-seq 数据167 例,LGG的mRNA-seq 数据524 例,利用Rx643.4.1 整理。纳入标准:(1)基因表达数据和临床数据完整;(2)患者经手术治疗且病理结果确诊为GBM或LGG。将患者信息整合为包含mRNA-seq 表达量、临床参数和生存资料的数据集,与GTEx 数据库内正常脑组织中PARD6A的mRNA-seq表达量进行差异性分析。
1.3 PARD6A 表达与临床病理学的相关性 对TCGA 和GTEx 数据库内胶质瘤相关PARD6A 的mRNA 和临床数据加以分析得出,GBM 和LGG 两组数据的PARD6A-mRNA表达量总的中位值为9.78,以PARD6A-mRNA 表达量的中位数为标准,设定≤9.78=1,>9.78=2,将数据分为高表达组和低表达组,研究PARD6A 表达量与年龄、性别、胶质瘤病理级别及生存状态的关系。
1.4 对PARD6A 进行生存分析 表达谱动态分析(Gene Expression Profiling Interactive Analysis,GEPIA) (http://gepia. cancer-pku.cn/) 是 一 款TCGA 和GTEx 数据库在线可视化网页工具,可以对目的基因进行Kaplan⁃Meier Plotter在线数据生存分析。本研究中,选择GBM-Tumor 和LGG Tumor 数据集后,利用GEPIA对PARD6A进行总生存期(overall survival,OS)和无病生存期(disease free survival,RFS)分析。
1.5 对PARD6A 进行单基因富集分析 以TCGA 数据库内PARD6A-mRNA 表达量的中位值为基准,将胶质瘤内PARD6A-mRNA 表达量的数据分为高表达(H)组和低表达(L)组,从分子特征数据库(Molecular Signatures Database,MsigDB)下载参考集2.cp.kegg.v5.1.symbols.gmt,根据缺省加权富集法,随机组合的数量定为1 000 次,名义P 值(nominal P-values,NOM P)<0.05,错 误 发 现 率(false discovery rates,FDR)<0.25,GSEA2.2.1 软件进行基因富集分析,分析PARD6A参与的信号通路。
1.6 PARD6A与关键基因的基因的相关性 根据PARD6A 富集分析得出的信号通路,寻找这些通路中已知的关键基因,通过GEPIA对基因PARD6A和这些关键基因进行相关性分析。
1.7 临床标本的收集和随访 本研究所采用临床标本共68例,其中胶质瘤样本58例,均源于在2015年1 月至2017 年10 月期间在临沂市人民医院微创与功能神经外科接受手术治疗的原发脑胶质瘤患者;对照组脑组织样本10例,来自于同时期急性重型颅脑损伤颅内减压患者。所有胶质瘤标本均按照世界卫生组织(WHO 2007)神经上皮肿瘤分类标准对胶质瘤组织标本进行分级,其中Ⅰ级胶质瘤10 例,Ⅱ级胶质瘤18例,Ⅲ级胶质瘤15例,Ⅳ级胶质瘤15例。所纳入胶质瘤标本均经我院病理科证实,Ⅲ~Ⅳ级胶质瘤患者术后均进行了替莫唑胺标椎化疗方案。所有脑组织的获取均已通过医院伦理委员会的通过。随访纳入实验的58例胶质瘤患者的1年和2年肿瘤复发情况。
1.8 实验试剂 PAR-6家族细胞极性调节因子α抗 体(Afffinity 公 司,DF7166-100);磷 酸 盐 缓 冲 液(PBS,pH 7.2~7.4),0.01 mol/L 柠檬酸钠抗原修复液(50X),增强型DAB 显色试剂盒(20X) (索莱宝),通用二步法检测试剂盒(中衫,FV-9000-6)。
1.9 免疫组织化学法 按照通用二步法检测试剂盒说明书对石蜡切片依次放入二甲苯、梯度乙醇中进行脱蜡、水化。微波炉高火加热0.01 mol/L 柠檬酸钠缓冲液(pH6.0)至沸腾后将组织切片放入进行抗原修复,低火维持20 min,自然冷却至室温后,置入蒸馏水中浸泡10 min;加入适量的内源性过氧化物酶阻断剂,室温孵育10 min;滴加50 μL一抗(1∶50 稀释),4℃冰箱过夜孵育;滴加100 μL反应增强液,室温孵育20 min,滴加100 μL 增强酶标兔抗山羊IgG 聚合物,室温孵育20 min。每次操作前后均用PBS 冲洗3 min×3次,加入适量新鲜配制的DAB显色液,室温孵育5~8 min。自来水冲洗,苏木素染色液孵育20 s;分化、冲洗返蓝,然后脱水、封片,对照组不加入一抗,以等量PBS代替,阅片由两名经验丰富的病理医师依据双盲法独立进行。
1.10 免疫组化染色结果评判标准 高倍镜视野下,每个组织样本随机选取5个视野,依据高倍镜视野下染色呈阳性结果的细胞在整个视野中所占比例以及阳性细胞的染色强度判定。染色结果按显色细胞数记分:无阳性染色细胞为0分,阳性细胞数≤25%为1 分,阳性细胞数26%~50%为2 分,阳性细胞数>50%为3 分;按细胞显色深浅记分,无阳性反应细胞为0分,浅黄色为1分,棕黄色为2分,棕褐色为3分。两项积分相乘,0~2为阴性表达,3~9为阳性表达。
1.11 统计学方法 应用SPSS21.0统计学软件进行数据分析。计量资料以均数±标准差表示,三样本的比较采用单因素方差分析,两两比较采用LSD检验;计数资料采用以百分率(%)来表示,PARD6A 免疫组化结果与肿瘤级别的关系分析采用χ2检验,目的基因与关键基因的相关性分析采用皮尔逊(Pearson)法。以P<0.05 为差异具有统计学意义。生存分析采用Kaplan⁃Meier在线分析。采用GSEA2.2.1版软件从分子标签数据库(Molecular Signatures Database,Msig-DB)中获得参照基因集2.cp.kegg.v5.1.symbols.gmt,然后按缺省加权富集统计的方法对PARD6A 进行单基因富集分析,设置随机组合次数为1 000 次,GSEA 中选取NOM P<0.05、错误发现率(false discovery rates,FDR)<0.25的基因集作为显著富集基因集。
2 结果
2.1 PARD6A RNA-seq 表达水平比较 对TCGA 和GTEx 数据库中PARD6A mRNA-seq 的表达数据进行检验发现,GBM组PARD6A mRNA-seq表达量最低,其次为LGG 组,Normal 组PARD6A mRNA-seq表达量最高,三组间差异具有统计学意义(F=195.548,P<0.05)。三组经LSD 法两两比较显示,Normal 与GBM、Normal 与LGG 以及LGG 与GBM 任意两组间的差异均具有统计学意义(P<0.05),见表1、图1。
表1 TCGA 数据库和GTEx 数据库中PARD6A mRNA-seq 表达水平比较

表1 TCGA 数据库和GTEx 数据库中PARD6A mRNA-seq 表达水平比较
注:与Normal组比较,aP<0.05;与LGG组比较,bP<0.05。
组别GBM组LGG组Normal组数量167 524 114 PARD6A mRNA-seq 9.31±4.98ab 10.78±4.30a 19.58±5.63b F值195.548 P值<0.05

图1 PARD6A mRNA-seq在TCGA和GTEx数据库中的基因差异性表达
2.2 PARD6A mRNA-seq 的表达与临床病理学参数的关系 采用χ2检验对PARD6A 低表达量组和PARD6A 高表达量组的年龄、性别、肿瘤级别及术后2 年的生存状态进行数据分析。结果显示,PARD6A RNA-seq 表达量与患者的年龄和性别无关(P>0.05),但是PARD6A RNA-seq 表达量与胶质瘤病理级别以及术后2 年的生存状态有关(P<0.05),见表2。
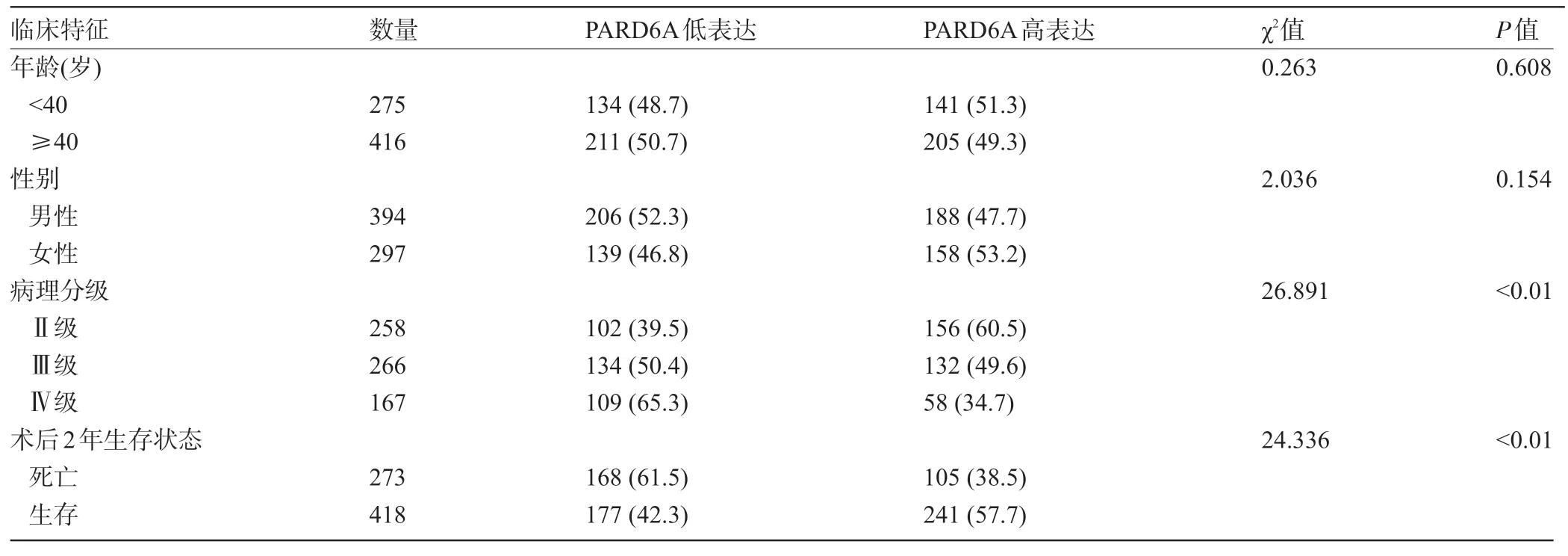
表2 PARD6A mRNA-seq的表达与临床病理学参数比较[例(%)]
2.3 生存分析 PARD6A Kaplan-Meier Plotter在线数据生存分析显示:无论是OS 还是DFS,PARD6A高表达组(红线)生存曲线均在低表达组(蓝线)之上,且在50 个月左右时差别明显(图2),即PARD6A 低表达组OS 及RFS 较PARD6A 高表达组均明显缩短,差异有统计学意义(P<0.05)。

图2 PARD6A在TCGA数据库中的生存分析
2.4 基因富集分析 使用GSEA 分析PARD6A对其他生物功能基因集的影响,结果显示高表达PARD6A的肿瘤样本富集了35条有意义的信号通路,差异均有统计学意义(P<0.05),其中包括Wnt 信号通路(图3A)、GnRH 信号通路(图3B)、ErbB 信号通路(图3C)和CALCIUM信号通路(图3D)。
2.5 PAR6蛋白在外伤脑组织和胶质瘤组织中的表达水平比较 免疫组化实验结果显示,Par6蛋白主要位于细胞核中,阳性染色为棕色。外伤脑组织中Par6蛋白阳性占90.0% (9/10);Ⅰ~Ⅱ级中阳性占53.6% (15/28);Ⅲ~Ⅳ级中阳性占20.0% (6/30)。经χ2检验分析表明,Par6蛋白在GBM组织中含量最低,其次为LGG组织,脑外伤组织样本中最高,差异有统计学意义(χ2=16.632,P<0.001)(表3、图4)。以PAR6 蛋白表达情况分组,统计其术后1 年和2 年的肿瘤复发率,结果显示,PAR6低表达组术后1年和2年肿瘤复发率分别为45.9%和71.8%,PARD6A 高表达组术后1 年和2 年肿瘤复发率分别为14.3%和23.8%,无论术后1 年还是2年的肿瘤复发率,低表达组相比高表达组均明显升高,差异有统计学意义(P<0.05),见表4。
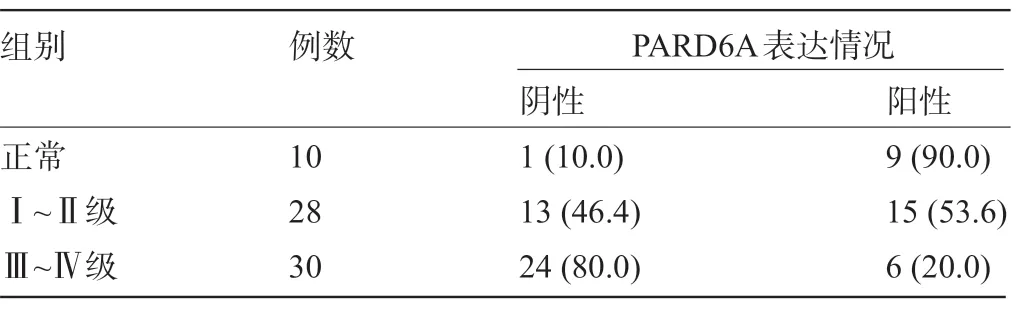
表3 正常脑组织及不同级别胶质瘤组织中PARD6A 表达水平比较[例(%)]
2.6 基因相关性表达 TGFB1、Twist1、Snail 和Slug 是Wnt 信号通路中的关键基因,本研究中利用GEPIA 对 基 因PARD6A 和TGFB1、Twist1、Snail 和Slug进行了Pearson相关性分析,结果显示PARD6A与TGFB1、Twist1、Snail 和Slug 的Pearson 相关系数R 分别为-0.44、-0.28、-0.34和-0.28,R值均为负数,且差异具有统计学意义(P<0.05),提示PARD6A 与TGFB1、Twist1、Snail和Slug均呈负相关,见图5。

图3 采用GSEA 2.2.1版软件对PARD6A进行单基因富集分析得到的部分信号通路
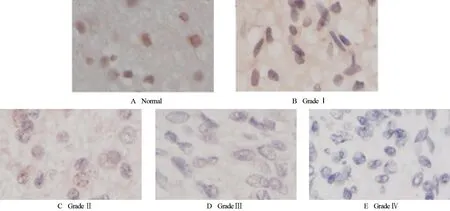
图4 正常脑组织(A),WHOI~IV级胶质瘤(B~E)的免疫组化图像(油镜400×)

表4 不同PARD6A表达水平患者术后胶质瘤复发情况比较[例(%)]

图5 PARD6A与TGFB1、Twist1、Snail和Slug的相关性
3 讨论
神经胶质瘤是最常见的原发性颅内肿瘤,起源于神经上皮组织,这些肿瘤的特征在于无限的细胞增殖和极强的侵袭性[6]。随着生物信息学技术的发展,世界多个国家相继建立了多个大型数据库,数据库的建立为理解肿瘤发生的机制以及寻找有效的的生物标志物提供了极其巨大的支持[7]。本研究通过生物信息学技术,利用TCGA 和GTEx 以及KEGG 等数据库内的数据资料,首次报道了PARD6A 在胶质瘤中的作用。PARD6A 在肿瘤发生发展中所扮演的重要作用,有研究显示PARD6A在乳腺癌[9]、肺癌[10]和前列腺癌[11]的发生发展中有着重要作用,然而尚未发现关于PARD6A在胶质瘤中的研究。
在本研究中,通过分析TCGA 和GTEx 数据库中胶质瘤内PARD6A-mRNA和临床数据,发现PARD6A在胶质瘤中呈明显低表达,并且其表达量与胶质瘤的病理分级有关,其中GBM组PARD6A表达量最低,其次为LGG 组,Normal组PARD6A表达量最高,经两两比较和三组数据的方差分析,差异均具有统计学意义。由于TCGA和GTEx数据库包含的是mRNA水平的表达数据,无法反应蛋白水平的表达情况,因此本研究通过收集临床外伤脑组织和胶质瘤组织标本,对PARD6A进行了蛋白水平的检测。免疫组化实验结果显示,PAR6 蛋白主要位于细胞核中,阳性染色为棕色。PAR6蛋白在脑外伤组织样本中明显高于LGG和GBM组织,且胶质瘤病理级别越高,PAR6表达越低,染色越浅(P<0.001)。免疫组化的结果从蛋白水平验证了TCGA和GTEx数据库中胶质瘤内PARD6A-mRNA数据,因此本实验从mRNA水平和蛋白水平均证明基因PARD6A在胶质瘤中低表达,且胶质瘤病理级别越高,PARD6A 表达越低,染色越浅。通过对数据库内数据分析还发现,PARD6A 的表达量还与患者的生存状态有关(P<0.001),高表达组患者的生存率显著高于低表达组,提示低表达的PARD6A 可能影响胶质瘤的预后。为探究PARD6A对胶质瘤患者生存的影响,利用GEPIA对PARD6A进行了Kaplan⁃Meier Plotter在线数据生存分析,结果显示PARD6A 低表达组患者的OS及RFS均较PARD6A高表达组低,并且在术后50个月左右时差别明显,即PARD6A 表达越低,患者的生存率越低。为了探究PARD6A 表达水平与患者预后的关系,本研究根据免疫组织化学实验中PARD6A表达情况的结果,结合对纳入实验的58例胶质瘤患者的随访数据,统计其术后1年和2年胶质瘤复发率,结果显示无论是术后1年还是2年的肿瘤复发率低表达组相比高表达组均明显升高,差异均有统计学意义(P<0.05)。综合Kaplan⁃Meier Plotter在线数据生存分析的结果,以及对胶质瘤患者术后1 年和2 年的肿瘤复发率的统计,认为PARD6A的表达与预后之间具有显著的相关性,PARD6A表达下调明显影响胶质瘤的预后。
本研究为寻找PARD6A 在胶质瘤中可能参与的机制,从KEGG 数据库内下载MsigDB 分子参考集(2.cp.kegg.v7.0.symbols.gmt),利用基因富集分析技术和GSEA 4.0.0 软件对PARD6A 在胶质瘤中涉及的信号通路进行基因富集分析。结果富集得到包括Wnt 信号通路和ErbB 信号通路、GnRH 信号通路和CALCIUM 信号通路,其中Wnt 信号通路在胶质瘤中的研究较为透彻,本研究将以该通路为主要研究内容。
在PAR 家族中,PAR3 与PAR6 关系最为密切。PAR3、PAR6 与非典型蛋白激酶C(aPKC)是极性复合体的重要组成部分,共同协调建立细胞极性[11]。这些极性复合物定位于上皮细胞的细胞连接(紧密连接和粘附连接)处,可为极性和上皮功能的建立提供一个中心调控通路[13]。在本研究中通过基因富集分析发现PARD6A 参与Wnt 信号通路。Wnt 通路的失调与乳腺[14]、结肠[15]和中枢神经系统[16]等多种组织的癌变有关。在肿瘤转化过程中,Wnt/β-catenin途径常常异常激活,并且通过促使β-catenin 磷酸化和(或)核定位增加影响肿瘤的迁移和侵袭[17]。此外,Wnt/β-catenin 途径还可以诱导上皮-间质转化(EMT),因为激活的Wnt/β-catenin信号触发了一组EMT激活因子的表达,包括Twist1、Snail和Slug[18]。EMT与癌细胞的迁移和侵袭能力密切相关,是肿瘤侵袭和转移的关键步骤[1]。在EMT 过程中,上皮细胞失去顶端-基底极性和细胞间连接,从而获得间充质运动表型[19-20]。巧合地是,EMT 的特征之一就是通过抑制相关极性基因的转录而致使细胞极性的丧失[21]。在EMT 过程中,由基因TGFB1 编码的转化因子β(TGF-β)因作为调控者而具有关键作用。TGF-β可诱导PAR6 发生磷酸化,通过TGF-β-PAR6 极性途径抑制细胞极性形成,进而发生EMT,最终引起肿瘤的无序增殖和转移[22]。当前对EMT 在上皮性肿瘤中发挥的作用已有较多研究。EMT是上皮来源的肿瘤获得侵袭、转移以及耐药表型的重要原因[23]。然而恶性胶质瘤是否发生EMT,过去一直存在争议[24],但近年来越来越多的证据显示,恶性胶质瘤细胞也能够发生EMT或EMT样改变[25]。因此有理由推测在胶质瘤中TGF-β可能通过TGF-β-PAR6极性途径抑制PARD6A的表达,进而影响细胞极性的建立,引起Wnt 信号通路失调,Twist1、Snail 和Slug 的大量激活,进而诱导发生EMT,最终引起肿瘤侵袭和转移的增强。不仅如此,本研究还利用GEPIA对基因PARD6A 和TGFB1、Twist1、Snail 和Slug 进 行 了Pearson相关性分析,结果显示PARD6A与这些基因呈负相关,这从侧面也为本研究提供了支持。此外,本研究通过富集分析还富集到了ErbB信号通路、GnRH信号通路和CALCIUM 信号通路在内的32 条信号通路,有研究表明这些信号通路在胶质瘤的发生和发展中扮演者重要角色[26-29]。
综上所述,本文通过分析TCGA 数据库和GTEx数据库内的mRNA和临床数据,同时结合免疫组化实验,首次发现并证实了PARD6A基因在胶质瘤中表达下调,并且可能通过Wnt 信号通路、ErbB 信号通路、GnRH信号通路和CALCIUM信号通路影响胶质瘤的进展,进一步研究其影响胶质瘤增殖、侵袭和转移的机制可以为脑胶质瘤的诊断及治疗提供新的思路。

