定点突变对低温脂肪酶活性和热稳定性的影响
陈海清,于 凡,王红静,张先舟,亢春雨,马 雯,檀建新
(河北农业大学 食品科技学院 / 河北省农产品加工工程技术中心,河北 保定 071000)
近年来,通过定点突变的理性设计来达到改造酶分子的目的,已被人们广泛采用且证明是可行的[11]。张晓凤等人利用理性设计的方法提高了来源于土曲霉(Aspergillus terreus)的脂肪酶的催化活力,突变体ATL-Lid 和ATL-V218W 的酶活性分别比野生型提高了2.26 倍和3.38 倍[12]。郭静等[13]使用在线预测软件PoPMuSiC-2.1 预测了源自Streptomyces kathirae SC-1 的酪氨酸酶的自由能变化,进行单点突变,证明点突变Arg95Tyr 和Gly123Trp 在60 ℃下的半衰期分别比野生型提高了1.35 和1.82 倍[14]。Mohammadi 等[15]报道采用定点突变技术,获得了粘质沙雷氏菌的脂肪酶的突变体G2P 和G59P,突变后脂肪酶酶蛋白的t1/2分别比野生型高2.3 倍和2.9 倍,但最适温度和最适pH 并没有改变。实践证明,蛋白质分子内作用力的种类和数量的改变对酶分子的催化活性和稳定性会有重要影响[16-17]。因此,通过点突变可改变酶分子局部区域氨基酸残基之间、酶与环境之间的相互作用,或可提高酶的活性或稳定性。
粘质沙雷氏菌(Serratia marcescens)菌株L1是本实验室前期从土壤样品中筛选获得的脂肪酶高产菌株,该菌株所产胞外脂肪酶(lipase A,lipA)活性可达3.6 U/mL,经克隆测序后证明脂肪酶基因由1 845 碱基组成,编码614 氨基酸,其三维模型结构与SML 2QUB.pdb 具有高度相似性,活性位点由氨基酸残基Ser207、Asp256 和His314 组成[18],该酶属于I.3 脂肪酶家族。本试验拟采用定点突变技术进一步提高粘质沙雷氏菌脂肪酶的酶活力或热稳定性,为该酶的开发与应用奠定基础。
1 材料与方法
1.1 试验材料及试剂
克隆宿主菌E. coli DH5α、表达宿主E. coli Rosetta(DE3)购于博迈德,脂肪酶生产菌粘质沙雷菌菌株L1 由本实验室保存。
T4 连接酶、限制性内切酶BamH I 和Hind III 购于宝生物工程(大连)有限公司。2×Easy Taq DNA supermix、质粒提取试剂盒和DNA 胶回收试剂盒购于北京全式金生物技术有限公司。DNA marker 购于天根生化科技(北京)有限公司。蛋白Marker 购自北京康为世纪生物科技有限公司。氨苄青霉素、IPTG 和镍-琼脂糖凝胶为北京索莱宝公司产品。对硝基苯棕榈酸酯(pNPP)购自阿拉丁试剂有限公司。其他试剂均购买自本地经销商。本研究所用PCR 引物由金唯智生物科技有限公司合成,见表1。
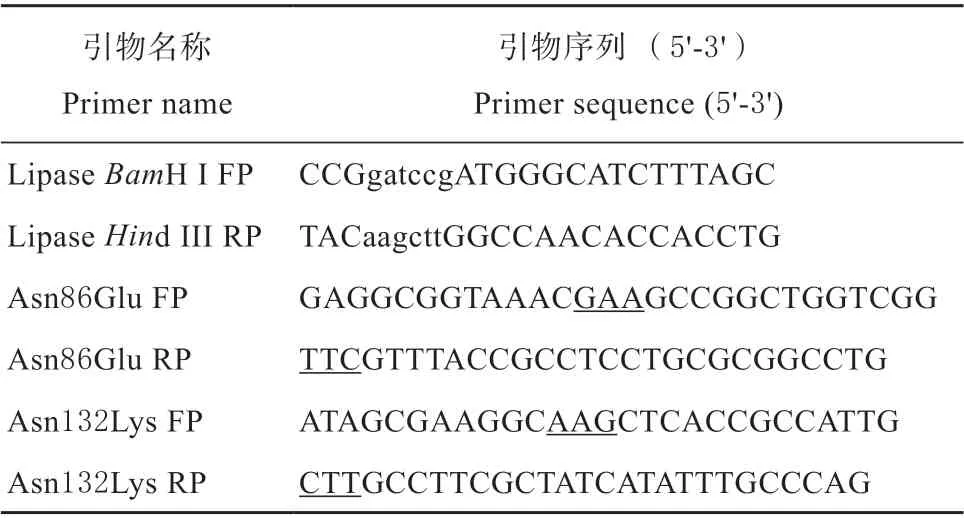
表1 重叠延伸PCR 扩增引物Table 1 Primers for overlap extension PCR amplification
1.2 主要仪器
PCR 仪:德 国Biometra 公 司;GL-20G- Ⅱ冷冻离心机:上海安亭科学仪器厂;DYY-10C 型电泳仪:北京市六一仪器厂;BL-300-X 电子天平:厦门佰伦斯电子科技有限公司;双层恒温振荡器ZHWY-2102C,上海智城分析仪器制造有限公司;恒温金属浴:上海一恒科技有限公司;JY04S-3E 凝胶成像分析系统:北京君意东方电泳设备有限公司。
1.3 同源建模和突变氨基酸选择
将菌株L1 脂肪酶蛋白序列提交至SWISSMODEL 进行同源建模,同源搜索比对表明,脂肪酶lipA 与脂肪酶SML(PDB 登录号:2QUB)同源性最高(97%),并以2QUB 为模版进行同源建模。之后将菌株L1 脂肪酶3D 结构提交至PoPMuSiC 在线服务器,预测脂肪酶每个突变氨基酸的去折叠自由能变化(ΔΔG),Asn 突变为Glu 或Lys 后,去折叠自由能为负值并且变化显著,故选Asn86 和Asn132 氨基酸残基作为突变位点。
1.4 定点突变及构建重组表达菌株
构建过程采用北京全式金生物技术有限公司的重叠引物法进行构建。以pET-22b(+)-lipase 质粒为模板,用Asn86Glu FP(或Asn132Lys FP)/ Lipase HindIII RP 引物对,Lipase BamHI FP /Asn86Glu RP(Asn132Lys RP)引物对进行第一步PCR 反应,分别扩增出脂肪酶突变点前后2 个片段,PCR 反 应体系为10 μL,包括模板pET-22b(+)-lipase 质粒1 μL,上、下游引物各1 μL,pfu mix 5 μL,ddH2O 2 μL。PCR 反应程序为:95 ℃预变性5 min;95 ℃变性30 s,58 ℃退火30 s,72 ℃延伸3 min 30 s,25 个循环后,72 ℃延伸10 min。将上述PCR 产物电泳并胶回收后得到突变点上、下游片段,以此为引物和模板(各2.5 μL),pfumix 5.0 μL,进行第二步PCR 反应,反应程序为:95 ℃预变性5 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸3 min,14个循环后,72 ℃延伸10 min。随后,以此步得到的PCR 产物做模版(4 μL),Lipase BamHI FP /HindIII RP 为上下游引物(各1 μL),pfumix 8 μL,ddH2O 6 μL,总体积20 μL,进行第三步PCR 反应,程序为:95 ℃预变性5 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸3 min 30 s,25 个循环后,72 ℃延伸10 min。第三步PCR 产物经BamH I/Hind III 双酶切后与pET22b连接构建表达载体pET22b-Asn86Glu(或pET22b-Asn132Lys),转到E. coli DH5α 中,在抗性平板上挑选阳性转化子并送至深圳华大基因股份有限公司测序,将测序正确的突变质粒转到E. coli Rosetta(DE3)中,挑选阳性转化子诱导表达。
1.5 脂肪酶基因lipA 在E. coli Rosetta(DE3)中表达和lipA 酶蛋白纯化
将重组的阳性转化子接种于含有氨苄青霉素(50 μg/mL)的LB 培养基中,37 ℃振荡培养至OD600为0.6 ~0.8 时,添加IPTG 至其终浓度为0.5 mmol/L,16 ℃继续培养16 h,10 000 r/min 离心10 min 收集菌体。超声波破碎后10 000 r/min 离心20 min 后,得到的上清液即为蛋白粗酶液。将粗酶液加入到镍柱中混匀后静置10 min,4 ℃下用咪唑浓度为20 和200 mmol/L 的洗脱缓冲液(50 mmol/L Tris-HCl,500 mmol/L NaCl,10% 甘油,pH 8.0)洗脱带有组氨酸标签的目的蛋白质,将200 mmol/L 咪唑浓度冲洗下来的蛋白经透析12 h 后进行浓缩,得到的即为纯化后浓缩的脂肪酶蛋白。
This is Christmas night, whether you're in a snowy winter night or under the starry sky. I wish you are around by the people you loved, having a bottle of your favorite wine, get slightly drunk and be happy from your heart. Merry Christmas!
1.6 脂肪酶的酶活性和蛋白质含量测定
参照文献[12]采用对硝基苯酚棕榈酸酯法(pNPP)测定酶活。酶活定义:每分钟催化生成 1 μmol/L 产物p-NP 的酶量定义为一个酶活单位(U)。p-NP 的测量在410 nm 的吸光度下进行。脂肪酶的蛋白含量测定根据BCA 试剂盒来测定纯化后脂肪酶的蛋白浓度。
1.7 突变脂肪酶的酶学性质
1.7.1 最适温度测定 根据酶活测定方法,将突变酶分别置于10、20、30、35、40、50 和60 ℃的温度下反应15 min 后测酶的活性。把酶活力最大值所对应的温度作为酶的最适温度,并且设定为100%,然后计算其余温度条件下的相对酶活力,即得到脂肪酶在不同温度下的活力变化。
1.7.2 最适pH 测定 在最适温度下,将脂肪酶置于不同的缓冲液体系中:柠檬酸-磷酸盐缓冲液(pH 2.0 ~7.0)、Tris-HCl 缓冲液(pH 8.0 ~9.0)、甘氨酸-NaOH 缓冲液(pH 10.0 ~11.0),测定酶活。酶活最高的pH 为最适pH,并将其设定为100%,计算其他pH 条件下的相对酶活力。
1.7.3 半衰期t1/2将脂肪酶分别置于50℃水浴中处理15、30、45 和60 min,然后置于冰浴10 min后在最适反应条件下测残余酶活。
酶的失活方程为:ln(Et/E0)=-kt,其中E0:孵育0 时刻的相对酶活,Et:t 时刻的相对酶活;半衰期t1/2计算公式为:t1/2=ln2/k
1.7.4 脂肪酶动力学研究 在最适反应条件下,以对pNPP 为底物,测定其不同浓度(0.25 ~2.0 μmol/L)下脂肪酶活性,根据米氏方程,使用Lineweaver-Burk 双倒数作图法,计算出不同突变脂肪酶动力学常数。
1.7.5 突变脂肪酶三级结构模拟 酶分子的折叠自由能变化值ΔΔG 用在线软件PoPMuSiC-2.1(http://dezyme.com)计算。将脂肪酶突变后的蛋白序列提交至SWISS-MODEL(http://swissmodel.expasy.org/)[19]网站,以PDB 数据(2QUB)的晶体结构为模板进行同源建模。
2 结果与分析
2.1 脂肪酶突变位点的选择和构建
将S. marcescens 脂肪酶lipA 解析的晶体结构提交至PoPMuSiC 在线服务器预测计算脂肪酶每个突变氨基酸的去折叠自由能变化(ΔΔG)。一般认为,ΔΔGmut 值是负值,表明突变后能量下降,酶稳定性提高,反之突变后能量上升,酶稳定性较 差[20],根据突变后每个突变点ΔΔG 的变化,选择 Asn86Glu(ΔΔG:-0.19 kcal/mol)、Asn132Lys(ΔΔG:-0.1 kcal/mol)作为突变位点。
如图1 所示,用PCR 方法先将lipA 基因突变点两侧的长、短片段分别扩增出来,使lipA 基因序列中编码Asn86 的密码子AAT 突变为编码Glu 的密码子GAA;编码Asn132 的密码子AAT 突变为编码Lys 的密码子AAG,再通过融合PCR 扩增获得突变后的全长lipA 基因,插入pET22b 表达载体中,构建了表达载体pET22b-Asn86Glu 和pET22b-Asn132Lys。用限制性内切酶BamH I 和Hind III 双酶切验证表明2 个突变体构建成功。
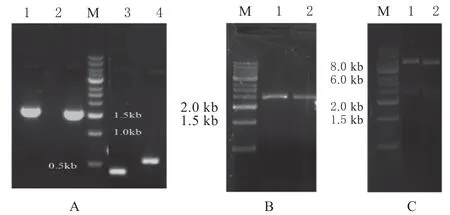
图1 Asn86Glu 和Asn132Lys 突变体的构建Fig.1 Construction of lipA Asn86Glu and Asn132Lys mutations
2.2 重组脂肪酶的表达和纯化
将野生型和2 个突变体的表达载体分别转化到E. coli Rosetta(DE3) 中获得阳性转化子,用IPTG诱导表达重组蛋白16 h 后,离心收集菌体并超声破碎,离心所得上清液含有可溶的脂肪酶蛋白(图2,泳道1 ~3),说明lipA 的野生型和突变体都得到了高效表达。使用Ni-NTA 分离纯化lipA,用咪唑浓度为200 mmol/L 的缓冲液洗脱目的蛋白,SDSPAGE 电泳结果(图2,泳道4 ~6)显示,纯化后的酶蛋白呈现单一条带,大小为65 kD,与野生型一致,表明lipA 脂肪酶的野生型和突变体均得到了完整高效的表达。
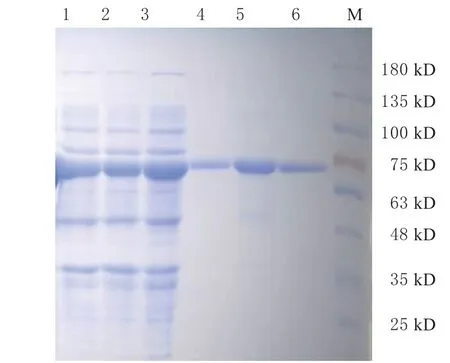
图2 脂肪酶lipA 粗提液及纯化后酶蛋白SDS-PAGE 分析Fig.2 MSDS-PAGE analysis of crude enzyme extracts and purified lipA
2.3 重组脂肪酶酶学性质
2.3.1 重组脂肪酶的酶活力 用纯化后的脂肪酶及其突变体测定了脂肪酶的活性,结果如图3 所示,测得野生型脂肪酶的比酶活为171.62 U/mg,lipAAsn86Glu 的比酶活为185.35 U/mg,lipA-Asn132Lys的比酶活为138.97 U/mg。lipA-Asn86Glu 的相对酶活比野生型提高了8%,lipA-Asn132Lys 的相对酶活较野生型下降了19%。表明点突变Asn86Glu 提高了酶的活性,而Asn132Lys 却降低了酶活性。

图3 野生型及突变体脂肪酶的酶活力Fig.3 The activity of the wild type and mutant lipases
2.3.2 重组脂肪酶的最适温度和最适pH 分别测定重组脂肪酶及突变体的最适温度,结果见图4。野生型脂肪酶的最适温度为30 ℃,2 个脂肪酶突变体Asn86Glu、Asn132Lys 的最适温度没有发生变化,均为30 ℃。10 ~30 ℃之间随温度提高,3 种酶的活力都在上升,野生型和Asn86Glu 都保持了较高的活力,10 ℃和20 ℃下分别为最适温度酶活力的50%和80%以上,而突变体Asn132Lys 的脂肪酶活性则有明显降低。35 ℃时,野生型和Asn86Glu 的活力略有下降,之后随着温度提高酶活力都大幅度降低,突变体Asn86Glu 较野生型的酶活力下降更为显著,表明Asn86Glu 突变可能导致lipA 在高温下容易发生构象变化,最终导致酶活力的丧失。对于突变体Asn132Lys 来说,尽管最适温度没有发生变化,但是所有被测温度下,其活性较野生型都有明显降低,表明该突变并未像预期的结果一样可以提高酶的稳定性,相反却降低了酶的稳定性。
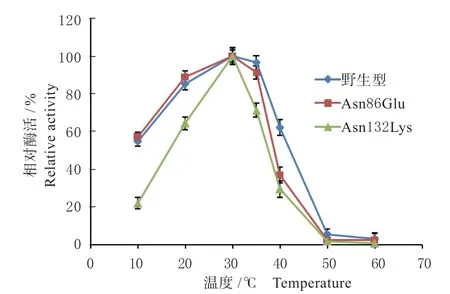
图4 野生型脂肪酶及突变体的最适温度Fig.4 Optimum temperature of the wild type and mutant lipases
如图5 所示,不同pH 下野生型和2 种突变体lipA 的活性呈现出相似的变化趋势,pH 8 时酶活性最高,为最适pH;pH 2 ~8 之间,随着pH 的增加酶活性也增加,pH 8 ~11 之间酶活性随pH 增加而显著下降,pH 5 ~9 之间可以表现出相对较高的活性。这结果证明Asn86Glu、Asn132Lys 这2 个突变体对脂肪酶活性的最适pH 没有影响,也没有影响其在高于或低于最适pH 前后酶活性的变化趋势。
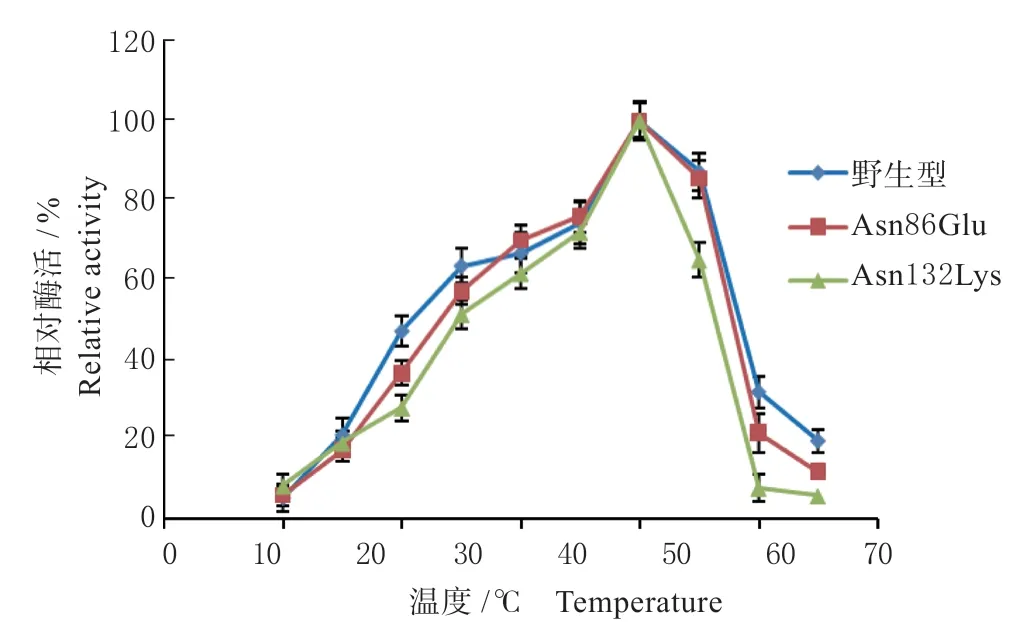
图5 野生型脂肪酶及突变体的最适pHFig.5 Optimal pH of the wild type and mutant lipases
2.3.3 脂肪酶的t1/250和动力学常数 将野生型和2种突变体脂肪酶在50 ℃下保温不同时间,检测半衰期,结果显示:与野生型相比,突变体Asn86Glu的半衰期与野生型几乎一致,而Asn132Lys 的半衰期则降低了51%(表2),说明前者突变并未造成热稳定性的剧烈变化,后者则正好相反,这一结果与最适温度测定中的结果一致。以0.5 ~2 mmol/L不同浓度的p-NPP 为底物测定脂肪酶及其突变体的动力学常数,结果见表2。相对于野生型脂肪酶,2个突变体的Km 值、Kcat 值都明显降低,说明底物与酶的亲和力变大了,但是最优条件下的催化效率却下降了。Asn86Glu 突变体的Kcat/Km 显著增加,Asn132Lys 的Kcat/Km 则显著减少,表明前者催化效率提高了,而后者则降低了,这是因2个突变对为Km 和Kcat 值有不同影响导致的。

表2 野生型脂肪酶及其突变体在50 ℃下的半衰期及动力学参数Table 2 Half-lives (50 ℃) and kinetic parameters of the wild type lipase and its mutants
2.3.4 突变脂肪酶的结构模拟与突变位点分析 为了探讨脂肪酶点突变对酶活性和热稳定性变化的影响,首先将野生型脂肪酶进行同源建模,根据突变点所在位置和氢键成键情况来分析突变之后酶活力或热稳定性改变的原因。如图6 所示,同源建模后发现野生型与突变体的三维结构没有明显的变化,而突变点处氨基酸残基改变后的成键情况则与突变前有所不同。如图6A 中的三级结构所示,位点Asn86 处在α 螺旋的末端,图7A、7B 表示Asn86 突变为Glu 后在该氨基酸残基及其周围氨基酸残基所在同一平面中的位置变化和氢键变化,可知突变后的氢键数量没变,但是所占空间结构变小,这一特点可能导致酶在与底物结合时更加有利,进而有利于突变后酶活提高,同时结构的变化也可能导致了酶的热稳定性变差。从图6B中的三级结构可知,位点Asn132 处在连接2 个β 叠片的拐角处,图7C 和7D 中所展示了Asn132 突变前后在空间结构中的二维截面上该氨基酸残基与周围氨基酸之间的位置变化和氢键变化,发现突变后氢键个数由原先的3 个变为2 个,这可能导致酶结构不稳定,使酶的活性和热稳定性均降低。
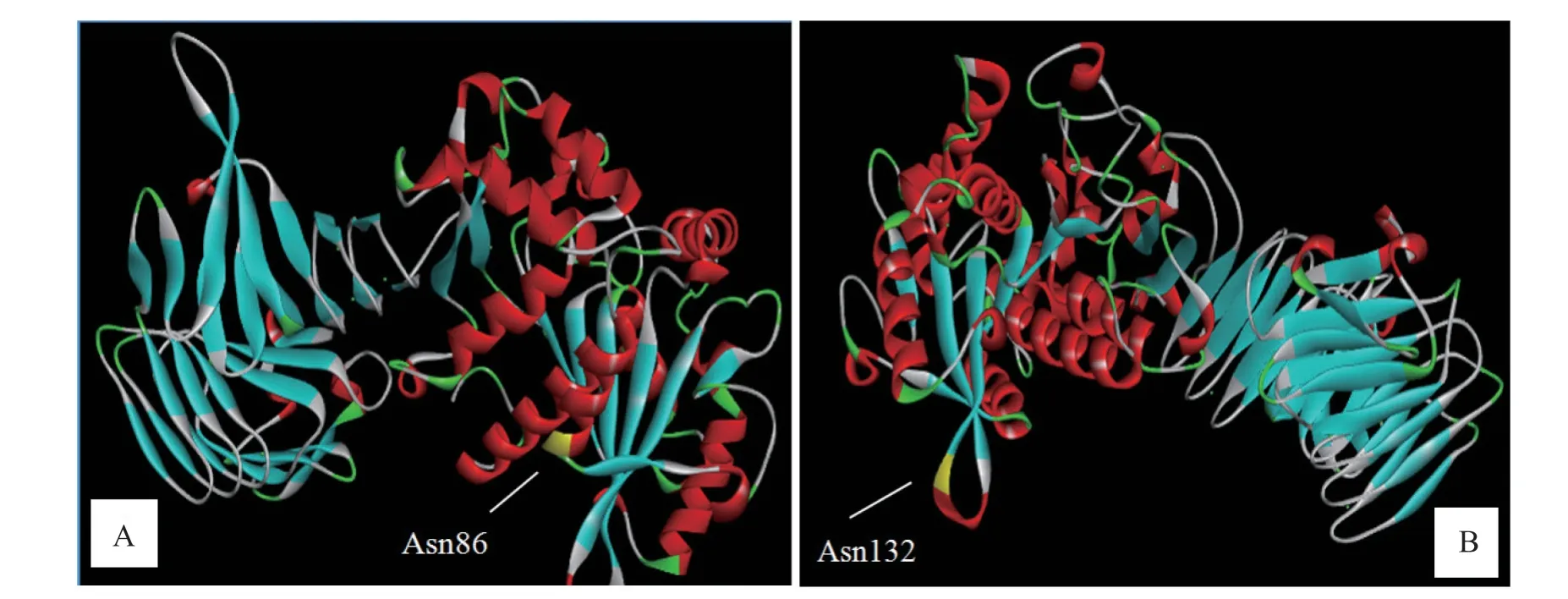
图 6 突变氨基酸在野生型脂肪酶3D 模型中的位置Fig.6 Location of the mutant residues in the 3D model of wild-type lipase

图7 氨基酸残基Asn86 和Asn132 突变前后在二维平面中的位置Fig.7 The location of residues Asn86 and Asn132 before and after the mutation on the 2D map
3 结论与讨论
利用在线预测软件 PoPMuSiC 计算酶蛋白序列中氨基酸替换点的去折叠自由能变化(ΔΔG)来辅助设计酶蛋白的定点突变,以预测氨基酸点突变后对酶的稳定性的变化,是点突变理性设计的一种常用方法。童理明等利用PoPMuSiC-2.1 在谷氨酰胺转氨酶上预测了2 个突变点P132I 和P132G,比酶活亦分别较野生酶提高了24%和12.4%[21]。由本试验对野生型和突变体的蛋白质结构预测分析可知,突变前后脂肪酶的蛋白结构及其酶活性中心基本没有发生改变。通过Asn 替换成Glu 以及Asn 替换成Lys 的方法得到2 个突变体,Asn86Glu 的酶活性较野生型提高了8%,而Asn132Lys 较野生型却下降了19%。lipA 及其突变体酶学特征的结果证明,突变体最适温度和最适pH 都和野生型一样,没有明显的变化。动力学分析结果表明,突变酶的Km 值比野生型的要小,说明突变酶对底物的亲和力有所提高。最后利用SWISS-MODEL 在线服务器将S. marcescens L1 脂肪酶野生型和突变后的蛋白序列输入并对其进行同源模拟,发现突变点Asn86Glu 和Asn132Lys 分别通过构象改变和氢键减少导致酶的热稳定性降低,但是Asn86 的酶活力高于野生型,热稳定性在低温下相差不大。这一结果暗示了,试图通过理性设计提高酶的热稳定性时,可选择突变后建模模拟氢键增加的氨基酸位点。
通过在线预测软件PoPMuSiC 预测并成功构建了脂肪酶lipA 的2 个突变体,对野生型和突变体表达体系构建、重组脂肪酶lipA 的表达、纯化和酶学特性的表征,证明突变体lipA-Asn86Glu 可以提高酶活力和催化效率,高温下稳定性略有降低,说明理性设计的定点突变对酶的活力和热稳定性有一定的影响,为脂肪酶的分子改造提供了实验依据和途径。

