水稻典型品种日本晴和IR24根系微生物组的解析
胡雅丽,戴睿,刘永鑫,张婧赢,胡斌,储成才,袁怀波,白洋
研究报告
水稻典型品种日本晴和IR24根系微生物组的解析
胡雅丽1,2,3,戴睿2,3,4,5,刘永鑫2,3,4,张婧赢2,3,4,胡斌2,储成才2,袁怀波1,白洋2,3,4,5
1. 合肥工业大学,食品与生物工程学院,合肥 230009 2. 中国科学院遗传与发育生物学研究所,植物基因组学国家重点实验室,北京 100101 3. 中国科学院大学,生物互作卓越创新中心,北京 100049 4. 中国科学院遗传与发育生物学研究所,中国科学院–英国约翰英纳斯中心植物和微生物科学联合研究中心,北京 100101 5. 中国科学院大学现代农学院,北京 100049
植物的各项生命活动与其根系微生物组密不可分,且根系微生物组的组成易受到植物生长环境和基因型的影响。为进一步探究中国北方地区种植的不同品种水稻根系微生物组的差异及其相互作用机制,本研究以种植于北京昌平和上庄农场的水稻典型品种日本晴(Nipponbare)和IR24为研究对象,基于16S rRNA基因扩增子测序技术获得根系微生物组序列,利用多样性分析、组成型分析、机器学习的随机森林和网络分析等方法,对旺盛生长期的两种不同品种的水稻根系微生物组进行详细比较。研究发现,种植地点和水稻基因型显著影响了水稻根系微生物组的群落结构,不同基因型导致了根系微生物组在物种分类组成上以及细菌间相互关系的差异,而且根系微生物组能作为生物标记跨地点区分宿主的基因型。本研究结果为深入理解我国北方种植的水稻根系微生物组的组成规律以及从根系微生物与植物互作的角度对品种进行改良提供了数据和理论基础。
水稻根系生物组;多样性分析;组成型分析;机器学习;网络分析
在自然界中,陆生植物的根部聚集着特定种类的微生物,被称为根系微生物组[1~4]。根系微生物组与植物之间的相互作用对植物的各项生命活动具有重要影响,如促进植物生长[5~7]、防止微生物病原体的侵害[8~10]等。根系微生物还能够转化、分解植物难以吸收利用的营养物质,进而提高植物对营养物质的利用效率[11]。例如,拟南芥()根际微生物群落介导了磷胁迫反应,参与了自身的免疫调控[12];富集在玉米()气生根中的细菌参与了玉米的固氮过程[13,14]。同时植物根系分泌物能为根际细菌提供生命活动所需的代谢产物进而招募有益菌,而这些代谢产物也能够抑制某些菌在植物根系的富集[15,16]。
随着高通量测序技术的发展和微生物组研究的深入,拟南芥[17~20]、水稻()[21~23]、小麦()[24~26]、玉米[27]、番茄()[28,29]、谷子()[30]等多种植物根系微生物组的结构得到了解析。研究表明,在单子叶和双子叶植物中,根系微生物组主要包括放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)[3,31]。影响根系微生物组结构的因素很多,其中包括种植环境的土壤条件(如pH值、土壤水分、碳、氮和锌等重要元素的含量)以及植物自身的调节[32~34]。不同物种的植物根系微生物组结构存在差异[15],此外不同基因型的植物对环境响应不同,其根系微生物组的结构也可能不同[11]。
水稻是世界上重要的粮食作物,约有1/3的世界人口以水稻为主食[35,36]。亚洲栽培稻主要包含两个亚种:粳稻()和籼稻(),这两个品种是目前国内最广泛种植的两个水稻亚种[37]。二者在形态特征、抗虫抗病性和营养利用效率上有着显著区别。日本晴(Nipponbare)和IR24分别为典型的粳稻和籼稻品种,二者的根系微生物组随着生长时期动态变化,并在旺盛生长期逐渐稳定[22]。然而,对该时期种植于不同地区下的这两种水稻的根系微生物组的结构仍然缺乏系统解析。本研究以种植于北京昌平和上庄的日本晴和IR24的根系微生物组为材料,利用机器学习和网络分析等方法,对两种基因型的水稻根系微生物组进行详细比较。
1 材料与方法
1.1 植物材料
选取典型粳稻代表品种日本晴以及籼稻代表品种IR24作为实验材料,实验所用材料均由中国科 学院遗传与发育生物学研究所储成才研究员实验室提供。
1.2 水稻种植
水稻材料种植于北京上庄农场(116.206E, 40.122N) (SZ)和北京昌平农场(116.424E, 40.109N) (CP),种植时间为2017年6月下旬,并于8月下旬取样。昌平农场的土质为泥质壤土,而上庄农场的土质为粘壤土。此外两个农场的土壤肥力不同,水稻长势存在差异。这两个农场的田地近几年只用于种植水稻,有助于在我国北方典型的水稻农业土壤中研究不同基因型间水稻根系微生物的异同。为了避免种子内生菌和表面相关微生物的影响,将水稻种子脱壳,在75%乙醇中表面消毒30 s,用2.5%次氯酸钠消毒3次,每次持续15 min,最后用无菌去离子水清洗5遍,然后在MS琼脂培养基中促进萌发,得无菌组培苗。约15 d后移栽至农场的水稻田中,在昌平农场中每个基因型各设置15个重复,在上庄农场,IR24有17个重复,日本晴有15个重复,共计62个样品。田里的灌溉水面处于刚浸没土壤状态,同时设置保护行。
1.3 样品收集
在移栽8周后,即水稻旺盛生长期,收集0~ 10 cm深度的水稻根。先用无菌水洗掉粘在水稻根上的松散的土壤颗粒,接着放入装有25 mL PBS缓冲液的50 mL管中,180 r/min摇晃清洗3次,然后用无菌滤纸吸干根,最后将根和相应的土壤放入−80℃中保存。
1.4 DNA提取、16S扩增子文库构建和测序
将冷冻的土壤样品用Precellys Evolution生物样品研磨器(Bertin Technologies,法国)在7200 r/min下匀质4次,每次持续30 s,然后根据制造商的说明使用FastDNA SPIN试剂盒(MP Biomedicals,美国)提取DNA。将冷冻的根化冻后在7200 r/min下匀质2次,每次30 s,之后放入液氮中冷冻5 s,在7200 r/min下匀质2次,每次30 s,再加入1100 µL缓冲液,在7200 r/min下匀质4次,每次30 s。使用FastDNA SPIN试剂盒(MP Biomedicals,美国)提取DNA。DNA浓度用PicoGreen dsDNA检测试剂盒(Life Technologies,美国)测定,最后将DNA浓度稀释到3.5 ng/µL。
用简并PCR引物799F和1193R对16S rRNA基因的可变区V5~V7进行聚合酶链式反应(polymerase chain reaction, PCR)扩增(表1)。每个样品在30 µL体系中一式3份(与体系作为对照)扩增,该反应体系包括3 µL模板,0.3 µL PrimeSTAR HS DNA聚合酶,1×PrimeSTAR缓冲液(TaKaRa,日本),2.4 µL dNTPs和正反向引物各0.75 µL。在98℃进行30 s初始变性后,将目标区域在98℃进行10 s,55℃进行15 s,72℃进行1 min的25个循环下扩增,最终72℃下进行5 min。用琼脂凝胶电泳验证PCR结果,若阴性未出带,则将3份样品合并,并用AMPure XP试剂盒(Beckman Coulter,美国)进行纯化,纯化后用Nanodrop (NanoDrop 2000C, Thermo Scientific)检测DNA浓度,并将DNA浓度稀释至10 ng/µL作为模板,用于使用Illumina兼容引物进行第二步PCR。在与第一轮PCR相同的条件下,将DNA样品一式3等份扩增8个循环。若阴性对照没有出带,则合并该样品的这3个技术重复。在1.2%琼脂糖凝胶上分离,并使用QIAquick凝胶提取试剂盒(Qiagen,美国)根据制造商的说明提取细菌16S rRNA基因扩增子。用PicoGreen dsDNA检测试剂盒(Life technologies,美国)测定DNA浓度,每个样品取200 ng。使用Agencourt AMPure XP试剂盒(Beckman Coulter GmbH,美国)将最终的扩增子文库清洗2次,并在HiSeq 2500平台(Illumina Inc.,美国)上进行测序[38]。
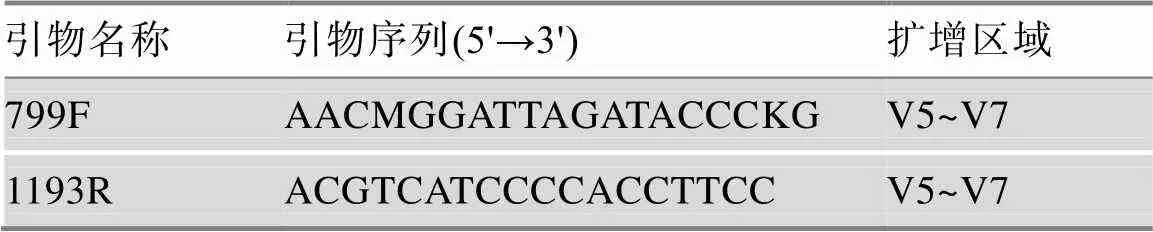
表1 用于扩增16S rRNA基因的引物序列
1.5 生物信息学分析
16S rRNA基因扩增子数据分析主要使用QIIME 1.9.1[39]、QIIME 2[40]、USEARCH 10.0[41]以及本研究团队编写的R脚本(https://github.com/raydai0218/ Hu2020Hereditas)[11,22,42,43]。主要步骤包括使用QIIME 1.9.1脚本extract_barcodes.py和split_libraries_ fastq.py对测序数据进行拆分,USEARCH对序列进行处理,用-fasteq_mergepairs命令合并双端序列,然后切除引物(-fastx_truncate),过滤低质量序列(-fastq_filter)并去冗余(-fastx_uniques)。之后用-cluster_otus命令基于UPARSE算法按97%相似度聚类为可操作分类单元(operational taxonomic unit, OTU)。最后基于SILVA数据库去除嵌合体和宿主后获得特征序列,再使用USEARCH的-otutab命令(-id 0.97)比对生成OTU表。此外用USEARCH的-sintax命令进行物种注释(-sintax_cutoff 0.6)。基于OTU表的下游分析和可视化主要在R 3.6.1语言(https://cran. r-project.org/)环境下实现,包括Alpha-和Beta-多样性分析采用QIIME 2和R包vegan 2.5-6实现[44]、差异比较主要基于秩和检验(Wilcoxon rank sum test)、可视化采用R包ggplot2 3.2.1[45]和在线绘图网站imageGP (http://www.ehbio.com/ImageGP)实现。同时,在探索水稻根系细菌微生物组作为潜在生物标注的研究中,采用R包randomForest 4.6-14中的随机森林分类算法实现[46,47]。网络分析采用R包igraph 1.2.4.2实现[48],网络的比较方法和可视化方案参考之前的研究[32,49,50]。
1.6 数据和可重复分析代码
本研究涉及的原始序列数据已保存在中国科学院北京基因组研究所BIG数据中心[51]的基因组序列档案库(Genome Sequence Archive)[52]中,编号CRA001372,可在https://bigd.big.ac.cn/gsa上公开访问。本研究的计算分析过程使用Rmarkdown可重复分析标准,分析中使用的数据表、代码和结果图表详见https://github.com/raydai0218/Hu2020Hereditas。
2 结果与分析
2.1 种植地点和品种差异影响水稻根系微生物组成
高通量测序最终从62个样品中共生成了3,376,530条高质量序列(平均54,460条,每个样品中读段数从32,390条到76,887条不等,54,460 ± 11,193.17)。用USEARCH对高质量序列进行质控分析,去冗余、去除嵌合体及宿主序列后,得到19,073个OTU。
多样性分析显示水稻根系微生物组受种植地点和基因型的影响。本研究对样品间Bray-Curtis距离做非限制性主坐标分析(principal coordinate analysis, PCoA) (图1A),结果显示在第一主坐标轴(PCo1)上水稻的根系微生物组按照地区被分为了两簇,表明种植地区差异导致了水稻根系微生物的组成不同,这是本研究中最主要的微生物差异来源。基因型是导致第二主坐标轴(PCo2)上样品差异的主要因素,来自两个种植地区的同一基因型在第二轴上的位置是一致的,说明水稻根系微生物组受到基因型的影响。通过比较样品中的物种丰富度,本研究发现种植地区和水稻基因型没有显著影响根系微生物的物种丰富度。种植于上庄的水稻根系物种丰富度较昌平的稍高,同一地区的两个基因型间丰富度指数(richness index)无明显差别,最小显著差异法(least significant difference, LSD)比较各组间差异的结果显示种植地区和基因型间的差异均不显著(> 0.05) (图1B)。
2.2 日本晴和IR24的根系微生物组在不同分类水平上存在差异
本研究对日本晴和IR24的根系微生物组在不同分类水平进行差异比较(图2)。在门和纲水平上,两个种植地区中拟杆菌门(Bacteroidetes)和gamma-变形菌纲(Gammaproteobacteria)在日本晴根系的相对丰度都显著高于IR24,而beta-变形菌纲(Betaproteobacteria)则在IR24根系中相对丰度更高(Wilcoxon秩和检验,< 0.05,< 0.2)。在上庄,放线菌门(Actinobacteria)在IR24根系的相对丰度比日本晴更高,但在昌平差异不显著;而delta-变形菌纲(Deltaproteobacteria)则在昌平的日本晴中较IR24相对丰度更高,在上庄无显著区别,这可能与种植环境对土壤微生物在水稻根系定植的影响有关(图2)。
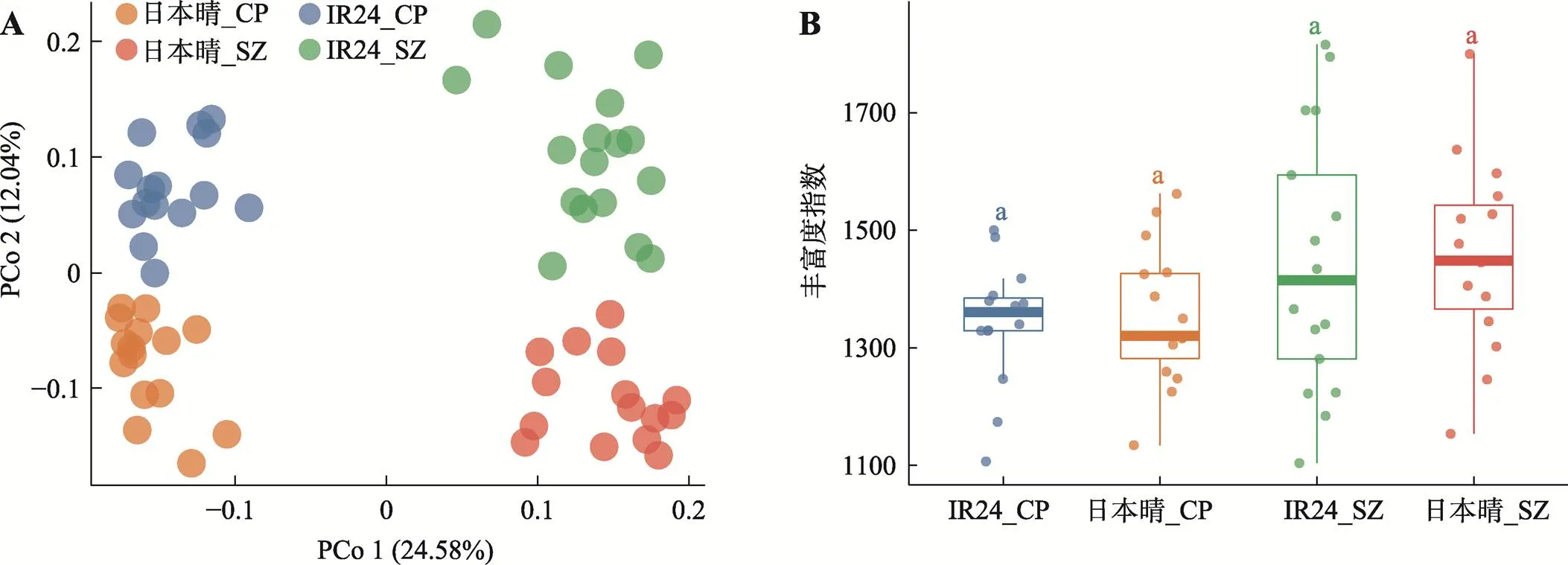
图1 种植地点和品种差异影响水稻根系微生物组成
A:基于样品间Bray Curtis距离的主坐标分析(PCoA)。日本晴和IR24的根系微生物组在PCoA的第一轴上(PCo 1)按种植地点开,第二轴上(PCo 2)按基因型分开,组间差异显著(PERMANOVA,< 0.001)。CP:昌平;SZ:上庄;坐标轴标题括号中显示整体差异的解释率百分比。B:日本晴和IR24根系微生物组的丰富度指数(richness index,样品内物种多样性)。箱线图中框内横线表示中位数,上下边缘分别代表上下四分位数,边缘上的延长线在无异常值时至极值,但最长不超过1.5倍上下四分位数的分布区间。各组间丰富度指数差异不明显,最小显著差异法(least significant difference, LSD)比较组间差异,4组均为a表示地点和基因型对根系微生物组的Alpha多样性影响不显著(> 0.05)。
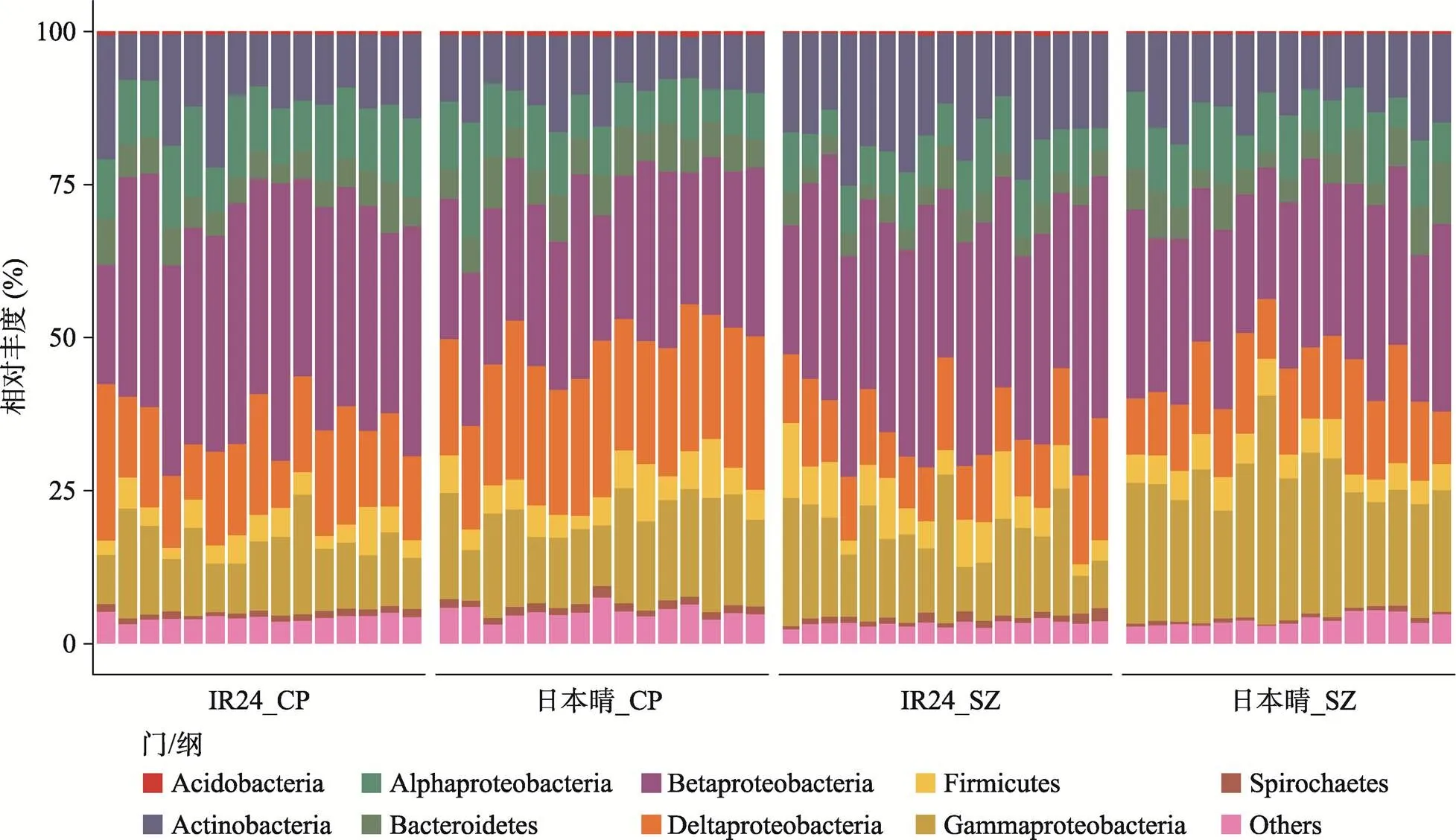
图2 日本晴和IR24的根系微生物组在门/纲级分布上存在差异
柱状图展示每个样品中根系微生物组的物种组成(门和纲水平的相对丰度)。变形菌门(Proteobacteria)因丰度较高在纲水平展开为alpha-、beta-、gamma-和delta-变形菌4个纲,其余为门水平。Acidobacteria:酸杆菌门;Actinobacteria:放线菌门;Bacteroidetes:拟杆菌门;Firmicutes:厚壁菌门;Spirochaetes:螺旋体门。在两个种植地区,IR24根系微生物中的beta-变形菌纲显著高于日本晴,而拟杆菌门和gamma-变形菌纲在日本晴的根系微生物组中丰度更高,此外放线菌门(上庄)和delta-变形菌纲(昌平)只在一个地区在两基因型间有显著差异(Wilcoxon秩和检验,< 0.05,FDR < 0.2)。CP:昌平;SZ:上庄。每组样品的数量为:IR2_CP (= 15),日本晴_CP (= 15),IR24_SZ (= 17),日本晴_SZ (= 15)。
在OTU水平,日本晴和IR24中富集的OTU在昌平和上庄存在重叠:有66个OTU在两个地区的日本晴中共同富集,占昌平中富集的39.8%、上庄中富集的31.8% (图3A)。这66个OTU主要属于拟杆菌门(Bacteroidetes,9个),厚壁菌门(Firmicutes,16个)和gamma-变形菌纲(Gammaproteobacteria,15个) (图3B)。有45个OTU在两个地区的IR24中共同富集,占昌平中富集的26.2%、上庄中富集的33.3% (图3C)。这45个OTU主要属于beta-变形菌纲(Betaproteobacteria,17个),厚壁菌门(Firmicutes,8个)和delta-变形菌纲(Deltaproteobacteria,5个) (图3D)。总体来说,OTU水平在两基因型间差异富集的菌在门水平主要属于放线菌门、拟杆菌门、厚壁菌门以及变形菌门。
2.3 基于物种组成的随机森林分类模型精准预测水稻基因型
为了探究能否利用根系微生物作为生物标记菌来预测宿主基因型,本研究用机器学习的随机森林方法对上庄和昌平各一半的日本晴和IR24根系微生物组(共34个样品)在科水平上建立判别模型,并用十倍交叉验证重复4次以评估根系菌对于判定水稻基因型的准确性。从交叉验证误差曲线可以看出,当使用14个菌时,曲线的错误率趋于稳定,故选择对模型重要性最高的前14个菌作为生物标记菌(图4A)。其中重要性较高的生物标记菌主要是杆菌纲(Bacilli)以及beta-、gamma-变形菌纲。这14个标记菌有13个菌的平均相对丰度都在3/‰以上,只有假衣原体科(Parachlamydiaceae)的相对丰度较低(图4B)。为了验证该模型的准确性,本研究用未参与模型训练的28个样品进行测试(图4C),测试结果准确率较高(96.4%),仅有一个样品出错。说明两个种植地区的日本晴和IR24的根系微生物组存在一致差异,随机森林模型能够依据该差异对水稻基因型作出较准确预测。
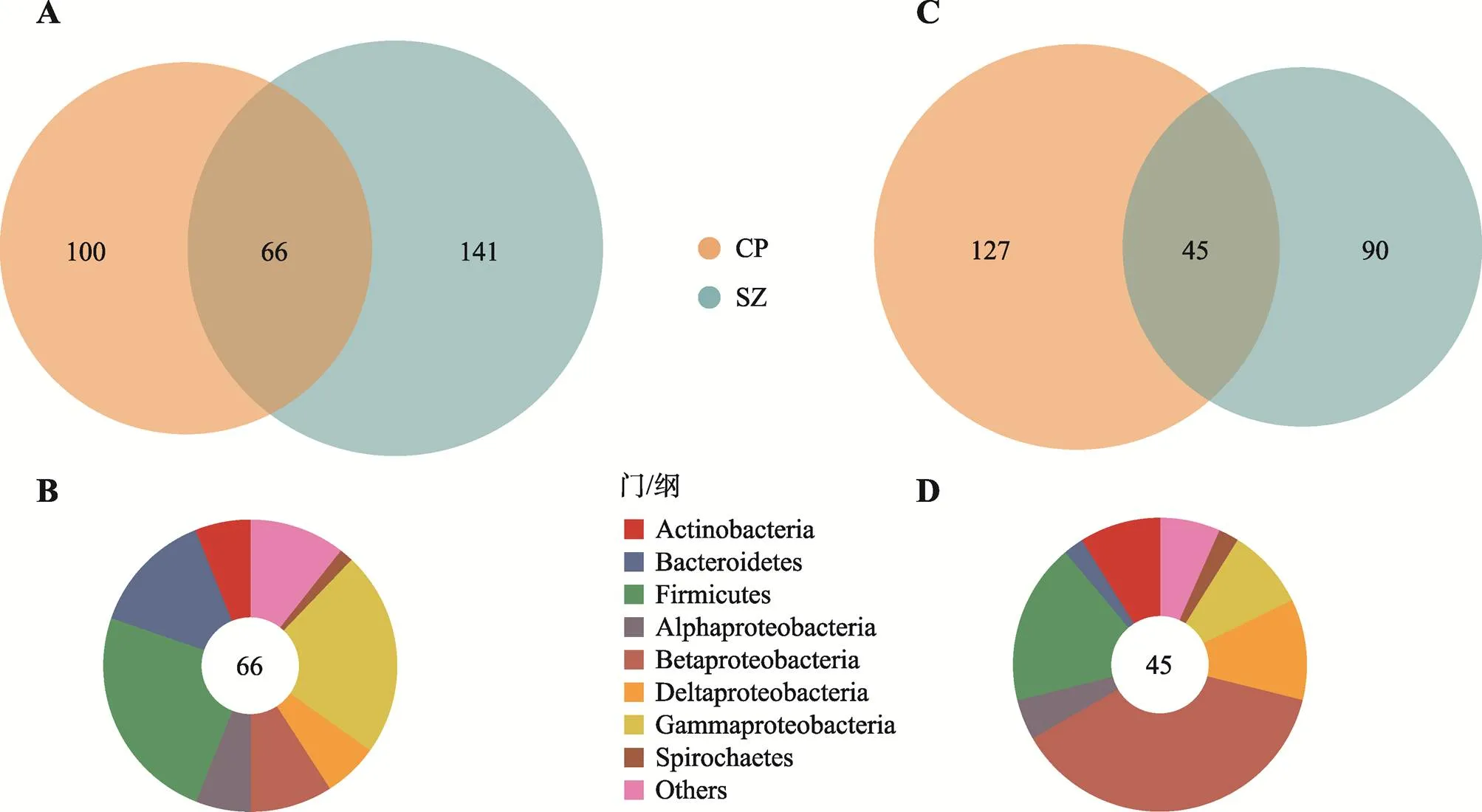
图3 日本晴和IR24根系的差异OTU及对应门纲水平分类组成
A:在两个种植地点中日本晴根系富集的OTUs。与IR24相比,日本晴在昌平(CP)和上庄(SZ)分别有166个和207个OTU富集,且其中有66个OTU在这两个种植地区都富集(Wilcoxon秩和检验用于比较差异OTUs,当< 0.05、< 0.2且差异倍数 > 1.2时,认为该OTU在两组样品间存在差异)。B:日本晴在两个种植地区均富集的根系OTUs的分类组成。共同富集的66个OTU大部分属于拟杆菌门,厚壁菌门和gamma-变形菌纲。C:在两个种植地点中IR24根系富集的OTUs。与日本晴相比,IR24在昌平和上庄分别有172个和135个富集OTUs,且其中有45个OTUs在这两个种植地区都富集。D:IR24在两个种植地区均富集的根系OTU的分类组成。共同富集的45个OTU大部分属于beta-变形菌纲,厚壁菌门和delta-变形菌纲。从图中可知,水稻基因型影响了微生物在根系的富集。Actinobacteria:放线菌门;Bacteroidetes:拟杆菌门;Firmicutes:厚壁菌门;Spirochaetes:螺旋体门;其余为alpha-、beta-、delta-、gamma-变形菌纲。
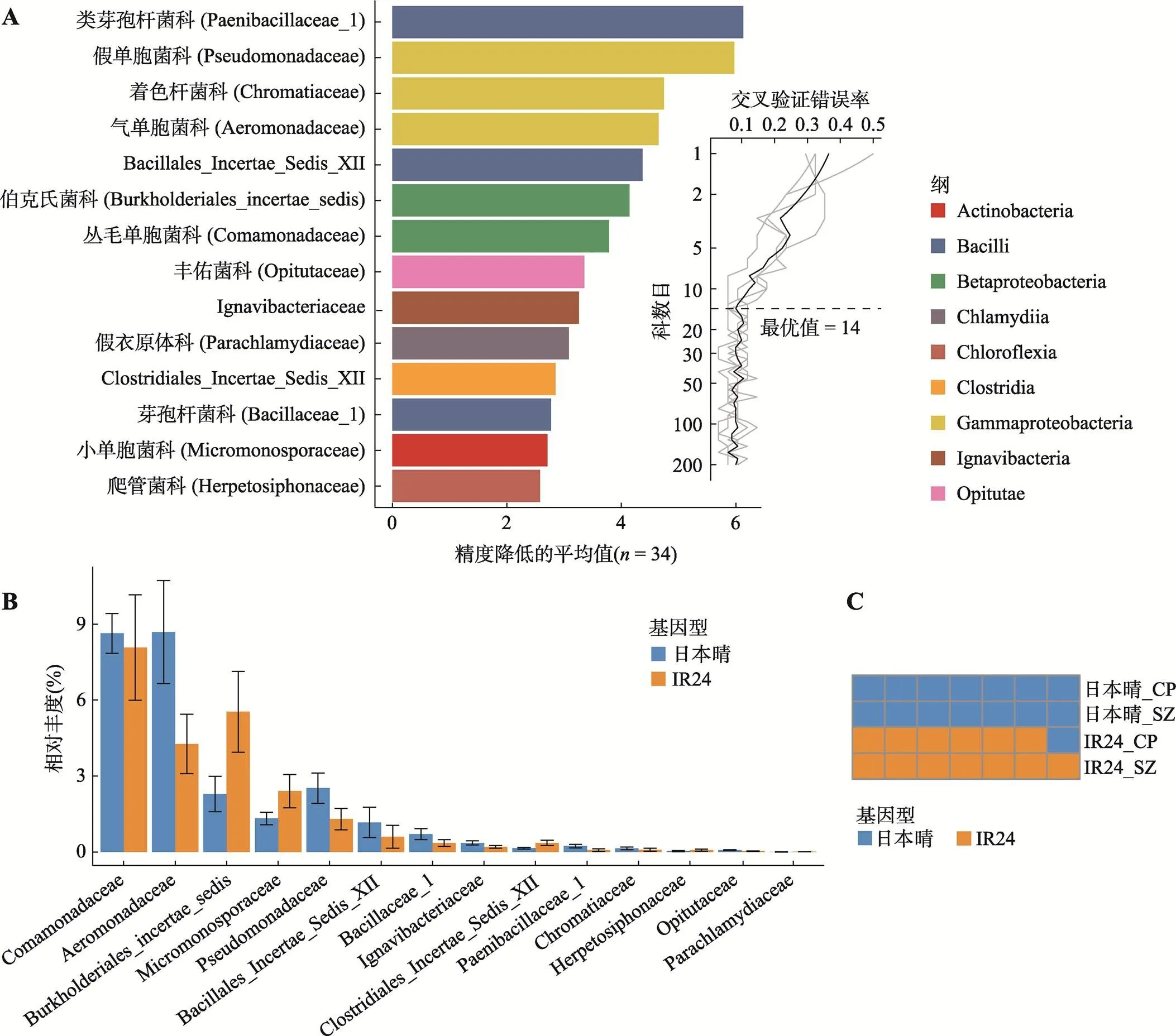
图4 基于根系微生物组的随机森林分类模型能较精准预测日本晴
A:随机森林分类模型中重要性排前14的生物标记菌。采用随机森林分类法,对昌平和上庄地区的IR24和日本晴根系微生物组(共34个样品)在科水平进行了分类,并在纲水平着色。生物标记菌按照对模型准确度的重要性由高到低排序,展示前14个。图右侧小图表示十倍交叉验证错误率,当标记菌个数超过14个时,模型的错误率较低且稳定。B:标记菌在日本晴和IR24根系微生物组中的相对丰度。蓝色为日本晴,橙色为IR24。C:用随机森林分类模型对两个基因型做分类预测。该模型基于样品的根系微生物组成判断样品的基因型。前两行样品为日本晴,后两行样品为IR24,蓝色表示样品基因型被预测为日本晴,橙色则是预测为IR24。CP:昌平;SZ:上庄。Actinobacteria:放线菌纲;Bacilli:杆菌纲;Betaproteobacteria:beta-变形菌纲;Chlamydiia:衣原体纲;Chloroflexia:绿弯菌纲;Clostridia:梭菌纲;Gammaproteobacteria:gamma-变形菌纲;Opitutae:丰佑菌纲。
2.4 日本晴和IR24的根系微生物网络存在明显差异
为了比较两个基因型中菌的相互关系,对日本晴和IR24的根系菌在科水平构建了相关性网络,网络中细菌间相关性及共有菌的连接度揭示了基因型对根系微生物共存网络的影响。两个网络的结构不同,日本晴的网络(图5A)复杂度比IR24 (图5B)的更低,科水平两基因型间的差异菌在两个网络中也各不相同。例如,两个网络中共有的科水平差异菌有地杆菌科(Geobacteraceae),其中地杆菌科在日本晴网络中与硝化螺旋菌科(、螺旋体科(正相关,而在IR24的网络中只与正相关关系。为了量化两个基因型间网络的差异,本研究比较两个网络中共有菌及其网络关系(连接度) (图5C),可见两个网络中共有的菌有34个(占日本晴网络中菌的65.4%,占IR24的44.7%),其中有部分菌在两个网络中有明显差别。梭菌科(和在日本晴网络中的连接度都在6以上,而在IR24中连接度较低,分别为3和1;相反,唇形科(和动球菌科(在IR24网络中连接度都为6,但在日本晴中只与一个菌有相互关系(图5C)。
3 讨论
植物根系微生物组与植物的关系密不可分,它参与调控植物的生长、免疫、繁殖等重要生命活动[3,53~56]。多项研究表明,定植在植物根系的微生物受多种因素影响,包括土壤微生物组[53]、土壤的pH值和元素含量等理化性质[32,33,57,58]、植物的物种及次级代谢产物等[31,59~61]。植物的基因型也是影响其根系微生物组的重要因素,在拟南芥[17,18]、玉米[27,62]、水稻[21~23]等重要植物中,都有相关研究阐明基因型对根系微生物带来的影响。日本晴和IR24分别属于粳稻和籼稻,在形态、籽粒以及抗病性等方面都有不同[63]。本研究以这两个水稻典型品种为研究对象,在两个不同种植地区对它们的根系微生物从多个角度进行比较,发现日本晴和IR24的根系微生物组有明显差异:多样性分析证明种植地区和水稻基因型影响了根系群落间的差异(图1A);两基因型在不同分类水平上都存在差异富集的细菌类群(图2,图3);随机森林分类模型能够按照根系生物标记菌对宿主进行分类,并在种植地区不同的前提下准确地预测日本晴(图4);日本晴和IR24的共存网络也有明显差别(图5)。
主坐标分析的结果表明种植地区影响了水稻根系菌群的结构(图1A),这与先前的研究结果一致[11,21]。土壤中真菌和细菌的相互作用参与塑造了宿主相关的根系菌群[5,64],土壤中的有益菌会在植物根中定植,因而土壤中微生物的结构同样会影响水稻根系微生物组的结构。本研究发现在两个种植地区中水稻基因型均分开,且相对位置一致。在一项对多品种的籼稻和粳稻根系微生物组的分析中,同样发现两块田间中不同品种的水稻分开趋势一致[21]。这说明水稻基因型对根系微生物组的影响在不同地区间存在一定共性和差异。变形菌门为在水稻根系主要富集的菌[1,2],其中beta-变形菌纲(Betaproteobacteria)在IR24根系的相对丰度显著高于日本晴,而gamma-变形菌纲(Gammaproteobacteria)在日本晴中丰度更高;此外拟杆菌门(Bacteroidetes)在两个品种间也存在差异(图2),且这些差异在两个地区均显著。而放线菌门(Actinobacteria)和delta-变形菌纲(Deltaproteobacteria)则分别只在上庄或昌平的两基因型间存在显著差异。
水稻的根系微生物受基因型影响,并在生长发育过程中有动态的变化[22,23]。因此,水稻根系菌能够像水稻表型特征一样可以被用来判断水稻的生长时期和基因型。本研究采用随机森林分类法建立了水稻日本晴和IR24的判别模型,尽管种植地区会影响水稻根系微生物组的结构(图1,图2),但该模型依旧能精准地根据水稻根系微生物预测出水稻基因型(图4)。增加样本数量并设置更多种植地区将有助于建立更为稳定、适用性更加广泛的分类预测模型,该模型能够降低种植地区带来的干扰,基于植物根系微生物组的组成特点判断植物的基因型,并有利于后续通过对标记菌的深入研究探索不同基因型的植物与根系微生物的相互作用关系。微生物共存网络(co-occurrence network)能够直观地展现各个微生物间的相互关系,从而找到按样品丰度所做的差异比较中被忽略的菌间作用[65]。通过比较日本晴和IR24的共存网络(图5),本研究发现二者网络中存在相互关系的菌有差异,且IR24的网络拓扑结构复杂性更高。这些相互作用可能与水稻的生长发育等生命活动相关。因此,可以挖掘网络中关键差异菌的潜在功能,进而探究基因型导致的根系微生物组差异会如何作用于植物进而产生调控作用。本研究结果从多角度反映了我国北方农田中日本晴和IR24的根系微生物组的差异,为从根系微生物与植物互作的角度对品种进行改良提供理论基础。
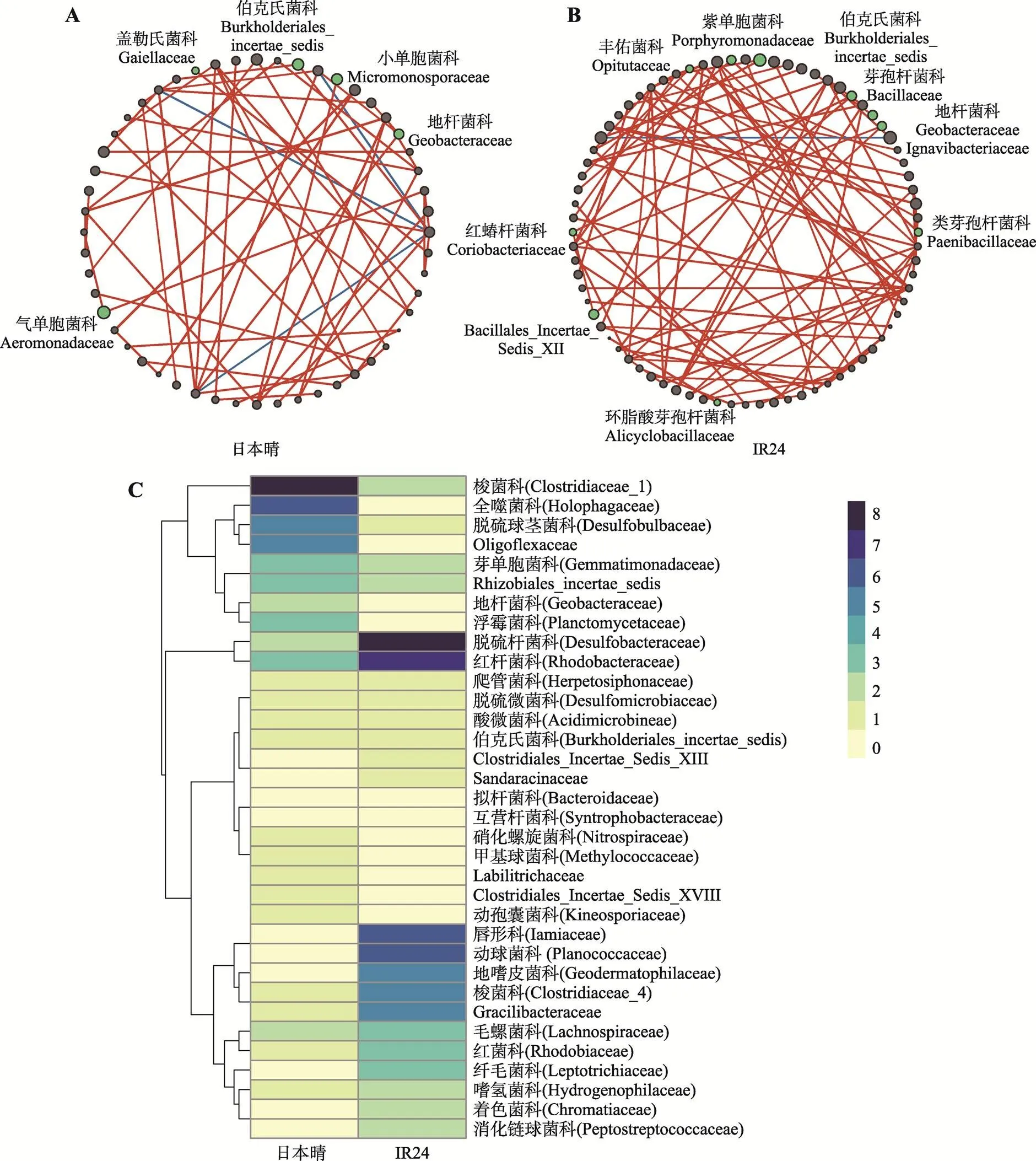
图5 日本晴和IR24的根系菌群形成的共存网络有明显区别
A:日本晴的微生物群落在科水平形成的共存网络。网络中共有52个节点,59条边。B:IR24的微生物群落在科水平形成的共存网络。网络中共有76个节点,93条边。共存网络关系利用Pearson相关系数判断科水平菌间相关性(< 0.05,|| > 0.7),网络中每个节点代表一个科,节点大小表示该科在每组样品中的平均相对丰度。其中绿色节点为科水平在两个基因型间存在差异的根系菌(Wilcoxon秩和检验,< 0.05、FDR < 0.2且差异倍数 > 1.2)。连接节点的边表示该两个节点间的相关性,红色为正相关,蓝色为负相关,线条粗细与相关性高低成正比。C:两个基因型网络间共有菌的连接紧密度比较。两个网络中共有34个菌,统计其中每个菌作为节点与网络中其他菌的关联度(即连接该节点的边的个数)。CP:昌平;SZ:上庄。
[1] Lundberg DS, Lebeis SL, Paredes SH, Yourstone S, Gehring J, Malfatti S, Tremblay J, Engelbrektson A, Kunin V, Rio TGd, Edgar RC, Eickhorst T, Ley RE, Hugenholtz P, Tringe SG, Dangl JL. Defining the coreroot microbiome.,2012, 488(7409): 86–90.
[2] Bulgarelli D, Rott M, Schlaeppi K, Ver Loren van Themaat E, Ahmadinejad N, Assenza F, Rauf P, Huettel B, Reinhardt R, Schmelzer E,Peplies J, Gloeckner FO, Amann R, Eickhorst T, Schulze-Lefert P. Revealing structure and assembly cues forroot-inhabiting bacterial microbiota.,2012, 488(7409): 91–95.
[3] Müller DB, Vogel C, Bai Y, Vorholt JA. The plant microbiota: systems-level insights and perspectives.,2016, 50(1): 211–234.
[4] Bai Y, Qian JM, Zhou JM, Qian W. Crop Microbiome: breakthrough technology for agriculture., 2017, 32(3): 260–265.白洋, 钱景美, 周俭民, 钱韦. 农作物微生物组:跨越转化临界点的现代生物技术. 中国科学院院刊,2017, 32(3):260–265.
[5] Durán P, Thiergart T, Garrido-Oter R, Agler M, Kemen E, Schulze-Lefert P, Hacquard S. Microbial interkingdom interactions in roots promotesurvival.,2018, 175(4): 973–983.
[6] Hiruma K, Gerlach N, Sacristán S, Nakano RT, Hacquard S, Kracher B, Neumann U, Ramírez D, Bucher M, O’Connell RJ, Schulze-Lefert P. Root endophyte colletotrichum tofieldiae confers plant fitness benefits that are phosphate status dependent.,2016, 165(2): 464–474.
[7] Wang XL, Wang ET. NRT1.1B connects root microbiota and nitrogen use in rice., 2019, 54(3): 285–287.王孝林, 王二涛. 根际微生物促进水稻氮利用的机制. 植物学报,2019, 54(3): 285–287.
[8] Kwak MJ, Kong HG, Choi K, Kwon SK, Song JY, Lee J, Lee PA, Choi SY, Seo M, Lee HJ, Jung EJ, Park H, Roy N, Kim H, Lee MM, Rubin EM, Lee SW, Kim JF. Rhizosphere microbiome structure alters to enable wilt resistance in tomato.,2018, 36: 1100–1109.
[9] Wang XF, Wei Z, Yang KM, Wang JN, Jousset A, Xu YC, Shen QR, Friman VP. Phage combination therapies for bacterial wilt disease in tomato.,2019, 37(12): 1513–1520.
[10] Carrión VJ, Perez-Jaramillo J, Cordovez V, Tracanna V, de Hollander M, Ruiz-Buck D, Mendes LW, van Ijcken WFJ, Gomez-Exposito R, Elsayed SS, Mohanraju P, Arifah A, van der Oost J, Paulson JN, Mendes R, van Wezel GP, Medema MH, Raaijmakers JM. Pathogen-induced activation of disease-suppressive functions in the endophytic root microbiome.,2019, 366(6465): 606–612.
[11] Zhang JY, Liu YX, Zhang N, Hu B, Jin T, Xu HR, Qin Y, Yan PX, Zhang XN, Guo XX, Hui J, Cao SY, Wang X, Wang C, Wang H, Qu BY, Fan GY, Yuan LX, Garrido-Oter R, Chu CC, Bai Y. NRT1.1B is associated with root microbiota composition and nitrogen use in field-grown rice.,2019, 37(6): 676–684.
[12] Castrillo G, Teixeira PJPL, Paredes SH, Law TF, de Lorenzo L, Feltcher ME, Finkel OM, Breakfield NW, Mieczkowski P, Jones CD, Paz-Ares J, Dangl JL. Root microbiota drive direct integration of phosphate stress and immunity.,2017, 543(7646): 513–518.
[13] Van Deynze A, Zamora P, Delaux PM, Heitmann C, Jayaraman D, Rajasekar S, Graham D, Maeda J, Gibson D, Schwartz KD, Berry AM, Bhatnagar S, Jospin G, Darling A, Jeannotte R, Lopez J, Weimer BC, Eisen JA, Shapiro HY, Ané JM, Bennett AB. Nitrogen fixation in a landrace of maize is supported by a mucilage-associated diazotrophic microbiota.,2018, 16(8): e2006352.
[14] Wang C, Bai Y. Maize aerial roots fix atmospheric N2by interacting with nitrogen fixing bacteria (in Chinese)., 2019, 49(1): 89–90.王超, 白洋. 玉米可利用气生根进行高效生物固氮.中国科学:生命科学, 2019, 49(1): 89–90.
[15] Huang AC, Jiang T, Liu YX, Bai YC, Reed J, Qu B, Goossens A, Nützmann HW, Bai Y, Osbourn A. A specialized metabolic network selectively modulatesroot microbiota.,2019, 364(6440): eaau6389.
[16] Yuan J, Zhao J, Wen T, Zhao M, Li R, Goossens P, Huang Q, Bai Y, Vivanco JM, Kowalchuk GA, Berendsen RL, Shen Q. Root exudates drive the soil-borne legacy of aboveground pathogen infection.,2018, 6(1): 156.
[17] Bai Y, Müller DB, Srinivas G, Garrido-Oter R, Potthoff E, Rott M, Dombrowski N, Münch PC, Spaepen S, Remus-Emsermann M, Hüttel B, McHardy AC, Vorholt JA, Schulze-Lefert P. Functional overlap of theleaf and root microbiota.,2015, 528(7582): 364– 369.
[18] Chen QW, Jiang T, Liu YX, Liu HL, Zhao T, Liu ZX, Gan XC, Hallab A, Wang XM, He J, Ma YH, Zhang FX, Jin T, Schranz ME, Wang Y, Bai Y, Wang GD. Recently duplicated sesterterpene (C25) gene clusters inmodulate root microbiota.,2019, 62(7): 947–958.
[19] Wang W, Yang J, Zhang J, Liu YX, Tian CP, Qu BY, Gao CL, Xin PY, Cheng SJ, Zhang WJ, Miao P, Li L, Zhang XJ, Chu JF, Zuo JR, Li JY, Bai Y, Lei XG, Zhou JM. Ansecondary metabolite directly targets expressionof the bacterial type III secretion system to inhibit bacterial virulence.,2020, 27(4): 601–613.
[20] Chen T, Nomura K, Wang XL, Sohrabi R, Xu J, Yao YL, Paasch BC, Ma L, Kremer J, Cheng YT, Zhang L, Wang N, Wang ET, Xin XF, He SY. A plant genetic network for preventing dysbiosis in the phyllosphere.,2020, 580(7804): 653-657.
[21] Edwards J, Johnson C, Santos-Medellín C, Lurie E, Podishetty NK, Bhatnagar S, Eisen JA, Sundaresan V. Structure, variation, and assembly of the root-associated microbiomes of rice.,2015, 112(8): E911–E920.
[22] Zhang JY, Zhang N, Liu YX, Zhang XN, Hu B, Qin Y, Xu HR, Wang H, Guo XX, Qian JM, Wang W, Zhang PF, Jin T, Chu CC, Bai Y. Root microbiota shift in rice correlates with resident time in the field and developmental stage.,2018, 61(6): 613–621.
[23] Edwards JA, Santos-Medellín CM, Liechty ZS, Nguyen B, Lurie E, Eason S, Phillips G, Sundaresan V. Compositional shifts in root-associated bacterial and archaeal microbiota track the plant life cycle in field-grown rice.,2018, 16(2): e2003862.
[24] Chen SM, Waghmode TR, Sun RB, Kuramae EE, Hu CS, Liu BB. Root-associated microbiomes of wheat under the combined effect of plant development and nitrogen fertilization.,2019, 7(1): 136.
[25] Shi Y, Li YT, Xiang XJ, Sun RB, Yang T, He D, Zhang KP, Ni YY, Zhu YG, Adams JM, Chu HY. Spatial scale affects the relative role of stochasticity versus determinism in soil bacterial communities in wheat fields across the North China Plain.,2018, 6(1): 27.
[26] Sun RB, Li WY, Dong WX, Tian YP, Hu CS, Liu BB. Tillage changes vertical distribution of soil bacterial and fungal communities.,2018, 9: 699.
[27] Walters WA, Jin Z, Youngblut N, Wallace JG, Sutter J, Zhang W, González-Peña A, Peiffer J, Koren O, Shi QJ, Knight R, Glavina del Rio T, Tringe SG, Buckler ES, Dangl JL, Ley RE. Large-scale replicated field study of maize rhizosphere identifies heritable microbes.,2018, 115(28): 7368–7373.
[28] Wei Z, Gu Y, Friman VP, Kowalchuk GA, Xu YC, Shen QR, Jousset A. Initial soil microbiome composition and functioning predetermine future plant health.,2019, 5(9): eaaw0759.
[29] Xiong W, Song YQ, Yang KM, Gu Y, Wei Z, Kowalchuk GA, Xu YC, Jousset A, Shen QR, Geisen S. Rhizosphere protists are key determinants of plant health.,2020, 8(1): 27.
[30] Jin T, Wang YY, Huang YY, Xu J, Zhang PF, Wang N, Liu X, Chu HY, Liu G, Jiang HG, Li YZ, Xu J, Kristiansen K, Xiao L, Zhang YZ, Zhang GY, Du GH, Zhang HB, Zou HF, Zhang HF, Jie ZY, Liang SS, Jia HJ, Wan JW, Lin DC, Li JY, Fan GY, Yang HM, Wang J, Bai Y, Xu X. Taxonomic structure and functional association of foxtail millet root microbiome.,2017, 6(10): 1–12.
[31] Liu YX, Qin Y, Bai Y. Reductionist synthetic community approaches in root microbiome research.,2019, 49: 97–102.
[32] Fan KK, Weisenhorn P, Gilbert JA, Shi Y, Bai Y, Chu HY. Soil pH correlates with the co-occurrence and assemblage process of diazotrophic communities in rhizosphere and bulk soils of wheat fields.,2018, 121: 185–192.
[33] Mori A, Kirk GJD, Lee JS, Morete MJ, Nanda AK, Johnson-Beebout SE, Wissuwa M.Rice genotype differences in tolerance of zinc-deficient soils: evidence for the importance of root-induced changes in the rhizosphere.,2016, 6: 1160.
[34] Fan KK, Delgado-Baquerizo M, Guo XS, Wang DZ, Wu YY, Zhu M, Yu W, Yao HY, Zhu YG, Chu HY. Suppressed N fixation and diazotrophs after four decades of fertilization.,2019, 7(1): 143.
[35] Zhao Q, Feng Q, Lu HY, Li Y, Wang AH, Tian QL, Zhan QL, Lu YQ, Zhang L, Huang T, Wang YC, Fan DL, Zhao Y, Wang ZQ, Zhou CC, Chen JY, Zhu CR, Li WJ, Weng QJ, Xu Q, Wang ZX, Wei XH, Han B, Huang XH. Pan-genome analysis highlights the extent of genomic variation in cultivated and wild rice.,2018, 50(2): 278–284.
[36] Wang W, Hu B, Yuan DY, Liu YQ, Che RH, Hu YC, Ou SJ, Liu YX, Zhang ZH, Wang H, Li H, Jiang ZM, Zhang ZL, Gao XK, Qiu YH, Meng XB, Liu YX, Bai Y, Liang Y, Wang YQ, Zhang LH, Li LG, Sodmergen S, Jing HC, Li JY, Chu CC. Expression of the nitrate transporter geneconfers high yield and early maturation in rice.,2018, 30(3): 638–651.
[37] Stein JC, Yu Y, Copetti D, Zwickl DJ, Zhang L, Zhang C, Chougule K, Gao D, Iwata A, Goicoechea JL, Wei S, Wang J, Liao Y, Wang M, Jacquemin J, Becker C, Kudrna D, Zhang J, Londono CEM, Song X, Lee S, Sanchez P, Zuccolo A, Ammiraju JSS, Talag J, Danowitz A, Rivera LF, Gschwend AR, Noutsos C, Wu CC, Kao SM, Zeng JW, Wei FJ, Zhao Q, Feng Q, El Baidouri M, Carpentier MC, Lasserre E, Cooke R, Rosa Farias Dd, da Maia LC, dos Santos RS, Nyberg KG, McNally KL, Mauleon R, Alexandrov N, Schmutz J, Flowers D, Fan C, Weigel D, Jena KK, Wicker T, Chen M, Han B, Henry R, Hsing YC, Kurata N, de Oliveira AC, Panaud O, Jackson SA, Machado CA, Sanderson MJ, Long M, Ware D, Wing RA. Genomes of 13 domesticated and wild rice relatives highlight genetic conservation, turnover and innovation across the genus.,2018, 50(2): 285–296.
[38] Guo XX, Zhang XN, Qin Y, Liu YX, Zhang JY, Zhang N, Wu K, Qu BY, He ZS, Wang X, Zhang XJ, Hacquard S, Fu XD, Bai Y. Host-associated quantitative abundance profiling reveals the microbial load variation of root microbiome.,2020, 1(1): 100003.
[39] Caporaso JG, Kuczynski J, Stombaugh J, Bittinger K, Bushman FD, Costello EK, Fierer N, Peña AG, Goodrich JK, Gordon JI, Huttley GA, Kelley ST, Knights D, Koenig JE, Ley RE, Lozupone CA, McDonald D, Muegge BD, Pirrung M, Reeder J, Sevinsky JR, Turnbaugh PJ, Walters WA, Widmann J, Yatsunenko T, Zaneveld J, Knight R. QIIME allows analysis of high-throughput community sequencing data.,2010, 7(5): 335–336.
[40] Bolyen E, Rideout JR, Dillon MR, Bokulich NA, Abnet CC, Al-Ghalith GA, Alexander H, Alm EJ, Arumugam M, Asnicar F, Bai Y, Bisanz JE, Bittinger K, Brejnrod A, Brislawn CJ, Brown CT, Callahan BJ, Caraballo- Rodríguez AM, Chase J, Cope EK, Da Silva R, Diener C, Dorrestein PC, Douglas GM, Durall DM, Duvallet C, Edwardson CF, Ernst M, Estaki M, Fouquier J, Gauglitz JM, Gibbons SM, Gibson DL, Gonzalez A, Gorlick K, Guo J, Hillmann B, Holmes S, Holste H, Huttenhower C, Huttley GA, Janssen S, Jarmusch AK, Jiang L, Kaehler BD, Kang KB, Keefe CR, Keim P, Kelley ST, Knights D, Koester I, Kosciolek T, Kreps J, Langille MGI, Lee J, Ley R, Liu YX, Loftfield E, Lozupone C, Maher M, Marotz C, Martin BD, McDonald D, McIver LJ, Melnik AV, Metcalf JL, Morgan SC, Morton JT, Naimey AT, Navas-Molina JA, Nothias LF, Orchanian SB, Pearson T, Peoples SL, Petras D, Preuss ML, Pruesse E, Rasmussen LB, Rivers A, Robeson MS, Rosenthal P, Segata N, Shaffer M, Shiffer A, Sinha R, Song SJ, Spear JR, Swafford AD, Thompson LR, Torres PJ, Trinh P, Tripathi A, Turnbaugh PJ, Ul-Hasan S, van der Hooft JJJ, Vargas F, Vázquez-Baeza Y, Vogtmann E, von Hippel M, Walters W, Wan Y, Wang M, Warren J, Weber KC, Williamson CHD, Willis AD, Xu ZZ, Zaneveld JR, Zhang Y, Zhu Q, Knight R, Caporaso JG. Reproducible,interactive, scalable and extensible microbiome data science using QIIME 2.,2019, 37(8): 852–857.
[41] Edgar RC. Search and clustering orders of magnitude faster than BLAST.,2010, 26(19): 2460– 2461.
[42] Liu YX, Qin Y, Guo XX, Bai Y. Methods and applications for microbiome data analysis., 2019, 41(9): 845–826.刘永鑫, 秦媛, 郭晓璇, 白洋. 微生物组数据分析方法与应用. 遗传,2019, 41(9): 845–826.
[43] Zheng MS, Zhou N, Liu SF, Dang CY, Liu YX, He SS, Zhao YJ, Liu W, Wang XK. N2O and NO emission from a biological aerated filter treating coking wastewater: main source and microbial community.,2019, 213: 365–374.
[44] Oksanen J, Kindt R, Legendre P, O’Hara B, Stevens MHH, Oksanen MJ, Suggests M. The vegan package.,2007, 10: 631–637.
[45] Wickham H. Ggplot2: Elegant Graphics for Data Analysis. Springer, 2016.
[46] Liaw A, Wiener M. Classification and regression by randomForest.,2002, 2(3): 18–22.
[47] Qian XB, Liu YX, Ye XH, Zheng WJ, Lv SX, Mo MJ, Lin JJ, Wang WQ, Wang WH, Zhang XN, Lu MP. Gut microbiota in children with juvenile idiopathic arthritis: characteristics, biomarker identification, and usefulness in clinical prediction.,2020, 21(1): 286.
[48] Csardi G, Nepusz T. The igraph software package for complex network research.,2006, 1695(5): 1–9.
[49] Wang JF, Zheng JY, Shi WY, Du N, Xu XM, Zhang YM, Ji PF, Zhang FY, Jia Z, Wang YP, Zheng Z, Zhang HP, Zhao FQ. Dysbiosis of maternal and neonatal microbiota associated with gestational diabetes mellitus.,2018, 67(9): 1614–1625.
[50] Deng Y, Jiang YH, Yang YF, He ZL, Luo F, Zhou JZ. Molecular ecological network analyses.,2012, 13(1): 113.
[51] National Genomics Data Center Members and Partners. Database resources of the national genomics data center in 2020.,2020, 48(D1): D24–D33.
[52] Wang YQ, Song FH, Zhu JW, Zhang SS, Yang YD, Chen TT, Tang BX, Dong LL, Ding N, Zhang Q, Bai ZX, Dong XN, Chen HX, Sun MY, Zhai SA, Sun YB, Yu L, Lan L, Xiao JF, Fang XD, Lei HX, Zhang Z, Zhao WM. GSA: Genome sequence archive.,2017, 15(1): 14–18.
[53] Zhang CL, Zhang YM, Ding ZJ, Bai Y. Contribution of microbial inter-kingdom balance to plant health.,2019, 12(2): 148–149.
[54] Toju H, Peay KG, Yamamichi M, Narisawa K, Hiruma K, Naito K, Fukuda S, Ushio M, Nakaoka S, Onoda Y, Yoshida K, Schlaeppi K, Bai Y, Sugiura R, Ichihashi Y, Minamisawa K, Kiers ET. Core microbiomes for sustainable agroecosystems.,2018, 4(5): 247–257.
[55] Xie JP, Han YB, Liu G, Bai LQ. Research advances on microbial genetics in China in 2015., 2016, 38(9): 765–790.谢建平, 韩玉波, 刘钢, 白林泉. 2015年中国微生物遗传学研究领域若干重要进展. 遗传,2016, 38(9): 765– 790.
[56] Wen CL, Sun CJ, Yang N. The concepts and research progress: from heritability to microbiability.,2019, 41(11): 1023–1040.文超良孙从佼, 杨宁. 从遗传力到肠菌力:概念及研究进展. 遗传,2019, 41(11): 1023–1040.
[57] Liu BB, Zhang XJ, Bakken LR, Snipen L, Frostegård Å. Rapid succession of actively transcribing denitrifier populations in agricultural soil during an anoxic spell.,2019, 9: 3208.
[58] Zhang KP, Shi Y, Cui XQ, Yue P, Li KH, Liu XJ, Tripathi BM, Chu HY. Salinity is a key determinant for soil microbial communities in a desert ecosystem.,2019, 4(1): e00225–00218.
[59] Lebeis SL, Paredes SH, Lundberg DS, Breakfield N, Gehring J, McDonald M, Malfatti S, Glavina del Rio T, Jones CD, Tringe SG, Dangl JL. Salicylic acid modulates colonization of the root microbiome by specific bacterial taxa.,2015, 349(6250): 860–864.
[60] Voges MJEEE, Bai Y, Schulze-Lefert P, Sattely ES. Plant-derived coumarins shape the composition of ansynthetic root microbiome.,2019, 116(25): 12558–12565.
[61] Zamioudis C, Korteland J, Van Pelt JA, van Hamersveld M, Dombrowski N, Bai Y, Hanson J, Van Verk MC, Ling HQ, Schulze-Lefert P, Pieterse CMJ. Rhizobacterial volatiles and photosynthesis-related signals coordinate MYB72 expression inroots during onset of induced systemic resistance and iron-deficiency responses.,2015, 84(2): 309–322.
[62] Niu B, Paulson JN, Zheng X, Kolter R. Simplified and representative bacterial community of maize roots.,2017, 114(12): E2450–E2459.
[63] Hu B, Wang W, Ou SJ, Tang JY, Li H, Che RH, Zhang ZH, Chai XY, Wang HR, Wang YQ, Liang CZ, Liu LC, Piao ZZ, Deng QY, Deng K, Xu C, Liang Y, Zhang LH, Li LG, Chu CC. Variation incontributes to nitrate-use divergence between rice subspecies.,2015, 47(7): 834–838.
[64] Getzke F, Thiergart T, Hacquard S. Contribution of bacterial-fungal balance to plant and animal health.,2019, 49: 66–72.
[65] Kim PJ, Price ND. Genetic co-occurrence network across sequenced microbes.,2011, 7(12): e1002340.
Analysis of rice root bacterial microbiota of Nipponbare and IR24
Yali Hu1,2,3, Rui Dai2,3,4,5, Yongxin Liu2,3,4, Jingying Zhang2,3,4, Bin Hu2, Chengcai Chu2, Huaibo Yuan1, Yang Bai2,3,4,5
The root-associated bacterial microbiota is closely related to life activities of land plants, and its composition is affected by geographic locations and plant genotypes. However, the influence of plant genotypes on root microbiota in rice grown in northern China remains to be explained. In this study, we performed 16S rRNA gene amplicon sequencing to generate bacterial community profiles of two representative rice cultivars, Nipponbare and IR24. They are planted in Changping and Shangzhuang farms in Beijing and have reached the reproductive stage. We compared their root microbiota in details by Random Forest machine learning algorithm and network analysis. We found that the diversity of rice root microbiota was significantly affected by geographic locations and rice genotypes. Nipponbare and IR24 showed distinct taxonomic composition of the root microbiota and the interactions between different bacteria. Moreover, the root bacteria could be used as biomarkers to distinguish Nipponbare from IR24 across regions. Our study provides a theoretical basis for the in-depth understanding of rice root microbiota in Northern China and the improvement of rice breeding from the perspective of the interaction between root microorganisms and plants.
rice root microbiota; diversity analysis; taxonomic composition; machine learning; network analysis
2020-03-15;
2020-04-16
中国科学院前沿科学重点研究项目(编号:QYZDB-SSW-SMC021)和国家自然科学基金面上项目(编号:31772400)资助[Supported by the Key Research Program of Frontier Sciences of the Chinese Academy of Science (No. QYZDB-SSW-SMC021), and the National Natural Science Foundation of China (No. 31772400)]
胡雅丽,在读硕士研究生,专业方向:根系微生物组。E-mail: 2017111262@mail.hfut.edu.cn戴睿,在读博士研究生,专业方向:根系微生物组,生物信息学。E-mail: raydai@genetics.ac.cn 胡雅丽和戴睿为并列第一作者。
白洋,博士,研究员,研究方向:根系微生物组。E-mail: ybai@genetics.ac.cn袁怀波,博士,副教授,研究方向:农产品资源综合开发利用研究。E-mail: yuanhuaibo001@163.com
10.16288/j.yczz.20-070
2020/4/28 15:50:23
URI: http://kns.cnki.net/kcms/detail/11.1913.R.20200427.1715.001.html
(责任编委: 赵方庆)

