新型冠状病毒棘突蛋白免疫原性分析及其多肽疫苗设计研究
许志强,鲁 陈,胡晓军,郭 有,胡巧丽,朱 颖,4
(1.南京医科大学第一附属医院,江苏 南京 210000;2.赣南医学院第一附属医院;3.赣州市疾病预防控制中心,江西 赣州 341000;4.南昌大学生命科学学院,江西 南昌 330031)
至2020年3月4日,我国被确诊感染新型冠状病毒肺炎患者超过8万人,并导致超过3千名患者死亡,且确诊和死亡人数仍在每日增加(http://www.nhc.gov.cn/),对国民生活、经济秩序造成极其严重的影响。目前,国际病毒分类委员会已将这种新型冠状病毒命名为“SARS-CoV-2”,而由此病毒感染的肺炎也被命名为“COVID-19”(Corona Virus Disease 2019)。
冠状病毒是一种具有包膜的单股正链RNA病毒。SARS-CoV-2属于β-冠状病毒,具有典型的冠状病毒基因组结构。棘突蛋白(spike protein, S)位于SARS-CoV-2病毒包膜表面,可与宿主细胞膜特定受体结合,在病毒侵入和感染细胞中具有至关重要的作用。尽管SARS-CoV-2的S蛋白在多种冠状病毒中表现出高度的变异,但是它与引起严重急性呼吸道综合征(severe acute respiratory syndrome,SARS)的SARS-CoV病毒表面的S蛋白具有较高的同源性。SARS-CoV-2与SARS-CoV的S蛋白在其受体结合域(receptor-binding domain, RBD)具有几乎相同的三维空间结构,表明SARS-CoV-2与SARS-CoV作用的受体相同,即都是通过S蛋白-血管紧张素转化酶2(angiotensin-converting enzyme 2, ACE2)结合途径感染支气管上皮细胞和肺上皮细胞[1]。截至2020年3月5日,SARS-CoV-2感染病例仍在增加,因此,有效的疫苗研发至关重要。
目前,已有大量的药物可作为SARS-CoV-2感染后的辅助治疗,包括广谱的病毒靶向药物,宿主靶向药物和中药等[2-3]。然而,市场上还没有特异性针对SARS-CoV-2的药物或疫苗,一些新型药物的研制也处于早期阶段。基于S蛋白在SARS-CoV-2病毒感染中的关键作用,其有望成为治疗药物设计靶点。已发表的针对SARS-CoV S蛋白结构的研究表明,其N末端区域(N terminal domain, NTD),融合肽区域(fusion peptide, F),七肽重复区(heptad region, HR)和中央螺旋区(central helix, CH)是激发体内产生中和抗体(neutralizing antibody)的潜在靶标[4]。因此,对SARS-CoV-2 S蛋白结构和表位的分析可在一定程度上为免疫原疫苗的研制提供依据。
本研究基于SARS-CoV S蛋白结构和SARS-CoV-2的基因组序列,对其S蛋白的功能域、空间结构进行分析,并通过DNAStar、ABCpred、Bepipred、ElliPro、DiscoTope和SEPPA对SARS-CoV-2的线性和构象B细胞表位进行了综合预测。在抗击COVID-19的紧迫形势下,本研究期望为新型疫苗的开发和研制节省时间和成本。
1 材料和方法
1.1序列检索与SARS-CoV-2S蛋白的物理化学性质分析从美国国家生物技术信息中心数据库(https://www.ncbi.nlm.nih.gov/)检索SARS-CoV-2和SARS-CoV 两种冠状病毒S蛋白的氨基酸序列,在GenBank中的检索号分别为QHD43416和P59594。利用Protparam对SARS-CoV-2 S 蛋白的分子量,氨基酸组成,亲水性均值和不稳定指数进行分析。
1.2SARS-CoV-2与SARS-CoVS蛋白的氨基酸序列比对和体系结构分析我们通过Clustal序列比对工具将SARS-CoV-2与SARS-CoV的S蛋白进行氨基酸序列比对,明确两种病毒中S蛋白的同源性,并根据已报道的SARS-CoV S蛋白结构域[4],进一步分析SARS-CoV-2 S蛋白的信号肽区域(signal peptite, SP)、N末端区域(N-terminal domain, NTD)、连接区域(linker or connect region, L or CR)、受体结合区域等结构(receptor-binding domain, RBD)。此外,通过TMHMM 2.0(http://www.cbs.dtu.dk/services/TMHMM/)对S蛋白的跨膜区域进行了预测[5]。
1.3二级结构和三级结构的构建与分析根据SARS-CoV-2的氨基酸序列,利用SWISS-MODEL构建了SARS-CoV-2 S蛋白的三级结构[6]。在SIWSS-MODEL服务器(https://swissmodel.expasy.org/)中输入SARS-CoV-2 S蛋白的氨基酸序列进行同源模拟,选择具有最大序列一致性、最大序列覆盖率、最高全局模型质量评估(Global Model Quality Estimation, GMQE)得分进行进一步分析。使用pymol展示最终获得的SARS-CoV-2的S蛋白模型[7],并分析其中α-螺旋和β-折叠结构。
1.4SARS-CoV-2线性B细胞表位分析B细胞表位是抗原蛋白中能被B细胞受体或随后由免疫系统产生的抗体所识别的特殊区域。一般来说,B细胞表位可分为两种类型,即线性表位(linear epitope)和构象表位(conformational epitope)。线性表位又称连续性表位,是由抗原的连续性的氨基酸组成。对于SARS-CoV-2的S蛋白的线性表位,我们利用三种不同类型的免疫信息学工具:DNAStar protean、ABCpred、Bepipred,全面分析线性B细胞表位。在DNAStar中,选择S蛋白氨基酸具有良好亲水性、柔韧性、可及性和高抗原性的区域作为初步筛选结果。随后使用ABCpred[8]和Bepipred 2.0[9]对S蛋白的线性表位进一步分析,分别从中选择得分高于0.8和0.55结果。综合上述三种工具所得结果,选择由两种工具以上筛选的相同序列作为最终的候选线性B细胞表位。
1.5SARS-CoV-2构象B细胞表位分析不同于线性B细胞表位,构象B细胞表位可由分布在三维结构中位置临近的非连续性的氨基酸组成。首先结合S蛋白三维结构,使用ElliPro筛选最小分数为0.8和最大距离为5埃的构象表位[10]。随后,使用DiscoTope 1.1和SEPPA 3.0区分S蛋白氨基酸序列中的表位和非表位残基[11-12]。综合ElliPro、DiscoTope 2.0、SEPPA 3.0的筛选结果,确定最终的构象B细胞表位。
2 结 果
2.1SARS-CoV-2S蛋白的序列检索和物理化学性质根据SARS-CoV-2 S蛋白的Genbank检索号(QHD43416)获取其氨基酸序列。通过Protparam分析,SARS-CoV-2的S蛋白包含1 273个氨基酸,分子量为141.18 kDa。其氨基酸中,110个带负电荷(D+E),103个带正电荷(R+K)。理论等电点(pI)为6.24,表明它在天然条件下是带负电荷的蛋白质。其亲水性均值和不稳定指数分别为33.01和-0.079,表明S蛋白具有稳定和亲水性特性。
2.2序列对比和体系结构分析通过Clustal软件对SARS-CoV-2和SARS-CoV S蛋白的氨基酸序列进行比对,它们的氨基酸一致性为76%,具有较高的同源性。 通过与SARS-CoV的序列相应区域比较,在SARS-CoV-2的S蛋白可分为两个部分:S1和S2。富含β折叠结构的S1亚基主要由N末端区域(氨基酸残基18-306)、受体结合区域(氨基酸残基331-527)、两个亚区域(氨基酸残基528-589和590-676)组成。 S2亚基则主要含有α螺旋结构,包括一个上游螺旋结构(upstream helix, UH)位于747-783、一个螺旋融合区域(fusion region, F)位于816-827、七肽重复区(hepated region, HR)位于912-984、中央α螺旋区域(central helix)位于985-1 034。随后紧接一个“发卡”结构(β-hairpin, βH)位于1 035-1 071和一个亚区域位于1 072-1 112。信号肽区、跨膜区和膜内区则分别位于1-17、1 214-1 236和1 237-1 273(图1)。
2.3SARS-CoV-2S蛋白的空间结构和折叠特征在SWISS-MODEL中,与SARS-CoV-2 S蛋白氨基酸序列一致性和覆盖率最高的模板为SARS-CoV S蛋白(PDB号:6ACC)。通过模型构建,SARS-CoV-2的S蛋白为同源三聚体结构,其中一个单体的受体结合区存在两种构象,即“横卧”和“立式”(图2B~C)。S1亚基含有35个β-折叠和6个α-螺旋结构,而S2亚基包含14个α-螺旋和7个β-折叠结构,其中第39和40个β-折叠共同形成β-发夹结构(图2D)。
2.4线性B细胞表位分析鉴定抗原中的B细胞表位是表位疫苗设计的重要步骤。综合亲水性、柔韧性、表面可及性和抗原性在内的序列特性,通过DNAStar protean得到40条多肽。ABCpred和Bepipred 2.0分别筛选出43条和35条符合条件的多肽。综合三种工具的肽段结果,最终确定出31个线性的SARS-CoV-2 S蛋白表位,其首-尾残基位置分别为:73-81、94-100、110-115、146-160、204-208、251-257、280-285、312-320、354-362、402-406、415-419、439-444、476-483、526-540、552-556、565-579、602-609、634-645、652-661、674-689、689-704、741-749、772-780、807-818、883-889、931-946、1 084-1 091、1 117-1 127、1 157-1 164、1 180 -1 186、1 191-1 196,每个表位对应的氨基酸序列和在S蛋白空间结构上的位置分别见表1和图3A,其中7个线性表位B1-B7位于SARS-CoV-2 S蛋白的NTD区域(氨基酸残基18-306),5个线性表位B9-B13位于RBD区域(氨基酸残基331-527)。
2.5构象B细胞表位分析对SARS-CoV-2 S蛋白的构象表位分析中,EliiPro共得出14条分值高于0.8且最大距离不超过5埃的表位。此外,通过DiscoTope 1.1和SEPPA 3.0分别鉴定出184和235个表位残基。整合这三种方法,共获得9条构象B细胞表位:72-75、(146-147)-(149-155)、246-257、(404-409)-(413-414)、443-448、458-463、474-481、498-506、556-571,相应的氨基酸序列见表2,其中3条表位CB1-CB3位于S蛋白NTD区域,5条表位C4-C8位于RBD区域(图3B)。
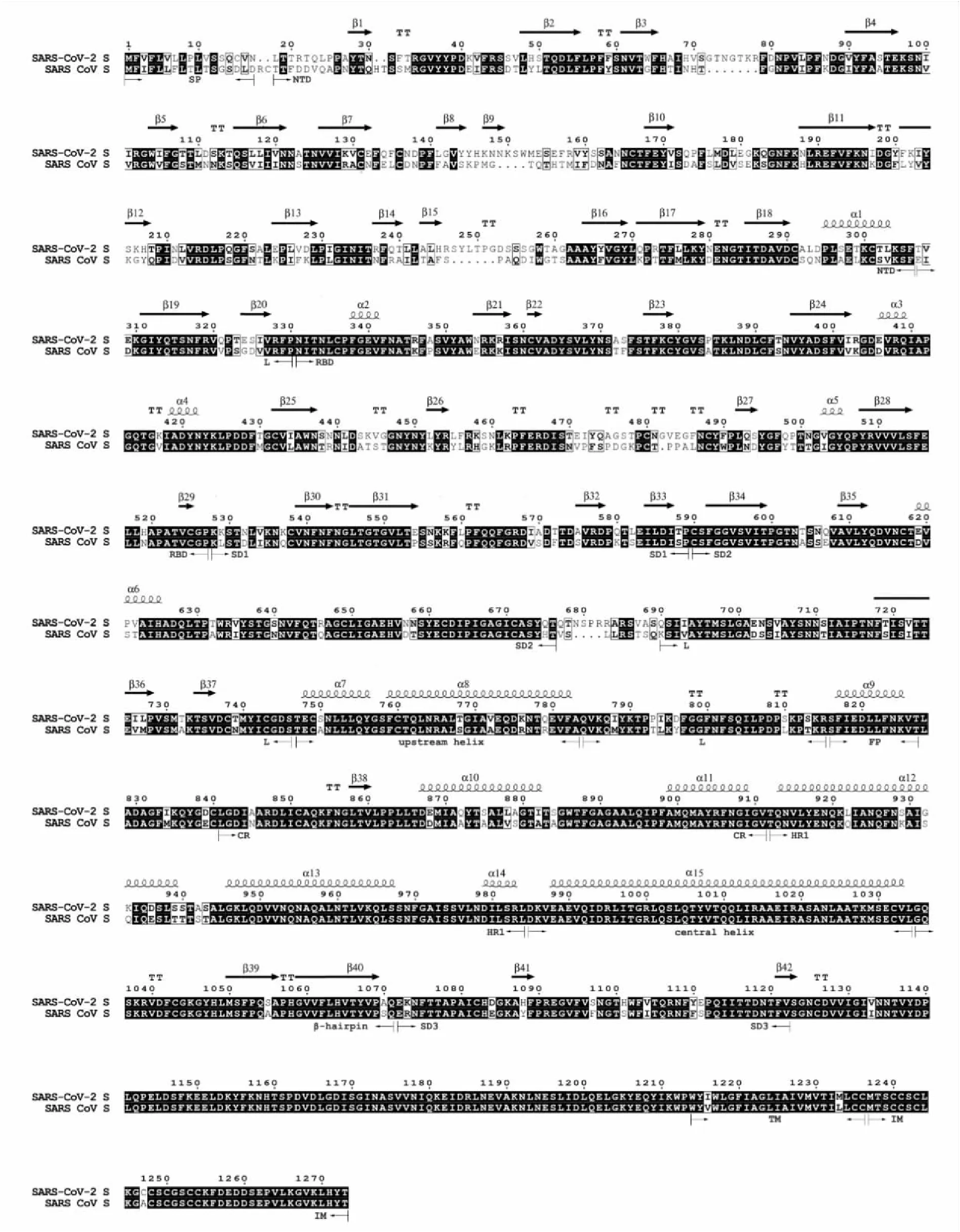
图1 SARS-CoV-2与SARS-CoV S蛋白的氨基酸序列比对结果
SP: 信号肽;NTD:N末端区域;L:连接区域;RBD:受体结合区域;SD1:亚区1;SD2:亚区2;upstream helix:向上螺旋结构;FP:融合肽;CR:连接区域;HR:七肽重复区;central helix:中央螺旋结构;β-hairpin:β发卡结构;SD3:亚区3;α1-α15:α螺旋结构;β1-β42:β折叠结构。

图2 SARS-CoV-2 S蛋白的空间结构
A.不同体系结构的平面分布图示。缩略词释义见图1。B~C.S蛋白的三聚体结构和两种构象的蛋白结构。lying RBD:“卧式”受体结合区;standing RBD:“立式”受体结合区。D.S蛋白不同体系结构的空间位置。cartoon:以二级结构展示;surface:以表面形式展示。

表1 线性B细胞表位筛选结果
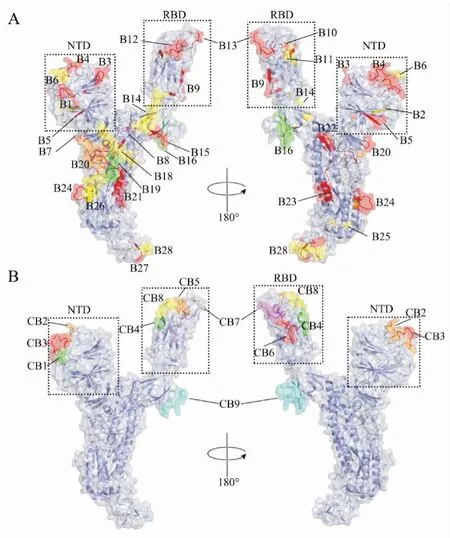
图3 B细胞表位在SARS-CoV-2 S蛋白空间结构上的位置
A.线性B细胞表位在S蛋白结构上位置。B1-B31:31个不同的B细胞表位。B.构象B细胞表位在S蛋白结构上的位置。CB1-CB9:9个不同的构象B表位。
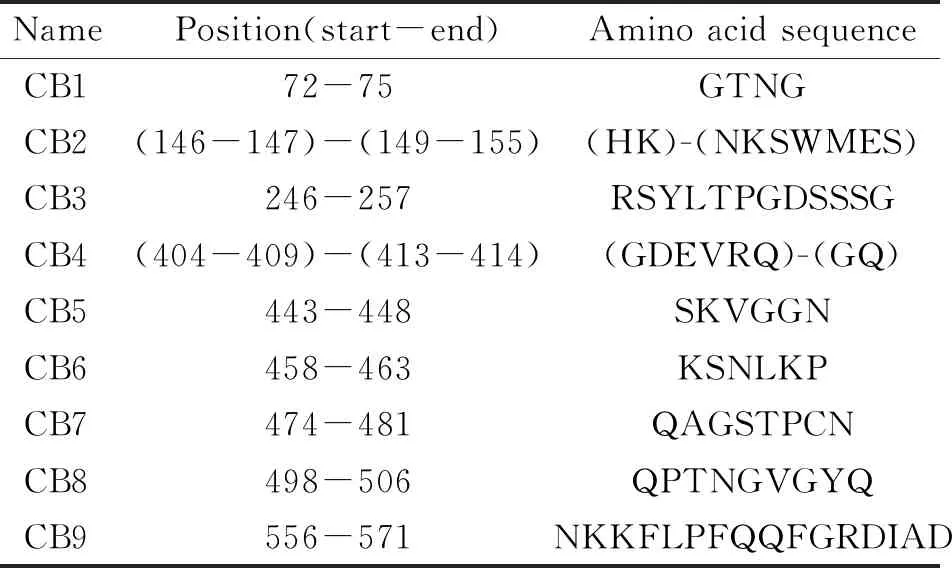
表2 构象B细胞表位筛选结果
3 讨 论
自2019年12月至今,新型冠状病毒在国内已引起超过8万人感染并导致3千名以上患者死亡,对公众健康造成极其严重的危害。目前,2019新型冠状病毒的基因组信息已经明确,与引起2003年严重呼吸综合征的SARS冠状病毒有非常高的同源性,并且在感染模式上也具有相似性。由于S蛋白在新型冠状病毒传播过程中起着至关重要作用,因此也是诱导体内中和抗体产生的首要靶点。鉴于疫情防控的严峻形势,迫切地需要明确SARS-CoV-2 S蛋白的结构特征和B细胞表位,以至于能够快速地研制免疫原疫苗和中和性抗体。
我们通过与SARS-CoV中的S蛋白进行氨基酸序列和体系结构的比较,发现虽然SARS-CoV-2的S蛋白与SARS-CoV在总体序列上具有高达76%的序列一致性,但在S蛋白的S1亚基中两者序列差异较大,并存在较多的缺口(gap),序列一致性为64%。S1亚基含有NTD和与ACE2受体结合的RBD,表明S1亚基是SARS-CoV-2特异性疫苗研制的重要区域。相较于S1亚基,SARS-CoV-2与SARS-CoV S蛋白的S2亚基的序列高度相似,氨基酸一致性达到90%。虽然CHAN等比较了SARS-CoV-2和SARS-CoV基因组的相似性并且也针对S蛋白进行了序列比对,但是他们主要针对进化层面进行分析,对于SARS-CoV-2 S蛋白也仅明确了S1、S2、SP、NTD和RBD区域[13]。而先前针对SARS-CoV S蛋白的研究显示S2亚基含有融合肽区域、七肽重复区和中央螺旋结构,均为诱使广谱中和抗体(broadly neutralizing antibodies)产生的重要位点[4]。因此,本研究还根据SARS-CoV的研究结果,同源比较分析得到其他多种功能区所在的位点。
随后,我们通过同源模拟成功构建了S蛋白的三维结构模型。与SARS-CoV相同,SARS-CoV-2的S蛋白也是一个同源三聚体,并且具有两种构象:其一是“卧式”,三个RBD全部都是隐蔽构象,由于位阻效应不易与ACE2受体结合;其二是“立式”,三聚体中的一个RBD转变为立式构象,是结合至ACE2受体的必要条件[14]。根据SARS-CoV-2的S蛋白结构模型,我们对其螺旋结构和折叠特征进行了分析。S1亚基主要为β-折叠结构而S2亚基主要为α-螺旋结构,这两种结构通常含有较高的化学键能量,不利于表位的形成。Wrapp等使用冷冻电镜对SARS-CoV-2 S蛋白的27-1 146位氨基酸之间的结构的实际测定结果(PDB ID:6VSB)更是进一步证实了上述结构特征[15]。我们通过同源模拟所得到的S蛋白的结构与电镜实测结构具有较高的一致性,通过均方根偏差计算(RMSD),两种方法所得S蛋白的“立式”构象的RMSD值为1.365 Å(750个原子),“卧式”构象为1.494 Å(788个原子)。两者结构模型之间的差异主要在两个方面:(1)两种模型S蛋白RBD区域所旋转的角度不同引起,先前对SARS病毒S蛋白的结构研究中也表明RBD区域存在多种旋转角度[14];(2)虽然冷冻电镜得到的SARS-CoV-2 S蛋白的实测结构,但作者表示其中缺少了一些柔性卷曲结构[15]。由于B细胞表位常处于蛋白的无规则卷曲和β-转角部分,明确完整的S蛋白的二级结构和三维结构对表位疫苗的研制尤为必要。
对于抗原B细胞表位的预测一般都基于氨基酸序列的物理化学特性,但仅适于线性B表位的分析。然而,构象表位在机体免疫反应过程中同样重要。因此,本研究中不仅对SARS-CoV-2的线性B细胞表位进行了全面分析,还基于“立式”构象的S蛋白结构对构象表位进行了综合预测。利用DNAStar、ABCpred、BepiPred三种工具共得到31条线性B细胞表位,分布于S蛋白各处,包括NTD、RBD、S2亚基。除B2以外,其余30条表位均位于S蛋白表面和无规则卷曲处。在构象表位的分析中,结合ElliPro得到的表位序列、DiscoTope和SEPPA所得阳性氨基酸残基,综合出9条构象表位,主要分布于S1的NTD和RBD,并且和线性B细胞表位具有部分重合,如B1与CB1、B4与CB2、B6与CB3、B13与CB7等。综合先前已报道对SARS-CoV S蛋白的B表位研究结果,其310-535、604-625、1 023-1 189均为验证后的表位区域[16-18],其中471-503、604-625、1 164-1 191在恒河猴中诱导产生的抗体可有效阻止SARS-CoV的感染[17]。这些位点在SARS-CoV-2 S蛋白中相应的位置为323-549、618-639、1 041-1 207,通过对表位的进一步筛选,位于这些区域的线性表位共有11条(B9-B14和B27-B31)、构象表位共有5条(CB4-CB8),均为潜在的疫苗研发候选表位。在这16条表位中,B9-B13、CB4-CB8均位于SARS-CoV-2 S蛋白与人ACE2受体结合的RBD区域,针对此10条表位疫苗产生的抗体在理论上能直接干扰S蛋白与人ACE2的结合。虽然BARUAH等也对SARS-CoV-2 S蛋白的B细胞表位进行了分析,但是缺少全面性和综合性。在线性表位筛选中没有考虑氨基酸自身的物理化学特性,在构象表位中也仅依靠ElliPro单一工具所得结果,最终只获得了3条线性和5条构象B细胞表位[19]。较少表位结果既丧失了灵敏性也缺失准确性,不利于后续疫苗的研究。本研究不仅综合了多种工具在多种S蛋白的特性下对表位进行了综合分析,而且根据先前针对SARS S蛋白B表位已有的研究和SARS-CoV-2的功能区,对所得表位结果进行了进一步的筛选,旨在加速研发针对SARS-CoV-2的中和抗体。
综上所述,我们不仅对SARS-CoV-2 S蛋白的结构域进行了分析,还对潜在表位进行了综合筛选,共获得40条B细胞表位。综合SARS-CoV的表位研究结果,我们从中筛选出16条可作为潜在的新型冠状病毒肺炎免疫原疫苗研发的候选表位,且其中的10条位于SARS-CoV-2 S蛋白RBD区,是较为有进一步研究价值的肽段。这些结果不仅能减少疫情防控所需疫苗的研制时间,更能进一步节约其研发成本。

