浙江近无柄雅榕古树叶绿体基因组特征分析
池 伟,池雷均,潘贺威,夏小李,黄振华
(1.瑞安市自然资源和规划局,浙江 瑞安 325200;2.浙江省海洋水产养殖研究所,浙江 温州 325005;3.浙江省近岸水域生物资源开发与保护重点实验室,浙江 温州 325005)
近无柄雅榕(Ficus concinna Miq.var.subsessilis Corner)又称无柄小叶榕[1],属桑科榕属,是小叶榕的一个变种,主要分布在中国浙江南部、江西南部、广东、云南等地,在泰国西北部、印度东北部也有分布。近无柄雅榕树冠大,枝叶繁茂,四季常青,耐低温,耐盐碱,是生态景观和泥质海岸防护林的重要构成树种,是福建省树,也是福州、赣州、温州等地的市树。
叶绿体是植物进行光合作用的细胞器,含有少量的遗传物质。1986年,烟草和地钱的叶绿体基因组全序列被首次公布,开创了叶绿体全基因组测序的先河。之后,陆续有其他物种的叶绿体基因组序列被公布。目前,NCBI 收录的叶绿体基因组序列已经超过4200 个。研究表明,叶绿体基因组在结构和功能上较为保守,含有大量的功能基因,且其序列片段的变异速率适中,可用作DNA条形码,开展分子鉴定、物种进化、迁徒等方面的研究[2-4]。目前叶绿体基因组研究多侧重于农作物、经济作物、药用植物和具有进化意义的古老物种,关于榕属植物叶绿体基因组序列的研究较少。本研究拟以一株树龄近千年的近无柄雅榕古树为研究对象,测定其叶绿体基因组序列并对其特点进行分析,以期为榕属种间的分子标记开发、桑科植物系统发育研究提供参考。
1 材料与方法
1.1 材料
本研究中的近无柄雅榕古树位于浙江省瑞安市塘下镇上马村,是瑞安市文物保护古树。树龄917年(至2019年),树高14 m,胸围1000 cm,平均冠幅29 m。
1.2 基因组提取和测序
利用Plant DNAzol试剂提取新鲜幼嫩叶片DNA,用plasmid-safe ATP-Dependent DNase 降解线性的染色体DNA。采用超声法Covaris 将大片段叶绿体基因组DNA 随机打断并产生300~500 bp DNA 片段,然后用T4 DNA Polymerase、Klenow DNA Polymerase和T4 PNK将打断形成的粘性末端修复成平末端,再在3′端加碱基“A”,以使DNA片段随后能与3′端带有“T”碱基的特殊接头连接,用电泳法回收所需长度的DNA片断,并加接头进行cluster制备。最后利用诺禾致源科技股份有限公司的Illumina HiSeq平台上机测序。
1.3 叶绿体基因组数据分析
对下机数据进行过滤质控,包括去除质量值连续≤20的碱基数达到一定程度的reads(默认40%,设置为36个),去除含N的碱基数目总和达到一定比例的reads(默认10%,设置为9 bp),去除adapter污染(默认adaper序列与read序列有15 bp的overlap,设置为10 bp),去除duplication污染等,得到clean data。利用CLC Genomics Workbanch、SOAP、SSPACE 等软件组装叶绿体基因组,组装后的完整序列利用Dogma 网站在线工具进行注释;用OGDRAW 软件[5]呈现叶绿体基因组序列图;用MISA(MIcroSAtellite identification tool)软件[6]分析SSR;用MEGA X 软件构建进化树。
2 结果与分析
2.1 叶绿体基因组结构特征
样品经Illumina HiSeq 平台测序得到的下机数据为1.0 G,过滤后的数据为0.9 G,平均长度为150 bp,覆盖度为158×。叶绿体基因组大小为160 292 bp,CDS 的总长度为76 521 bp,GC 含量为35.8%。与其他大多数被子植物一样,近无柄雅榕叶绿体基因组是典型的环状结构[7-8],包括1个大单拷贝区(LSC)、1个小单拷贝区(SSC)和一对分开的反向重复区域(IR)。LSC区长度为88 565 bp,GC含量为33.5%;SSC区长度为20 145 bp,GC含量为28.9%;IR 区长度为25 791 bp,GC 含量为42.7%(见图1、表1)。由于4个rRNA 基因均分布在IR区,导致IR区的GC含量高于2个单拷贝区。
植物叶绿体基因组一般有110~130个基因[9],本研究中的叶绿体基因组共注释出118个基因,包括83个蛋白质编码基因、31个tRNA基因和4个rRNA基因,其中IR区有11个蛋白质编码基因、7个tRNA基因和4个rRNA基因。上述118个基因主要分为3类:与光合作用相关的基因有45个,主要包括光合系统Ⅰ(psa)、光合系统Ⅱ(psb)、细胞色素b/f复合体(pet)、ATP合成酶(atp)、NAD(P)H脱氢酶(ndh)等基因;与叶绿体转录、翻译、表达相关的基因有60 个,主要包括转运RNA(trn)、核糖体RNA(rrn)、RNA 聚合酶(rpo)和核糖体蛋白(rpl/rps)等基因;与开放式阅读框和其他蛋白编码相关的基因有13个,主要包括ycf、matk等基因。
与大部分叶绿体一样,近无柄雅榕叶绿体基因组内含子数量较少[10-11],仅注释出11 个含内含子的基因(见表2),包括7个蛋白质编码基因和4个tRNA 基因。其中,9个基因仅含1个内含子;2个基因含有2个内含子的,分别为ycf3和clpP基因;ndhA基因的内含子最长,长度为1179 bp。

图1 近无柄雅榕叶绿体基因组图谱Figure 1 Map of the chloroplast genome of Ficus concinna
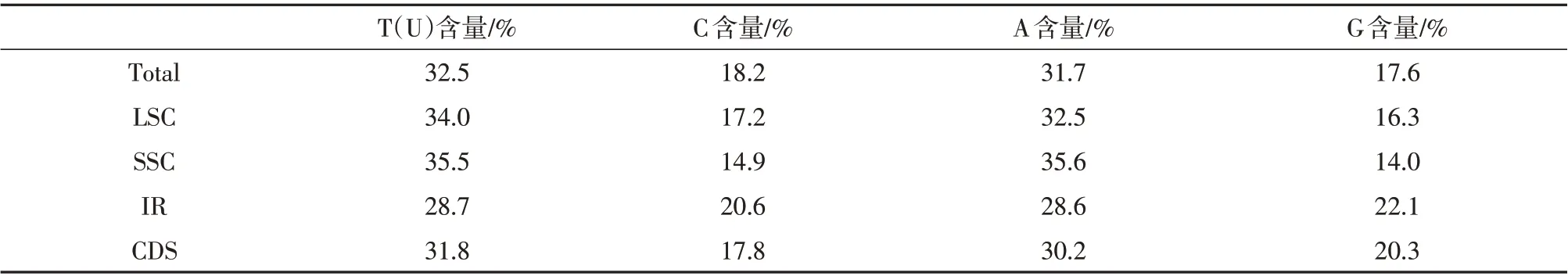
表1 近无柄雅榕叶绿体基因组碱基组成Table 1 Base composition of chloroplast genome of Ficus concinna

表2 近无柄雅榕叶绿体基因组中含内含子的基因信息Table 2 The genes including introns in the Ficus concinna chloroplast genome
2.2 微卫星的分布特征
微卫星(Simple Sequence Repeats,SSR)标记具有标记数量丰富、共显性遗传、重复性好等特点,被广泛用于构建遗传图谱、分析亲缘关系和种群结构等[12-16]。本研究从近无柄雅榕叶绿体基因组中检索到68个SSR,其中包括11个复合型SSR、52个单碱基重复、4个二碱基重复和1个三碱基重复,出现次数最多的SSR是单碱基重复,约占76.47%;叶绿体基因组中平均每2.36 kb有一个SSR位点(见表3)。检索到的SSR中共有4种重复单元类型,其中2种为单碱基重复,分别为A/T和C/G,绝大多数为A/T类型,占98.61%;二碱基重复和三碱基重复各有1种,主要重复单元类型分别为AT/TA和TTA/AAT(见表4)。
SSR的序列长度为10~133 bp,平均长度为19.06 bp,其中复合型SSR的平均长度为59.09 bp,单碱基重复的平均长度为11.1 bp,二碱基重复的平均长度为12 bp,三碱基重复的平均长度为21 bp;重复序列长度为10~20 bp的最多(占82.35%),重复序列长度为21~40 bp的占10.3%,重复序列长度大于40 bp的占7.35%。SSR重复单元的重复次数为6~14次(不计算复合型SSR),其中重复次数为5~10次的有26个(占SSR总数的38.24%),重复次数为11~15次的有31个(占SSR总数的45.59%)。

表3 近无柄雅榕叶绿体基因组中SSR重复单元的分布特征Table 3 Distribution of SSR repeat units in chloroplast genome for Ficus concinna

表4 近无柄雅榕叶绿体基因组中SSR不同重复单元的出现频数Table4 Frequency of SSR different repeat units in chloroplast genome of Ficus concinna
2.3 密码子偏好性分析
叶绿体基因组的蛋白编码基因共包含53 430个密码子,其中编码亮氨酸(Leu)的密码子最多,有5471个(占10.24%);其次为丝氨酸(Ser)和异亮氨酸(Ile),分别为4957 个(占9.28%)和4946 个(占9.26%);编码色氨酸(Trp)的密码子最少,仅672个(占1.26%)。在3个终止密码子中,UAA使用最频繁,其数量占终止密码子总数的43.33%;UGA的数量占终止密码子总数的32.84%;UAG的数量占终止密码子总数的23.83%。同义密码子相对使用度(RSCU)指某个密码子在编码对应氨基酸的同义密码子中出现的相对概率,在进行密码子编好性分析时可去除氨基酸组成的影响。RSCU>1表示该密码子为偏好密码子,RSCU<1表示该密码子使用率较低,RSCU=1表示该密码子没有偏好性(见表5)。近无柄雅榕叶绿体基因组的密码子偏好使用A/T碱基,其中第一个、第二个和第三个碱基为A/T 碱基的密码子分别占密码子总数的64.53%、63.50%和64.35%。

表5 近无柄雅榕叶绿体基因组密码子使用率Table 5 Codon usage of chloroplast genome of Ficus concinna
2.4 系统进化分析
选取Genbank 数据库中Ficus religiosa L.、Ficus carica L.、Ficus racemosa L.等10 个桑科植物叶绿体基因组数据(见表6),以榆科醉翁榆(Ulmus gaussenii)的叶绿体基因组作为外群,采用邻接(Neighbor-joining)法构建系统进化树,结果见图2。自举分析(Bootstrap)1000次重复检测各分支的置信度,结果显示进化树的置信值均大于90%,表明聚类结果的可信度较高。在系统发育树中,桑科桑属的5个物种聚到一个大分支中;桑科榕属和构属的5 个物种共同聚到另一个大分支中,该分支又分为2 个亚分支,分别是榕属分支和构属分支。近无柄雅榕在发育树上与菩提树(Ficus religiosa)的亲缘关系最近,同源性达99%以上。

表6 物种信息表Table 6 Species Information

图2 基于桑科叶绿体全基因组序列构建的系统发育树Figure 2 Phylogenetic tree based on chloroplast genome sequence of Moraceae
3 讨论
3.1 植物叶绿体基因组特征
大多数叶绿体基因组具有典型的四段式环状结构,即2个反向重复区(IR)被一个大单拷贝区(LSC)和小单拷贝区(SSC)分开;大小一般为100~218 kb;GC含量高度保守,通常在30%~40%。近无柄雅榕叶绿体基因组大小为160 292 bp,IR区长25 791 bp,GC含量为35.8%,密码子偏好使用A/T碱基,结果与典型的被子植物叶绿体基因组特征吻合[17-22]。在系统进化分析中,榕属植物的叶绿体基因组大小、结构、GC含量相差不大,说明叶绿体基因组具有高保守性[23]。
3.2 叶绿体微卫星标记特征及应用
高等植物叶绿体微卫星(chloroplast SSR,cpSSR)既具有核基因组SSR的高多态性、多等位性、共显性等特点,还兼有单亲遗传模式的结构简单、相对保守等特点[24-25],具有良好的种间、种内遗传变异区分能力,被广泛应用于农作物和林业资源研究[26-27]。基于叶绿体特定基因片段的DNA 条形码技术在植物的快速识别和物种鉴定中也发挥了巨大作用[28]。在近无柄雅榕叶绿体基因组中检索到68个SSR,共有4种类型的重复单元,单碱基重复中绝大多数为A/T类型(98.61%),具有碱基偏好性。下一步,笔者将利用近无柄雅榕叶绿体基因组中的SSR标记深入研究榕属植物的起源、迁徙和进化等。
3.3 桑科榕属种群遗传特征研究展望
榕树主要分布于热带和亚热带地区,属热带植物区系中最大的木本属之一——桑科榕属。中国榕属植物数量虽不多,但多样性丰富,榕属的亚属种类均有不同程度的分布[1],其独特的地理分布特征对全世界榕属植物进化史和种间协同进化研究具有重要参考价值。近年来,科研人员已在榕树种类及其生理、群落生态等方面开展了相关研究[29-35],但关于中国榕属植物分子遗传特征及其地域性信息等方面的研究还非常有限。本研究通过系统进化分析发现,近无柄雅榕与菩提树的亲缘关系最近,同源性达99%以上。由于NCBI数据库中的叶绿体基因组序列有限,本研究构建的进化关系还不够全面,有待于进一步完善。

