基于TCGA数据库分析ALAS1在肝癌中的表达及临床意义*
王杨燕,汪自然,马筱玲
(安徽医科大学附属省立医院检验科,合肥230001)
5-氨基酮戊酸合酶(5-aminolevulinate synthase 1,ALAS) 是血红素生成过程中的一个重要的限速酶。在线粒体中,甘氨酸和琥珀酰辅酶A在ALAS作用下生成δ-氨基-γ-酮戊酸(δ-aminplevulinic acid,ALA)。ALAS包括ALAS1和ALAS2两亚型。ALAS1(也称为ALAS-N)在红细胞和非红细胞中广泛表达,而ALAS2(也称为ALAS-E)在红细胞中呈特异性表达[1]。目前,ALAS1在肝癌发生发展中的具体作用仍不清楚。本研究通过癌症基因图谱 (The Cancer Genome Atlas,TCGA)公共数据库分析ALAS1在肝癌中的表达及其临床意义,以期为肝癌的靶向治疗提供依据。
1 材料与方法
1.1数据资料收集 从TCGA数据库(https://tcga-data.nci.nih.gov/tcga/)中下载并预处理肝癌数据集中423例的mRNA表达RNASeqV2数据(数据时间为2006年1月1日至2017年12月31日,资料收集时间为2019年10月15日)。同时在TCGA数据库中下载临床资料数据和预后数据。
1.2数据整理和临床病理特征相关性分析 筛选数据得到含有ALAS1mRNA表达量的正常肝组织样本50例,肝癌组织样本371例。对肝癌组织和正常肝组织的ALAS1表达量进行正态性检验并分析两组间的表达差异。选取性别、年龄、T分期、N分期、M分期、AJCC分期作为肝癌临床病理特征指标,去除临床病理特征缺失或者不完全的病例。根据ALAS1mRNA表达量的均值(12.39)进行分组,以表达量高于均值的为高表达组(173例),反之为低表达组(169例)。
1.3生存分析 选取数据集中包含预后随访数据的病例,使用KaPlan-Meier模型对ALAS1低表达组和高表达组之间的生存期差异进行分析并进行Log-rank检验,以P<0.05为差异有统计学意义。
1.4ALAS1相关基因筛选 登录LinkedOmics数据库(http://www.linkedomics.org/)首页,选择肿瘤类型为“肝癌”,靶点为“ALAS1”,数据集选择TCGA数据集,分析与ALAS1表达呈正相关和负相关的基因。
1.5基因集富集分析(gene set enrichment analysis,GSEA) 采用GSEA 4.0.1版软件进行分析。根据ALAS1mRNA的表达水平分为高表达组和低表达组。进一步利用GSEA网站MsigDB数据库(http://software.broadinstitute.org/gsea/msigdb)中获得c2.cp.kegg.v7.1.symbols.gmt数据集。采用缺省加权富集统计的方法进行富集分析并设置随机组合次数为1 000次。
1.6统计学分析 使用SPSS 16.0软件进行统计学分析,Graphpad Prism 7.0软件绘图。正态性检验采用Kolmogorov-Smirnov法,两组间数据比较使用非配对t检验。ALAS1表达量与肝癌患者临床病理特征的相关性分析使用χ2检验及Fisher确切概率法,不同ALAS1表达量患者预后分析使用Kaplan-Meier和log-rank检验法。以P<0.05为差异有统计学意义。在GSEA分析中以错误发现率(false discovery rates,FDR)<0.25的基因集作为显著富集基因集。
2 结果
2.1肝癌组织与正常肝组织中ALAS1的表达差异 采用Kolmogorov-Smirnov法对肝癌组织和正常肝组织的ALAS1表达量进行正态性检验,结果表明肝癌组织(P=0.05)和正常肝组织(P>0.10)中ALAS1mRNA表达量均符合正态分布。进一步进行非配对t检验,结果发现肝癌组织中ALAS1mRNA表达水平(12.39±1.20)显著低于正常肝组织(13.93±0.72),差异具有统计学意义(t=8.87,P<0.01)。
2.2ALAS1表达与肝癌患者临床病理特征相关性 筛选后共获得具有完整临床病理特征数据的肝癌病例342例,其中ALAS1低表达组169例,ALAS1高表达组173例。ALAS1的表达水平与性别(P<0.01)、T分期(P=0.01)以及AJCC分期(P=0.01)显著相关,而与年龄、N分期和M分期无相关性。见表1。
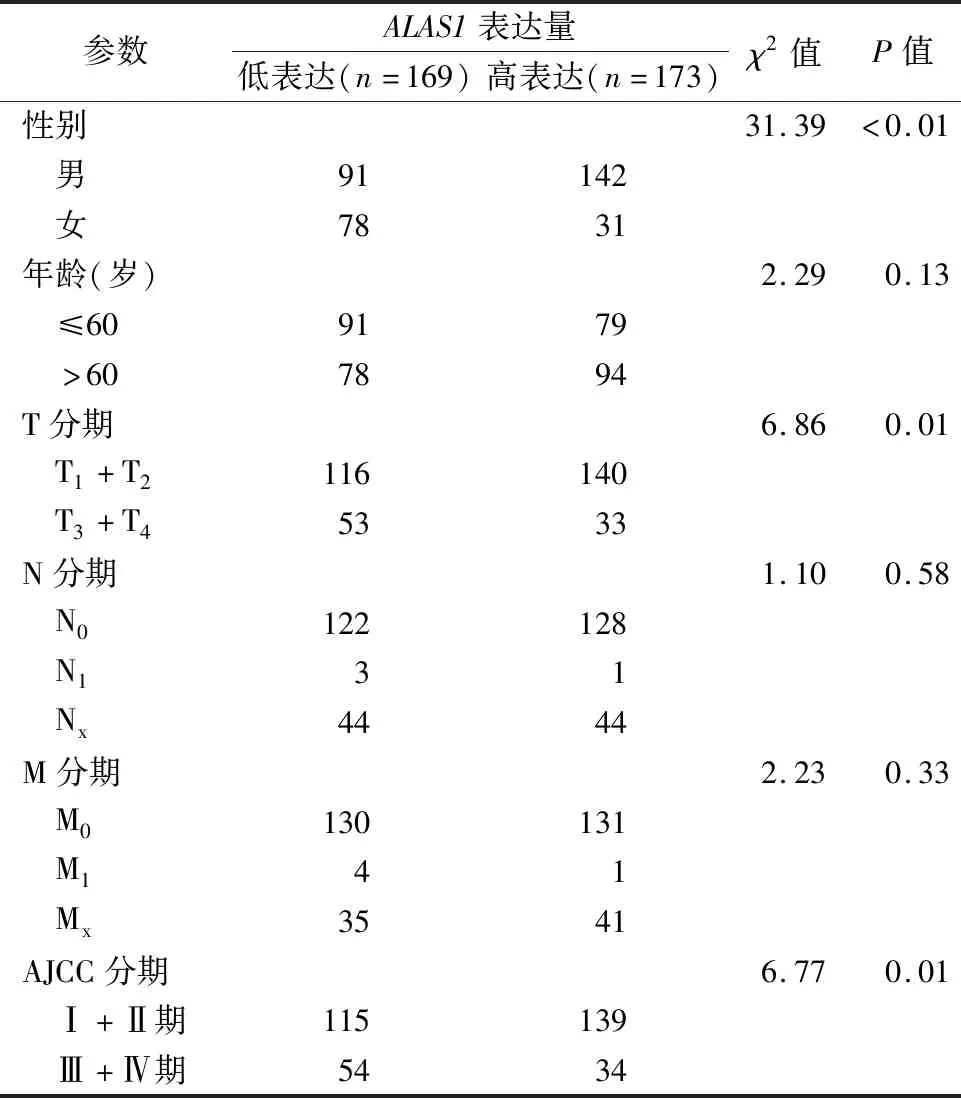
表1 ALAS1表达水平与肝癌患者临床病理特征相关性分析
2.3ALAS1表达肝癌患者预后相关性ALAS1表达水平与肝癌患者预后相关(Log-rankχ2=4.48,P=0.03)。此外,低表达ALAS1患者的中位生存期(1 372 d)显著低于高表达患者(2 456 d)。见图1。

图1 ALAS1表达肝癌患者预后相关性
2.4ALAS1相关基因分析 使用LinkedOmics数据库对ALAS1进行相关分析,以P<0.01为界限,共检索到2 433个正相关基因,6 109个负相关基因(图2A)。肝癌中ALAS1的表达与POR(r=0.70)、SORD(r=0.68)、AQP9(r=0.67)基因呈显著正相关,与PKM2(r=-0.65)、VEGFB(r=-0.60)、PLCB3(r=-0.60)基因呈显著负相关,见图2B、2C。

注:A,与ALAS1相关的基因分布散点图;B,与ALAS1呈正相关基因(前50位)热图;C,与ALAS1呈负相关基因(前50位)热图。
图2ALAS1相关基因分析
2.5ALAS1的功能富集分析 GESA富集分析结果表明,ALAS1低表达样本显著富集到细胞周期(FDR=0.03,P<0.01)、T细胞受体信号通路(FDR=0.06,P<0.01)、GnRH信号通路(FDR=0.06,P<0.01)以及趋化因子信号通路(FDR=0.154,P<0.01)等基因集。见图3。
3 讨论
虽然肝癌的诊断和治疗技术有了持续进展,但肝癌的发病机制仍未完全阐明。因此,寻找新的靶点对于肝癌的诊断和治疗有着重要的意义。TCGA组学数据库具有样本量大,数据信息丰富完整,随访时间长等优点,得到的结论较为可靠,有利于充分认识基因在肿瘤发生、发展中的作用[2-5]。通过生物医学大数据分析,可以为进一步研究基因在肿瘤的功能和调控机制提供依据。
ALAS1是血红素合成过程中的一种重要的限速酶,目前的研究主要集中在ALAS1在血红素合成中的调控作用。研究表明,在肿瘤细胞中,血红素的合成能力明显高于正常细胞[6]。在非红细胞中,血红素的生物合成受到严格的负反馈机制控制,过度的血红素合成通过降低转录和翻译、破坏mRNA的稳定、抑制前体蛋白线粒体转运等途径抑制ALAS1的表达[7]。Chen等[8]报道青蒿琥酯和组蛋白去乙酰化酶抑制剂能够促进ALAS1的表达进而发挥抗肿瘤作用。因此,探究ALAS1在肝癌中的作用和具体调控机制对于肝癌的靶向治疗意义重大。
本研究首先下载TCGA数据库中肝癌RNASeq数据集、病例临床数据和预后数据。通过对肝癌组织和正常肝组织中ALAS1表达量比较分析发现,ALAS1在肝癌组织中明显低表达,提示ALAS1可能是肝癌中的一个重要的抑癌基因。进一步分析ALAS1与肝癌患者临床特征的关系,结果发现ALAS1表达水平与性别、T分期以及AJCC分期显著相关,而与年龄、N分期和M分期无相关性。同时,生存曲线分析表明,低表达ALAS1的肝癌患者明显预后不良。以上结果表明ALAS1基因低表达可能是肝癌预后的一种不利因素。为了进一步探索ALAS1在肝癌中可能的作用机制,我们通过LinkedOmics数据库分析ALAS1密切相关的基因,结果显示ALAS1与POR、SORD、AQP9基因呈显著正相关,与PKM2、VEGFB、PLCB3基因呈显著负相关,提示以上分子可能是ALAS1潜在的上、下游分子。GSEA分析首先运用预定义的基因集,将基因按照在两组样本中的表达差异程度进行排序,而后检验预定义的基因集是否富集在排序表的顶端或者底部,是当下研究基因表达差异富集信号通路的理想生物信息学方法。本研究利用GSEA方法预测ALAS1可能的调控信号通路,发现ALAS1低表达样本显著富集到细胞周期、T细胞受体信号通路、GnRH信号通路以及趋化因子信号通路等基因集,推测ALAS1低表达可能通过调控细胞周期、免疫调节以及趋化因子信号通路等促进肝癌的进展。
ALAS1在肝癌中的作用及机制目前仍不清楚,本研究主要通过TCGA数据库数据分析ALAS1在肝癌中的表达及临床意义,证实ALAS1基因可能为肝癌临床预后评估的指标以及靶向治疗的新靶点,并且初步探讨ALAS1在肝癌中的可能分子机制与信号通路。但本研究也存在一些不足,比如TCGA数据库中为RNASeq数据,可能无法完全代表ALAS1蛋白的表达水平,以及研究数据部分分组样本量较少,可能造成统计学偏差。后期我们将围绕ALAS1进一步研究验证其分子机制与信号通路,为肝癌的靶向治疗提供新的思路。

