胃癌顺铂抵抗相关免疫标记基因的富集分析
汪圣毅,程 彦,李旭升,闫亚飞,张尚鑫,闫 强,李永翔
胃癌在我国高发,手术是主要治疗方法,辅助化疗可改善预后[1],但化疗抵抗制约疗效,与基因、通路的作用有关[2-3],免疫标记基因(immunologic signature genes, ISGs)与化疗抵抗有关[4]。外周髓磷脂蛋白22(peripheral myelin protein 22,PMP22)增强胃癌顺铂抵抗[3],是否通过ISGs调控尚不清楚。常规富集分析可发现PMP22相关的化疗抵抗通路和基因[2],但预设差异基因的倍数和统计P值,不能较为全面地反映整体信息[5]。基因集富集分析(gene set enrichment analysis, GSEA)纳入全部的差异基因,具有不预筛组间差异基因的优点[6],可发现细微变化。为明确ISGs与PMP22相关胃癌顺铂抵抗的关系,用GSEA法分析GSE 94714数据集,寻找PMP22下游的ISGs,验证新发现ISGs对胃癌总生存的影响,为深入揭示胃癌化疗抵抗的机制提供参考。
1 材料与方法
1.1 表达谱数据用基因表达数据库(gene expression omnibus,GEO)的GSE 94714数据集,包含6个样本,3个样本(GSM2481329~GSM2481331)的胃癌细胞MGC-803进行了PMP22基因敲减,另外3个样本(GSM2481332~GSM 2481334)的PMP22未行敲减处理。GEO2R分析差异基因,设置分组:PMP22基因敲减3个样本为A组,未敲减3个样本为B组,保存所有差异基因至excel表,得到差异基因表。
1.2 芯片数据及探针基因名转换下载GSE94714的soft文件,导入excel,复制探针和对应基因名称(GENE_SYMBOL),形成新的excel表即芯片数据表,探针名称升序排序。差异基因表中的探针名称进行相同的升序排序,复制ID、adj.P.Val、logFC共3列到芯片数据表,excel数据高级筛选,匹配出探针对应的基因名称,包含差异基因表,形成探针基因名转换表,定位GENE_SYMBOL列的空值,删除没有GENE_SYMBOL的所有行,另存为包含GENE_SYMBOL的差异基因表,用于后续分析。
1.3 差异表达分析包含GENE_SYMBOL的差异基因表,导入在线分析工具(Easy Tools v 2.2 by Chris Lou)中(www.chrislifescience.club:3838/R/AnnoE2)的Volcano Online,绘制火山图。差异倍数(fold change, FC)以2为底的对数(Log2FC或Log FC)分别设置为1、1.5,P值<0.05,计算两种Log FC条件下,有差异(上调、下调)和无差异的基因数目,并计算无差异基因数目的差值。
1.4GSEA未筛选的所有差异基因,用含GENE_SYMBOL的差异基因表,导入在线工具(www.chrislifescience.club:3838/R/AnnoE2)的GSEA中,列名中分别填写导入表格中对应各列的名称,分子标签数据库(MSigDB,molecular signatures database)中ID选择c7。标准化富集评分(normalized enrichment score,NES)降序排列,取NES前6位的基因集进一步分析。
1.5 免疫标记基因集的交集基因NES前6位的ISGs条目导入MSigDB(http://software.broadinstitute.org/gsea/msigdb/search.jsp),查询并下载基因,在线工具分析交集基因(http://bioinformatics.psb.ugent.be/cgi-bin/liste/Venn/),绘制韦恩图。
1.6 统计学处理在线工具的R软件进行统计,富集评分标准化的值(NES)进行排列检验(permutation test),错误发现率(false discovery rate,FDR)方法判断NES的统计学意义;组间基因表达差异进行t检验;交集基因导入KM-plotter数据库(http://kmplot.com/analysis/),根据基因表达水平自动选择临界值,分割为高、低表达组,Kaplan-Meier法分析基因表达水平与胃癌患者总生存(overall survival,OS)的关系,Log-rank检验比较组间差异,给出风险比(hazard ratio,HR)、95 %的可信区间(confidence interval,CI),P<0.05为差异有统计学意义。
2 结果
2.1 差异表达矩阵GEO2R分析共获取差异基因34 183个,用芯片数据表对表达数据进行整理,转换基因名,删除无GENE_SYMBOL的行,获差异表达基因共29 833个。COUNTIF函数计算Log FC>0的上调基因共12 452个,Log FC<0的下调基因共17 381个。
2.2 差异基因表达差异倍数的对数(Log FC)为1时,结果表格中,COUNTIF函数计算上调(UP)、下调(DOWN)、无差异(NOT)基因数目,分别为3 819个、3 730个、22 284个;Log FC为1.5时,UP、DOWN、NOT基因数分别为2 300个、1 492个、26 041个。两种条件下的火山图见图1。两种条件下,UP、DOWN、NOT基因数的差值分别为1 519个、2 238个、3 757个。上下调基因数目与筛选条件的关系见图2。
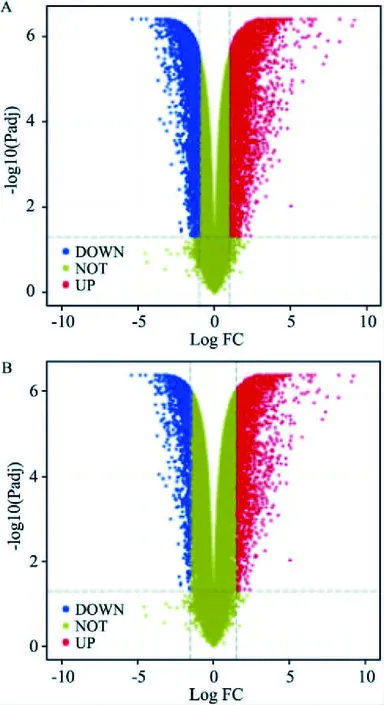
图1 差异表达基因的火山图A:Log FC为1,P<0.05的火山图;B:Log FC为1.5,P<0.05的火山图
2.3 富集分析MSigDB中的c7作为富集分析的基因集,富集得到条目4 872个,NES降序排序,排序前6的结果见表1,前6位的条目作GSEA图,见图3。
2.4 免疫标记基因集的交集基因NES前6位的ISGs条目导入MSigDB,查询、下载基因,在线工具分析交集,A、C、D基因集得到的交集基因1个,为线粒体核糖体蛋白L12(mitochondrial ribosomal protein L12,MRPL12),见图4a。E、F基因集的交集基因2个:富含脯氨酸蛋白13(proline rich 13,PRR13)、富含嘌呤元素结合蛋白A(purine rich element binding protein A,PURA);A、E的交集基因5个:艾杜糖2-硫酸酯酶(iduronate 2-sulfatase,IDS)、LIM结构域2(LIM domain containing 2,LIMD2)、小视觉叶同源物(small optic lobes homolog,SOLH)、CCR4-非转录复合物亚单位3(CCR 4-NOT transcription complex subunit 3,CNOT3)、转化生长因子β1(transforming growth factor beta 1,TGFβ1);A、F的交集基因4个:毛状蛋白样F-肌动蛋白结合蛋白1(coactosin like F-actin binding protein 1,COTL1)、聚(RC)结合蛋白1(poly(rC) binding protein 1,PCBP1)、二酰甘油激酶ζ(diacylglycerol kinase zeta,DGKZ)、接头蛋白2(docking protein 2,DOK2),见图4b。

表1 基因集富集分析NES前6的条目

图2 上调和下调基因数与筛选条件的关系
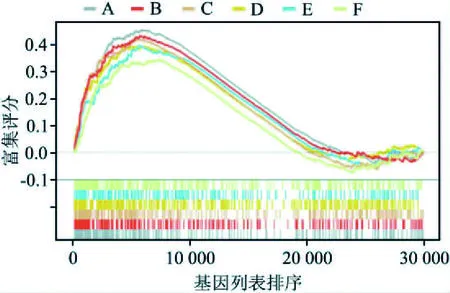
图3 前6位免疫标记相关条目的GSEAA~F:对应表1中条目编码
2.5 相关基因对胃癌总生存的影响上述12个交集基因,导入KM-plotter,分析发现:MRPL12、PRR13、COTL1、PCBP1的高表达与胃癌OS延长关联(P<0.05),IDS、LIMD2、PURA、SOLH、CNOT3、TGFB1、DGKZ、DOK2的高表达与胃癌OS降低关联(P<0.05),见表2,部分基因的生存曲线见图5。
3 讨论
胃癌的综合治疗可提高疗效,作为综合治疗重要组成的化疗,在胃癌手术前后的辅助治疗中发挥重要作用,明确化疗耐药机制、降低化疗抵抗、提高化疗敏感性,具有重要的临床意义。顺铂通过多种机制抑制胃癌细胞,其耐药性的产生可由PMP22介导,但其下游是否与ISGs存在关联,尚无文献报道。为探索PMP22下调后减弱胃癌顺铂抵抗是否通过下游的ISGs发挥作用,本研究对GEO数据库的GSE94714数据集进行了GSEA分析,发现PMP22可通过多种ISGs相关的下游机制,对胃癌顺铂抵抗的表型进行调节。
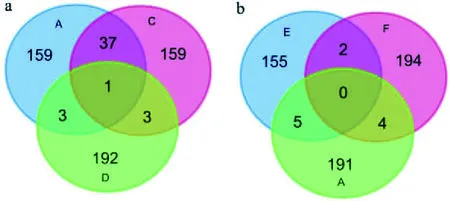
图4 交集基因韦恩图a:基因集A、C、D的交集;b:基因集A、E、F的交集
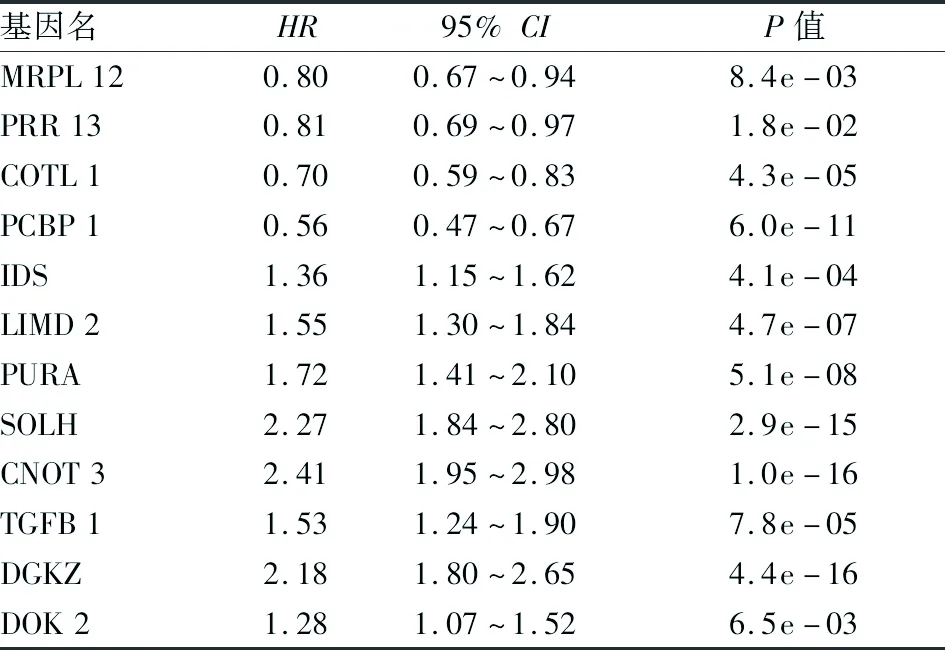
表2 交集基因与胃癌总生存的关系
本研究发现,根据差异倍数(fold change, FC)和统计P值进行筛选,PMP22基因敲减、未敲减组差异表达基因的数目减少,因此采用未筛选的差异基因,GSEA分析发现了NES前6位的ISGs条目,其组成基因用韦恩图方法取交集,发现了12个ISGs,均与胃癌的总生存(OS)有关。
随着差异倍数FC或logFC的增大,统计分析无差异的基因数逐渐增加,被排除的基因增多,筛选出有差异的基因数目逐渐减少,即上下调基因的数目均减少,与Xiao et al[7]进行条件筛选后、使组间差异表达基因数减少的研究结果一致。预筛选可遗漏部分基因,造成后续分析无法找到有意义、但是改变细微的重要基因。GSEA方法可避免遗漏,较为全面地分析差异基因,通过预定义基因集,可获取有分类的基因标记信息。笔者通过GSEA分析获得的12个基因,既与免疫有关,又与胃癌的预后有关,并且在前期进行的常规富集分析中未能被发现[2],表明GSEA方法是常规富集分析方法的重要补充。
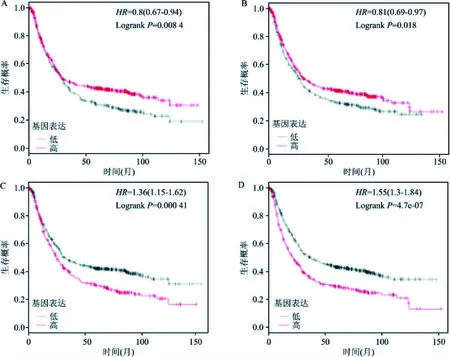
图5 部分交集基因对胃癌OS的影响A:MRPL12高、低组的OS生存曲线;B:PRR13高、低组的OS生存曲线;C:IDS高、低组的OS生存曲线;D:LIMD2高、低组的OS生存曲线
新发现的ISGs通过不同机制参与肿瘤细胞的化疗抵抗和发生发展,部分基因与胃癌化疗抵抗的关系尚未见文献报道。MRPL12基因可通过改变线粒体代谢、调节细胞的氧耗量,影响肿瘤生长,MRPL12敲减后,胰腺肿瘤细胞内的线粒体活性降低,糖酵解加强,肿瘤细胞体外生长减慢,但体内生长加速[8]。本研究显示PRR13高表达胃癌的OS延长,但Pontikakis et al[9]发现卡铂、紫杉醇治疗后,PRR13高表达卵巢上皮性癌的OS缩短,与化疗抵抗有关,表明PRR13表达对不同肿瘤生存时间的作用存在差异,其机制有待进一步阐明。COTL1基因可增强乳腺癌对化疗的敏感性,与白细胞介素24、非经典TGFβ1通路有关[10]。PCBP1可降低豆荚蛋白表达、增强胃癌腹膜转移细胞对多西他赛的敏感性[11]。LIMD2与甲状腺乳头状癌的转移有关[12]。TGFβ1可能通过细胞黏附、转移相关分子通路影响胃癌预后[13],本研究结果与之相似。DGKZ作为关键基因,与非小细胞肺癌的进展和预后有关[14]。上调因启动子甲基化而沉默的DOK2表达,可增强卵巢癌对铂类药物的敏性[15]。IDS、PURA、SOLH、CNOT3与胃癌化疗抵抗的关系,目前尚未见文献报道,是进一步研究的方向。
本研究的局限性:新发现的ISGs与胃癌顺铂抵抗的关系尚需进一步的体内外实验验证,其作用方向和机制依然存在不确定性,初步结果仅为继续研究的基础和线索。

