高通量测序技术在转基因植物分子特征评价中的应用
马硕, 焦悦, 王旭静, 翟勇, 王志兴*
(1.中国农业科学院生物技术研究所, 农业农村部农业转基因生物安全评价(分子)重点实验室,北京 100081; 2.农业农村部科技发展中心, 北京 100122)
分子特征是指转基因植物中外源DNA片段在受体基因组中的整合(包括插入外源基因的拷贝数、插入位点的侧翼序列、插入序列的完整性、是否有载体骨架序列的插入等)、表达以及遗传稳定性等方面的信息,是建立转基因植物精准检测技术的依据,也是转基因植物安全评价的主要内容和核心。目前,插入外源基因的拷贝数可以通过Southern杂交、荧光定量PCR或数字PCR来进行解析[1-3],插入位点的侧翼序列可以通过基于PCR的染色体步移技术获得[4-9]。但这些技术仅能对外源插入片段整合情况比较简单的转基因作物的分子特征进行有效解析,难以获得同一位点插入多个拷贝和多个外源基因复合性状的转基因作物以及基因编辑、RNAi等新型转基因作物的精准分子特征信息。
随着测序技术的发展和大数据时代的到来,高通量测序成为精准解析转基因生物分子特征的一项新技术,其具有高通量、灵敏度高、可操作性强和可重复性强等特点。基于高通量测序分析转基因生物分子特征获得的数据已经被美国、加拿大、欧盟等国家监管部门所接受,用于转基因生物的安全评价。在我国转基因生物安全评价中,可否接纳高通量测序数据,以何种方式接纳数据以及测序数据以何种方式呈现等都是目前需要解决的主要问题。鉴于此,本文综述了测序技术的发展、高通量测序技术在转基因植物分子特征解析中的应用、国外转基因植物安全评价中对高通量测序数据的要求等,并提出了我国采用高通量测序数据进行转基因生物安全评价的建议,为其在我国转基因生物安全评价中的应用提供理论支撑。
1 转基因植物分子特征解析常用方法
目前,分析转基因植物分子特征的常用方法包括Southern杂交、实时荧光定量PCR、数字PCR、基于PCR的染色体步移技术等(表1),这些方法都得到了有效的验证和使用。其中,Southern杂交、实时荧光定量PCR和微滴数字PCR是分析外源基因拷贝数的常用方法。Southern杂交是当前转基因生物安全监管部门认可的分析方法,具有准确度高、重复性好等优点,但无法正确分析同一位点插入多个拷贝的复杂情况,也无法明确插入片段是否有缺失或突变。实时荧光定量PCR是一种相对定量分析外源基因拷贝数的方法,需要利用标准物质制定标准曲线,还需要选择已知拷贝数的基因作为参考基因,因此,结果易受引物和探针浓度、DNA纯度等因素的影响[10]。数字PCR操作流程相对简单,在测定过程中无需绘制标准曲线,实现了对外源基因的绝对定量。但存在对荧光探针的特异性要求高、成本高、微滴形成卡的通量低等不足。
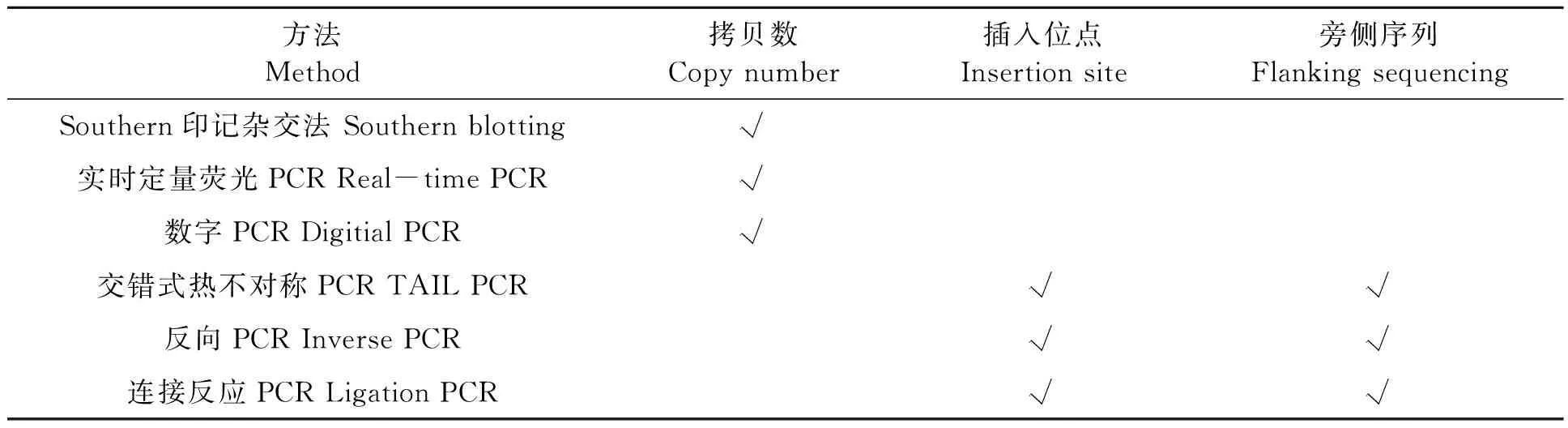
表1 常用转基因植物分子特征解析方法Table 1 Common method for molecular character analysis of transgenic plant
基于PCR的染色体步移技术包括TAIl-PCR(thermal asymmetric interlaced)[11]、反向PCR(inverse PCR)[7]和连接介导的PCR(ligation-mediated PCR)[9]等,是分析转基因生物中外源基因测序序列的常用方法。利用这些方法,成功的获得了转基因大豆、玉米、棉花等转基因作物的侧翼序列[12-16]。但这些技术在扩增过程中经常会产生非特异条带,不适合用于多位点插入转化事件的侧翼序列分析[17],而且当插入片段发生序列重排时,上述方法很难确定外源基因在受体生物基因组中的位置和插入片段的完整性[18]。
2 DNA测序技术的发展
DNA测序技术始于20世纪70年代, Sanger和Coulson于1975年发明了链终止测序法(Sanger测序)[19]。1977年,Maxam等[20]发明了化学降解测序法。1977年,首次利用Sanger测序测定了噬菌体X174的长度为5 375 bp的基因组全序列[21]。自此,人类开始利用基因组学大数据探究生命遗传差异的本质,步入基因组学时代。
Sanger测序被称为第一代测序技术,其原理是利用双脱氧核苷三磷酸(ddNTP)在DNA合成过程中不能形成磷酸二酯键而中断DNA合成,即链终止法测序。在测序过程中需要进行PCR扩增,具有测序读长较长、准确度高达99.999%的特点,但存在测序成本高和通量低的缺点。
2005年,454公司推出了基于焦磷酸测序法的GS-20测序系统,标志着高通量测序开始,是新一代测序技术的里程碑事件。此技术不需要荧光标记的引物或核酸探针,也不需要进行电泳,具有分析结果快速、准确、高灵敏度和高自动化等特点[22]。 2006年,Illumina推出了基于可逆链终止物和合成测序法的HiSeq技术。2007年,ABI公司推出了基于连接酶法的Solid测序技术,此技术的最大优点就是每张玻片能容纳更高密度的微珠,在同一系统中轻松实现更高的通量[23-24]。这些高通量测序技术被称为第二代测序技术,具有通量高、成本低等特点[25]。
2009年,由PacBio公司研发的SMRT测序技术问世,步入单分子测序时代[26-27]。随后出现的单分子测序技术还包括Oxford Nanopore 公司的纳米孔单分子测序技术[28-29]。这些技术不需要进行PCR扩增,能有效避免因PCR偏向性而导致的系统错误,同时读长明显提高,可以达到2 000 bp以上,被称为第三代测序技术(表2)。
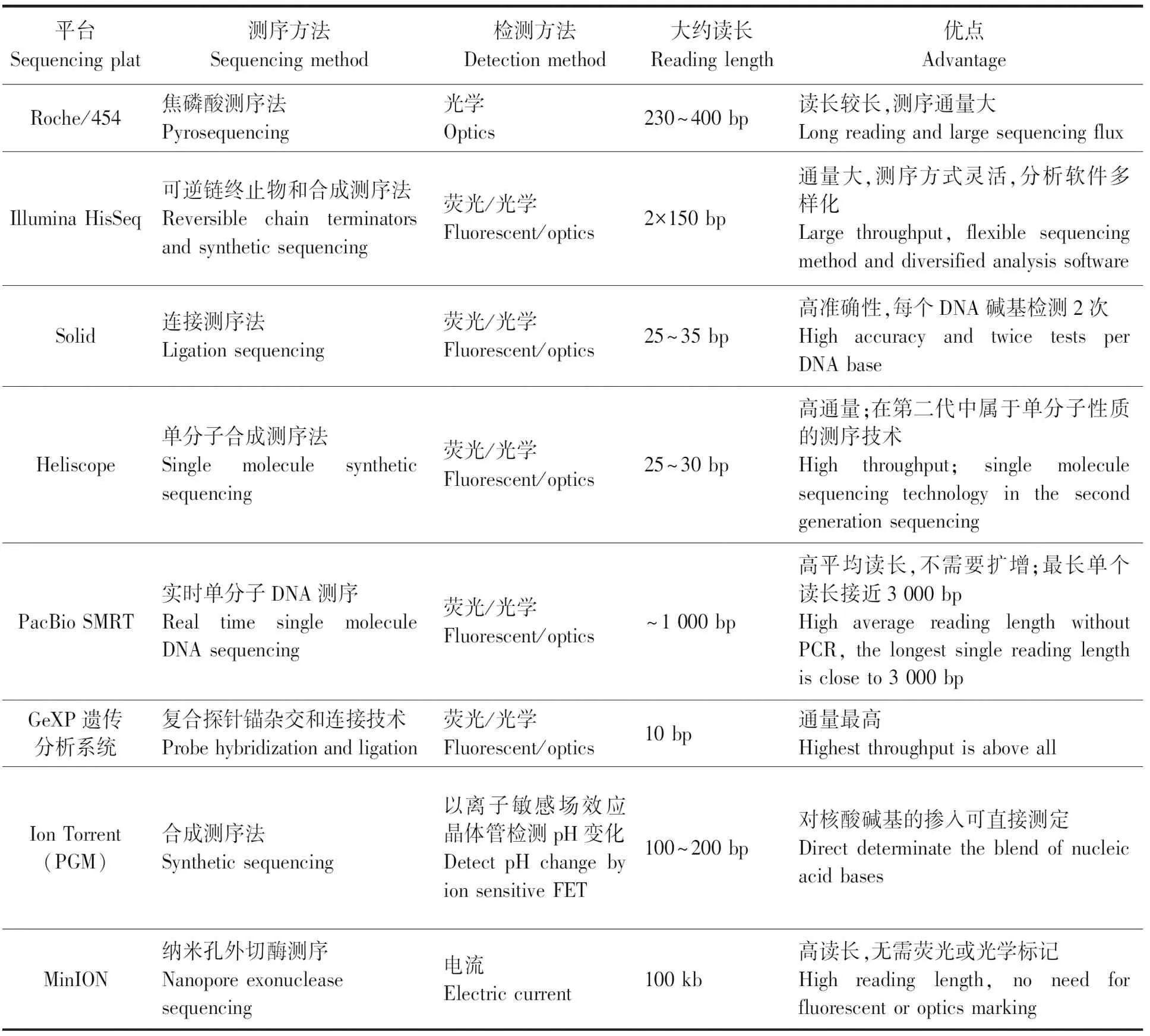
表2 测序平台间比较[19-30]Table 2 Comparison of different sequencing plat[19-30]
3 高通量测序技术解析转基因分子特征
3.1 捕获测序解析转基因生物分子特征
捕获测序是指将基因组DNA片段化后,根据转化载体的已知序列设计探针,利用探针捕获目标DNA片段,富集后再进行测序的一项技术。目前根据设计探针的位置不同,可以分为T-DNA捕获测序和SBS(Southern by sequencing)两种技术体系。
3.1.1T-DNA捕获测序技术 T-DNA 捕获测序技术利用T-DNA边界序列作为探针捕获T-DNA和基因组的结合区,然后通过测序获得外源DNA片段插入信息的一种技术(图1)。探针长度一般为70 bp,5’端生物素化。Lepage等[31]利用T-DNA捕获和454测序技术获得了55个拟南芥突变体的T-DNA插入信息。Inagaki等[32]利用T-DNA捕获和Illumina测序技术对29个拟南芥样品进行了分子特征分析,获得了22个样品的T-DNA插入信息,其中有4个样品为多位点插入,证明T-DNA捕获测序技术是分析转基因生物分子特征的快速有效方法。但此技术只能用于农杆菌介导法获得的转基因生物中外源插入片段侧翼序列的分析,无法解析插入片段的完整性和是否有载体骨架的插入。
3.1.2SBS技术 SBS技术是依据转化载体的DNA序列设计一系列带有标记的探针,利用这些探针与受体基因组DNA片段杂交,对捕获到的DNA片段进行测序,通过分析结合区序列来确定外源基因在转基因生物基因组中的整合情况(图2)。
Zastrow-Hayes等[33]构建了115 kb的包含有89个转化载体序列的捕获探针库,利用SBS技术对79个转基因玉米中外源基因的拷贝数进行了分析,获得的结果与Southern结果一致,证明SBS技术是分析转基因生物分子特征的有效方法。
3.2 全基因组测序解析转基因生物分子特征
全基因组测序首先通过对转基因生物进行基因组测序,然后再利用载体DNA序列从测序数据中捕获外源插入DNA序列及外源DNA与受体基因组结合区序列,根据结合区序列分析转基因生物分子特征。此方法既可以用于分析转基因生物中外源插入序列的侧翼序列,也可以分析外源插入序列的拷贝数,是目前常用的分子特征解析方法(图3)。
Kovalic等[35]利用全基因组测序技术和接合区序列分析获得转基因大豆MON17903和MON87704的分子特征信息,通过与Southern杂交结果进行比较,二者的结果一致,证明全基因组测序技术是分析转基因生物插入拷贝数的有效方法。Wahler等[18]利用HiSeq GAII测序平台获得了转基因水稻LLRice62中外源插入序列的侧翼序列,明确了外源基因在基因组中的插入位点。Guo等[36]利用HiSeq 2500测序平台获得了耐除草剂转基因大豆中外源插入片段的侧翼序列,测序深度为21×。Guttikonda等[37]以转基因大豆TE1、TE2和TE1×TE2为试验材料,证明全基因组测序和捕获测序是分析转基因生物分子特征的有效方法。Park等[38]利用HiSeq 2500测序平台获得了抗虫转基因水稻SNU-Bt9-5、SNU-Bt9-30和SNU-Bt9-109中外源插入片段的侧翼序列,测序深度大于54×。Cade等[39]以转基因玉米5323和MIR152为试验材料,证明全基因组测序技术在拷贝数分析、非预期插入的鉴定和小片段插入分析方面,与Southern杂交具有相同的灵敏性,是分析转基因玉米分子特征的有效方法。
4 基因组测序数据在转基因生物安全评价中的应用
随着基因组测序技术的发展,已有越来越多的国家认可利用基因组测序技术来分析转基因生物分子特征,并在安全评价中接受利用基因组测序技术获得的分子特征数据和信息。目前,美国、澳大利亚、阿根廷、欧盟和加拿大等12个国家或地区接受基因组测序技术数据用于分析转基因生物的分子特征。其中欧盟和加拿大已出台了相应的指南,对测序要求、数据分析和数据呈现形式等进行了规定和说明。
4.1 欧盟在转基因生物安全评价中对提交基因组测序数据的要求
2018年6月,欧盟发布了《用于鉴定转基因植物分子特征的DNA测序质量的技术说明》,对采用测序技术解析转基因生物分子特征时的DNA样品准备、测序要求、提交数据格式等方面进行了明确规定[40]。
首先,DNA样品必须来自待评估的转基因植物。提交的评价材料中应详细描述转基因植物中的包含的转化事件、植物材料的来源、取样的具体过程和时间、取样的部位、取样的样本数、DNA的提取过程等信息。为了便于重测序检验,申请者应储存至少能满足三次测序所需的样品DNA量。
在测序方面,需提供测序文库的制备过程、测序方法和测序平台的详细信息。如果使用了捕获技术,还应提供探针的设计、杂交过程、如何评估杂交条件和捕获效率等方面的详细资料。为评估测序数据的可靠性,应提供测序深度方面的信息。当利用全基因组测序(whole genome sequencing, WGS)识别插入DNA和可能插入的骨架序列时,必须估计全基因组中的平均读序深度。当用Illumina等短读序技术测定插入DNA和侧翼区的序列时,最小读序深度不应低于40 bp。
申请者需提交压缩(比如gzip)FASTQ格式的原始NGS读序,采用Sequence Alignment/Map(SAM)格式、Binary Alignment/Map(BAM)格式或CRAM格式提交经过比对/匹配后用于生成最终转化体序列的序列,最终转化体序列必须提交电子版的ASCII文本文件,格式可以为EMBL/GenBank或NCBI’s Sequin(ASN.1)格式。
另外,此指导文件还对测序技术用于分析不同分子特征提出了不同的要求:①当分析DNA插入序列的侧翼序列时,如果申请者此前已向欧盟委员会(EFSA或EURL-GMFF)提交过转化体的序列,则申请时必须将正在评估的GM转化体的序列与所有之前提交过的该转化体的序列进行比对。申请者必须提供包括所有那些序列在内的比对结果,并报告所发现的差异,同时探讨出现差异的原因。申请者必须详细描述和解释所用的方法,提供测序深度信息并说明理由。②当分析DNA插入的拷贝数时,可以用接头序列分析(junction sequence analysis, JSA)的方法,需要对任何丢弃的接头读序进行解释说明,需要提供读序深度的详细信息。③当分析遗传稳定性时,可以通过比对基于测序的读序(或重叠群)与插入DNA及侧翼区的序列来实现这个目的。对于包含多个转化体的GM植物,应证明复合性状转基因植物中每个转化体的完整性。
4.2 加拿大在转基因生物安全评价中对提交基因组测序数据的要求
2017年,针对全基因组测序在转基因分子特征中的应用,加拿大公布了《提交全基因组测序(WGS)数据支持新型食品、新型饲料及新性状植物上市前评估指南》(简称《指南》)。《指南》指出,申请者可以只提交用传统方法获得的数据,也可在此基础上增加与分子表征有关的WGS数据。此《指南》是首个针对提交WGS数据支持转基因植物上市前评估的文件(https://www.canada.ca/en/health-canada/services/food-nutrition/legislation-guidelines/guidance-documents/guidance-submit-wgs-data.html)。《指南》要求,申请者应对WGS研究的目的、原理、实验方案、DNA样品制备、测序仪品牌和型号、分析软件和版本以及所带软件的版本等进行详细描述。如果试验设计用到对照,应说明与对照有关的信息。
WGS数据分析应包括如下内容:数据处理过程;数据质量报告(即FASTQC),应提供基本的统计数据,如读序长度范围、读序次数、GC含量等,以及显示量化总体数据质量的指标的图表;数据处理软件,如果申请者开发了新的计算软件,应包含该软件的验证研究;每一步数据分析的目的,并提供每个计算步骤中的参数选择理由;说明分析中的每一步所获得的结果,如果使用了参考序列来匹配所读取的转化体基因组序列,申请者应明确所用的参考品种并说明理由。
5 我国转基因作物安全评价中采用高通量测序数据的建议
综上所述,高通量测序技术具有通量高、成本低、标准化、精确度高等特点,在转基因生物分子特征解析中得到应用,并且测序数据已被美国、欧盟、加拿大等国家认可用于转基因生物安全评价。当前,在国家转基因生物新品种培育重大专项的多年资助下,我国复合性状、RNAi、基因编辑等新型转基因作物不断涌现,给转基因作物分子特征解析技术带来了新的挑战。将高通量测序技术应用于我国转基因生物分子特征解析,并在转基因生物安全评价接受测序数据是必然趋势。为保证测序数据在我国转基因生物安全评价中应用的科学有效性,吸取其他国家的相关经验,对高通量测序技术在我国转基因生物安全评价中的应用提出几点建议: ①制定高通量测序技术在我国转基因生物安全评价中应用的标准和技术规范,对数据质控要求、测序覆盖度、提交的数据类型及格式、数据呈现形式等进行明确规定。②采取个案分析原则。对数据要求、测序深度等的要求不应统一要求,而应根据测序的目的、转基因作物的类型、转基因作物受体基因组大小等进行具体情况具体分析。个案分析原则也是当前我国转基因生物安全评价中一直遵循的主要原则之一。③任何技术都有其自身的特点和适用范围,相互之间是相辅相成的,不存在相互替代的问题。同理,高通量测序技术获得的分子特征数据与Southern杂交、染色体步移等常规技术获得的数据之间是相互验证和补充的关系。因此,在转基因生物安全评价过程中,可以只提供Southern杂交结果,高通量测序数据可以作为辅证。当Southern杂交不能明确转化事件的分子特征时,也可以只提供高通量测序数据。
6 结语
高通量测序技术与传统分析方法相比,具有通量高、标准化、准确性好等优点,在转基因玉米、大豆、水稻等作物分子特征解析方面已得到应用,已成为转基因作物分子特征解析的有效方法之一。
当前,生物技术发展迅猛,基因编辑、RNAi、mRNA、复合性状等新型转基因作物不断涌现,Southern杂交、染色体步移技术等传统分析方法难以正确解析这些新型转基因作物的分子特征。高通量测序技术能够精确获得DNA序列信息,在解析这些新型转基因作物分子特征方面具有明显的优势,相信伴随测序技术的不断发展,高通量测序技术在解析转基因作物分子特征方面将发挥更大的作用,具有良好的应用前景。

