大鼠骨骼肌挫伤后血清生物标志物的时序性变化
(四川大学华西基础医学与法医学院,四川 成都 610041)
骨骼肌挫伤是法医学实践中常见的机械性损伤,挫伤时间推断是目前法医学鉴定中的难点之一。既往推断骨骼肌挫伤时间的研究大多是对单一物质的表达变化进行检测,存在物质的某一表达值对应两个甚至多个时间点的现象,因此难以准确界定挫伤时间的范围。
代谢组学是在20世纪90年代后期,继蛋白组学、转录组学、基因组学之后发展起来的系统生物学的一个新分支,以生物样本中相对分子质量<1000的小分子物质为研究对象,如糖类、脂类、氨基酸、核苷酸等,通过气相色谱-质谱法(gas chromatography-mass spectrometry,GC-MS)、液相色谱-质谱法(liquid chromatography-mass spectrometry,LC-MS)和核磁共振(nuclear magnetic resonance,NMR)等技术获得大量的机体代谢物随时间变化的信息,实现对机体受刺激或扰动前后代谢产物谱图及其动态变化的整体监测,利用主成分分析(principal components analysis,PCA)、偏最小二乘判别分析(partial least square-discriminant analysis,PLS-DA)、正交偏最小二乘判别分析(orthogonal partial least square-discriminant analysis,OPLSDA)等多元统计分析方法对数据进行降维分析,在已知高维数据中找到规律对未知样本进行判别分析[1]。近年来,代谢组学广泛应用于各学科领域的研究,HAO等[2]利用超高效液相色谱-质谱(ultra-high performance liquid chromatography-mass spectrometry,UPLC-MS)代谢组学方法对慢性低剂量高灭磷中毒的大鼠尿液进行分析,认为可以将二甲基硫代磷酸盐作为高灭磷中毒的敏感性指标。朱士胜[3]运用1HNMR代谢组学方法研究了弥漫性轴索损伤(diffuse axonal injury,DAI)大鼠血浆中代谢物质的变化,筛选出32种可能为DAI早期诊断提供可靠依据的特异性标志物,说明了代谢组学用于进行DAI早期诊断的可能。
本研究在建立大鼠骨骼肌不同挫伤时间模型基础上,利用GC-MS技术获取大鼠的血清代谢物数据,比较PCA、PLS-DA和OPLS-DA 3种模式识别方法的识别预测能力,选择识别能力高的方法筛选生物标志物;利用支持向量机(support vector machine,SVM)对所获取的生物标志物数据进行分析及建模,初步探讨其在大鼠骨骼肌挫伤时间推断中应用的可能性,以期为大鼠骨骼肌挫伤时间的推断提供新思路。
1 材料与方法
1.1 实验动物分组
健康成年雄性SD大鼠60只(购自成都达硕实验动物有限公司)被随机分为10个实验组(伤后0、2、4、8、12、24、48、96、144、240 h组),1个对照组和1个验证组(伤后192 h组),每组5只。所有实验过程均遵守四川省实验动物管理委员会的规定,符合《实验动物管理和使用指导》。
1.2 实验方法
1.2.1 模型制备与样本收集
所有大鼠均适应性喂养3d。参照自由落体打击装置[4]将500 g砝码从90 cm高处自由下落,打击1次造成大鼠左后肢骨骼肌挫伤。确保打击处皮肤完整,皮下出血、肿胀伴爬行时患肢拖拉,胫腓骨未扪及骨折。实验组大鼠分别在伤后0、2、4、8、12、24、48、96、144、240h脱颈椎处死,验证组大鼠在伤后192h脱颈椎处死。对照组大鼠不造成损伤,在适应性喂养3 d后脱颈椎处死。大鼠处死后立即从腹主动脉采血分离上清液冻存。取挫伤区中心肌肉标本行常规苏木精-伊红(hematoxylin-eosin,HE)染色,于光学显微镜下观察骨骼肌的病理学改变。
1.2.2 样本前处理
血清样本:上述冻存的血清样本解冻后取50μL于微量离心管中,与150 μL的-20℃冰乙腈混匀、涡旋,以离心半径10cm,12000r/min,离心10min,取上清液吹干,肟化、硅烷化后加入内标硬脂酸甲酯庚烷溶液,涡旋静置,取上清液进行GC-MS分析。
质量控制(quality control,QC)样本:取所有血清样本各50 μL于10 mL离心管中混匀,制得QC样本。取QC样本50 μL于1.5 mL微量离心管中,加入冰乙腈150 μL,其余操作同血清样本。QC样本用于物质定性,检验仪器和样本的稳定性。
空白对照样本:在空的杜氏小管中加入100 μL冰乙腈,用氮吹仪吹干,其余操作同血清样本。与血清样本、QC样本代谢产物数据进行对比,去除干扰物数据。
1.2.3 GC-MS检测条件
使用7890A-5795C气相色谱-质谱联用仪(美国Agilent公司),色谱分离采用DB-5MS毛细管柱(30 m×0.25 mm,0.25 μm),载气为高纯氦气,流速为1.0mL/min,进样口温度为250℃,辅助加热器温度为230℃,分流比为10∶1;起始温度为60℃,保持1min,以8℃/min升至300℃,最后保持5min。质谱离子源温度为230℃,最大值为250℃,四极杆温度为150℃,最大值为200℃,电离能量为70 eV,全扫描模式,扫描范围m/z 50~600。
1.3 数据分析与统计学处理
利用AMDIS 6.51软件(美国NIST公司)对GC-MS数据去卷积,以峰面积>10000、匹配度>80%[5]定性色谱峰,比对美国国家标准与技术研究所(National Institute of Standards and Technology,NIST;https://www.nist.gov)14质谱库、结合人类代谢组学数据库(Human Metabolome Database,HMDB;https://hmdb.ca)进行色谱峰定性,通过色谱峰对齐、污染物排除、缺失值填补、数据归一化等预处理后初步筛选生物标志物,获得代谢物名称、保留时间、相对含量等信息,导入SIMCA-P 13.0软件(瑞典Umetrics公司)进行PCA、PLS-DA、OPLS-DA,比较3种模式识别方法的识别预测能力。
为避免过拟合,采用交叉验证评估模型的稳定性及预测能力[6],Q2>0.5为可靠模型,Q2>0.9为优秀模型,且 R2、Q2值差距不大于 0.2~0.3 为好[7],R2、Q2值越接近1说明模型可靠性越强。以R2、Q2值比较3种模式识别方法的识别预测能力。置换验证用于验证模型的可靠度,R2与Q2值小于得分图中原始值,并且Q2回归直线与Y轴交点小于0说明该模型有效[8]。
通过以上标准选择最佳模式识别方法,以变量投影重要性(variable importance in projection,VIP)>1.0且单因素方差分析P<0.05为条件进一步筛选生物标志物。分别以初步筛选的生物标志物和进一步筛选出的生物标志物相对含量作为输入向量,以实际损伤时间为输出向量建立SVM回归模型,为防止模型过拟合,选择70%数据(36个样本)作为训练集,30%数据(14个样本)作为测试集。以验证组(伤后192h组)数据为验证集,通过预测损伤时间与真实损伤时间的差值及预测损伤时间均方根误差(root mean square error,RMSE)初步判断模型预测能力。
2 结 果
2.1 大鼠骨骼肌挫伤的病理学改变
对照组大鼠骨骼肌大体观察及组织病理学检验均未见明显异常。
实验组大鼠大体观察见损伤处皮肤完整,皮下出血、肿胀,解剖确定无胫腓骨骨折。组织病理学检验发现,骨骼肌挫伤后即可见皮下出血,部分肌纤维断裂。伤后2~4h出血加重,肌纤维变性,间质水肿。伤后8h部分骨骼肌出现不同程度坏死,横纹不清,间质水肿加重,较多中性粒细胞浸润。伤后12~24h骨骼肌进一步坏死,横纹消失,间质水肿明显,中性粒细胞较前增多,可见较多单核巨噬细胞和少量淋巴细胞(图1)。伤后48h组织疏松水肿仍较明显,中性粒细胞较前明显减少,单核巨噬细胞和淋巴细胞增多,挫伤区大片肌细胞坏死解离。伤后96h损伤区周边红细胞溶解,周围可见新生毛细血管出现,中性粒细胞进一步减少,以单核巨噬细胞和淋巴细胞为主的炎症细胞散在浸润。伤后144~240h损伤区各炎症细胞进一步减少,可见肉芽组织形成。

图1 对照组及伤后24h组大鼠骨骼肌的组织病理学变化(HE×400)Fig.1 Histopathological changes of skeletal muscle of rats in control group and 24h after injury group(HE×400)
2.2 生物标志物的筛选
2.2.1 QC样本分析
将QC样本、对照组样本和实验组样本的所有代谢物数据一起导入SIMCA-P 13.0软件中进行PCA分析,发现QC样本分布较为集中,提示系统误差小,实验结果可靠(图2)。
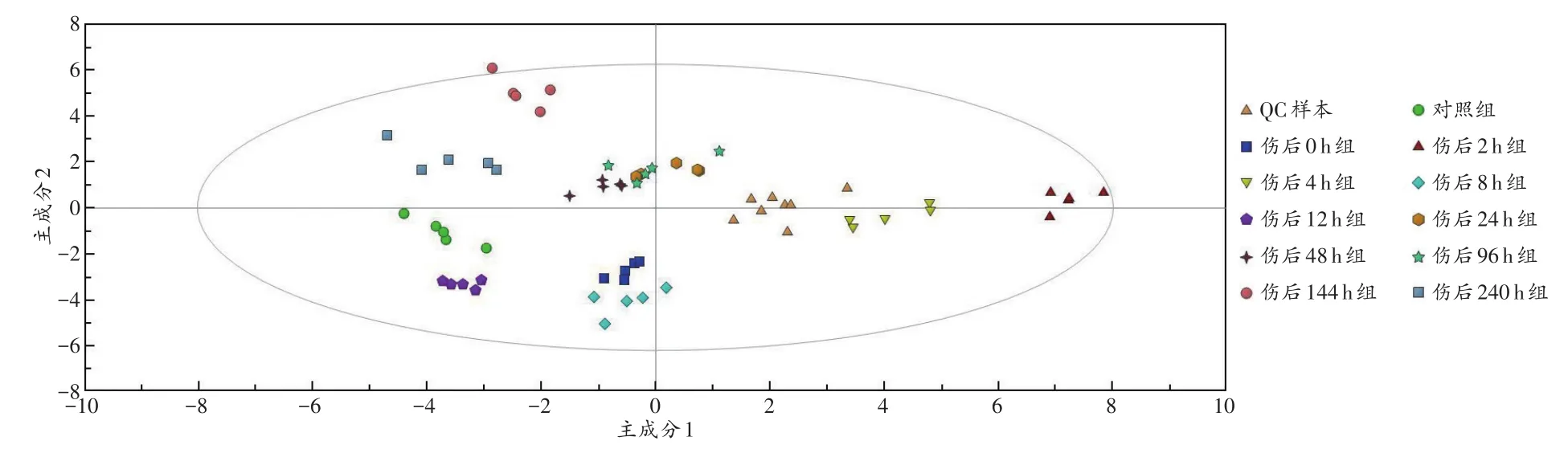
图2 PCA分析结果Fig.2 Results of PCA analysis
2.2.2 初步筛选生物标志物
将前处理后的所有样本进行GC-MS检测,获得实验组与对照组代谢产物的原始谱图(图3,实验组以伤后48h组为例),因图中色谱峰数量过多并有重叠现象,难以凭借肉眼区分谱图的差异。

图3 GC-MS检测原始谱图Fig.3 The original spectrum of GC-MS
将QC样本与NIST 14质谱库比对,结合HMDB数据库共选出60种内源性代谢产物。各实验组样本和对照组样本经数据预处理后,筛选出31种代谢产物,主要是酸类、糖类、脂类等物质(表1)。

表1 筛选出的代谢物及其保留时间Tab.1 Selected metabolites and their retention time(时间/s)
2.2.3 进一步筛选生物标志物
分别以PCA、PLS-DA、OPLS-DA 3种模式识别方法对各实验组与对照组、相邻时间实验组两两之间进行多元统计分析,结果模型均验证成功(图4~5,以对照组和伤后24 h组比较为例),且3种模式识别方法均能将各样本明显分离,其中OPLS-DA模式下R2、Q2值更接近1,提示该方法的识别预测能力最佳。故对全部实验组和对照组进行OPLS-DA分析,进一步筛选生物标志物。

图4 对照组和伤后24h组得分图Fig.4 Score plots of 24h after injury group and control group

图5 对照组和伤后24h组比较的模型置换验证结果Fig.5 Model replacement test in comparison of 24h after injury group and control group
在OPLS-DA模式下,以VIP>1.0且P<0.05为标准筛选出6种生物标志物,分别为单硬脂酸甘油酯、乙醇胺、缬氨酸、来苏糖、酪氨酸、棕榈酸。6种生物标志物的相对含量在骨骼肌挫伤后的变化见表2。单硬脂酸甘油酯含量在挫伤当时明显高于对照组,伤后4 h迅速下降至最低值,而后呈现逐渐上升趋势。乙醇胺含量在伤后2、24、96h出现3次峰值,在8、48h出现2次谷值。缬氨酸含量在挫伤当时低于对照组,后逐渐升高,至2h达第1个峰值后开始下降,至伤后12h降至谷值后开始上升,并于48h出现第2个峰值,于144h降至最低值后再次上升。来苏糖含量在挫伤当时高于对照组,随后开始下降,至8h达最低值,后续在24h和144h出现2次峰值,在48h出现一次谷值。酪氨酸含量在挫伤当时低于对照组,后上升,至4h达峰值后开始下降,于12h时降至最低值后再次上升,于96h时达最高峰。棕榈酸含量在伤后2h达到峰值,之后随时间延长出现波动,至伤后48h降至最低值。由上述结果可发现,6种生物标志物含量随时间变化的规律性欠佳,难以用于进行骨骼肌挫伤时间的推断。
2.3 SVM回归模型的建立
2.3.1 建立SVM回归模型
分别以31种和6种生物标志物数据为输入向量,实际损伤时间为输出向量进行SVM建模,训练集和测试集的预测结果见表3。发现相较于6种生物标志物数据,使用31种生物标志物数据作为输入向量时,训练集和测试集所识别的挫伤时间都更接近真实挫伤时间。
表2 实验组及对照组6种代谢物相对含量变化Tab.2 Changes of 6 metabolites in experimental group and control group[n=5,±s,浓度(/μmol/L)]

表2 实验组及对照组6种代谢物相对含量变化Tab.2 Changes of 6 metabolites in experimental group and control group[n=5,±s,浓度(/μmol/L)]
注:1)与对照组比较,P<0.05;2)与相邻上组比较,P<0.05。
组别对照伤后0h伤后2h伤后4h伤后8h伤后12h伤后24h伤后48h伤后96h伤后144h伤后240h单硬脂酸甘油酯45.54±8.15 164.24±1.281)35.00±2.571)2)33.39±4.531)37.86±4.111)38.57±1.83 47.85±4.922)36.78±6.281)2)41.53±7.30 50.26±8.702)52.88±4.701)乙醇胺41.13±3.03 43.90±5.04 66.57±1.441)2)59.51±2.941)2)43.50±2.862)48.33±1.341)2)64.80±4.951)2)47.94±1.881)2)71.56±4.611)2)67.59±2.391)58.15±3.411)2)缬氨酸366.30±5.98 280.29±5.141)587.08±5.761)2)501.08±14.811)2)312.29±8.561)2)221.17±11.431)2)276.72±22.701)2)781.10±21.311)2)612.30±12.511)2)128.41±12.481)2)629.76±16.111)2)来苏糖68.61±5.20 118.16±5.451)115.94±7.051)84.58±18.711)2)48.14±9.241)2)60.72±6.20 110.53±18.141)2)74.56±11.002)81.97±13.01 113.88±7.421)2)92.76±16.141)2)酪氨酸229.04±16.66 122.30±5.551)240.14±13.522)243.65±19.16 164.34±48.011)2)128.38±21.241)2)185.69±19.731)2)216.64±10.64 268.55±6.161)2)225.32±37.092)222.59±32.742)棕榈酸389.80±54.96 494.80±12.251)641.33±77.741)2)501.54±9.011)2)567.95±51.641)2)498.70±48.281)2)502.68±8.501)340.77±3.901)2)385.79±6.74 406.62±37.25 404.59±18.84
表3 SVM回归模型训练集和测试集的预测结果Tab.3 Prediction results of training set and test set of SVM regression model (±s,时间/h)
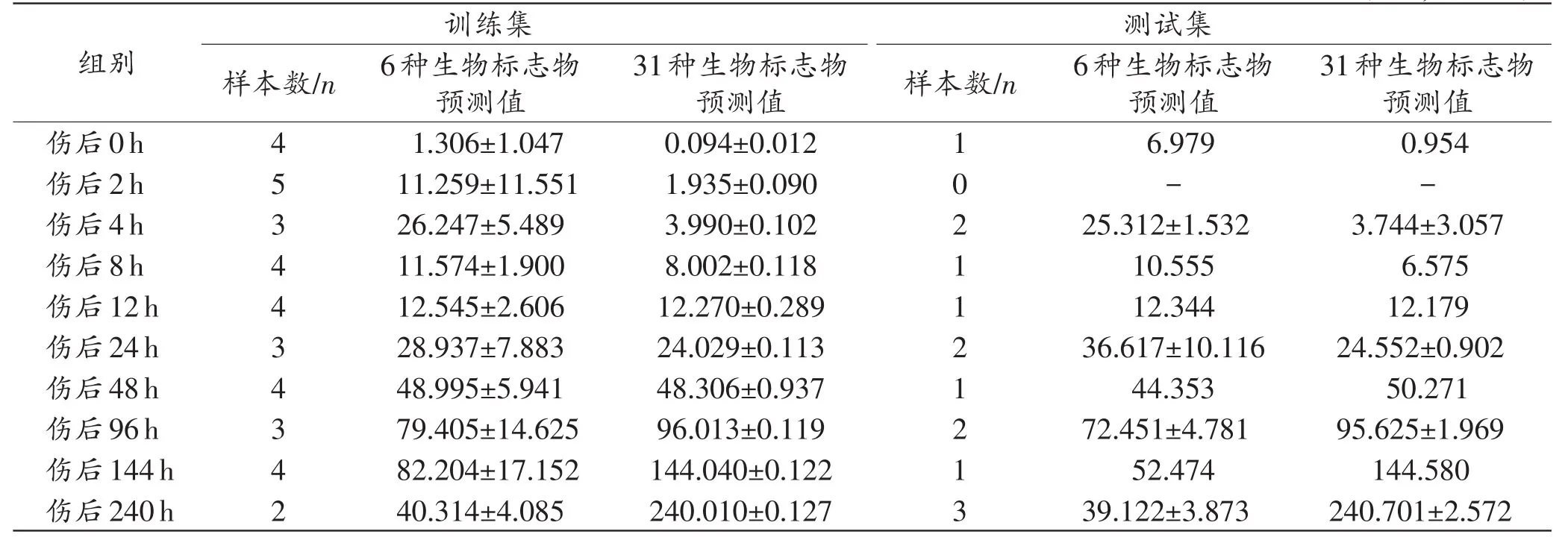
表3 SVM回归模型训练集和测试集的预测结果Tab.3 Prediction results of training set and test set of SVM regression model (±s,时间/h)
注:“-”表示无数据。
训练集样本数/n测试集样本数/n组别伤后0h伤后2h伤后4h伤后8h伤后12h伤后24h伤后48h伤后96h伤后144h伤后240h 4534434342 6种生物标志物预测值1.306±1.047 11.259±11.551 26.247±5.489 11.574±1.900 12.545±2.606 28.937±7.883 48.995±5.941 79.405±14.625 82.204±17.152 40.314±4.085 31种生物标志物预测值0.094±0.012 1.935±0.090 3.990±0.102 8.002±0.118 12.270±0.289 24.029±0.113 48.306±0.937 96.013±0.119 144.040±0.122 240.010±0.127 1021121213 6种生物标志物预测值6.979-25.312±1.532 10.555 12.344 36.617±10.116 44.353 72.451±4.781 52.474 39.122±3.873 31种生物标志物预测值0.954-3.744±3.057 6.575 12.179 24.552±0.902 50.271 95.625±1.969 144.580 240.701±2.572
2.3.2 SVM回归模型验证
将验证组(伤后192h组)血清样本的相应代谢物数据分别代入6种和31种生物标志物的SVM回归模型中进行骨骼肌挫伤时间推断。结果显示,以31种生物标志物建立的SVM回归模型的推断准确性较高(表4)。
表4 SVM回归模型验证结果Tab.4 Validation results of SVM regression model(n=5,±s,时间/h)

表4 SVM回归模型验证结果Tab.4 Validation results of SVM regression model(n=5,±s,时间/h)
注:RMSE为均方根误差。
RMSE 136.819 4.052生物标志物6种31种预测损伤时间55.344±7.485 195.781±1.629
3 讨 论
3.1 骨骼肌挫伤动物模型及GC-MS数据可靠性评价
建立骨骼肌挫伤动物模型的方法有多种,不同的方法造成骨骼肌挫伤的机制相同,但骨骼肌挫伤的程度并不一致,实际应用中以重物自由落体打击法应用较多。本研究在以往研究基础上通过研究自由落体打击装置所建立的骨骼肌挫伤模型,其病理学改变典型,适用于骨骼肌挫伤的相关研究。
在代谢组学研究中,仪器状态不稳定、操作误差等均会降低代谢组学数据的可靠性,所以有必要设置QC样本对仪器稳定性和数据可靠性进行检测[9]。本研究中QC样本由所有样本等比例混合所得,所含成分相同,在正常样本之间穿插进样,利用PCA模式对包括QC样本在内的所有样本进行分析,如果QC样本在PCA得分图上紧密聚集,则说明仪器稳定,代谢物数据可靠。本研究发现,QC样本分布较为集中,提示本研究所用仪器稳定,所获代谢物数据可靠,可用于后续分析。
3.2 生物标志物含量在骨骼肌挫伤时间推断中的应用价值
本研究应用OPLS-DA模式共筛选出单硬脂酸甘油酯、乙醇胺、缬氨酸、来苏糖、酪氨酸、棕榈酸6种生物标志物,其含量在骨骼肌挫伤后均随时间延长出现变化,但是并无明显规律,难以用于骨骼肌挫伤时间的推断。原因可能为OPLS-DA主要是基于线性回归的方法在不同类别之间建立数学模型,使不同类别的样本分离最大化,从而发现不同类别之间的差异性代谢物[10],而代谢组学数据并不能用简单的线性关系来表述[11]。综上,OPLS-DA模式能够较好地解决代谢组学研究中的分类问题,并能寻找出导致类别不同的生物标志物,但不能根据生物标志物的含量变化直接进行骨骼肌挫伤时间的推断。鲁翰霖[12]对大鼠挫伤后0.5、1、2 h的骨骼肌进行代谢通路分析,发现各对应时间点及损伤后2h内均有特征代谢差异物及持续表达的差异代谢产物,差异代谢物与三羧酸循环、糖酵解、蛋白质等多个代谢途径相关,参与了损伤早期骨骼肌抗氧化、清除坏死组织、能量供给、挫伤区蛋白质降解与生成等过程。本研究中,生物标志物为血清中脂类代谢物及氨基酸,其含量波动可能与损伤后能量代谢有关,相关代谢通路尚待进一步研究,以明确其含量发生差异性变化的代谢机制。
3.3 SVM回归模型在骨骼肌挫伤时间推断中的应用评价
本研究中,以6种生物标志物和31种生物标志物均成功建立了推断挫伤时间的SVM回归模型。根据6种生物标志物数据建立SVM回归模型时,在训练集、测试集中预测损伤时间与实际损伤时间差距均较大;对验证组大鼠的骨骼肌挫伤时间进行推断时结果为(55.344±7.485)h,与实际损伤时间(192 h)之间差值较大且RMSE值较高。根据31种生物标志物数据建立SVM回归模型时,在训练集、测试集中预测损伤时间与实际损伤时间差距均较小;对验证组大鼠的骨骼肌挫伤时间进行推断时结果为(195.781±1.629)h,与实际损伤时间(192 h)较为接近且RMSE值较低。以上结果提示,根据31种生物标志物数据建立的SVM回归模型的准确性远高于根据6种生物标志物数据建立的SVM回归模型的准确性,利用31种生物标志物数据建立的SVM回归模型用来进行骨骼肌挫伤时间推断的可靠性较高,其原因可能是31种代谢物的信息含量多于6种生物标志物的信息含量。顾嘉运等[13]发现,样本量达到一定值时继续增加样本量并不能增加SVM回归预测的准确度,本研究中特征量的多少与SVM回归预测准确度之间是否存在类似的关系,以及具体需要多少特征量以使SVM回归模型推断骨骼肌挫伤时间的能力达到最大化需后续进一步分析研究。近年来,许多研究[14-17]结果表明,大鼠骨骼肌损伤后相关蛋白的表达及炎症因子水平随损伤时间延长呈现时序性变化,有望用于损伤时间的推断,但随着损伤时间的延长,机体代偿、恢复,可能会出现同一表达水平对应多个时间点的情况。DU等[18]研究了大鼠骨骼肌挫伤后相关愈合基因的表达,构建预测模型并引入外部数据进行验证,结果显示基因表达模型和多变量分析有望用于损伤时间的推断。
本研究利用代谢组学和SVM回归模型对骨骼肌挫伤时间进行推断,为寻找、探究生物标志物用于推断骨骼肌挫伤时间提供了新的思路。但本研究发现,不同数量代谢物信息推断损伤时间差异较大,且验证组样本过少未能进一步评估预测模型的泛化能力,后续研究可进一步明确生物标志物参与的代谢通路、优化构建SVM回归模型的代谢物数量、引入多时间组的外部验证,以提高该方法推断骨骼肌挫伤时间的准确性和可行性。

