蔗糖转化酶SUC2在商品化酿酒酵母中的高水平表面展示
孙 大 禹, 周 海 龙, 杜 元 元, 李 宪 臻, 杨 帆
( 1.大连工业大学 生物工程学院, 辽宁 大连 116034;2.中国生物发酵产业协会, 北京 100833 )
0 引 言
菊糖是由多个D-呋喃果糖分子通过β-2,1-糖苷键连接而成的果糖聚合物,呈直链结构,其还原末端通过α-1,2糖苷键连有一个葡萄糖残基[1]。菊糖的聚合度一般在2~60,平均聚合度为10,水解后可生成少量葡萄糖和果糖[2-3]。菊糖作为贮存性果聚糖类碳水化合物,广泛存在于菊芋等植物中[4]。菊芋在我国的亩产可达3~5 t,是生产燃料乙醇的优质能源生物质[5]。
研究表明,仅有少数微生物能够直接利用菊糖生产乙醇,且其乙醇生产能力十分有限[6]。酿酒酵母(Saccharomycescerevisiae)作为生产乙醇的传统菌株,其乙醇生产能力强、易于培养且遗传操作平台成熟,成为转化菊糖产乙醇的首选微生物[7]。然而,绝大多数天然酿酒酵母菌株都不能有效将菊糖水解为可发酵糖[8],严重阻碍了菊糖基乙醇的工业化生产进程。研究发现,酿酒酵母KCCM50549在5 L发酵罐中,利用180 g/L菊芋粉(菊糖聚合度小于15)生产乙醇时的乙醇产量为36.2 g/L[9]。中科院青岛生物能源研究所微生物资源团队获得了一株温度耐受性菊糖代谢酿酒酵母菌株,该菌株在40 ℃下乙醇得率为79.7%,这是目前报道的非工程酿酒酵母中菊芋乙醇发酵的最高得率。
作为酿酒酵母水解菊糖的关键酶,蔗糖转化酶SUC2的表达水平直接影响酿酒酵母水解菊糖的能力[10]。本工作克隆获得酿酒酵母蔗糖转化酶SUC2的编码基因,并利用AGA1-AGA2锚定蛋白复合体将SUC2表面展示于商品化酿酒酵母菌株EBY100中,旨在为理性改造酿酒酵母使其高效转化菊糖产乙醇提供理论基础。
1 材料和方法
1.1 材料与仪器
S.cerevisiaeEBY100及酵母表面展示载体pYD1,美国英杰生命技术有限公司;E. coli DH10B及pMD18-T Simple Vector获赠于大连化学物理研究所赵宗保研究员课题组。酿酒酵母BY-BS是含有质粒pYC230-SUC2的酿酒酵母BY4741重组菌株,由本室前期构建并保藏。
PrimeSTAR HS DNA 聚合酶,250 bp DNA Marker,大连宝生物有限公司;DpnⅠ,NEB公司。
酵母发酵液体培养基YPD:葡萄糖20 g/L,酵母浸粉10 g/L,蛋白胨20 g/L,pH 6.0;固体YPD培养基:琼脂粉15.0 g/L,其余同YPD液体培养基;YNB-CAA液体培养基:YNB 6.7 g/L,酪蛋白氨基酸5 g/L,葡萄糖或半乳糖20 g/L;LB液体培养基:胰蛋白胨10.0 g/L,酵母提取物5.0 g/L,氯化钠10.0 g/L,pH 7.0;LB固体培养基:琼脂粉15.0 g/L,其余同LB液体培养基。
Eppendorf AG22331电击融合仪,德国艾本德股份有限公司;MD spectramax paradigm酶标仪,美谷分子仪器(上海)有限公司。
1.2 方 法
1.2.1 引物设计
蔗糖转化酶编码基因SUC2的克隆及表达载体的构建、鉴定所用引物均合成于大连宝生物有限公司,引物序列及功能如表1所示。
1.2.2 蔗糖转化酶编码基因的克隆
取摇床培养24 h的酿酒酵母新鲜发酵菌液,于4 ℃、4 000 r/min离心收集菌体,采用基因组抽提试剂盒提取基因组DNA并以之为模板进行蔗糖转化酶全长编码基因的扩增。基因扩增上游引物为SUC2-F,下游引物为SUC2-R。PCR反应体系:100 ng酿酒酵母基因组DNA,10 μL 5×PrimeSTAR buffer,20 μmol/L引物,2.5 mmol/L dNTPs,1.25 U PrimeSTAR HS DNA聚合酶,加水至总体积为50 μL。PCR反应条件:94 ℃ 5 min,94 ℃ 30 s,52 ℃ 45 s,72 ℃ 1.5 min,30个循环,72 ℃ 10 min,4 ℃结束反应。扩增产物用1%琼脂糖凝胶电泳检测,回收扩增产物。将回收的SUC2基因片段通过TA克隆连接至pMD18-T,构建质粒pT-SUC2并进行测序。

表1 SUC2引物序列及功能
1.2.3 pYD1-SUC2表面展示载体的构建
采用RF克隆策略(Restriction-free cloning)将SUC2基因连接到表面展示载体pYD1上。RF I反应所用引物为pYD-SF和pYD-SR,以质粒pT-SUC2为模板,反应条件:94 ℃ 5 min,94 ℃ 30 s,52 ℃ 45 s,72 ℃ 1.5 min,30个循环,72 ℃ 10 min,4 ℃结束反应。回收试剂盒对RFI扩增产物进行回收。RF Ⅱ反应体系:280 ng RF Ⅰ回收产物,100 ng pYD1,5 μL 5×PrimeSTAR buffer,2.5 mmol/L dNTPs,1.25 U PrimeSTAR HS DNA聚合酶,加水至总体积为25 μL。RF Ⅱ反应条件:94 ℃ 5 min,94 ℃ 1 min,65 ℃ 1 min,68 ℃ 10 min,15个循环(每个循环退火温度降低1℃),随后,94 ℃ 1 min,55 ℃ 1 min,68 ℃ 10 min,20个循环,68 ℃ 15 min,4 ℃结束反应。
取8.5 μL RF Ⅱ反应产物,加入1.5 μL 10 U/μLDpnⅠ,37 ℃消化6 h以上降解模板质粒。取3 μL消化产物电击转化至E.coliDH10B中,转化菌液涂布于含终浓度100 μg /mL氨苄青霉素的LB平板,挑选阳性克隆进行酶切验证。酶切体系:0.5 μg质粒DNA,0.5 μLXbaⅠ,1 μL 0.1%×BSA,1 μL 10×M buffer,加水至体积为10 μL,37 ℃温育2 h,1%琼脂糖凝胶电泳对酶切产物进行分析。酶切鉴定正确的质粒送至上海生工测序。测序正确的质粒命名为pYD1-SUC2。
1.2.4 表面展示蔗糖转化酶SUC2的重组酿酒酵母的构建
将鉴定正确表达质粒pYD1-SUC2电击转化至S.cerevisiaeEBY100感受态细胞中,电压为1.5 kV。电击结束后转化菌液加入1 mL山梨醇(1 mol/L),30 ℃温育2 h后将转化菌液涂布于色氨酸选择性筛选平板上,30 ℃培养至出现转化子。挑取阳性转化子提取质粒DNA并进行PCR鉴定,正确的重组酿酒酵母命名为EBY-S。
1.2.5 表面展示有SUC2的酿酒酵母重组菌株酶活测定
重组菌EBY-S在YNB-CAA液体培养基中于20 ℃、200 r/min摇床培养96 h。不同时间点取样测定菌体表面展示SUC2的蔗糖酶活和菊糖酶活。酶活测定方法:1 mL发酵菌液5 000g、4 ℃ 离心10 min收集菌体,重悬于1 mL 0.1 mol/L PBS缓冲溶液(pH 6.8)。取50 μL重悬菌液与450 μL菊糖或蔗糖底物(20 g/L,pH 5.0)混匀,50 ℃水浴反应15 min。采用DNS法测定反应释放的还原糖。菊糖酶及蔗糖酶酶活定义:在pH 5.0、50 ℃的反应条件下,每分钟水解菊糖或蔗糖释放1 μmol葡萄糖或果糖所需的酶量为1 U。对照菌株分别为本实验室前期构建的SUC2分泌表达菌株BY-BS和含有空pYD1载体的酿酒酵母EBY-C。
2 结果与讨论
2.1 蔗糖转化酶编码基因SUC2的克隆
以酿酒酵母基因组DNA为模板,PCR扩增蔗糖转化酶编码基因SUC2,琼脂糖凝胶电泳结果如图1所示。在1 500 bp附近扩增出一条特异性条带,大小与蔗糖转化酶目的基因片段一致。将回收的PCR产物送至上海生工测序,测序结果经NCBI数据库比对证明为蔗糖转化酶的编码基因,大小为1 592 bp,该基因编码的蔗糖转化酶含有533个氨基酸残基,理论分子质量为60.6 ku。该蔗糖转化酶的N端含有一段大小为20 aa的分泌信号肽,其催化结构域隶属于糖苷水解酶32c家族。
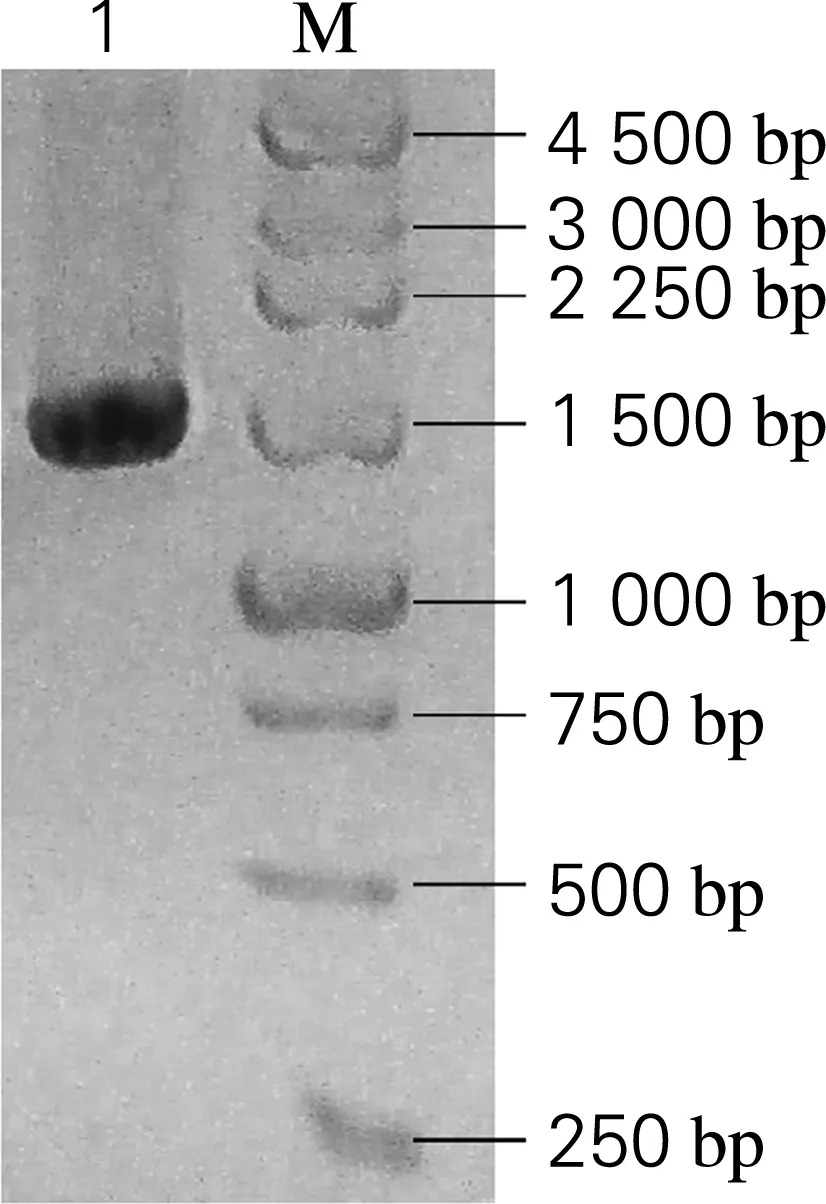
1, SUC2基因目的条带;M, DNA marker
2.2 SUC2表面展示表达载体及菌株的构建
利用RF克隆策略将SUC2基因连接到表面展示载体pYD1上,连接产物转化至E.coliDH10B感受态细胞中。挑取阳性克隆进行质粒提取,XbaⅠ酶切验证。电泳结果如图2所示,1号重组质粒经酶切后显示有大小为1 800和4 700 bp的两条特异性条带,与预测正确重组质粒的酶切结果基本一致。将酶切鉴定结果正确的质粒送上海生工测序,结果表明本工作成功构建了蔗糖转化酶表达载体pYD1-SUC2。将表达载体pYD1-SUC2转化至S.cerevisiaeEBY100感受态细胞中,30 ℃静置培养,挑取阳性克隆提取重组质粒并进行PCR鉴定。电泳结果如图3所示,重组质粒经酶切后显示有大小为1 800 bp的特异性条带,与理论PCR结果基本一致。该结果表明,成功构建了表面展示蔗糖转化酶的重组酿酒酵母EBY-S。
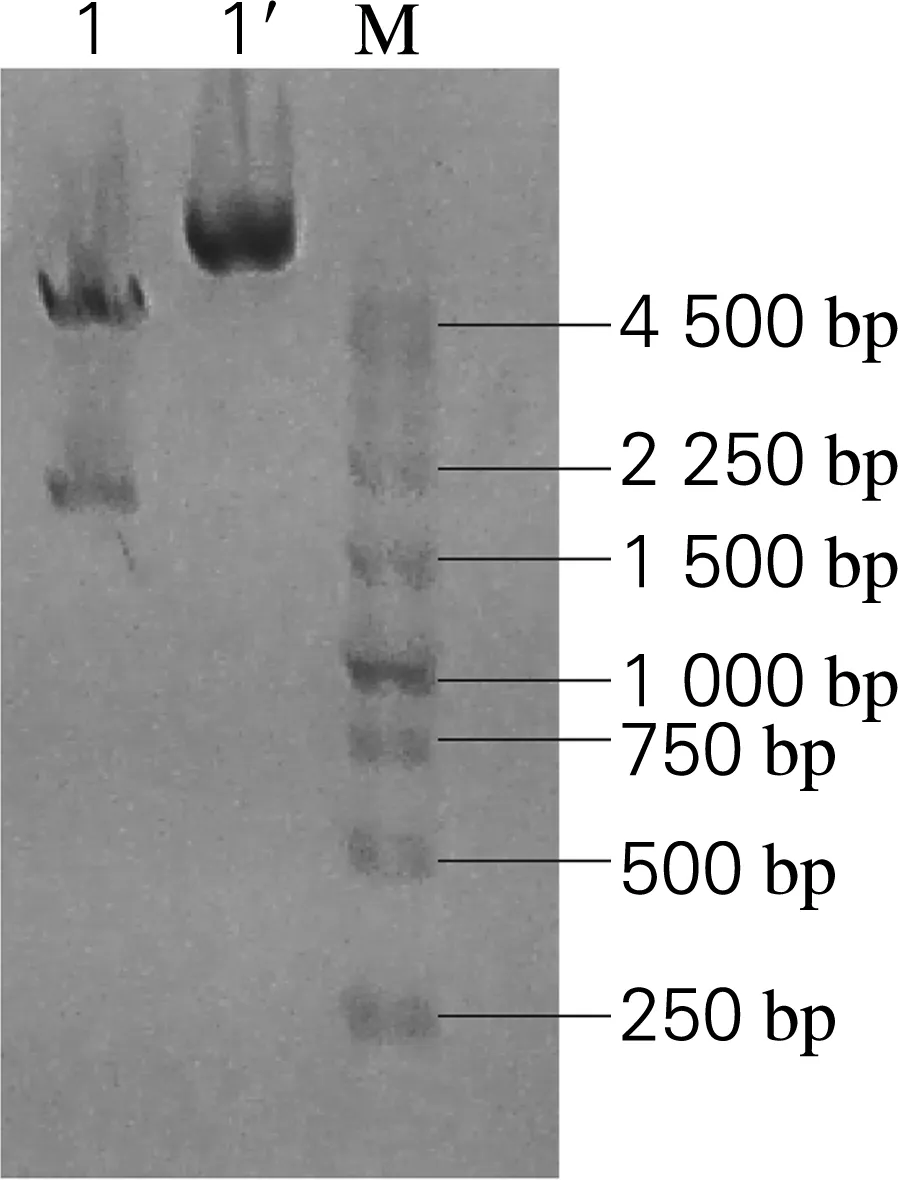
1, 重组质粒酶切条带; 1′, 重组质粒; M, DNA marker
图2 重组质粒pYD1-SUC2的XbaⅠ酶切验证结果
Fig.2 The electrophoresis results of the recombinant plasmid pYD1-SUC2 digested byXbaⅠ
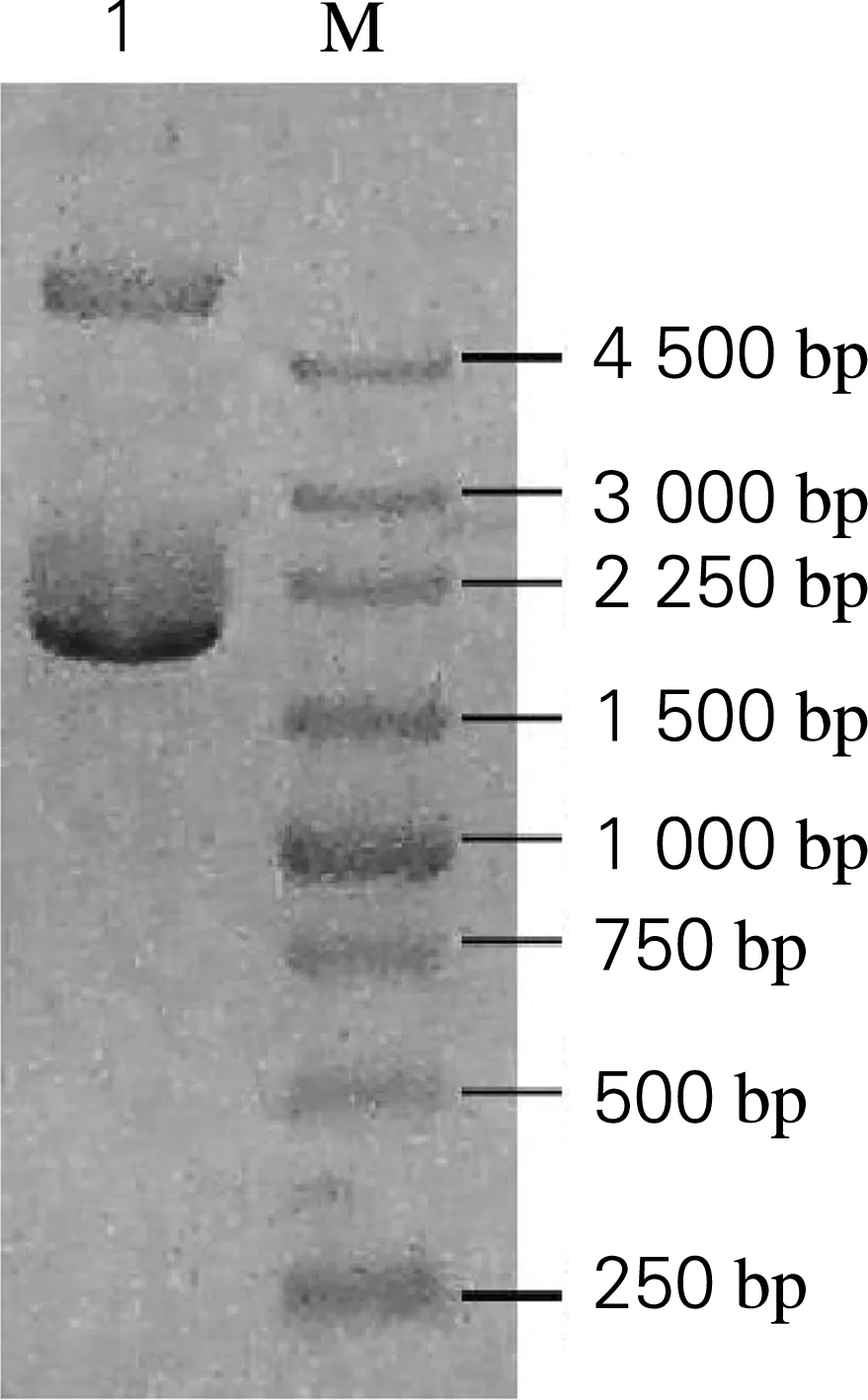
1, PCR鉴定结果; M, DNA marker
图3 转化有pYD1-SUC2的酿酒酵母重组菌株EBY-S的鉴定结果
Fig.3 PCR identification of EBY-S transformed with pYD1-SUC2
2.3 表面展示蔗糖转化酶SUC2的酿酒酵母重组菌株最佳产酶时间的确定
将表面展示蔗糖转化酶SUC2的酿酒酵母重组菌EBY-S在含有2%半乳糖的YNB-CAA液体培养基中诱导表达96 h,每12 h取样,结果如图4所示。随着发酵时间的延长,细胞表面逐渐展示出蔗糖酶活力,72 h达到最高值241.6 U/mL,之后发酵菌液细胞表面SUC2酶活力逐渐下降。因此,EBY-S重组菌表面展示SUC2的最佳发酵时间为72 h。
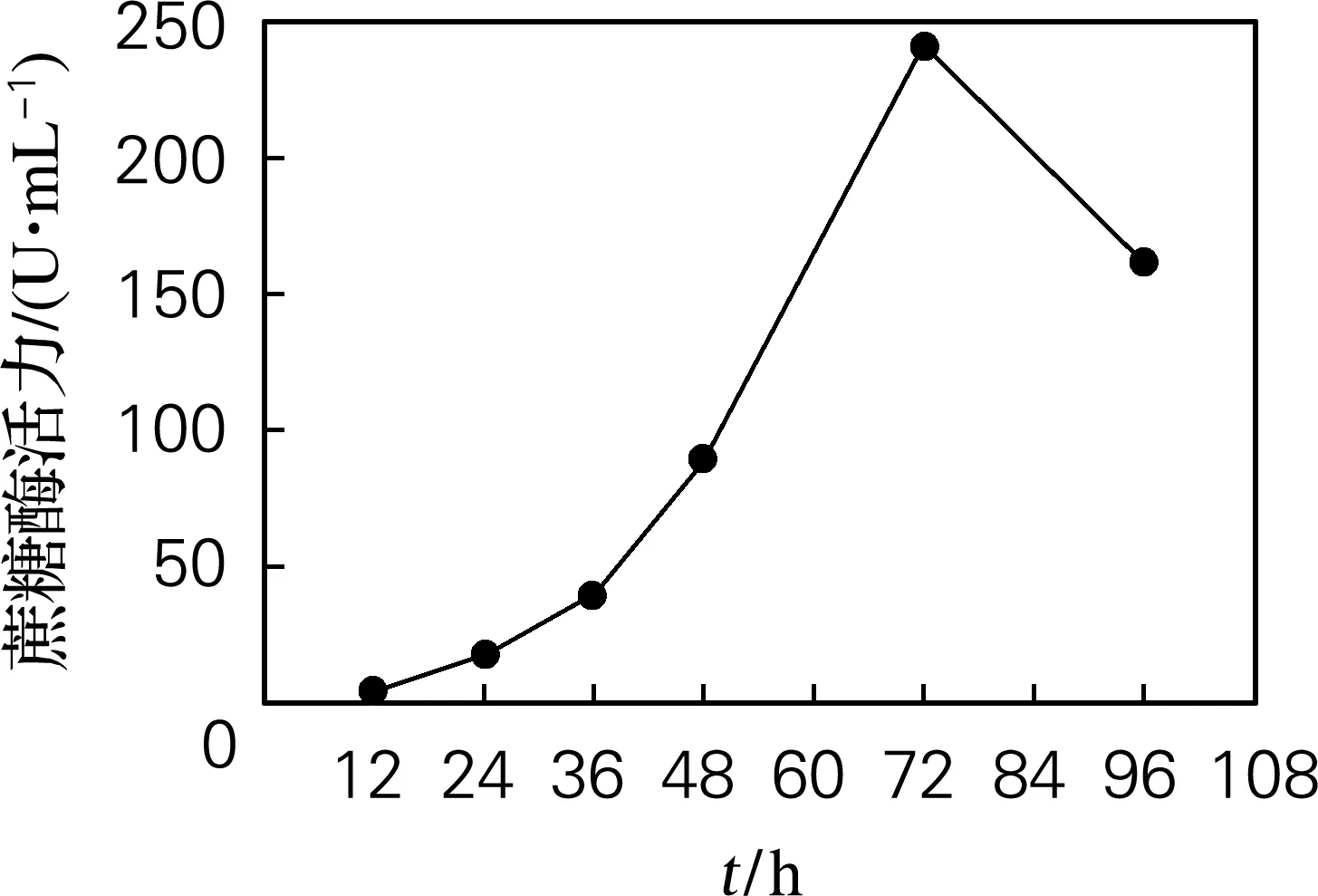
图4 重组菌株EBY-S蔗糖酶的最佳发酵时间
2.4 表面展示SUC2的酿酒酵母重组菌株酶活力分析
如图5所示,在最适发酵时间条件下,EBY-S重组菌菌体表面展示SUC2的蔗糖酶酶活力为241.6 U/mL,菊糖酶酶活力为9.4 U/mL,显著高于阴性对照菌株EBY-C。说明SUC2能够借助锚定蛋白复合体AGA1-AGA2在EBY-100细胞表面成功展示表达。
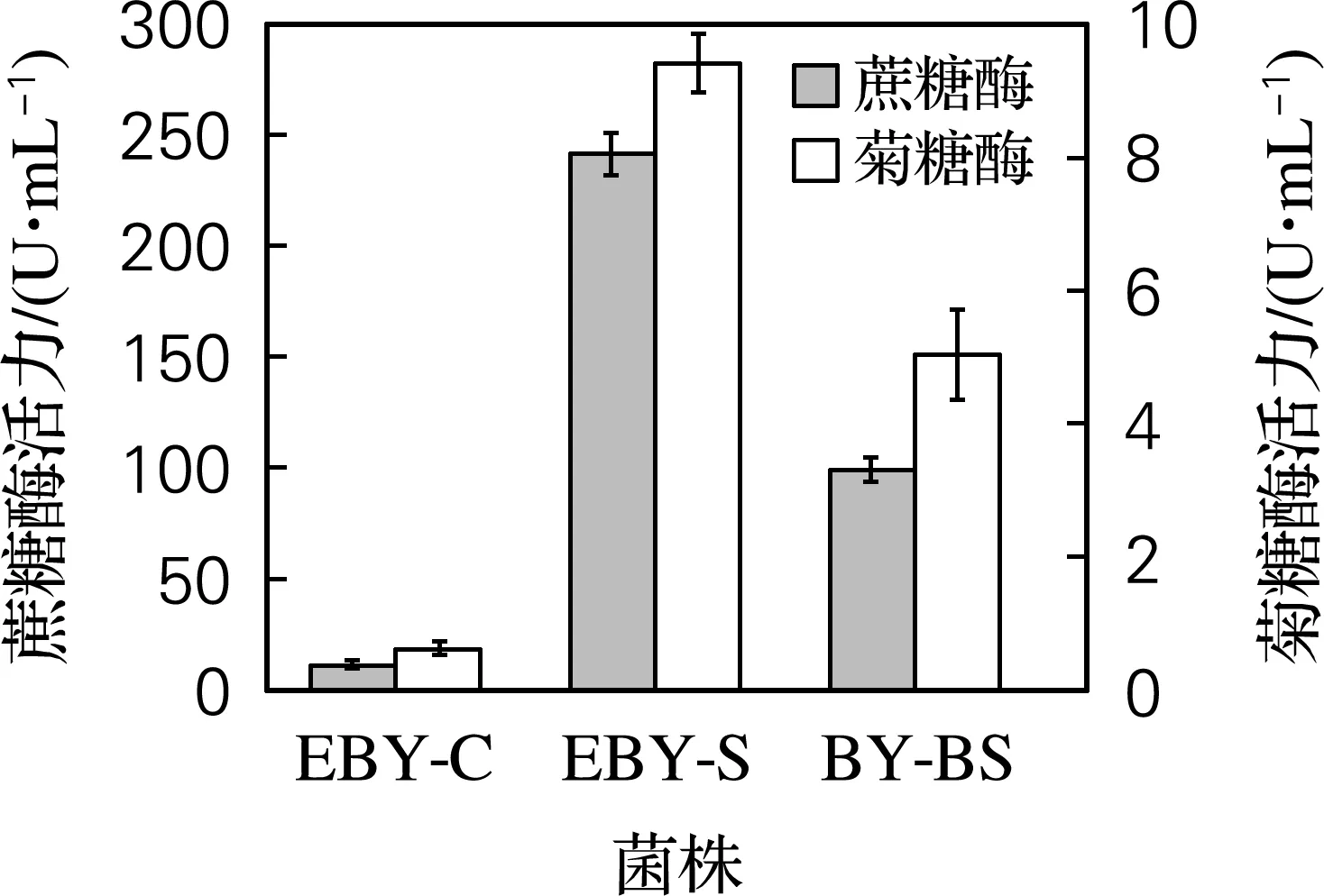
图5 SUC2酿酒酵母重组菌株酶活力分析
潘志友等[11]将脂肪酶B表面展示于酿酒酵母,产物乙酸乙酯的产率提高到98%。唐语谦等[12]将人源蛋白酶体α亚基6(α6)成功地展示于酿酒酵母中,结果显示人源蛋白酶体α亚基6(α6)有很好的抗原性和特异性。本课题组前期工作中,将SUC2分泌表达在酿酒酵母BY4741中,获得重组菌株发酵液上清的蔗糖酶及菊糖酶活力分别为100和5 U/mL[10]。本实验中,当SUC2成功的展示于酿酒酵母上后,EBY-S重组菌菌体表面蔗糖酶活力是BY-BS的2.4倍,菊糖酶活力是BY-BS的1.9倍。结果表明,较之分泌表达技术,表面展示技术能够显著的提高酶在酿酒酵母中的酶活力。
3 结 论
本工作以酿酒酵母为出发菌株,从中克隆出蔗糖转化酶SUC2编码基因,并通过融合锚定蛋白AGA2实现其在酿酒酵母细胞表面的展示表达。重组菌在发酵72 h时,SUC2能够通过锚定蛋白复合体AGA1-AGA2成功地表面展示于酿酒酵母细胞壁,且相对其他发酵时间表现出最高的表面展示水平,酶活力达241.6 U/mL。同时,相对于SUC2分泌表达型重组菌,SUC2表面展示菌株具有更高的蔗糖酶活力及菊糖酶活力。说明酿酒酵母表面展示系统对于提高菌株SUC2表达水平及酶活力具有很好的效果。

