不同环境下稻米品质性状QTL的检测及稳定性分析
王小雷 刘杨 孙晓棠 欧阳林娟 潘锦龙 彭小松 陈小荣 贺晓鹏 傅军如 边建民 胡丽芳 徐杰 贺浩华 朱昌兰
不同环境下稻米品质性状QTL的检测及稳定性分析
王小雷#刘杨#孙晓棠 欧阳林娟 潘锦龙 彭小松 陈小荣 贺晓鹏 傅军如 边建民 胡丽芳 徐杰 贺浩华*朱昌兰*
(江西农业大学作物生理生态与遗传育种教育部重点实验室, 南昌 330045;#共同第一作者;*通信联系人, E-mail: wxl0vip@163.com; zhuchanglan@163.com)
【目的】挖掘新的稻米品质性状QTL并利用分子育种技术改良稻米品质。【方法】利用构建的一套以籼稻香型恢复系昌恢121为背景亲本,以优质粳稻越光为供体亲本的染色体片段代换系(CSSLs)为材料,在4个环境下对稻米品质性状进行QTL检测及稳定性分析。【结果】在4个环境下共检测到44个QTL,其中6个QTL能在多个环境下被检测到;第2、3、5、6和10染色体上存在多效QTL簇,对稻米品质性状具有明显的调控作用;第1、6和12染色体上7个QTL能在不同环境下稳定表达。【结论】第1染色体RM3143−RM1117区间和第12染色体RM3331−RM5479区间是两个新的稳定表达QTL。
水稻;生态环境;品质性状;稳定性分析
水稻是世界近半数人口的主粮作物,也是我国种植面积最大的粮食作物之一。长期以来关于水稻的研究主要集中在产量的提高,而在稻米品质方面却不尽人意。随着人们生活条件的不断提高,人们对稻米品质要求也越来越高,为此,培育优质水稻显得愈加重要。
稻米品质性状容易受环境影响,多属于数量性状,遗传基础十分复杂。目前已经报道了一些稻米品质性状的QTL位点。Chen等[1]以籼稻PYZX和粳稻P02428自交系群体为材料,利用基于高通量基因测序分型技术构建高密度遗传图谱,结果发现影响垩白粒率的重要差异表达基因位于第5、7和8染色体上的QTL簇区。Gao等[2]利用培矮64S和9311构建的重组自交系,在第1、4、6、7、9和12染色体上鉴定出19个QTL与垩白有关,占表型变异的5.1%~30.6%。Xu等[3]以籼稻/粳稻构建的重组自交系为材料,在四个典型的水稻栽培区进行研究,共检测到19个与硬度、黏性和弹性有关的QTL。根据前人研究[4-8],在12条染色体上都检测到控制蛋白质含量的QTL,其中第6和第8染色体在不同群体中能被重复定位到;控制胶稠度的QTL广泛分布在各染色体上[9-13],第5、10、12染色体中检测出的较少;控制RVA谱特征值的QTL分布在12条染色体上[14-18],主要分布在第2、6、7、8和10染色体上。目前,通过构建不同群体和运用测序技术,大量的品质性状QTL被检测到,但仅有少数品质性状QTL被克隆,如[19]、[20]、[21]、[22]、[23]、[24]、[25]、[26][27][28]。因此,需要通过构建不同遗传群体,挖掘更多稻米品质性状相关QTL,再通过分子聚合育种培育高产优质的品种。
本研究利用香型恢复系籼稻昌恢121为背景亲本和优质粳稻越光为供体亲本构建染色体片段代换系,在2015年江西南昌、2016年海南和江西南昌、2017年海南4个环境下对稻米品质性状进行QTL分析,以期阐明不同环境下稻米品质性状的表达规律,挖掘新的稳定表达的主效QTL位点,为优质水稻育种提供理论基础和基因资源。
1 材料与方法
1.1 实验材料与田间实验
以香型籼稻昌恢121为背景亲本、以粳稻越光为供体亲本构建染色体片段代换系,该群体包含208个BC3F8、BC4F7和BC5F6株系,从中挑选覆盖全基因组的59个株系材料用于品质鉴定。分别于2015年在江西南昌(E1)、2016年海南(E2)、江西南昌(E3)和2017年海南(E4)等4个环境下进行试验。种植行株距16.7 cm×18.7 cm,常规田间管理。
1.2 性状鉴定
成熟后对每个株系进行混收,分别对垩白粒率、胶稠度、蛋白质含量、RVA谱和糊化温度进行测定。垩白粒率的测定按照中华人民共和国国家标准GB/T17891−1999的方法进行。按照国家标准GB/T 17891−1999胶稠度的测定方法,测定样品的胶稠度。用万分之一电子分析天平准确称取0.5000 g精米粉,利用FOSS-2300型全自动凯氏定氮仪,按半微量凯氏法测定精米粉全氮含量,按系数5.95换算成蛋白质含量。稻米淀粉黏滞特性采用澳大利亚Newport Scientific仪器公司生产的TecMaster型RVA仪测定,用TCW(thermal cycle for Windows)配套软件进行数据分析;按照美国谷物化学协会规程和RACI标准方法测定。每份样品重复测定2次,取其平均值。
1.3 遗传图谱构建
齐穗期采集亲本和CSSL群体各家系叶片,采用CTAB法抽提基因组DNA。PCR扩增体系10 µL,包括DNA模板2.0 µL,10 µmol/L引物各0.5 µL,7 µL PCR混合体系(5.7 µL ddH2O;1 µL 10×缓冲液;0.2 µL dNTPs;0.1 µL酶)。扩增程序如下: 94℃下5 min,94℃下30 s,55℃~58℃下30 s,72℃下30 s,35个循环;72℃下10 min,4℃下保存。PCR扩增产物用6%~8%的琼脂糖凝胶电泳检测。利用两亲本有多态性的标记构建遗传图谱,遗传图谱包含139个标记,覆盖12条染色体,图谱覆盖基因组约1455.1 cM,标记平均距离10.5 cM。
1.4 数据统计及QTL分析
采用Microsoft Excel 2003和SPSS 17.0进行统计分析。利用QTL IciMapping 4.1软件,通过复合区间作图法进行QTL定位分析,若LOD≥2.5时,则认为对应区间存在目标性状的QTL。QTL命名遵循McCouch等[29]的原则,利用双样本等方差测验分析被检测QTL对应代换系相应表型与背景亲本昌恢121之间的差异显著性,验证目标QTL在不同环境中的效应显著性,如果在多个环境中差异都达到显著水平,则说明该QTL能够稳定表达。
2 结果与分析
2.1 亲本及CSSL群体品质性状的表型变异
在不同环境中昌恢121与越光两亲本内的各性状变异幅度不大。2016年江西南昌越光的垩白粒率非常高是由于扬花期遇高温。两亲本蛋白质含量不存在明显差异。在糊化和RVA谱特性方面,越光的胶稠度、崩解值及成糊温度高于昌恢121,而热浆黏度、冷浆黏度、消减值、回复值均低于昌恢121。测定的11个性状在群体中均呈连续分布,呈双向超亲分离现象,近似于连续的正态分布,符合QTL作图的要求(表1)。
2.2 各品质性状之间的相关性分析
不同栽培环境条件下,该染色体片段代换系群体的稻米品质性状之间呈显著或极显著相关,有些品质性状间的相关性在4个环境下方向一致,不同环境之间的相关性系数存在明显差异。其中,垩白粒率与蛋白质含量在4个环境中存在负相关;胶稠度与热浆黏度、冷浆黏度、消减值和回复值之间在4个环境中均达到显著或极显著负相关,与崩解值z在4个环境中均表现为极显著正相关;蛋白质含量与最高黏度、热浆黏度在两个环境中达到显著负相关,与成糊温度在4个环境中均达到显著或极显著正相关(表2)。
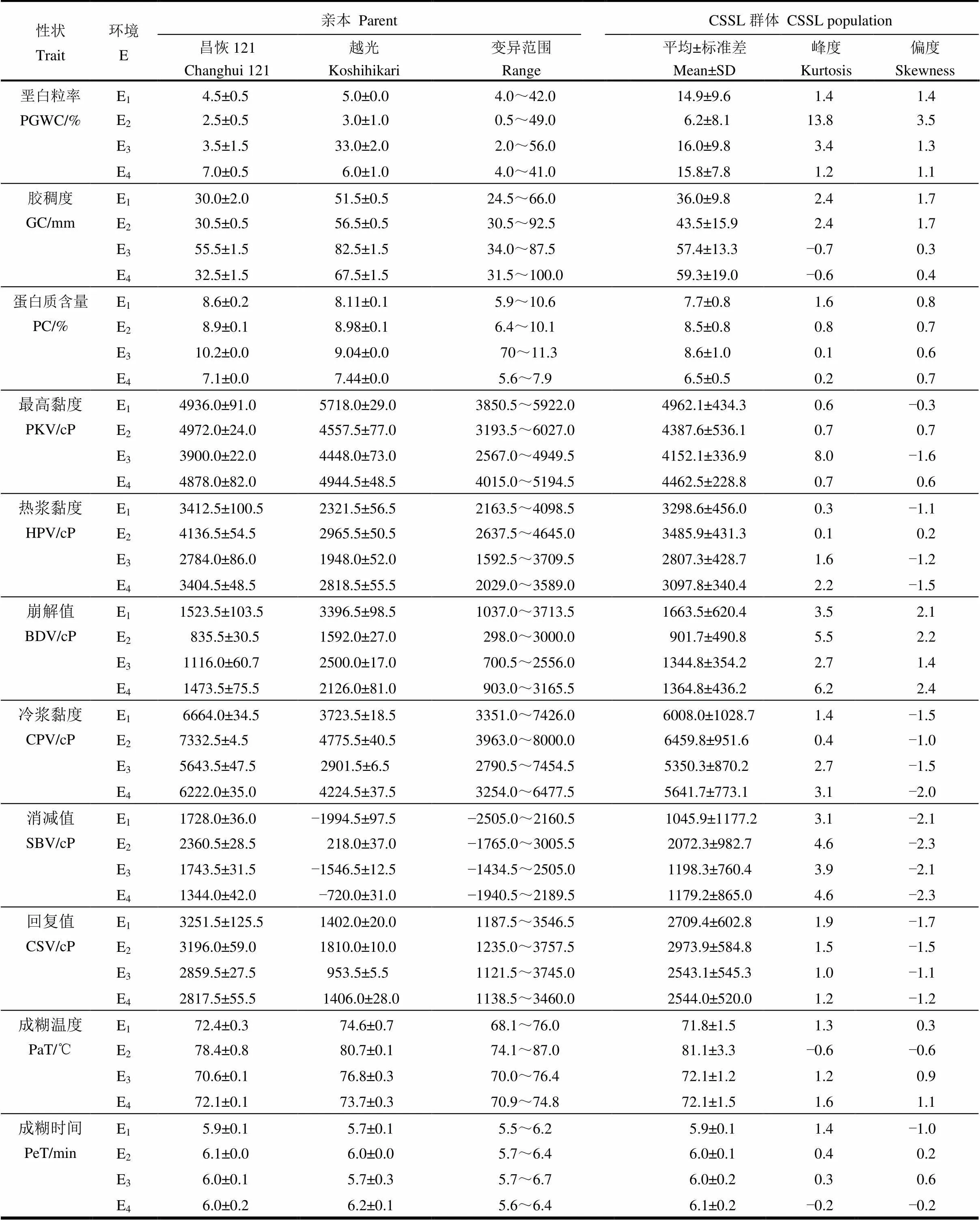
表1 亲本及CSSLs群体在4个环境中的稻米品质性状
E1–江西南昌(2015年); E2–海南(2016年); E3–江西南昌(2016年); E4–海南(2017年)。
PGWC, Percentage of grains with chalkiness; GC, Gel consistency; PC, Protein content; PKV, Peak viscosity; HPV, Trough viscosity; BDV, Breakdown viscosity; CPV, Final viscosity; SBV, Setback viscosity; CSV, Consistence viscosity; PaT, Pasting temperature; PeT, Peak time.
E1, Nanchang, Jiangxi Province(2015); E2, Hainan Province(2016); E3, Nanchang, Jiangxi Province(2016); E4, Hainan Province (2017).
2.3 稻米品质性状的QTL分析
共检测到稻米品质性状的QTL 44个(表3、4),在4个环境下分别检测到15、16、13和20个,分布于12条染色体上(图1),LOD值介于2.71~56.05,表型贡献率变幅为1.03%~94.34%,其中4个QTL能在2个环境中被同时检测到;6个QTL能在3个及以上环境中被同时检测到,其他35个QTL仅能在单一环境中检测到。
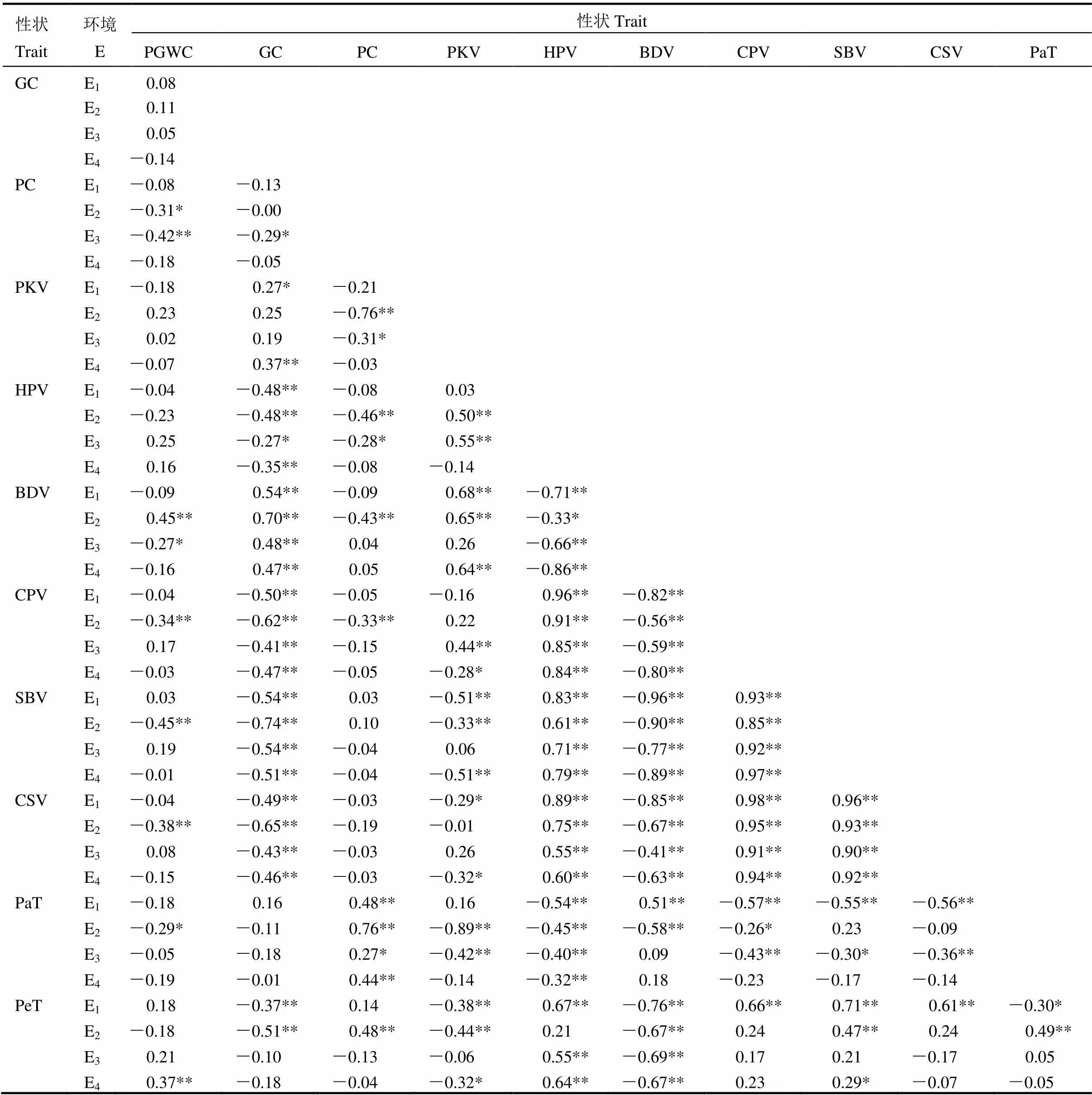
表2 昌恢121/越光CSSLs群体在4个环境中稻米各品质性状间的相关系数
*和**指相关分别达到0.05和0.01显著水平。E1–江西南昌(2015年); E2–海南(2016年); E3–江西南昌(2016年); E4–海南(2017年)。
PGWC–垩白粒率; GC–胶稠度; PC–蛋白质含量; PKV–最高黏度; HPV–热浆黏度; BDV–崩解值; CPV–冷浆黏度; SBV–消减值; CSV–回复值; PaT–成糊温度; PeT–成糊时间。
*and** indicate significant correlation at 0.05 and 0.01 levels, respectively. E1, Nanchang, Jiangxi Province(2015); E2, Hainan Province(2016); E3, Nanchang, Jiangxi Province(2016); E4, Hainan Province(2017). PGWC, Percentage of grains with chalkiness; GC, Gel consistency; PC, Protein content; PKV, Peak viscosity; HPV, Trough viscosity; BDV, Breakdown viscosity; CPV, Final viscosity; SBV, Setback viscosity; CSV, Consistence viscosity; PaT, Pasting temperature; PeT, Peak time.
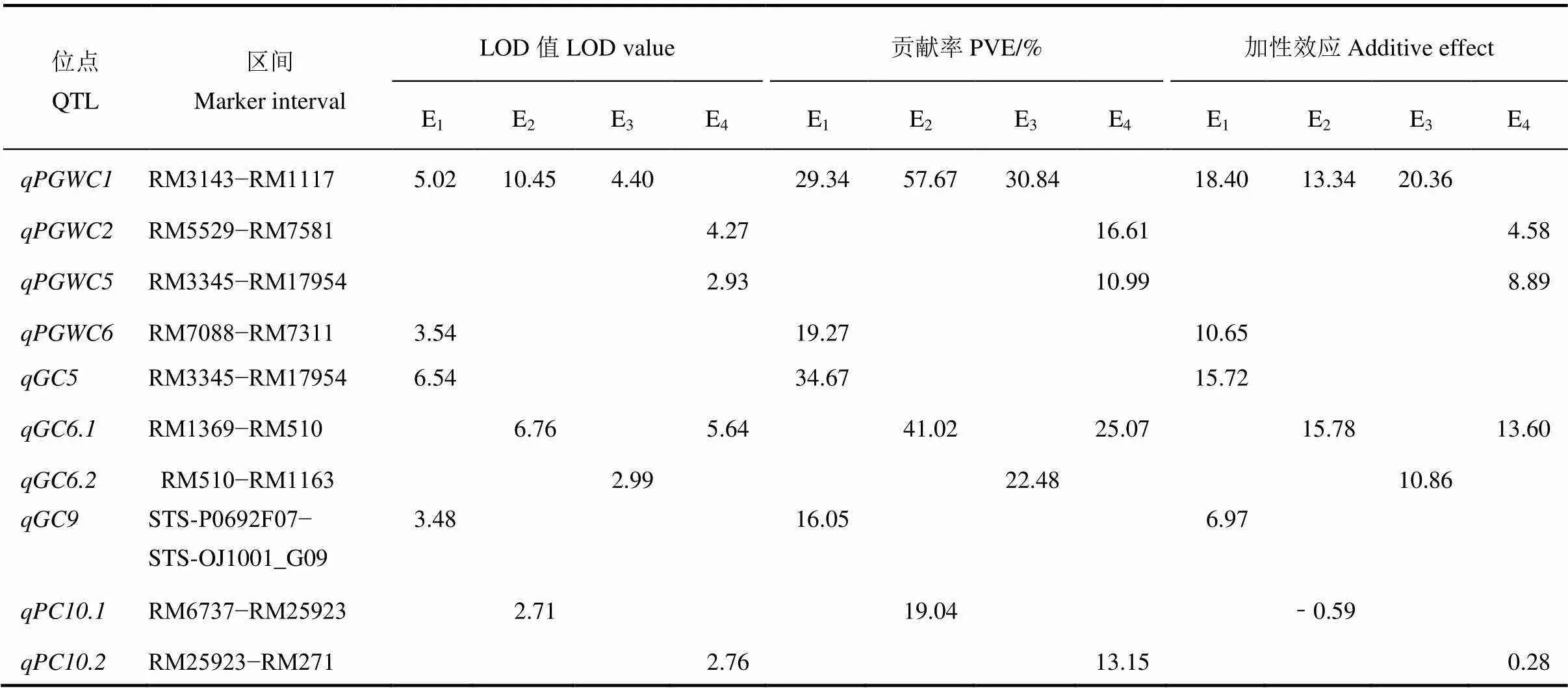
表3 昌恢121/越光CSSLs群体垩白粒率、胶稠度和蛋白质含量QTL分析
E1–江西南昌(2015年); E2–海南(2016年); E3–江西南昌(2016年); E4–海南(2017年)。
E1, Nanchang, Jiangxi Province(2015); E2, Hainan Province(2016); E3, Nanchang, Jiangxi Province(2016); E4, Hainan Province(2017).
PVE, Percentage of phenotypic variance explained.
2.3.1 垩白粒率
共检测到4个控制垩白粒率的QTL,分布于第1、2、5和6染色体上,其中在3个环境中都被检测到,表型贡献率分别为29.34%、57.67%和30.84%,该位点在越光中表现为正向加性效应。、和仅在一个环境中检测到,表型贡献率分别为16.61%、10.99%和19.27%,效应值分别为4.58、8.89和10.65,表明正向加性效应等位基因均来自于供体亲本越光。
2.3.2 胶稠度
共检测到4个控制胶稠度的QTL,分布于第5、6和9染色体上,其中第6染色体上检测到两个,在两个环境中被检测到,表型贡献率分别为41.02%和25.07%,效应值分别为15.78和13.60,说明该位点在越光中表现为正向加性效应。和仅能在一个环境中检测到,表型贡献率在16.05%~34.67%之间,效应值分别为15.72、10.86和6.97,这些QTL都表现为正向加性效应且等位基因均来自于供体亲本越光。
2.3.3 蛋白质含量
检测控制蛋白质含量的QTL共有2个,、表型贡献率分别为19.04%、13.15%,都位于第12染色体上,两者都是在海南检测到,而在江西南昌未能检测到,说明这些QTL可能易受到环境的影响。
2.3.4 RVA谱特征值
共检测到34个与RVA谱特征值相关的QTL,分布于除第9染色体外的11条染色体上,LOD值变化范围为2.70~56.05,表型贡献率变幅为1.03%~94.34%。
6个与PKV相关QTL,分别位于第1、3、6、10和11染色体;3个与HPV相关QTL,分别位于第3、6和8染色体;3个与BDV相关QTL,分别位于第3、5和6染色体;4个与CPV相关QTL,分别位于3、6和8染色体;6个与SBV相关QTL,分别位于2、3、5、6和8染色体;3个与CSV相关QTL,分别位于3、5和6染色体;5个与PaT相关QTL分别位于4、6、10和12染色体;4个与PeT相关QTL分别位于6和7染色体。其中第6染色体RM1369−RM510区间的、、、、在4个不同的环境中都能被重复检测到,表型贡献率分别为56.27%、65.17%、70.45%、85.20%、71.57%。
从QTL在染色体上的分布来看,控制RVA特征值的衍生数据BDV、SBV、CSV的QTL主要分布于第3、5、6染色体上。控制HPV,CPV的QTL主要分布在第3、6、8染色体上。而控制PKV、PaT、PeT的QTL均匀分布在各染色体上,重复检测到的QTL仅有,,,RVA谱特征值相关QTL的不稳定遗传说明了RVA谱特征值遗传在不同环境中的复杂性。
2.5 稻米品质性状相关QTL的稳定性分析
多个稻米品质性状QTL能在不同环境下检测到,其中包括、和。在4个环境中对稻米品质性状进行QTL分析,其中在3个环境中都能被重复检测到。定位于RM3143−RM1117区段内,对应的染色体片段代换系为X15,其垩白粒率与昌恢121在4个环境中表型差异均达到极显著水平(图2)。连续在2个环境下均能被重复检测到。定位于RM3331−RM5479区间,对应的染色体片段代换系为X125,其成糊温度与昌恢121连续在2个环境下表型差异达到极显著水平(图2)。利用X15与X125分别验证了与的加性效应,并说明了这2个QTL的环境稳定性。
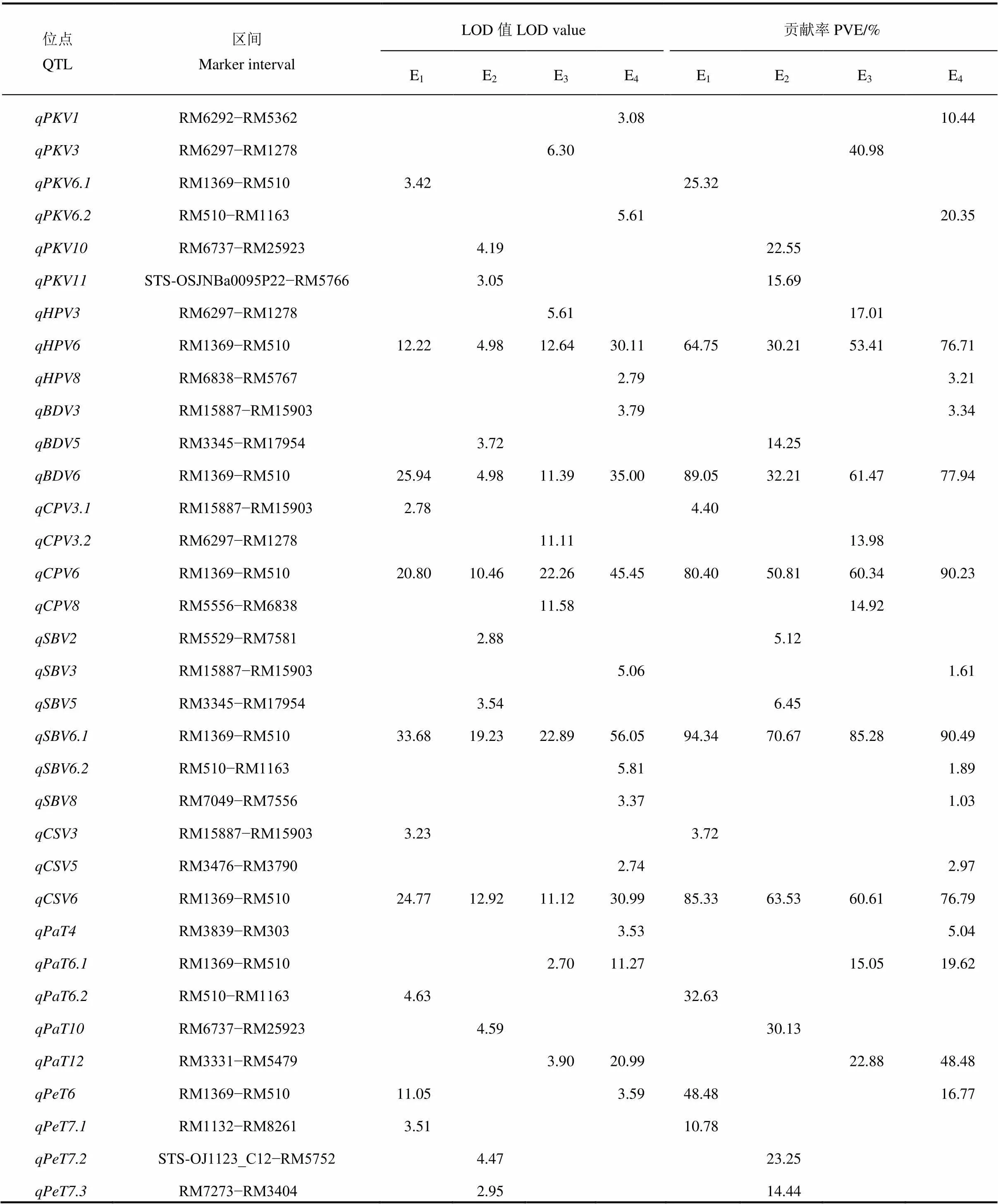
表4 昌恢121/越光CSSLs群体8个RVA谱特征值的QTL分析
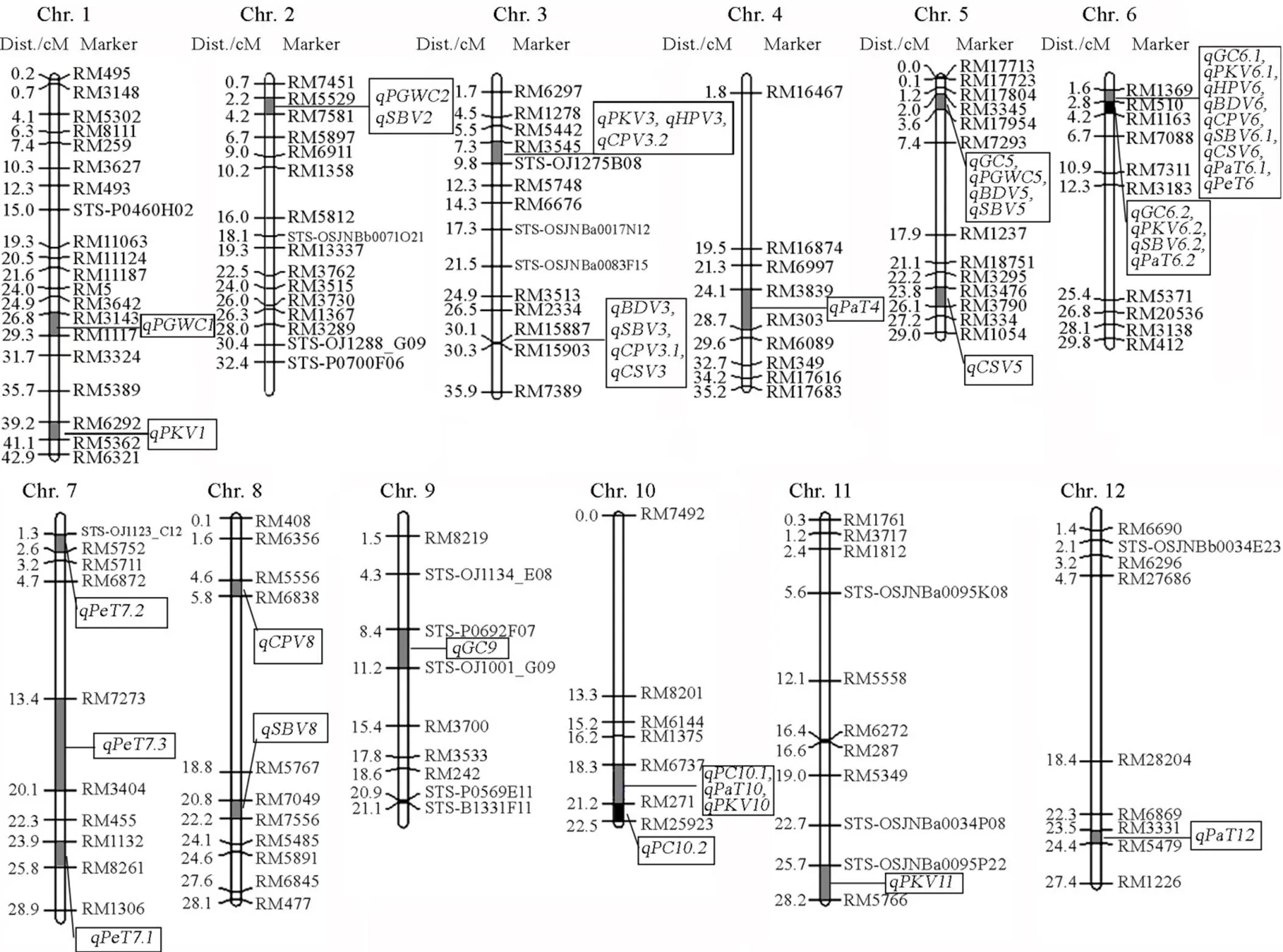
图1 水稻品质性状QTL在染色体上的分布
Fig. 1. Location of QTL detected for rice grain quality traits on chromosomes.
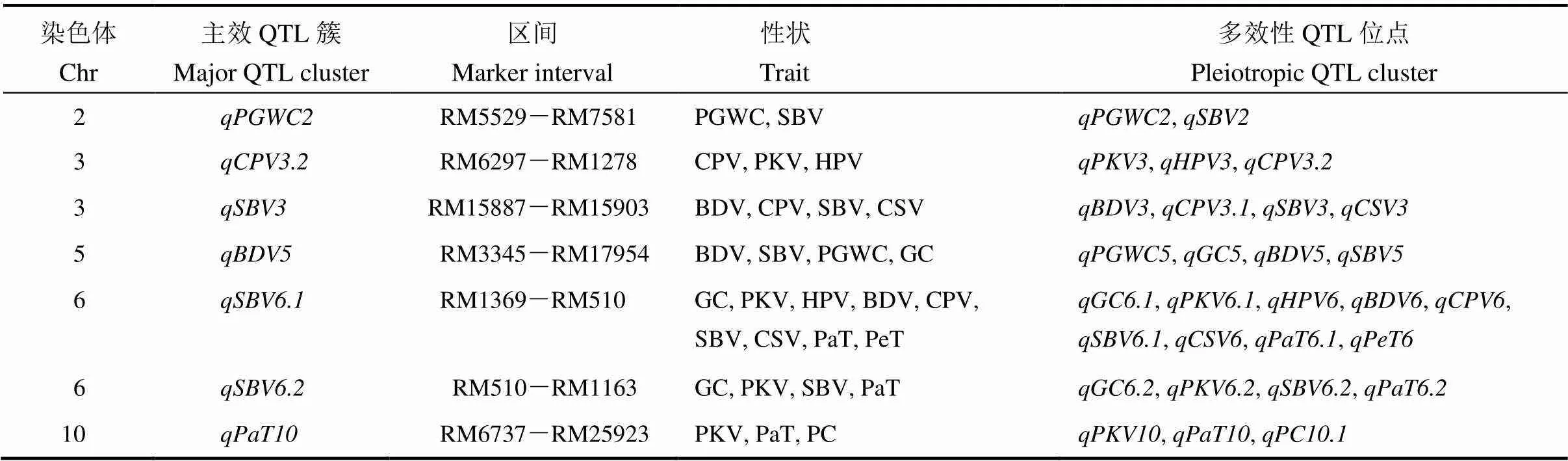
表5 水稻品质多效性区域分析
对RVA分析发现,、、、、在4个环境中都能被重复检测到,且都位于RM1369−RM510内,对应的染色体片段代换系为X19、X93和X198。将携带有、、、、的染色体片段代换系与背景亲本昌恢121进行差异比较,发现在4个环境中除2016年南昌环境下的热浆黏度,代换系X19、X93与昌恢121差异不显著,其余4个环境中每个代换系的表型与昌恢121之间都存在显著或极显著的差异(图2)。2016南昌的热浆黏度未能达显著差异可能是在扬花期期间遇到高温极端天气,这可能也说明高温会严重影响热浆黏度。总之,这5个QTL在多个环境中是稳定表达的,说明导入稳定的QTL可以用于分子育种改良水稻品质。
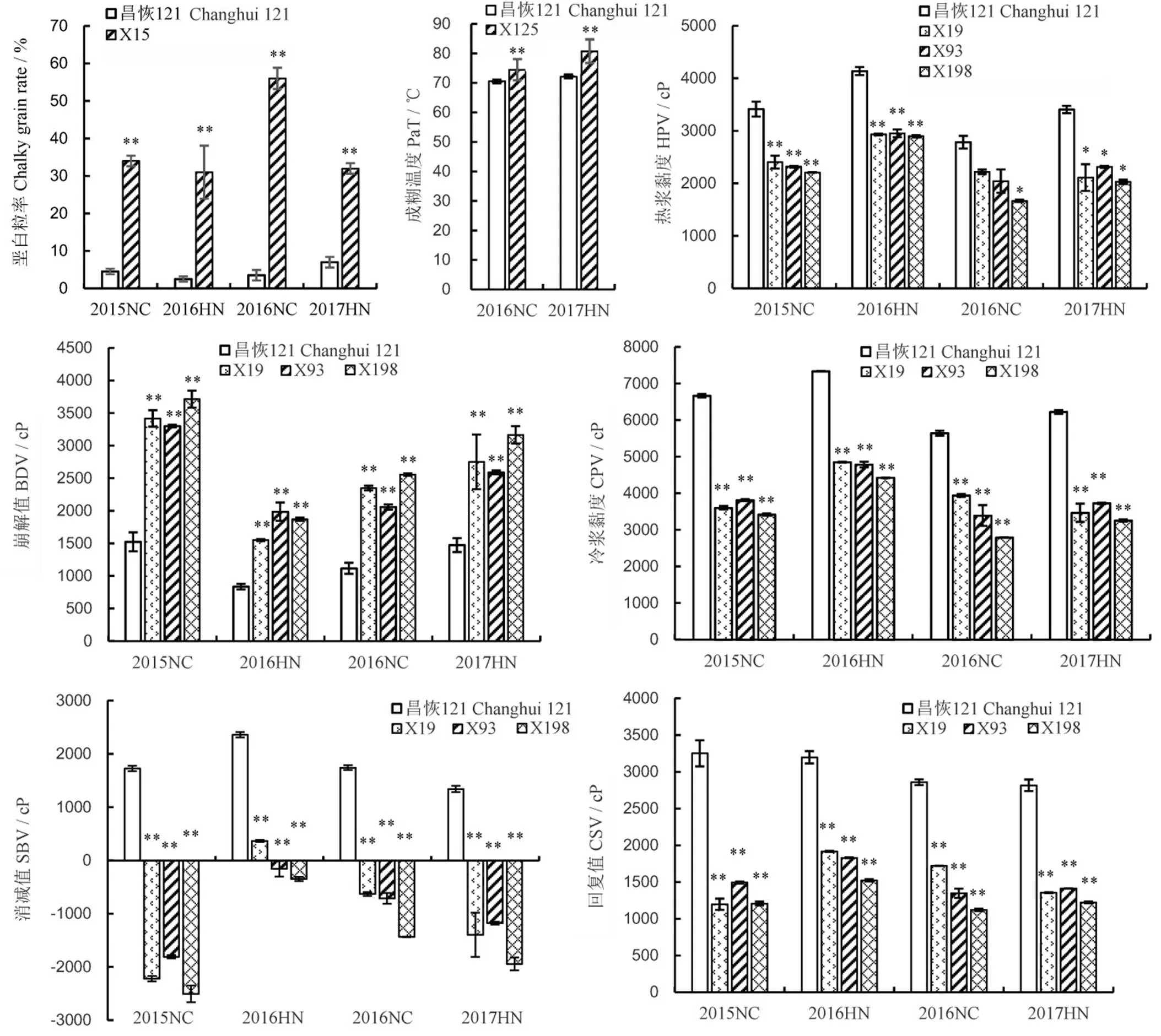
*和**分别表示昌恢121的品质性状与目标片段代换系之间的差异达5%和1%显著水平。NC和HN分别表示江西南昌和海南。
Fig. 2. Differences of phenotypic values of rice quality traits between genetic background parent Changhui 121 and the CSSLs harboring the QTL alleles.
3 讨论
4个环境中共检测到44个稻米品质相关的QTL,分布于12条染色体上,发现7个QTL簇,分布在第2、3、5、6和10染色体上(表5)。其中第2染色体RM5529−RM7581区域内的QTL簇控制着垩白粒率、消减值;第3染色体上有2个QTL簇,分别位于RM6297−RM1278和RM15887− RM15903区间,控制着冷浆黏度、最高黏度、热浆黏度、崩解值、消减值和回复值;第5染色体上的QTL簇位于RM3345−RM17954区间,控制着崩解值、消减值、垩白粒率和胶稠度;第6染色体上2个QTL簇,分别位于RM1369−RM510和RM510− RM1163区域,控制着胶稠度、最高黏度、热浆黏度、崩解值、冷浆黏度、消减值、回复值、成糊温度和成糊时间;第10染色体RM6737−RM25923区间的QTL簇控制着最高黏度、成糊温度和蛋白质含量。这些QTL成簇分布是一因多效或基因连锁引起,同时多个QTL能在两个及两个以上的环境中被重复检测到,说明这些QTL簇是确实存在的并且稳定表达。
与前人研究结果比较发现,本研究检测到的覆盖了Li等[22]克隆的的区间段,贡献率为16.61%;、、与RM190基因)紧密连锁;位于基因附近;位于基因附近。在稻米淀粉黏滞性方面,第6染色体RM1369−RM510区间定位到5个能稳定遗传的QTL(、、、、),位于McCouch等[29]克隆的基因附近。本研究发现在第5和第8染色体上的QTL簇与杨亚春等[16]和张杰等[18]的研究结果类似,说明控制RVA谱特征值的遗传表现为多基因紧密连锁。通过对RVA谱QTL多个环境进行检测,结果表明除在第6染色体上遗传效应大的位点能重复检测到外,其他微效基因检测重复性不好,说明稻米RVA谱特征值的遗传是由主效基因控制为主,多微效基因与环境互作协同控制,这与吴殿星等[30]、李欣等[31]和张巧凤等[32]的结论一致。在第12染色体上检测到控制成糊温度的基因与刘鑫燕等[33]用日本晴×9311染色体代换系检测结果一致。、、和位于第5染色体的RM3345−RM17954区间,与控制籽粒垩白基因位于同一染色体区域。、、、、、、、、、和都位于第6染色体的RM1369−RM510区间,与蜡质基因位于同一染色体区域。、、和都位于第6染色体的RM510−RM1163区间,都与淀粉合成酶基因位于同一染色体区域。此外,、和位于基因的上游区域。这些基因集合在同一区域是一因多效引起的还是多基因紧密连锁引起的,需进一步的验证。
水稻品质性状容易受环境影响,多属于数量性状,在不同群体、不同环境和不同栽培条件下定位到的位置可能不一致。本研究从香型恢复系籼稻昌恢121为背景亲本,以优质粳稻越光为供体亲本构建的染色体片段代换系中挑选59个家系,在其生育期基本一致和栽培方式相同条件下,对4个环境下稻米品质性状QTL进行检测,结果发现在4个环境下检测到44个稻米品质相关的QTL中,其中有10个 QTL能在两个及以上环境中重复检测到,其中仅有、和能在4个环境下重复检测到,这表明水稻品质性状受环境影响较大,不容易被重复检测到。通过对、、、、和在不同环境下的稳定性分析,发现除极端外界环境影响之外如(2016南昌环境下的HPV未能达显著差异可能是在扬花期期间遇到高温极端天气影响),这7个QTL都能在不同环境下稳定遗传,通过分子育种技术将这些稳定的QTL位点,导入到稳定遗传的育种材料中,可改善稻米食味品质。和能在两个及以上环境中重复检测到,与前人研究比较发现这两个QTL尚未被精细定位及克隆,通过对该QTL进一步的分离克隆将有助于改善稻米的食味品质,为粳稻改良籼稻品质提供理论依据。
[1] Chen L K, Gao W W, Chen S P, Wang L P, Zou J Y, Liu Y Z, Wang H, Chen Z Q, Guo T. High-resolution QTL mapping for grain appearance traits and co-localization of chalkiness-associated differentially expressed candidate genes in rice[J]., 2016, 9(1): 48-65.
[2] Gao Y, Liu C L, Li Y Y, Zhang A P, Dong G J, Xie L H, Zhang B, Ruan B P, Hong K, Xue, D W, Zeng D L, Guo L B, Qian Q, Gao Z Y. QTL analysis for chalkiness of rice and fine mapping of a candidate gene for[J]., 2016, 9(1): 41.
[3] Xu X M, Xu Z J, Matsue Y J, Xu Q. Effects of genetic background and environmental conditions on texture properties in a recombinant inbred population of an inter-subspecies cross[J]., 2019, 12(1): 32.
[4] Tan Y F, Sun M, Xing Y Z, Hua J P, Sun X L, Zhang Q F, Corke H. Mapping quantitative trait loci for milling quality, protein content and color characteristics of rice using a recombinant inbred line population derived from an elite rice hybrid[J]., 2001, 103(6/7): 1037-1045.
[5] 翁建峰, 万向元, 吴秀菊, 王海莲, 翟虎渠, 万建民. 利用CSSL群体研究稻米AC和PC相关QTL表达稳定性[J]. 作物学报, 2006, 32(1): 14-19.
Weng J F, Wan X Y, Wu X J, Wang H L, Zhai H Q, Wan J M. Stable expression of QTL for AC and PC of milled rice (L.) using a CSSL population[J]., 2006, 32(1): 14-19. (in Chinese with English abstract)
[6] 张涛, 郑家奎, 吴先军, 蒋开锋, 杨乾华, 陈温福, 杨莉. 水稻糙米蛋白质含量的QTL定位[J]. 分子植物育种, 2009, 7(1): 67-72.
Zhang T, Zheng J K, Wu X J, Jang K F, Yang Q H, Chen W F, Yang L. QTL mapping of brown rice protein content in a RIL population of rice[J]., 2009, 7(1): 67-72. (in Chinese with English abstract)
[7] 李晨, 潘大建, 孙传清, 周汉钦, 范芝兰, 陈建酉, 王象坤. 水稻糙米高蛋白基因的QTL定位[J]. 植物遗传资源学报, 2006, 7(2): 170-174.
Li C, Pan D J, Sun C Q, Zhou H Q, Fan Z L, Chen J Y, Wang X K. QTL location of high protein gene in rice grain[J]., 2006, 7(2): 170-174. (in Chinese with English abstract)
[8] 杨亚春, 倪大虎, 宋丰顺, 李莉, 冯光, 李泽福, 杨剑波. 两个环境下糙米和精米蛋白质含量的QTL分析[J]. 中国水稻科学, 2012, 26(3): 351-355.
Yang Y C, Ni D H, Song F S, Li L, Feng G, Li Z F, Yang J B. Identification of QTL for protein content in brown and milled rice in two environments[J]., 2012, 26(3): 351-355. (in Chinese with English abstract)
[9] Bao J S, Harold C, He P, Zhu L H. Analysis of quantitative trait loci for starch properties of rice based on an RIL population[J]., 2003, 45(8): 986-994.
[10] Fan C C, Yu X Q, Xing Y Z, Xu C G, Luo L J, Zhang Q F. The main effects, epistatic effects and environmental interactions of QTLs on the cooking and eating quality of rice in a double-haploid line population[J]., 2005, 110(8): 1445-1452.
[11] 吴长明, 孙传清, 王象坤, 李自超, 付秀林, 张强. 稻米食味品质性状的QTL分析[J]. 吉林农业科学, 2003, 28(2): 6-14.
Wu C M, Sun C Q, Wang X K, Li Z C, Fu X L, Zhang Q. Study on QTLs of grain eating quality characters in rice[J]., 2003, 28(2): 6-14. (in Chinese with English abstract)
[12] 邵高能, 唐绍清, 焦桂爱, 罗炬, 唐傲, 胡培松. 稻米蒸煮品质性状的QTL定位[J]. 中国水稻科学, 2009, 23(1): 94-98.
Shao G N, Tang S Q, Jiao G A, Luo J, Tang A, Hu P S. Mapping of QTL for cooking quality traits of rice[J]., 2009, 23(1): 94-98. (in Chinese with English abstract)
[13] Lanceras J C, Huang Z L, Naivikul O, Vanavichit A, Ruanjaichon V, Tragoonrung S. Mapping of genes for cooking and eating qualities in Thai Jasmine rice (KDML105) [J]., 2000, 7(2): 93-101.
[14] 张巧凤, 张亚东, 朱镇, 赵凌, 赵庆勇, 许凌, 王才林. 稻米淀粉黏滞性(RVA谱)特征值的遗传及QTL定位分析[J]. 中国水稻科学, 2007, 21(6): 591-598.
Zhang Q F, Zhang Y D, Zhu Z, Zhao L, Zhao Q Y, Xu L, Wang C L. Analysis of inheritance and QTLs of rice starch viscosity (RVA profile) characteristics[J]., 2007, 21(6): 591-598. (in Chinese with English abstract)
[15] 张永生, 江玲, 刘喜, 刘世家, 陈亮明, 翟虎渠, 万建民. 优质水稻品种越光(Koshihikari)中控制稻米淀粉RVA谱特征值的QTL分析[J]. 中国水稻科学, 2010, 24(2): 137-144.
Zhang Y S, Jiang L, Liu X, Liu S J, Chen L M, Zhai H Q, Wan J M. Analysis of QTLs for starch RVA profile properties in the superior rice cultivar Koshihikari[J]., 2010, 24(2): 137-144. (in Chinese with English abstract)
[16] 杨亚春, 倪大虎, 宋丰顺, 李莉, 陆徐忠, 李泽福, 杨剑波. 不同生态环境下稻米淀粉RVA谱特征值的QTL定位分析[J]. 作物学报, 2012, 38(2): 264-274.
Yang Y C, Ni D H, Song F S, Li L, Lu X Z, Li Z F, Yang J B. Identification of QTL for rice starch RVA profile properties under different ecological sites[J]., 2012, 38(2): 264-274. (in Chinese with English abstract)
[17] 张昌泉, 胡冰, 朱孔志, 张华, 冷亚麟, 汤述翥, 顾铭洪, 刘巧泉. 利用重测序的水稻染色体片段代换系定位控制稻米淀粉黏滞性谱QTL[J]. 中国水稻科学, 2013, 27(1): 56-64.
Zhang C Q, Hu B, Zhu K Z, Zhang H, Leng Y L, Tang S Z, Gu M H, Liu Q Q. Mapping of QTLs for rice RVA properties using high-throughput re-sequenced chromosome segment substitution lines[J]., 2013, 27(1): 56-64. (in Chinese with English abstract)
[18] 张杰, 郑蕾娜, 蔡跃, 尤小满, 孔飞, 汪国湘, 燕海刚, 金洁, 王亮. 稻米淀粉RVA谱特征值与直链淀粉、蛋白含量的相关性及QTL定位分析[J]. 中国水稻科学, 2017, 31(1): 31-39.
Zhang J, Zheng L N, Cai Y, You X M, Kong F, Wang G X, Yan H G, Jin J, Wang L. Correlation analysis and QTL mapping for starch RVA profile properties and amylose and protein contents in rice[J]., 2017, 31(1): 31-39. (in Chinese with English abstract)
[19] Matsushima R, Maekawa M, Kusano M, Kondo H, Naoko F, Kawagoe Y, Sakamoto W. Amyloplast- localized SUBSTANDARD STARCH GRAIN4 protein influences the size of starch grains in rice endosperm[J]., 2014, 164(2): 623-636.
[20] She K, Kusano H, Koizumi K, Yamakawa H, Hakata M, Imamura T, Fukuda M, Naito N, Tsurumaki Y, Yaeshima M, Tsuge T, Kudoh M, Itoh E, Kikuchi S, Kishimoto N, Yazaki J, Ando T, Yano M, Aoyama T, Sasaki T, Satoh H, Shimada H. A novel factor FLOURYENDOSPERM2 is involved in regulation of rice grain size and starch quality[J]., 2010, 22(10): 3280-3294.
[21] Wang E, Wang J J, Zhu X D, Hao W, Wang L Y, Li Q, Zhang L X, He W, Lu B, Lin H X, Ma H, Zhang G Q, He Z H. Control of rice grain-filling and yield by a gene with a potential signature of domestication[J]., 2008, 40(11): 1370-1374.
[22] Li Y B, Fan C C, Xing Y Z, Yun P, Luo L J, Yan B, Peng B, Xie W B, Wang G W, Li X H, Xiao J H, Xu C G, He Y Q.encodes a vacuolar H+translocating pyrophosphatase influencing grain chalkiness in rice[J]., 2014, 46(4): 398-404.
[23] Luo Q, Tang D, Wang M, Luo W X, Zhang L, Qin B X, Shen Y, Wang K J, Li Y F, Cheng Z K. The role ofin crossover formation during rice meiosis., 2013, 3(6): 729-742.
[24] Han X H, Wang Y H, Liu X, Jiang L, Ren Y L, Liu F, Peng C, Li J J, Jin X M, Wu F Q, Wang J L, Guo X P, Zhang X, Cheng Z J, Wan J M. The failure to express a protein disulphide isomerase-like protein results in a floury endosperm and an endoplasmic reticulum stress response in rice[J]., 2012, 63(1): 121-130.
[25] Wan Y H, Ren Y L, Liu X, Jiang L, Chen L, Han X H, Jin M N, Liu S J, Liu F, Lv J, Zhou K N, Su N, Wan J M.regulates endomembrane organization and storage protein trafficking in rice endosperm cells[J]., 2010, 64(5): 812-824.
[26] Yang R F, Sun C L, Bai J J, Luo Z X, Shi B, Zhang J M, Yan W G, Piao Z Z. A putative genefor resistant starch mutated from SBE3 for starch branching enzyme in rice (L.) [J]., 2012, 7(8): e43026.
[27] Cai Y C, Li S F, Jiao G A, Sheng Z H, Wu Y W, Shao G N, Xie L H, Peng C, Xu J F, Tang S Q, Wei X J, Hu P S.encodes a plastidic pyruvate kinase involved in rice endosperm starch synthesis, compound granule formation and grain filling[J]., 2018, 16(11): 1878-1891.
[28] Tuncel A, Kawaguchi J, Ihara Y, Matsusaka H, Nishi A, Nakamura T, Kuhara S, Hirakawa H, Nakamura Y, Cakir B, Nagamine A, Okita T W, Hwang S K, Satoh H. The rice endosperm ADP-glucose pyrophosphorylase large subunit is essential for optimal catalysis and allosteric regulation of the heterotetrameric enzyme[J]., 2014, 55(6):1169-83.
[29] McCouch S R. Gene nomenclature system for rice[J]., 2008, 1(1): 72-84.
[30] 吴殿星, 舒庆尧, 夏英武. 利用RVA谱快速鉴别不同表观直链淀粉含量早籼稻的淀粉黏滞特性[J]. 中国水稻科学, 2001, 15(1): 58-60.
Wu D X, Shu Q Y, Xia Y W. Rapid identification of starch viscosity property of early indica rice varieties with different apparent amylose content by RVA profile[J]., 2001, 15(1): 58-60. (in Chinese with English abstract)
[31] 李欣, 张蓉, 隋炯明, 梁国华, 沈新平, 严长杰, 顾世梁, 顾铭洪. 稻米淀粉黏滞性谱特征的表现及其遗传[J]. 中国水稻科学, 2004, 18(5): 10-16.
Li X, Zhang R, Sui J M, Liang G H, Shen X P, Yan C J, Gu S L, Gu M H. Performance and inheritance of rice starch viscosity (RVA Profile) characteristics[J]., 2004, 18(5): 10-16. (in Chinese with English abstract)
[32] 张巧凤, 张亚东, 朱镇, 赵凌, 赵庆勇, 许凌, 王才林. 稻米淀粉黏滞性(RVA谱)特征值的遗传及QTL定位分析[J]. 中国水稻科学, 2007, 21(6): 591-598.
Zhang Q F, Zhang Y D, Zhu Z, Zhao L, Zhao Q Y, Xu L, Wang C L. Analysis of inheritance and QTLs of rice starch viscosity (RVA profile) characteristics[J]., 2007, 21(6): 591-598. (in Chinese with English abstract)
[33] 刘鑫燕, 朱孔志, 张昌泉, 洪燃, 孙鹏, 汤述翥, 顾铭洪, 刘巧泉. 利用9311来源的粳型染色体片段代换系定位控制稻米糊化温度的微效QTL[J]. 作物学报, 2014, 40(10): 1740-1747.
Liu X Y, Zhu K Z, Zhang C Q, Hong R, Sun P, Tang S Z, Gu M H, Liu Q Q. Mapping of minor QTLs for rice gelatinization temperature using chromo-some segment substitution lines from9311 in thebackground[J]., 2014, 40(10): 1740-1747. (in Chinese with English abstract)
Identification and Stability Analysis of QTL for Grain Quality Traits Under Multiple Environments in Rice
WANG Xiaolei#, LIU Yang#, SUN Xiaotang, OUYANG Linjuan, PAN Jinlong, PENG Xiaosong, CHEN Xiaorong, HE Xiaopeng, FU Junru, BIAN Jianmin, HU Lifang, XU Jie, HE Haohua*, ZHU Changlan*
(Key Laboratory of Crop Physiology, Ecology and Genetic Breeding, Ministry of Education, College of Agronomy, Jiangxi Agricultural University, Nanchang 330045, China;#These authors contributed equally to this work;*Corresponding author, E-mail: wxl0vip@163.com; zhuchanglan@163.com)
【Objective】Our aim is to explore new QTLs controlling rice qualityand to use molecular breeding technology to improve rice quality.【Method】Usingfragrant restorer line Changhui 121 as the background parent and high-qualityrice Koshihikari as the donor parent, a set of chromosome segment substitution lines(CSSLs) was constructed for QTL detection and stability analysis of rice quality traits in four environments.【Result】 A total of 44 QTLs were detected in four environments, six of which could be detected in multiple environments. There were multiple QTL clusters on chromosome 2, 3, 5, 6 and 10, which have obvious regulatory effects on rice quality. Seven QTLs on chromosomes 1, 6 and 12 can be expressed stably in different environments. 【Conclusion】 Theof chromosome 1 (RM3143−RM1117) and theof chromosome 12 (RM3331−RM5479) are two new stable QTL.
rice; ecological environment; rice quality traits; stability analysis
Q343.1+5; S511.033
A
1001-7216(2020)01-0017-11
10.16819/j.1001-7216.2020.9052
2019-04-30;
2019-06-13。
江西创新驱动5511工程平台和人才团队计划资助项目(20165BCB19005);国家自然科学基金资助项目(31560507);江西省教育厅资助项目(GJJ150409)。

