水稻子预44和江南香糯基因组比较鉴定稻瘟病抗性相关基因
李金璐 张慧 焦泽宇 刘剑宇 韩光煜 卓晓轩 罗琼
水稻子预44和江南香糯基因组比较鉴定稻瘟病抗性相关基因
李金璐#张慧#焦泽宇 刘剑宇 韩光煜 卓晓轩 罗琼*
(云南农业大学 农业生物多样性与病虫害控制教育部重点实验室/省部共建云南生物资源保护与利用国家重点实验室, 昆明 650201;#共同第一作者;*通信联系人, E-mail: qiongbf@aliyun.com)
【目的】子预44是一具有广谱持久稻瘟病抗性的云南地方粳稻品种。为了鉴定子预44中候选稻瘟病抗性相关基因,【方法】本研究利用高通量测序技术(High-throughput sequencing)对子预44和感病水稻江南香糯进行了全基因组测序。而后使用软件GATK(3.4-46)对高质量测序结果进行SNP和InDel的检测和统计,进一步筛选出子预44和江南香糯DNA水平存在SNPs/InDels多态性抗病相关基因。【结果】通过Hiseq X10 PE150平台分别获得了4 118 170 045 bp和2 995 054 509 bp子预44和江南香糯的基因组数据,比对到参考基因组(Ensembl release 31)的比对率分别为98.56%和98.30%。在抗病水稻子预44和感病水稻江南香糯之间鉴定了922个纯合突变的差异抗病相关基因。结合基因定位结果,在子预44中鉴定了一个新的抗稻瘟病候选基因。【结论】研究结果为子预44中抗稻瘟病新基因的克隆提供了参考,对子预44广谱持久抗瘟分子机制的研究奠定了基础。
水稻;稻瘟病;基因组测序;抗病相关基因
水稻是世界上近一半人口的主粮,随着世界人口的不断增长,预计到2030年水稻生产至少需要增加40%的产量才能满足人们的生活所需[1]。由稻瘟病菌引起的稻瘟病是水稻生产上最具毁灭性的病害,全球每年因稻瘟病造成稻谷减产10%~30%,每年减产的粮食足够养活6千万人口[2]。目前,稻瘟病防治主要采用的是药剂防治和种植抗病品种。虽然药剂防治对稳定水稻产量起到了非常重要的作用,但农药的重复大量使用带来了严重的环境污染[3, 4]。利用抗病基因培育抗病品种是控制稻瘟病最经济有效和环保的方法[3-7]。目前已鉴定的主效抗性基因位点已超过100个,数量抗性位点已超过350个[5, 8, 9],已克隆的抗稻瘟病基因约30个[5, 6, 9-12]。基因介导的小种特异性抗性效果明显、抗性强,在水稻育种中容易被利用,所以目前生产上使用的抗病品种多属于这种抗性。然而,基因介导的抗性会由于病原的快速进化而丧失,因而抗性品种推广种植3~5年后其抗性就逐渐丧失[13, 14]。此外,我国新育成的一些水稻品种抗瘟效果并不太理想。例如,在2004−2008年间,国审的174个品种对稻瘟病的平均抗病指数仅为5.5,处于中感水平[7]。因此,优异的稻瘟病抗性新基因的发掘,尤其是广谱抗性基因的鉴定和利用对保证水稻产量具有重要意义。
子预44是一具有广谱持久抗瘟性的云南地方粳稻品种[16]。我们在子预44中鉴定了多个主效和微效抗瘟基因位点[17-22],发现子预44第6染色体短臂上10.008~11.043 Mb区间携带一个抗多个稻瘟病菌的主效基因。但由于水稻第6染色体短臂上抗稻瘟病基因成簇分布,且序列高度保守[23],通过常规的PCR方法进行该区域DNA片段的扩增和分析鉴定候选基因不仅耗时费钱,而且结果不理想。为了能快速有效地鉴定和克隆已定位的抗病基因,我们尝试在基因初步定位的基础上,进行抗病亲本子预44和感病亲本江南香糯的全基因组重测序和序列比较分析,筛选鉴定抗病候选基因。
1 材料与方法
1.1 水稻材料
抗病粳稻品种为子预44和中花11。感病粳稻品种为日本晴、台北309、江南香糯和丽江新团黑谷。抗病籼稻品种为9311和地谷。感病籼稻品种为Kasalath。以上水稻材料均由本实验室保存。
1.2 稻瘟病菌菌株
稻瘟菌株LP33、LP174和LP29-3由何月秋教授课题组提供,H53由本实验室从黑龙江采集的稻瘟病病样分离保存。
1.3 水稻育苗
选取健康饱满的成熟水稻种子,75%酒精消毒40 s,无菌水清洗3次,20%次氯酸钠消毒40 min,期间每10 min轻微晃动一次,消毒完成后用无菌水清洗3次。将消毒后的种子浸泡于无菌水中于37℃下吸胀,待种子露白后转移至铺有灭菌滤纸的培养皿中,加少量无菌水后于37℃下催芽。幼芽长至0.5 cm左右时播于特制的96孔PCR育苗板上,放置于营养液(参照国际水稻研究所的配方,并依据本实验室实际使用情况略作调整)中,室温、正常光照条件下培养。
1.4 水稻幼苗基因组DNA提取
采用稍作修改的CTAB法提取水稻幼苗基因组DNA。新鲜水稻幼苗剪碎后置于液氮预冷的研钵中,加入液氮快速研磨成粉,分装于2 mL离心管中,每管分装约0.1 g水稻样品。加入650 μL已于65℃下预热的CTAB提取液,充分震摇混匀。65℃下水浴30 min(每10 min摇晃混匀一次,以免组织结块)。加入等体积650 μL氯仿,颠倒混匀,12 000 r/min下离心10 min。上清转入新的1.5 mL离心管中,加入等体积−20℃预冷的异丙醇,轻轻颠倒混匀,于−20℃下静置30 min,12 000 r/min下离心5 min。弃上清,沉淀加500 μL 75%酒精洗涤,12 000 r/min下离心2 min,弃上清,洗涤2~3次,用无水乙醇洗一次。离心管开盖于超净工作台晾干。
晾干的DNA沉淀加入20~50 μL双蒸水充分溶解后,用琼脂糖凝胶电泳检测DNA质量,并同已知浓度的λ DNA分子量标准比较估计其浓度。于−20℃冰箱保存备用。
1.5 子预44和江南香糯全基因组重测序
子预44是云南地方品种麻早谷与城堡2号杂交选育而成的广谱抗稻瘟病的高原粳稻,对6群16个中国稻瘟病菌生理小种(ZA1,ZA49,ZA57,ZA61;ZBl,ZB13,ZB17,ZB25;ZCl,ZC3,ZCl3,ZC15;ZEI,ZE3;ZFl和ZGl)表现广谱抗性[16]。江南香糯是一低海拔高感稻瘟病的粳糯水稻品种,是抗稻瘟病基因克隆中常用的感病亲本[24]。在前期研究中,我们利用子预44和江南香糯作为杂交亲本构建的F7重组自交系(RIL F7)群体,进行了子预44中数量抗性位点分析[20];利用子预44和江南香糯杂交构建的F2群体,在子预44中定位了抗稻瘟病菌ZE1、LP33、LP11和LP36的主效抗瘟基因(t)[17]、(t)[18]、(t)[21]和(t)[22]。为了进一步克隆这些主效抗病基因,本研究利用高通量测序技术对子预44和江南香糯进行了基因组重测序和序列比较分析。
校园宣传语的汉译英不仅要基于原文,更要考虑翻译要求,现实翻译目的,它是检验翻译是否充分的标准;因而要对受众做分析,译文难度要适合中学生的英语水平,同时译者心中还应有虚拟受众,译文对他们必须有效;作为校园宣传语,有其独特的语体特征,译文要短小精悍、朗朗上口、便于记忆、利于宣传;另外,由于中英两种语言在语言特点、修辞手法上存在差异,基于翻译目的适时变通与创新。
参照文献[17]提取子预44和江南香糯单株基因组DNA,用酶随机打断成短的DNA片段后,进行末端修复。然后在DNA片段两端连接dA尾,并连接测序接头。加上接头的DNA片段经过AMPure XP磁珠纯化,选择300~400 bp范围的片段进行PCR扩增,构建测序文库。建好的文库经过纯化、库检,Hiseq X10 PE150上机测序。
1.6 数据过滤与质量分析
为了保证数据质量,在信息分析前对原始数据进行质控,通过数据过滤减少数据噪音。对下机的测序序列片段(clean reads)再进行更严格的过滤,得到高质量的测序序列片段(High quality clean reads),用于后续的信息分析。过滤的步骤包括去除含接头的,含N比例大于10%的以及低质量的(质量值Q≤10的碱基数占整条测序序列片段的50%以上)测序序列片段。
1.7 比对到参考基因组和覆盖度统计
使用比对软件0.7.12[25]和Mem算法,将过滤后的测序序列片段比对到参考基因组(Ensembl release 31)上,比对参数设置为-k 32 –M。
序列片段比对之后的结果用Picard 1.129 (Picard: http:// sourceforge.net/projects/picard/)进行标记,并统计插入片段数量。
用Bedtools 2.25.0[26]软件进行覆盖度统计。
1.8 SNPs和InDels统计
在基因组水平上由单个核苷酸或者几个核苷酸的插入或缺失所形成的DNA序列多态性,为变异。使用软件(3.4-46)[27]的UnifiedGenotyper模块将处理好的比对文件进行多个样本的变异检测,检测到的变异使用VariantFiltration进行过滤,过滤参数为-Window4, -filter “QD < 4.0 || FS > 60.0 || MQ < 40.0”,-G_filter “GQ < 20”。用ANNOVAR[28]对检测出的变异进行功能注释。
1.9 候选抗病相关基因鉴定
将子预44和江南香糯基因组测序获得的数据对应到日本晴基因组中,根据日本晴基因组中基因注释的结果,鉴定子预44和江南香糯中的抗病相关基因。筛选出子预44和江南香糯之间DNA水平上存在SNP/InDel多态性的基因,并统计差异位点。筛选出分布在抗病相关基因编码区的SNP/InDel差异位点,进一步筛选出非同义纯合突变的差异基因,作为初步候选抗病相关基因。结合前期基因定位结果,进一步鉴定候选抗病相关基因。
1.10 稻瘟病菌孢子悬浮液制备
参照文献[17],进行菌株活化培养和产孢培养,将孢子浓度调制为2×105个/mL,配成终浓度为0.4%的明胶孢子悬浮液,待用。
1.11 水稻幼苗期接种及调查
水稻幼苗长到3~4叶期,将其转移至恒温接种室,配制2×105/mL的孢子悬浮液进行喷雾接种。接种后在温度25℃,湿度95%的条件下,黑暗培育24 h后,12 h光照和12 h黑暗交替培育5~7 d,参照文献[29]采用6级分级法进行病斑类型调查。
1.12 DNA片段的PCR扩增、回收和测序
PCR扩增采用50 μL反应体系:DNA模板1 μL(10 ng/μL),正反引物各0.75 μL(10 μmol/L),10×缓冲液 5 μL,dNTPs 4 μL(2.5 mmol/L),MgSO42 μL,KOD-PLUS 0.4 μL,DMSO 0.5 μL,ddH2O 35 μL。95℃下变性5 min,然后95℃下15 s,57±2℃下15 s,68℃下30 s/1000 bp,35个循环,72℃下延伸5 min。
PCR扩增产物用1%琼脂糖凝胶,1×TAE电泳缓冲液中,120 V电压电泳~30 min,待DNA分子量标准各个条带区分明显后于切胶台切取目的条带。采用北京全式金公司的胶回收试剂盒(Easy PureQuick Gel Extraction Kit),按每0.1g回收胶加300 μL溶胶液(Binding Buffer),65℃下加热溶解。溶胶加入分离柱(最多700 μL),10 000 r/min下离心1 min,将液相加入分离柱重复此步骤。弃液相,再加300 μL新的溶胶液,10 000 r/min下离心1 min。弃液相,加入700 μL已加入无水乙醇的清洗液(Wash Buffer),10 000 r/min下离心1 min。弃液相,重复此步骤1次。弃液相,空管离心,10 000 r/min下离心2 min。弃液相,开盖于65℃下加热3 min。换干净1.5 mL离心管,加入30 μL 65℃下预热的ddH2O。65℃下闭盖保温3 min,1300 r/min下离心2 min,收集洗脱液。

表1 子预44和江南香糯基因组序列数据
连接反应:回收产物0.5~4 μL,北京全式金公司平末端克隆载体(pEasy-Blunt Simple Cloning Vector )1 μL,补充ddH2O到总体积5 μL,混匀。25℃下连接5~30 min(连接时间因连接片段大小而异:0.1~1 kb为5~10 min;1~2 kb为10~15 min;2~3 kb为15~20 min;大于3 kb为20~30 min)。
连接反应结束后,产物中加入50 μL大肠杆菌感受态DH5α,轻弹混匀。冰浴30 min, 42℃水浴热击1 min,立即放冰上2 min,然后加入1 mL的空白LB培养基,于37℃、200 r/min下摇床培养60 min,涂布于含有卡那霉素的LB平板,吹干,37℃下倒置培养过夜。挑取单菌落于含有卡那霉素的LB液体培养基中,37℃下培养4 h后,取菌液1 μL进行PCR检测,能扩增出目的片段的单菌落菌液送昆明硕擎生物有限公司测序。
2 结果与分析
2.1 子预44、江南香糯基因组重测序
通过Hiseq X10 PE150平台获得的子预44和江南香糯高质量测序片段(HQ Clean Reads)分别为 41 021 044条(4 118 170 045 bp)和29 931 088条 (2 995 054 509 bp),覆盖参考基因组(Ensembl release 31)单末端的测序片段分别为104 368条和 64 567条,覆盖参考基因组双末端的测序片段分别为20 163 591×2和14 678 401×2,未比对上的测序片段分别为589 494条和509 719条,比对率分别为98.56%和98.30%。数据读长为101 bp,Q30分别为95.39%和94.82%(Q30是指质量值大于30的碱基所占百分比,测序碱基正确率为99.9%,表1)。
2.2 子预44和江南香糯基因组比较分析及抗病相关基因鉴定
将子预44和江南香糯基因组测序获得的数据对应到日本晴基因组中,根据与日本晴基因组中的基因注释的比对结果,在子预44和江南香糯中共鉴定了4510个抗病相关基因对。这些基因广泛分布于水稻的12条染色体上。不同染色体上的基因数目存在差异,第1染色体上有640个基因,数量最多,占总数的14%。第9染色体上的基因数最少,为199个(5%,图1)。
进一步分析发现,4 510个抗病相关基因中有1769个基因在子预44与江南香糯之间存在DNA水平上的SNP/InDel多态性,差异位点达22 537个,广泛分布于基因的上游、下游、外显子以及内含子区域(图2)。
在这22 537个SNP/InDel多态性位点中,有5063个SNP/InDel差异位点分布在987个抗病相关基因的编码区,并导致了氨基酸水平的差异。其中,SNP/InDel包括四种类型,最多的为非同义SNP,共有4 732个(表2)。将包含不同SNP/InDel位点的抗性相关基因进行维恩分析,发现含有非同义SNP的基因数目最多,为728个(图3)。
978个差异基因中有922个为纯合突变的差异基因(附表1),它们不均匀地分布在水稻的12条染色体上,其中,第1、11和6染色体较多,分别有168个(17%)、158个(16%)和114个(12%)。第11和6染色体也是到目前为止鉴定和克隆抗稻瘟病基因最多的染色体。第1染色体虽然预测的抗病相关基因数量较多,但克隆的基因只有4个位于第1染色体上。该结果对子预44中抗稻瘟病基因的鉴定具有重要参考价值(图4)。
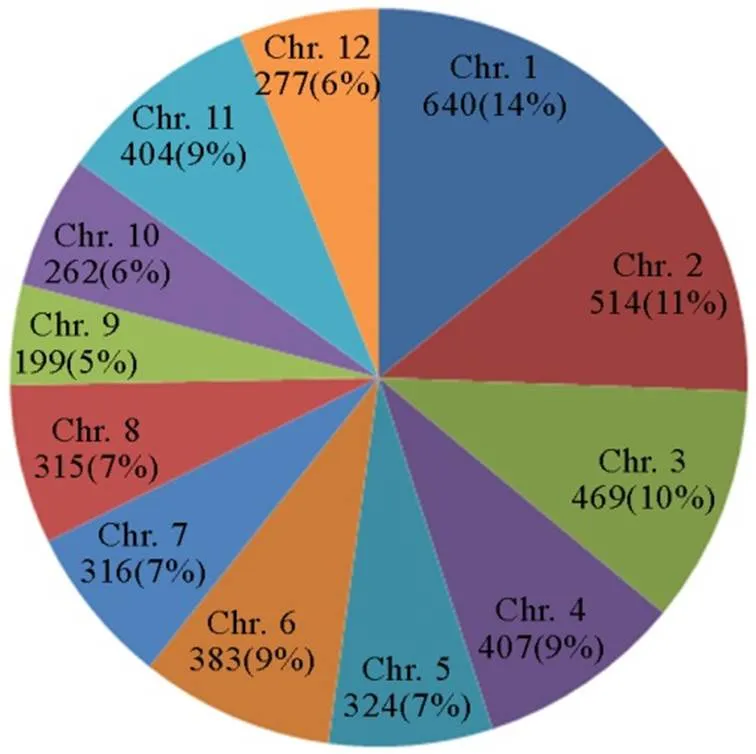
图1 抗病相关基因在水稻的分布
Fig. 1. Distribution of resistance related genes on rice chromosomes.
Fig. 2. Polymorphisms of disease resistance-related genes between Ziyu 44 and Jiangnanxiangnuo.
2.3 子预44中一个新的抗稻瘟病候选基因的鉴定
利用LP33、LP29-3、LP174、H53等多个稻瘟病菌株进行子预44中抗稻瘟病主效基因定位发现,子预44中抗这些菌株的基因均定位于水稻第6染色体短臂上10.008−11.043 Mb之间。进一步利用该定位区间的多态性标记和扩大的F2群体进行基因精细定位,缩小定位区间的工作却困难重重,进展缓慢。基于此,结合定位区间内抗病相关基因的预测和子预44与江南香糯基因组的比较分析发现,该区间内1个预测的丝/苏氨酸蛋白激酶编码基因,在子预44和江南香糯等位基因间有9个核苷酸的缺失/插入(GCCGCCGCC)突变(图5-A),导致了编码蛋白3个氨基酸(AAA)的缺失/插入差异(图5-B)。

表2 非同义突变的SNP/InDel
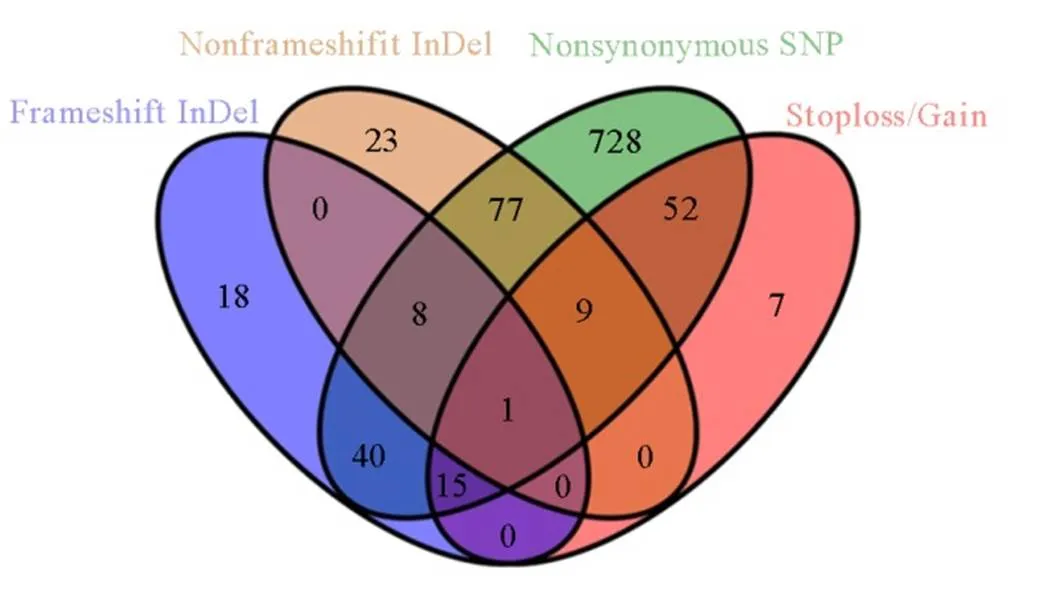
Frameshift InDel−引起氨基酸编码移码突变的插入缺失突变;Nonframeshift InDel−不导致氨基酸编码移码的插入缺失突变;Nonsynonymous SNP−引起氨基酸差异的单核苷酸多态性;Stoploss/gain−SNP/InDel突变引起功能缺失或获得的氨基酸翻译终止或延伸。
Fig. 3. Nonsynonymous mutation of SNP/InDel between Ziyu 44 and Jiangnanxiangnuo.
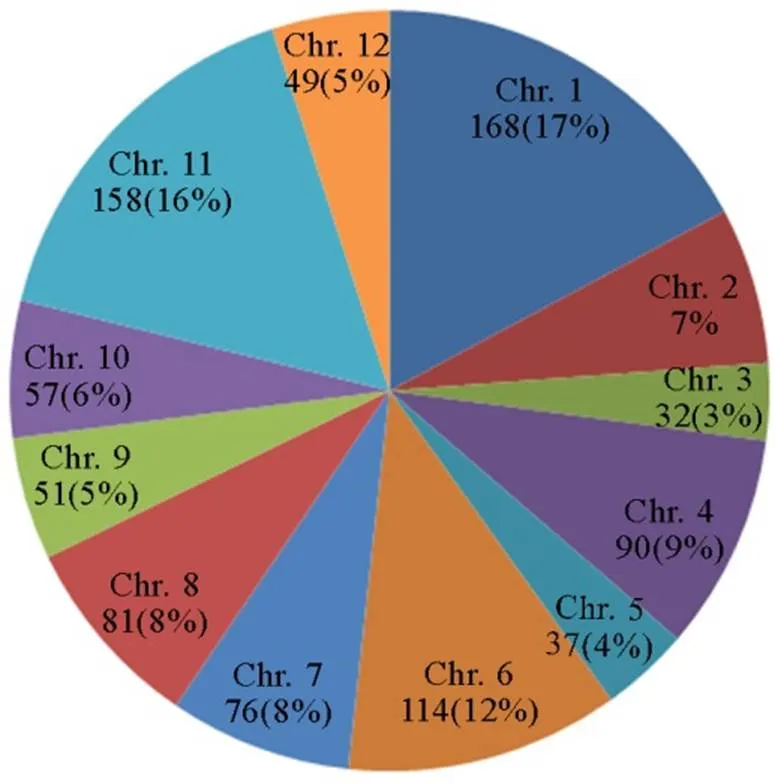
图4 922个抗病相关基因在水稻染色体上的分布
Fig. 4. Distribution of 922 resistance-related genes on rice chromosomes.
进一步根据基因序列设计特异的PCR引物,分别从3个抗病水稻(9311、中花11、地谷)和4个感病水稻(日本晴、丽江新团黑谷、Kasalath、台北309)中扩增其基因片段。序列分析显示,3个抗病水稻中基因的DNA序列与子预44一致,而4个感病水稻中该基因的DNA序列与江南香糯一致(图5-A)。
为进一步确定9个水稻材料中基因在DNA水平上的差异是否与其抗/感表型一致,利用稻瘟病菌株LP174,对9个水稻材料进行了苗期喷雾接种鉴定。结果表明,子预44、9311、中花11和地谷对LP174的抗病型类似,江南香糯、日本晴、丽江新团黑谷、Kasalath和台北309对LP174表现相似的感病特征(图6),抗/感表型与基因在分子水平的差异一致。因此,推测该基因可能是子预44中抗稻瘟病菌株LP174的候选基因。
3 讨论
到目前,鉴定的抗稻瘟病主效基因已超过100个,分布在水稻除第3染色体以外的其他所有染色体上。其中以第6、11和12染色体分布较多,占总数的一半以上[30]。本研究结果显示,第3染色体上预测的抗病相关基因有469个,占总数的10%,仅次于第1和2染色体,多于定位和克隆抗稻瘟病基因数量较多的第6(383)、11(404)和12(277)染色体,居第3位(图1)。这与目前定位和克隆的抗稻瘟病基因在水稻染色体上的分布不完全一致。是什么原因导致几乎没有能通过图位克隆的方法在第3染色体上鉴定和克隆到抗稻瘟病基因呢?对子预44和江南香糯间非同义突变的差异抗性相关基因的进一步分析发现,子预44和江南香糯间非同义突变的差异抗性相关基因主要分布在第1、6和11染色体上,也就是说,虽然第3染色体上预测的抗病相关基因数量较多,但仅有5%预测的抗病相关基因在两个抗、感水稻间存在非同义突变差异,差异基因的数目是12条染色体中最少的(图4),这与目前鉴定和克隆的抗稻瘟病基因主要分布在水稻第1、6、11和12染色体上,第3染色体上鉴定的抗病基因数量最少的结果一致[15]。我们推测第3染色体上预测的抗病相关基因很可能多为没有抗性功能的假基因,或微效基因位点,也有可能这些抗病基因与病原菌无毒基因间的相互作用不符合“基因对基因”假说。Li等[16]通过66份水稻材料的基因组测序和分析,在水稻第3染色体上鉴定了一个编码C2H2结构域转录因子的隐性广谱抗瘟基因,实现了水稻第3染色体上抗稻瘟基因克隆数量零的突破,为这一推论提供了一定的证据,但仍有待相关研究的进一步证实。

ZY44–子预44;Digu–地谷;ZH11–中花11;JNXN–江南香糯;Nip–日本晴;LTH–丽江新团黑谷;TP309–台北309。
Fig. 6. Symptoms of nine rice varieties inoculated withLP174.

ZY44–子预44;Digu–地谷;ZH11–中花11;JNXN–江南香糯;Nip–日本晴;LTH–丽江新团黑谷;TP309–台北309。
Fig. 5. Comparison of the nucleotide and amino acid sequences offrom nine rice varieties.
目前,已定位和克隆的抗稻瘟病基因除[31],(t)[32]和(t)[14]为隐性抗病基因外,其余都是显性抗病基因。图位克隆方法是定位和克隆水稻各种功能基因普遍采用的经典方法,并卓有成效。然而近年来利用这种方法克隆的抗稻瘟病基因大多数是已克隆基因的等位或是直系同源基因[10]。Shang等[33]在籼稻品种93-11和粳稻品种日本晴基因组中编码的NBS-LRR基因分析和比较的基础上,设计假基因化分子标记,从地谷中克隆了抗稻瘟病基因[33]。最近,Li等[16]通过66份水稻材料的基因组测序和分析,在水稻第3染色体上鉴定的隐性广谱抗瘟基因。我们在基因定位的基础上,结合两亲本基因组序列的比较分析,在水稻第6染色体短臂上鉴定一个新的编码丝氨酸苏氨酸蛋白激酶(serine threonine kinase)抗多个稻瘟病菌株的候选抗瘟基因()。这些结果表明,随着高通量基因组测序技术的快速发展,通过基因组测序和比较基因组学的方法鉴定和克隆新的水稻抗病基因,是一种省时有效的方法。
子预44是一具有广谱持久稻瘟病抗性的云南地方粳稻品种,利用高通量测序技术Hiseq X10 PE150进行了子预44和感病水稻江南香糯的全基因组重测序,分别获得了高质量的基因组数据 4 118 170 045 bp和2 995 054 509 bp,比对到参考基因组(Ensembl release 31)的比对率分别为98.56%和98.30%。在子预44和江南香糯之间鉴定了922个纯合突变的差异抗病相关基因,明确了这些基因的染色体分布,为子预44中抗病新基因的克隆和育种利用研究提供了有价值的信息,为研究子预44的广谱抗瘟分子机制奠定了重要的基础。
谢辞:感谢中国科学院遗传与发育生物学研究所程祝宽研究员和梁承志研究员在水稻基因组测序中提供的帮助,感谢广州基迪奥生物科技有限公司在数据分析中的帮助。
辅助信息:有一个辅助性表格S1放在《中国水稻科学》网站(http://www.ricesci.cn)上。
[1] Khush GS. What it will take to feed 5.0 billion rice consumers in 2030[J]., 2005,59(1):1-6.
[2] Skamnioti P, Gurr SJ. Against the grain: Safeguarding rice from rice blast disease[J]., 2009,27(3):141-150.
[3] Hu K M, Qiu D Y, Shen X L, Li X H, Wang S P. Isolation and manipulation of quantitative trait loci for disease resistance in rice using a candidate gene approach[J]., 2008,1(5):786-793.
[4] Miah G, Rafii MY, Ismail MR, Puteh AB, Rahim HA, Asfaliza R, Latif MA. Blast resistance in rice: A review of conventional breeding to molecular approaches[J]., 2013,40(3):2369-2388.
[5] Deng Y W, Zhai K, Xie Z, Yang D Y, Zhu X D, Liu J Z, Wang X, Qin P, Yang Y Z, Zhang G M, Li Q, Zhang J F, Wu S Q, Milazzo J, Mao B, Wang E T, Xie H A, Tharreau D, He Z H. Epigenetic regulation of antagonistic receptors confers rice blast resistance with yield balance[J]., 2017,355(6328):962-965.
[6] Zhou X G, Liao H C, Chern M, Yin J J, Chen Y F, Wang J P, Zhu X B, Chen Z X, Yuan C, Zhao W, Wang J, Li W T, He M, Ma B T, Wang J C, Qin P, Chen W L, Wang Y P, Liu J L, Qian Y W, Wang W M, Wu X J, Li P, Zhu L H, Li S G, Ronald PC, Chen X W. Loss of function of a rice TPR-domain RNA-binding protein confers broad-spectrum disease resistance[J]., 2018,115(12):3174-3179.
[7] 何峰, 张浩, 刘金灵, 王志龙,王国梁. 水稻抗稻瘟病天然免疫机制及抗病育种新策略. 遗传, 2014,36(8):756-765.
He F, Zhang H, Liu J L, Wang Z L, Wang G L. Recent advances in understanding the innate immune mechanisms and developing new disease resistance breeding strategies against the rice blast fungusin rice[J].(), 2014,36(8):756-765. (in Chinese with English abstract)
[8] Tanweer FA, Rafii MY, Sijam K, Rahim HA, Ahmed F, Latif MA. Current advance methods for the identification of blast resistance genes in rice[J]., 2015,338(5):321-334.
[9] Liang Z J, Wang L, Pan Q H. A new recessive gene conferring resistance against rice blast[J].2016,9(1):47. doi: 10.1186/s12284-016-0120-7.
[10] Xu X, Lv Q M, Shang J J, Pang Z Q, Zhou Z Z, Wang J, Jiang G H, Tao Y, Xu Q, Li X B, Zhao X F, Li S G, Xu J C, Zhu L H. Excavation of Pid3 orthologs with differential resistance spectra toin rice resource[J].2014,9(3):e93275. doi:10.1371/journal.pone.0093275.
[11] Li W T, Zhu Z W, Chern M, Yin J J, Yang C, Ran L, Cheng M P, He M, Wang K, Wang J, Zhou X G, Zhu X B, Chen Z X, Wang J C, Zhao W, Ma B T, Qin P, Chen W L, Wang Y P, Liu J L, Wang W M, Wu X J, Li P, Wang J R, Zhu L H, Li S G, Chen X W. A natural allele of a transcription factor in rice confers broad-spectrum blast resistance[J]., 2017,170(1):114-126.
[12] You Q Y, Zhai K, Yang D L, Yang W B, Wu J N, Liu J Z, Pan W B, Wang J J, Zhu X D, Jian Y K, Liu J Y, Zhang Y Y, Deng Y W, Li Q, Lou Y G, Xie Q, He Z H. An E3 ubiquitin ligase-BAG protein module controls plant innate immunity and broad-spectrum disease resistance[J]., 2016,20(6):758-769.
[13] Kou Y J, Wang S P. Broad-spectrum and durability: understanding of quantitative disease resistance[J]., 2010,13(2):181-185.
[14] Kou Y J, Wang S P. Toward an understanding of the molecular basis of quantitative disease resistance in rice[J]., 2012,159(4):283-290.
[15] Zhang X H, Yang S H, Wang J, Jia Y H, Huang J, Tan S J, Zhong Y, Wang L, Gu L J, Chen JQ, Pan Q H, Bergelson J, Tian D C. A genome-wide survey reveals abundant rice blast R genes in resistant cultivars[J]., 2015,84(1):20-28.
[16] 张锦文, 洪汝科, 范静华, 张祎颖,曾千春,罗琼. 一份云南地方稻广谱持久抗稻瘟病初步分析[J]. 西南农业学报, 2011,24(4):1323-1326.
Zhang J W, Hong R K, Fan J H, Zhang Y H, Zeng Q C, Luo Q. Analysis of broad spectrum and persistent rice blast resistance in Yunnan local rice variety[J]., 2011, 24(4): 1323-1326. (in Chinese with English abstract)
[17] 张锦文, 谭亚玲, 洪汝科,范静华,罗琼,曾千春. 高原粳稻子预44 抗稻瘟病基因遗传分析和定位[J]. 中国水稻科学, 2009,23(1):31-35.
Zhang J W, Tan Y L, Hong R K, Fan J H, Luo Q, Zeng Q C. Genetic analysis and gene mapping of rice blast resistance invariety Ziyu 44[J]., 2009, 23(1): 31-35. (in Chinese with English abstract)
[18] 樊琳琳, 姚波, 刘志涛, 王波,刘剑宇,王韵茜,汪秉琨,曾千春,罗琼. 子预44中抗稻瘟病基因(t)的定位[J]. 分子植物育种, 2015,13(5):961-967.
Fan L L, Yao B, Liu Z T, Wang B, Liu J Y, Wang Y Q, Wang B K, Zeng Q C, Luo Q. Identification of(t)gene conferring resistance to rice blast isolate LP33 in Ziyu 44[J]., 2015, 13(5): 961-967. (in Chinese with English abstract)
[19] 李书, 李权, 樊琳琳, 沙莎,曾千春,罗琼. 高原粳稻子预44中三个稻瘟病抗性基因的假基因化分子标记鉴定[J]. 分子植物育种, 2014,12(2):219-225.
Li S, Li Q, Fan L L, Sha S, Zeng Q C, Luo Q. Identification of the rice blast resistance genes by pesudogenization molecular markers in plateauvariety Ziyu44[J]., 2014,12(2):219-225.(in Chinese with English abstract)
[20] 周镕, 王波, 杨睿, 李书,范琳琳,曾千春,罗琼. 粳稻子预44中稻瘟病数量抗性位点分析[J]. 植物学报, 2015,50(6):691-698.
Zhou R, Wang B, Yang R, Li S, Fan L L, Zeng Q C, Luo Q. Quantitative trait locus analysis for rice blast resistance inrice variety Ziyu 44[J]., 2015, 50(6): 691-698. (in Chinese with English abstract)
[21] 胡朝芹, 刘剑宇, 王韵茜, 杨睿,汪秉琨,何月秋,曾千春,罗琼. 粳稻子预44抗LP11稻瘟病菌基因(t)的定位[J]. 植物学报, 2017,6(1):1-9.
Hu C Q, Liu J Y, Wang Y Q, Yang R, Wang B K, He Y Q, Zeng Q C, Luo Q. Mapping of(t), a gene conferring resistance to the rice blast strain LP11, insubsp.cultivar Ziyu 44[J].,2017, 52(1): 61-69. (in Chinese with English abstract)
[22] 卓晓轩, 樊琳琳, 安星宇, 郭敬玮, 杨睿, 曾千春, 罗琼. 云南地方品种子预44中一个新的抗稻瘟病基因的定位[J]. 中国水稻科学, 2019,33(1):12-19.
Zhuo X X, Fan L L, An X Y, Guo J W, Yang R, Zeng Q C, Luo Q. Mapping of a new rice blast resistance gene in Ziyu 44, a rice landrace from Yunnan Province, China[J]., 2019,33(1):12-19. (in Chinese with English abstract)
[23] Ballini E, Morel JB, Droc G, Price A, Courtois B, Notteghem JL, Tharreau D. A genome-wide meta-analysis of rice blast resistance genes and quantitative trait loci provides new insights into partial and complete resistance[J]., 2008,21(7):859-868.
[24] Chen X W, Li S G, Xu J C, Zhai W X, Ling Z Z, Ma B T, Wang Y P, Wang W M, Cao G, Ma Y Q, Shang J J, Zhao X F, Zhou K D, Zhu L H. Identification of two blast resistance genes in a rice variety, Digu[J]., 2004, 152: 77-85.
[25] Li H, Durbin R. Fast and accurate short read alignment with Burrows-Wheeler transform[J]., 2009,25(14):1754-1760.
[26] Quinlan AR, Hall IM. BEDTools: A flexible suite of utilities for comparing genomic features[J]., 2010,26(6):841-842.
[27] DePristo MA, Banks E, Poplin R, Garimella KV, Maguire JR, Hartl C, Philippakis AA, del Angel G, Rivas MA, Hanna M, McKenna A, Fennell TJ, Kemytsky AM, Sivachenko AY, Cibulskis K, Gabriel SB, Altshuler D, Daly MJ. A framework for variation discovery and genotyping using next-generation DNA sequencing data., 2011,43(5):491-498.
[28] Wang K, Li M Y, Hakonarson H. ANNOVAR: Functional annotation of genetic variants from high-throughput sequencing data., 2010,38(16):e164(https://doi.org/10.1093/nar/gkq603).
[29] 王韵茜, 苏延红, 杨睿, 李鑫, 李晶, 曾千春, 罗琼. 云南疣粒野生稻稻瘟病抗性. 植物学报, 2018, 53(4): 477-486.
Wang Y Q, Su Y H, Yang R, Li X, Li J, Zeng Q C, Luo Q. Rice blast resistance of wild rice in Yunnan., 2018, 53(4): 477-486. (in Chinese with English abstract)
[30] Ashkani S, Rafii MY, Shabanimofrad M, Ghasemzadeh A, Ravanfar SA, Latif MA. Molecular progress on the mapping and cloning of functional genes for blast disease in rice (L.): Current status and future considerations., 2014:1-15.
[31] Fukuoka S, Okuno K. QTL analysis and mapping of,a recessive gene for field resistance to rice blast in Japanese upland rice., 2001,103:185-190.
[32] He X Y, Liu X Q, Wang L, Wang L, Lin F, Cheng Y S, Chen Z M, Liao Y P, Pan Q H. Identification of the novel recessive gene(t) conferring resistance to., 2012,55(2):141-149.
[33] Shang J J, Tao Y, Chen X W, Zou Y, Lei C L, Wang J, Li X B, Zhao X F, Zhang M J, Lu Z K, Xu J C, Cheng Z K, Wan J M, Zhu L H. Identification of a new rice blast resistance gene,, by genomewide comparison of paired nucleotide-binding site-leucine-rich repeat genes and their pseudogene allelesbetween the two sequenced rice genomes.,2009,182(4):1303-1311.
Identification of Blast Disease Resistance-related Genes by Genomic Sequence Comparison of Rice Variety Ziyu 44 and Jiangnanxiangnuo
LI Jinlu#, ZHANG Hui#, JIAO Zeyu, LIU Jianyu, HAN Guangyu, ZHUO Xiaoxuan, LUO Qiong*
(State Key Laboratory for Conservation and Utilization of Bio-Resources in Yunnan, Ministry of Education Key Laboratory of Agricultural Biodiversity for Plant Disease Management, Yunnan Agricultural University, Kunming 650201, China;#These authors contributed equally to this work;*Corresponding author, E-mail: qiongbf@aliyun.com)
【Objective】Ziyu 44 is indigenousrice from Yunnan Province of Chinawith durable broad-spectrum resistance to. In order to identify novel candidate rice blast resistance-related genes from Ziyu44, 【Method】we performed the whole genome sequencing of Ziyu 44 and susceptible variety Jiangnanxiangnuo(JNXN). Then we checked and summarized SNPs/InDels of high-throughput sequencing data by software GATK(3.4-46), and screened out disease resistance-related genes with SNPs/InDels polymorphic loci at DNA level of Ziyu44 and JNXN.【Result】4118170045bp and 2995054509bp genomic data of Ziyu44 and Jiangnanxiangnuo were respectively produced using Hiseq X10 PE150 platform. The alignment rates to the reference genomes (Ensembl release 31) were 98.56% and 98.30%, respectively. A total of 922 resistance-related differential genes were identified between Ziyu44 and JNXN. Further, combined with the result of gene mapping, we identified a new blast resistance candidate gene in Ziyu44. 【Conclusion】Our results provide valuable information for cloning of new rice blast resistance new genes, and lay an important foundation for exploring the molecular mechanism of durable broad-spectrum resistance to rice blast in Ziyu44.
rice; rice blast; genome sequencing; resistance-related genes
S435.111.4+1; S511.034
A
1001-7216(2020)01-0008-09
10.16819/j.1001-7216.2020.9066
2019-06-12;
2019-06-28。
国家重点研发计划资助项目(2016YFD0100600);国家自然科学基金资助项目(31160223);云南省高校科技创新团队支持计划资助项目(IRTSTYN)。

