STIL对胃癌细胞基因表达谱的影响
王 举,窦忠霞,王永强,姜洪伟,袁宏伟
1内蒙古自治区人民医院胃肠外科,呼和浩特 0100172内蒙古医科大学病理解剖教研室(诊断室),呼和浩特 010010
中心体由一对中心粒和其周围物质组成,是动物细胞内有丝分裂期最主要的微管组织中心,参与纺锤体组装、纤毛形成和染色体迁移等重要生物学过程[1]。中心粒复制属于半保留复制,以母中心粒为模板形成子中心粒,如子中心粒不能形成,最终影响胚胎发育[2]。如果中心体过度复制,形成多个子中心粒,导致染色体分离异常,出现非整倍体,诱发肿瘤。中心粒复制相关蛋白STIL在此过程中发挥重要作用[3]。STIL从人类急性淋巴细胞白血病相关染色体重排中克隆而来[4],与鼠、斑马鱼胚胎及神经系统发育有关[5- 7]。此外,STIL诱导细胞进入有丝分裂期,加快细胞周期转换,促进细胞增殖。有报道STIL在肺癌、胰腺癌组织中呈高表达,高表达个体易发生转移且预后不良[8- 10]。前期研究显示,与癌旁相比,胃癌组织STIL表达显著增高,STIL敲减后,SGC- 7901细胞增殖能力下降,凋亡增加,表明STIL促进胃癌发生发展,但具体机制尚不清楚[11]。本研究采用基因芯片联合生物信息学分析STIL敲减后基因表达谱的变化,筛选差异表达基因(differentially expressed genes,DEGs),并对DEGs进行基因注释(gene ontology,GO)、京都基因和基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)通路富集、转录因子调控网络和蛋白互作网络等分析,初步阐明STIL促进胃癌发生发展的潜在分子机制。
材料和方法
材料人胃癌细胞株SGC- 7901购于中国科学院上海生命科学研究院细胞资源中心;RPMI 1640培养基、胎牛血清、胰蛋白酶购于美国Gibco公司;用于下调STIL的慢病毒RNAi(Lenti-KD)购于上海吉凯基因公司;总RNA提取试剂盒(HP Total RNA Kit)购于美国Omega Bio-Tek公司;第一链生成试剂盒(All-in-OneTMFirst-Strand cDNA Synthesis Kit)和实时定量逆转录试剂盒(All-in-OneTMqPCR Mix)购于美国GeneCopoeia公司;STIL和GAPDH引物由上海吉凯基因公司合成;生物素标记试剂盒(GeneChip 3’IVT Expression Kit)、杂交洗涤染色试剂盒(GeneChip hybridization Wash and Stain Kit)以及全人类基因组表达阵列(PrimeView Human Gene Expression Array)购于美国Affymetrix公司。
细胞培养及转染SGC- 7901胃癌细胞在含10%胎牛血清的RPMI 1640培养液,37℃、5%CO2恒温箱中培养。取对数生长期细胞按2×105/孔接种于6孔板,培养24 h后按说明书进行转染。实验组加入靶向STIL的RNA慢病毒干扰质粒Lv-ShSTIL,对照组加入乱码序列病毒质粒Lv-ShCon。
RNA提取、转录及探针制备继续培养48 h后,收集各处理组细胞,采用RNA试剂盒提取细胞总RNA,以Oligo(dT)为引物,采用反转录试剂盒合成cDNA第一链,再以此链为模板,采用实时定量PCR试剂盒生成双链cDNA,STIL上游引物:5’-CCCAACGCCAACTGGAGATTT- 3’,下游引物:5’-AGTCGGATGGTCTTCTCAGTC- 3’,扩增片段87 bp。 GAPDH上游引物:5’-TGACTTCAACAGCGACACCCA- 3’,下游引物:5’-CACCCTGTTGCTGTAGCCAAA- 3’,扩增片段121 bp。PCR反应体系为:95℃,预变性10 min,95℃,30 s;52℃,45 s;72℃,45 s,40个循环。再以cDNA为模板,采用因芯片表达试剂盒通过体外反转获得带生物素标记的核酸探针,将其纯化、片段化后用于后续杂交实验。
基因表达谱芯片杂交、扫描和分析采用基因芯片杂交染色试剂盒对核酸探针与基因表达谱芯片进行杂交16 h,用昂飞流体工作站450进行洗脱和染色,基因芯片扫描仪 3000 7G扫描生成图像。芯片扫描图像采用微阵列5.0软件进行数字化处理和分析。
实时定量PCR结合后续GO和KEGG分析结果,筛选显著富集于某通路的DEGs,采用实时定量PCR(quantitative real time polymerase chain reaction,qRT-PCR)验证。具体方法同RNA提取、转录,PCR反应40个循环后,使用自动PCR仪检测。采用2-△△Ct相对定量法计算ShSTIL、ShCon组各基因mRNA的相对表达水平。
生物信息学分析采用R“limma”包分析全基因组表达谱芯片检测结果,筛选DEGs,R“pheatmap”包绘制DEGs表达热图。GO分析利用DAVID 6.8(https://david.ncifcrf.gov/),KEGG富集分析采用R“clusterProfiler”包,R“ggplot2”包绘制柱状图,并采用Cytoscape软件绘图对KEGG通路及富集基因进行可视化。同样,将DEGs输入David网站,输出转录因子及调控基因文件,利用Cytoscape软件绘制转录因子调控基因网络图。此外,挑选显著富集于KEGG通路的DEGs用于后续蛋白互作分析。为构建蛋白互作网络图,于BioGRID网站(https://thebiogrid.org/)查询STIL互作蛋白并下载,联合KEGG筛选的DEGs、上游转录因子,输入String网站(https://string-db.org/)进行分析,下载蛋白互作关系文件,采用Cytoscape软件绘制以STIL为中心的蛋白互作网络图。
结 果
RNA质控结果总RNA经超微量分光光度计NanoDrop 2000检测,吸光度160/280比值在1.7~2.2;总RNA经凝胶电泳检测,RNA完整数≥7.0,28s/18s>0.7,说明样品合格,无RNA降解。两样品总RNA 28s/l8s峰值面积比值>7,说明RNA质量较好,符合基因芯片杂交检验的要求。
差异表达基因筛选结果人类全基因组表达芯片包含36 000个探针,将各处理组RNA探针与该芯片杂交,分析STIL敲减前后胃癌细胞基因表达谱的变化。结果显示与对照组相比,STIL基因沉默导致417个基因表达下降,87个基因表达上升(P<0.05,差异倍数>1或<-1)。选择差异最显著的20个DEGs,绘制基因表达热图(图1)。

ShSTIL:靶向STIL的短发夹RNA;ShCon:靶向乱码序列的短发夹RNA;DEGs:差异表达基因;红色代表高表达,蓝色代表低表达
ShSTIL:short hairpin RNA targeting STIL;ShCon:short hairpin RNA of negative control;DEGs:differentially expressed genes;red and blue colors represent high- and low-expression level,respectively
图1ShSTIL和ShCon处理组前20个DEGs热图
Fig1The heatmap of the top 20 DEGs between ShSTIL- and ShCon-treated groups
qRT-PCR验证基因芯片检测结果利用IPA(Ingenuity Pathway Analysis)在线整合分析软件,对DEGs进行上游调控分析,转化生长因子-β被预测为强烈抑制,该基因可能调控30个DEGs(SERP1、GNB4、OLR1、SRI、RAB31、SLC35A5、GLCE、CCT6A、CD46、TUBA1A、IGFBP3、MSMO1、MBNL2、BCL2L11、PTGS2、DSG2、MRFAP1L1、AHNAK、LOXL2、SMAD2、SNTB2、FBN1、CD44、ARL4A、ITGAV、PTPRK、KDELR2、PODXL、F2R、TPM1)。针对这些基因,分别设计引物,分别以Lv-ShSTIL和Lv-ShCon处理SGC- 7901细胞,提取RNA,采用qRT-PCR法检测各处理组DEGs的表达水平,证实26个DEGs表达与基因芯片检测结果相似,准确率达86.67%(图2)。
GO和KEGG富集分析结果GO细胞组成分析显示,DEGs位于胞浆、外泌体、高尔基复合体及生物膜。KEGG分析显示,DEGs参与P53信号通路、泛素介导的蛋白水解、干细胞多能性调控(图3)。以显著富集的信号通路和相应的DEGs为输入文件,采用Cytoscape软件绘制通路与基因关系图,筛选通路之间的节点(DEGs),IGF1R、GRB2和CDC42分别为hsa05205、hsa04510、hsa04550、hsa01521、hsa04144通路,hsa05205、hsa04510、hsa04550、hsa01521、hsa05206通路和hsa05130、hsa04144、hsa05205、hsa04510通路的共同节点(图4),相关通路描述见表1。
转录因子调控网络图为明确调控DEGs与其上游转录因子的关系,选择富集于KEGG通路的DEGs,利用DAVID6.8进行转录因子富集分析,筛选显著富集转录因子及调控基因,采用Cytoscape软件绘制调控网络图。图5描述了17个转录因子与DEGs的调控网络图,其中FOXO1调控CAV2、GALNT7、AP4E1、GRB2、SEC11A、PRKAG1、 PRKCI、ALG5、SMAD2、DCN、PMAIP1、TPM1、BCL2L11、CHMP2B、IGF1R、 IMMP2L、EI24、CCND3、CD44、ITGAV、RAB22A、JAK1、RCHY1、ACSL4、 FGF2、UBE2E2基因的表达。
STIL蛋白互作网络图为探讨STIL诱发胃癌的分子机制,构建蛋白互作网络图,预测STIL直接互作蛋白及信号通路。结果显示E3泛素连接酶STUB1、BTRC(FBXW1)、FBXW11、CUL1、SKP2、RCHY1、CDC46与周围蛋白连线较多,在STIL调控网络中发挥重要作用,其中STUB1、SKP2与FOXO1互作,而FOXO1是重要的转录因子(图6)。
讨 论
胃癌是严重威胁人类健康的消化道恶性肿瘤之一,发病率居恶性肿瘤第4位(仅次于肺癌、乳腺癌和结直肠癌),死亡率居第3位[12]。笔者前期研究显示,STIL促进胃癌细胞增殖、抑制凋亡[11]。为进一步探索STIL诱发胃癌的分子机制,通过基因表达谱芯片联合生物信息学分析,本研究初步证实STIL通过调控P53通路、干细胞多能性、泛素介导的蛋白水解、溶酶体等,促进胃癌发生发展。蛋白互作网络和转录调控网络分析显示STIL可能通过与E3泛素连接酶互作,抑制其活性,促进抑癌分子FOXO1的泛素化降解,从而诱发胃癌。
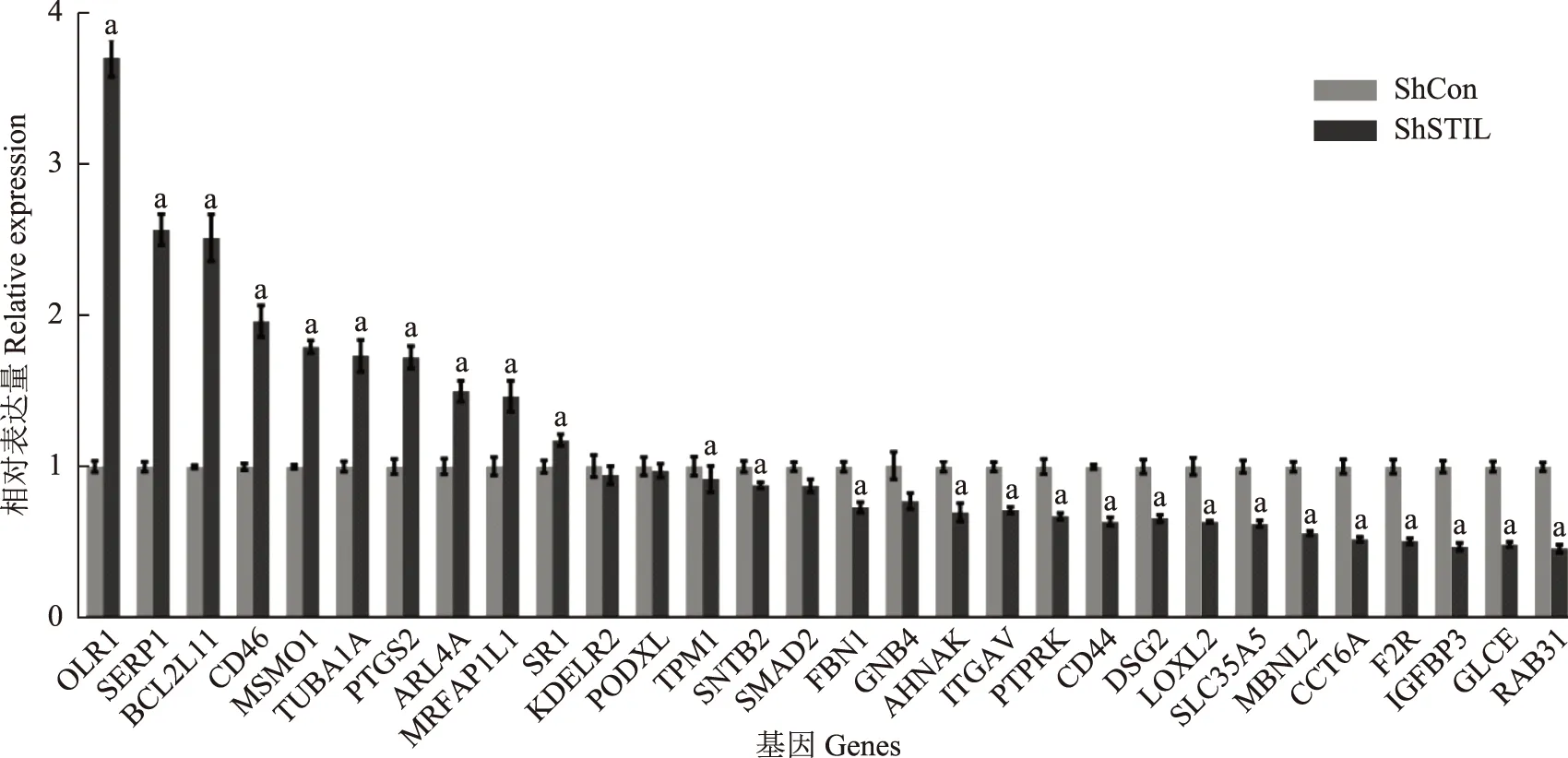
与ShCon比较,aP<0.05
aP<0.05 compared with ShCon
图2ShSTIL和ShCon处理的SGC- 7901细胞中转化生长因子-β调控基因mRNA的表达水平
Fig2The mRNA levels of genes regulated by transforming growth factor-β in the ShSTIL- and ShCon-treated SGC- 7901 gastric cancer cell line

GO:基因本体;KEGG:京都基因和基因组百科全书;ER:内质网;PEC:致病性大肠杆菌;PSC:干细胞多能性;EGFR-TKI:表皮生长因子受体-酪氨酸激酶抑制剂
GO:gene ontology;KEGG:Kyoto encyclopedia of genes and genomes;ER:endoplasmic reticulum;PEC:pathogenic escherichia coli;PSC:pluripotency of stem cells;EGFR-TKI:epidermal growth factor receptor-tyrosine kinase inhibitor
图3GO(A)和KEGG(B)富集分析
Fig3The enrichment analysis of GO(A)and KEGG(B)
中心粒复制相关蛋白STIL与肺癌、胰腺癌和结直肠癌发生密切相关[8- 9,13],笔者前期研究显示STIL在胃癌组织中呈高表达,与细胞增殖、克隆形成、细胞周期调控及凋亡有关[11]。然而,STIL诱发肿瘤的机制存在组织特异性。Kasai等[9]研究显示STIL在胰管上皮细胞的基底部少量表达,但在胰腺上皮内瘤变和胰腺导管腺癌中表达显著增高。进一步研究证实KRAS促进STIL与融合抑制因子(suppressor of fused,SUFU)N端结合并抑制其活性,而SUFU是转录因子GLI1的负性调控因子,因此GLI1被释放进入细胞核,调控相关基因表达。转录因子是一群能与基因5’端上游特定序列专一性结合,保证目的基因以特定强度、时间、空间去表达蛋白分子。基因在不同组织、时间具有表达差异性,转录因子调控发挥重要作用。同时,转录因子也是信号转导通路的靶标,也是被调控的对象。Castiel等[14]报道STIL促进检查点蛋白CHFR的自身泛素化降解,细胞G2/M转化时相缩短,增殖加快,诱发肿瘤。CHFR属于E3泛素连接酶,可介导其他底物的泛素化降解,包括Aurora A、HDAC1、TOPK、PARP1和Kif22等,这些蛋白均与恶性肿瘤的发生相关。因此,Chfr是一种抑癌基因,其启动子区域CpG岛超甲基化,导致Chfr表达沉默,与恶性肿瘤发生发展有关[15- 16]。如STIL介导CHFR发生自身泛素化降解,导致胞浆中促癌因子蓄积,诱发肿瘤。亲和捕获串联质谱分析进一步证实,STIL通过直接互作,激活泛素特异性蛋白酶- 7(ubiquitin specific protease 7,USP7)[17],而USP7可通过去泛素化,抑制CHFR的自身泛素化降解[18]。Wu等[19]敲减前列腺癌工具细胞STIL表达,细胞表型变化与笔者前期研究[11]相似,细胞增殖、克隆形成能力下降,凋亡增加,细胞信号抗体芯片显示STIL可能通过丝裂原活化蛋白激酶等信号通路调控肿瘤发生发展。
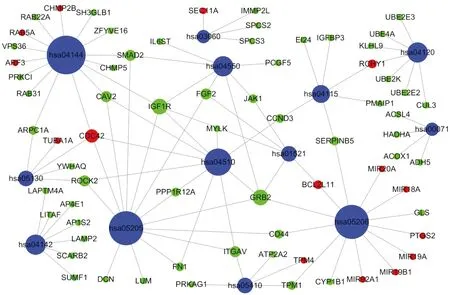
红色、绿色节点分别代表高、低表达基因,蓝色节点代表通路ID;节点大小代表连通性,连通性高的基因位于调控网络中心,即核心基因
Red and green nodes represents up- and down-regulated genes,respectively,while blue nodes represents IDs of pathways;the size of nodes represents connectivity,and the ones with higher connectivity,located in the centre of the network,are called hub genes
图4KEGG通路及相关基因
Fig4KEGG pathways and related genes

表1 KEGG通路描述Table 1 Descriptions of KEGG pathways

红色、绿色节点分别代表高、低表达基因,蓝色节点代表转录因子
Red and green nodes represent up- and down-regulated genes,respectively,and blue nodes represent transcription factors
图5转录因子调控靶基因网络图
Fig5The network of transcription factors regulating target genes
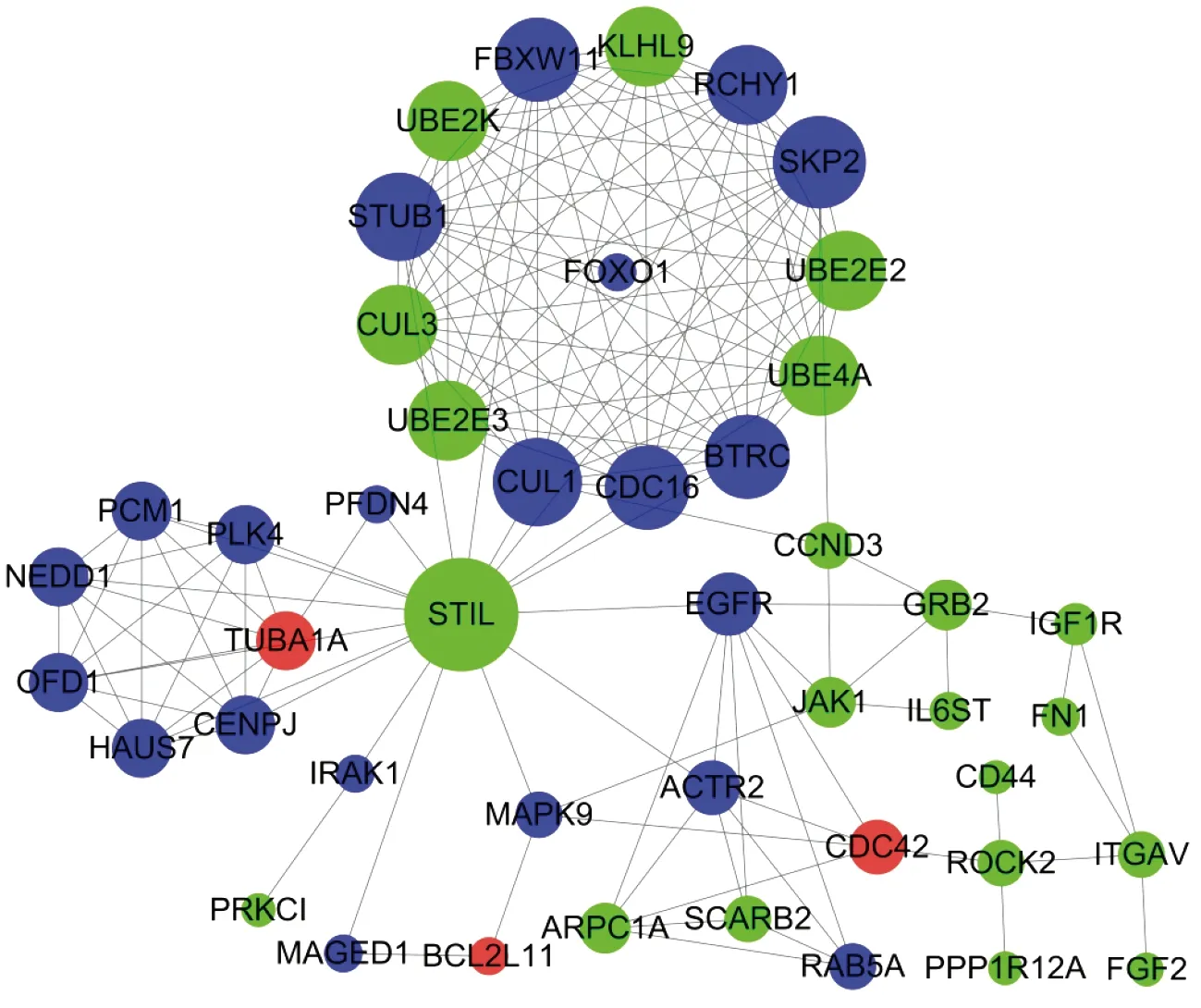
红色、绿色、蓝色节点分别代表高、低表达和无显著变化的基因,节点大小代表连通性
Red,green,and blue nodes represent up-,down- and non-significantly altered genes,the size of nodes represents connectivity
图6STIL为中心的蛋白互作网络图
Fig6Protein-protein interacting networks of STIL centred
本研究通过基因表达谱芯片串联生物信息学分析显示,STIL通过互作激活E3泛素连接酶SKP2、STUB1的活性,导致FOXO1发生泛素化降解。FOXO1属于转录因子,进入细胞核后与抑癌基因启动子结合,促进靶基因转录,负性调控消化道肿瘤的发生[20]。因此,STIL可能通过激活E3泛素连接酶,促进抑癌转录因子FOXO1的降解,促进胃癌的发生发展。

