日本结缕草衰老叶片全长cDNA文库构建及酵母单杂交筛选验证
滕 珂, 檀鹏辉, 范希峰, 岳跃森, 姜红岩, 武菊英*
(1.北京市农林科学院,北京草业与环境研究发展中心, 北京 100097; 2. 北京林业大学,草业与草原学院, 北京 100083)
植物cDNA文库是植物在特定发育时期或特定生长条件下转录的全部mRNA经过反转录而成的cDNA片段与文库载体连接而成的克隆的集合[1]。自从1976年HOFSTETTER构建了首个cDNA文库以来,cDNA文库的应用越发广泛,构建技术亦经历了多次升级[2]。经典cDNA文库受到克隆片段长度的限制,存在一定的天然缺陷[1]。全长cDNA文库可提供完整的mRNA信息,并且可以用来研究mRNA可变性剪切信息[3]。均一化cDNA文库,可增加获取低丰度mRNA的概率[4]。构建cDNA文库不仅可以保存濒危物种的基因资源,也可以用于批量克隆基因而进行基因功能研究[3]。近年来,随着分子生物学的不断发展,对基因上下游调控机制的研究已越发受到青睐。因此,获得一个高质量的cDNA文库成为深入研究基因功能及其调控机制的重要基础。
酵母单杂交系统是在酵母双杂交基础上衍生而来的,主要用来研究DNA与蛋白质的互作[5-6]。酵母单杂交的主要原理基于DNA结合域与转录激活域相互独立,将猎物蛋白融合到含有GAL4转录激活域的载体中,然后用诱饵序列调取猎物蛋白激活下游报告基因,从而达到筛选或验证的目的[7]。酵母单杂交系统的优点在于借助酵母体内表达,保证转录因子蛋白的自然结构,可更真实的模拟真核蛋白的表达模式[8]。目前,应用较为广泛的酵母单杂交系统主要有pHIS2系统和pAbAi系统;前者不需要对表达载体进行线性化进而整合到酵母基因组,具有操作方便的优点,而后者采用金担子素A(Aureobasidin A,AbA)作为筛选标记,具有灵敏度高的优点。但是,单纯的酵母单杂交筛选无法完全避免假阳性的存在,需要结合双荧光素酶(Dual-luciferase)分析和凝胶迁移(Electrophoretic mobility shift assays,EMSA)等手段进一步验证互作的真实性。
日本结缕草(Zoysiajaponica)是一种重要的暖季型草坪草,因其耐践踏、抗旱性强、耐盐等优点而广泛应用于运动场草坪的建植及城市绿化[9-11]。然而,较短的绿期成为制约结缕草在我国北方地区进一步推广应用的重要因素[12-13]。因此,从分子水平上揭示日本结缕草叶绿素降解的调控机制显得十分必要,可为延长绿期和延缓衰老提供理论依据。脱镁叶绿素脱镁叶绿酸水解酶(Pheophytin pheophorbide hydrolase,PPH)专一地催化脱镁叶绿素a脱植醇生成脱镁叶绿酸a,是调控叶绿素降解的一个关键酶[14]。但目前,有关PPH的上游调控机制仍不是十分清楚,在结缕草上更是如此。本研究旨在构建一个高质量的日本结缕草衰老叶片的全长cDNA文库,并利用酵母单杂交技术筛选ZjPPH1基因的上游调控转录因子,以期为更好地探索日本结缕草的叶绿素降解和衰老途径提供理论依据。
1 材料与方法
1.1 材料
研究所用的植物材料为日本结缕草‘Meyer’品种,其幼苗放置于人工气候箱(RXZ-380D-LED,宁波江南仪器)中培养。培养条件为28℃/25℃(昼/夜),湿度65%,光照16 h·d-1,光强600 μmol·m-2·s-1,种植基质为草炭,蛭石和珍珠岩(2∶1∶1)混合基质,培养8个月。
RNA提取试剂盒购于OMEGA公司(美国)。cDNA构建试剂盒、亮氨酸缺陷型培养基(SD/-Leu)、色氨酸缺陷型培养基(SD/-Trp)、亮氨酸-色氨酸缺陷型培养基(SD/-Leu-Trp)、色氨酸-组氨酸缺陷型培养基(SD/-Trp-His)、亮氨酸-色氨酸-组氨酸缺陷型培养基(SD/-Leu-Trp-His)等这些酵母缺陷型培养基购自Clontech公司(美国)。感受态细胞HST08、限制性内切酶SfiI购于TaKaRa公司(日本)。酵母单杂交系统为BD公司(美国)(www.bdbiosciences.com)的pHIS2系统,3-AT (3-AMINO-1,2,4-TRIAZOLE)购自Sigma-Aldrich公司(德国)。所用的电转仪为BIO-RAD公司(美国)的E.coli pulser。
1.2 方法
1.2.1总RNA的提取及全长cDNA的合成 选取健康生长的日本结缕草自然衰老的叶片,称取0.1 g,液氮中全部研磨,用试剂盒提取总RNA,得到的RNA用DNase I处理。用Nano Drop 2000C分析提取的RNA的OD 260/280和OD 260/230的比值,之后经1.5%的琼脂糖凝胶电泳进行检测,检测合格的RNA用于后续试验。首先,取1 μg RNA使用Clontech公司的SMART cDNA Library Construction Kit合成第一链cDNA;然后利用Advantage HF 2 PCR Kit (Clontech公司)取2 μL第一链cDNA进行LD-PCR扩增,合成双链cDNA。反应结束后,通过琼脂糖凝胶电泳检测cDNA合成效果。
1.2.2cDNA短片段去除 使用限制性内切酶SfiI对cDNA进行酶切处理,然后使用CHROMA SPIN-1000-TE对酶切后cDNA进行过柱处理,去除短片段。经PCI/CI净化处理后,用无水乙醇精制,最后用dd H2O 溶出。取1 μL cDNA进行琼脂糖凝胶电泳检测短片段去除效果。
1.2.3初级文库质粒的获得 使用DNA ligation Kit将pGADT7载体与适量过柱后的cDNA在12℃条件下过夜连接,将获得的连接液进行纯化精制,得到20 μL初级cDNA文库。取0.5 μL初级文库连接液,利用BIO-RAD公司的 E.coli pulser电转仪转化HST08感受态细胞(电转条件为:1.8 KV,200 Ω,25 μF)。取10 μL转化液涂布氨苄(Ampicillin,Amp)抗性的LB平板,37℃过夜培养,统计平板生长菌落的个数,按照文库滴度(cfu·mL-1)=单菌落数量×稀释倍数/涂板体积(mL)公式计算初级文库的滴度。随机挑取16个单菌落,使用通用引物pGADT7-F (5′-GGAGTACCCATACGACGTACC-3′)和pGADT7-R(5′-TATCTACGATTCATCTGCAGC-3′)进行插入片段检测。
1.2.4初级cDNA文库百万级扩增及质粒提取 根据初级文库的库容大小,取初级文库大于100万克隆的量,计算需要的连接液的体积,电转化至HST08感受态细胞中,涂布10个Amp抗性的LB大平板(24.5 cm×24.5 cm),37℃过夜培养,监测实际转化获得的扩增克隆数量。回收扩增菌落,用Nucleo Bond Xtra Midi EF试剂盒(Clontech公司)进行质粒提取,取100 ng扩增文库质粒进行1.5%琼脂糖凝胶电泳检测。
1.2.5酵母单杂交筛选cDNA文库 将前期试验克隆得到的ZjPPH1基因启动子的327 bp的序列通过同源重组的方法克隆到pHIS2载体中,获得pHIS2-ZjPPH1pro重组载体。为确定能够抑制HIS3表达背景的最低3-AT浓度,首先将pHIS2-ZjPPH1pro质粒通过LiAc的方法[15]转化Y187酵母感受态细胞,之后用0.9%的NaCl溶液将鉴定为阳性克隆的菌液OD值调到0. 2左右,然后取10 μL菌液稀释1,10,100倍,分别点在含有0,50,60,80,100,150 mmol·L-13-AT的SD/-Trp-His平板上,30℃下培养48 h。监测酵母的生长情况,然后确定最低3-AT的浓度。
在确定好3-AT浓度之后,利用共转化的方法进行酵母单杂交筛选。试验设置正对照为:pGAD-Rec2-53+p53HIS2;负对照为:pGAD-Rec2-53+pHIS2。取10 μLcDNA文库质粒(1 μg·μL-1)和5 μg pHIS2-ZjPPH1pro质粒,共转化600 μL的Y187酵母感受态细胞。用0.9%的NaCl溶液将转化后的Y187酵母感受态细胞稀释到OD值约0.2。分别取100 μL 1/10、1/100和1/1000转化后的酵母感受态细胞涂布于SD/-Leu,SD/-Trp和SD/-Leu-Trp的培养基上,以此监控转化效率。剩下500 μL酵母感受态涂布于10个含有最适3-AT的SD/-Leu-Trp-His培养基平板上,30℃下培养48 h。挑取能够正常生长的酵母单菌落,分别划线至新的含有适量3-AT的SD/-Leu-Trp-His培养基上,能够正常生长的即视为互作蛋白。最后,利用通用引物pGADT7-F和pGADT7-R进行菌落PCR,经测序后确定候选基因。
1.2.6候选基因的生物信息学分析 利用DNAMAN 8.0对PCR测序结果进行去载体片段处理,确定候选蛋白核苷酸序列。确定序列之后,制作FASTA格式文件,利用百迈客的云平台小工具(https://console.biocloud.net)进行Nr(NCBI non-redundant protein database),KEGG(Kyoto encyclopedia of genes and genomes),Cog(Cluster of orthologous genes),SwissProt (A manually annotated,non-redundant protein database),Kog(euKaryotic orthologous groups)等注释,获得候选基因的生物信息学信息。
2 结果与分析
2.1 RNA提取及质量检测
利用Nano Drop 2000C检测RNA的质量和浓度,结果显示OD260/280=2.07,OD260/230=2.13,RNA质量合格。经1.5%琼脂糖凝胶电泳再次检测RNA的质量,结果显示,18S和28S两条带清晰可见(图1),RNA质量可满足建库要求。

图1 RNA质量检测Fig.1 Quality verification of the RNA
2.2 全长cDNA合成
取5 μL cDNA进行1.5%琼脂糖电泳检测合成效果。结果显示,获得的cDNA长度分布在250~4 500 bp区间内(图2),表明已获得质量合格的cDNA,可用于下一步试验。
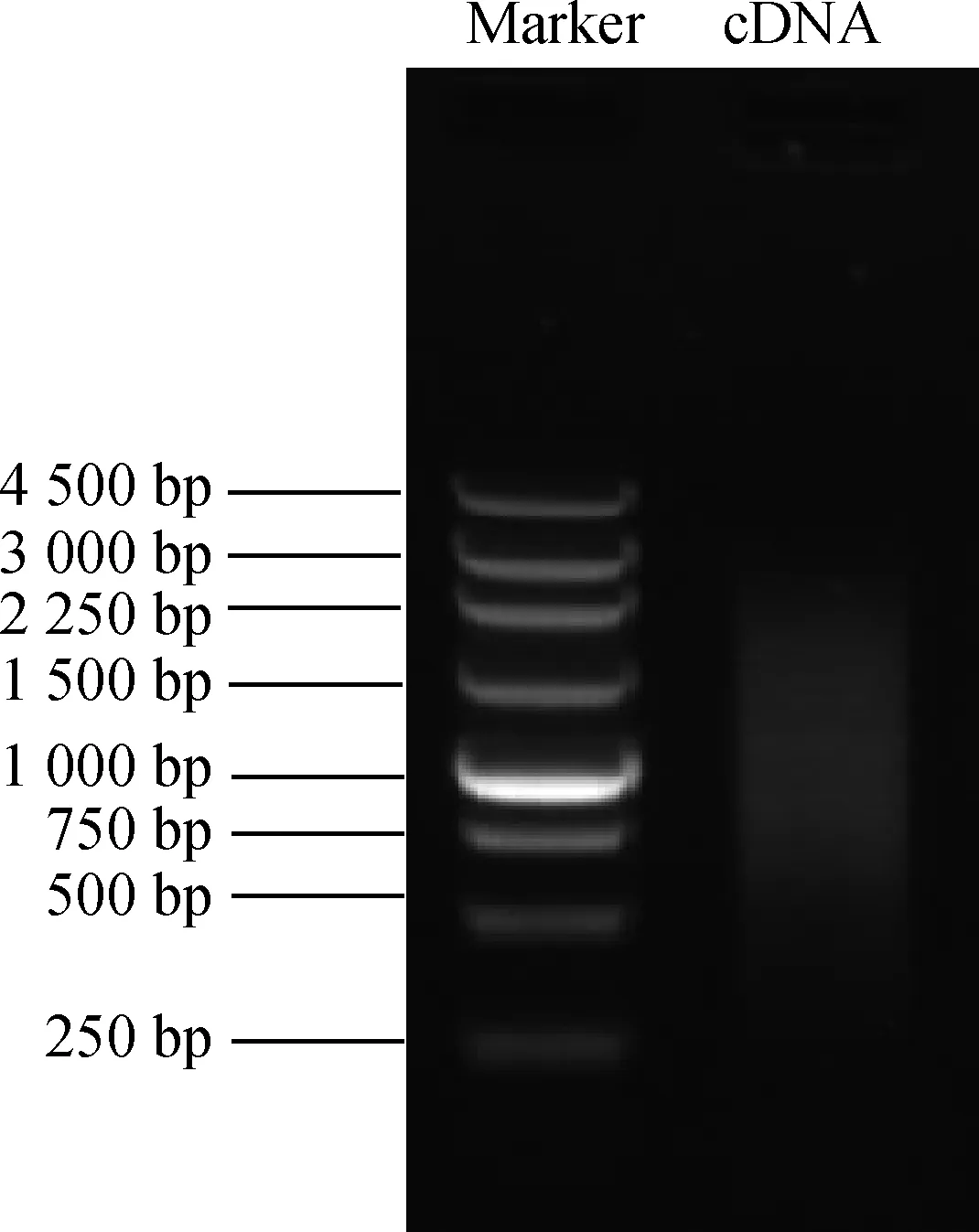
图2 全长ds cDNA检测Fig.2 Quality examination of the full length ds cDNA
2.3 短片段去除
用SfiI对上一步所获得的cDNA进行酶切处理后,利用CHROMA SPIN-1000-TE进行过柱处理,去除短片段。经PCI/CI净化处理后,取1 μLcDNA进行1.5%琼脂糖凝胶电泳。结果显示,小于500 bp的小cDNA片段和未使用完的引物、引物二聚体等得到有效去除,可用于下一步的连接转化(图3)。
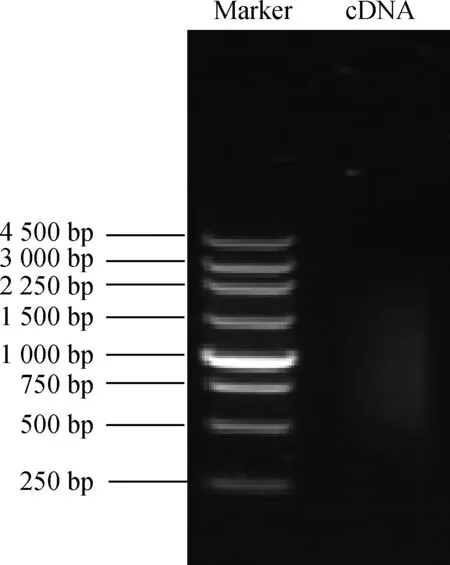
图3 短片段去除检测Fig.3 Purifying of the ds cDNA
2.4 载体连接及初级文库滴度检测
经计算,该研究所获cDNA初级文库的库容约2.0×106cfu·mL-1。随机挑取16个单克隆,利用引物pGADT7-F和pGADT7-R进行菌落PCR检测。电泳结果显示,插入片段的长度分布范围为500~3 000 bp,平均长度大于1 000 bp。16个克隆均可扩增出高亮条带,重组率为100%(图4)。

图4 插入片段检测Fig.4 Determination of the inserted cDNA segments注:1~16表示不同插入片段Note:1~16 representing different insert segments
2.5 初级cDNA文库百万级扩增及质粒提取
取初级文库约130万克隆的量电转化HST08感受态细胞,涂布10个24.5 cm×24.5 cm的LB平板,37℃过夜培养检测实际转化获得的扩增克隆数量。回收扩增菌落,使用NucleoBond Xtra Midi EF试剂盒进行质粒提取后,共获得扩增文库质粒约5 mg。取100 ng扩增文库质粒进行1.5%琼脂糖凝胶电泳,结果显示,文库质粒大小符合预期(图5)。该研究获得扩增文库质粒充足,可满足后续酵母单杂交等试验需求。
2.6 pHIS2-ZjPPH1pro质粒最低3-AT浓度筛选
为确定能够抑制HIS3表达背景的最低3-AT浓度,首先将pHIS2-ZjPPH1pro质粒通过LiAc的方法转化Y187酵母感受态细胞。用0.9%的NaCl水溶液将确定为阳性克隆的菌液OD调至0. 2左右,然后取10 μL梯度稀释,分别点在含有0,50,60,80,100,150 mmol·L-13-AT的SD/-Trp-His的平板上。30℃培养48 h后的结果如图6所示,在低浓度3-AT背景下,均发现有不同程度的酵母克隆生长;而在150 mmol·L-1的3-AT背景下,酵母生长明显受到抑制,表明150 mmol·L-1的3-AT浓度可以有效抑制背景HIS3的表达,该浓度可用于下一步的酵母单杂交筛库。
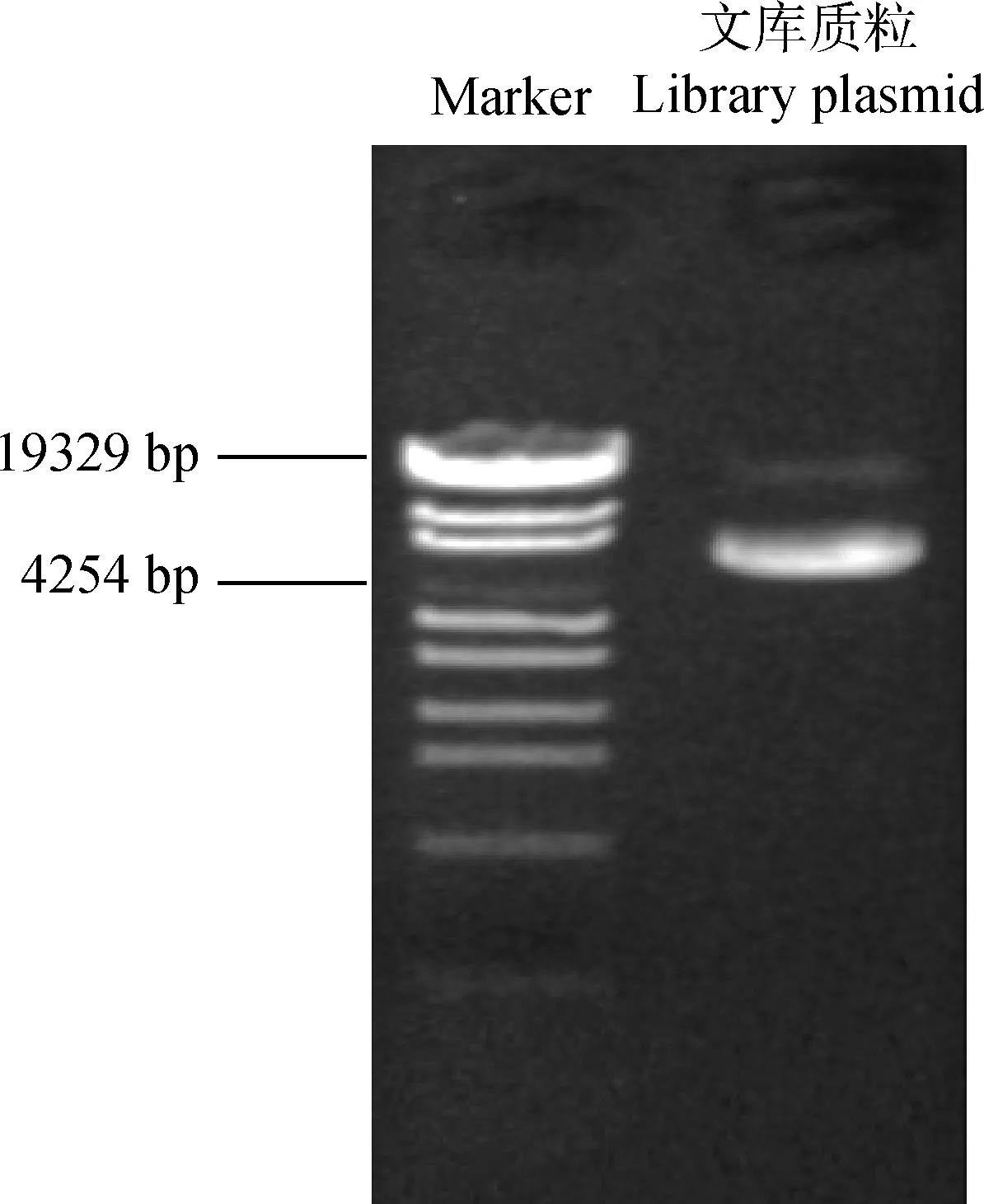
图5 文库质粒检测Fig.5 Examination of the constructed cDNA library plasmid
2.7 酵母单杂交筛选候选基因
本研究采取共转化的方法将10 μL文库质粒(1 μg·μL-1)和5 μg pHIS2-ZjPPH1pro质粒共转化600 μL准备好的Y187酵母感受态细胞。在含有150 mmol·L-13-AT的SD/-Leu-Trp-His平板上培养3 d后,检测酵母的生长情况。结果发现,阳性对照几乎长满整个平板,而阴性对照未发现有菌落长出。观察试验组的情况,发现有零星的单菌落分布。挑取48个单菌落进行菌落PCR验证,电泳结果显示,42个菌落可扩增出条带,其中有34个菌落可扩增出单一条带。另外8个含有多个条带,应该是转入了多个质粒所致,为酵母单杂交过程中的正常现象。然后挑选20个条带较亮的PCR产物送测序,分析候选基因。

图6 酵母单杂交最适3-AT浓度筛选Fig.6 Screening of the optimal concentration of 3-ATused for yeast one-hybrid screening
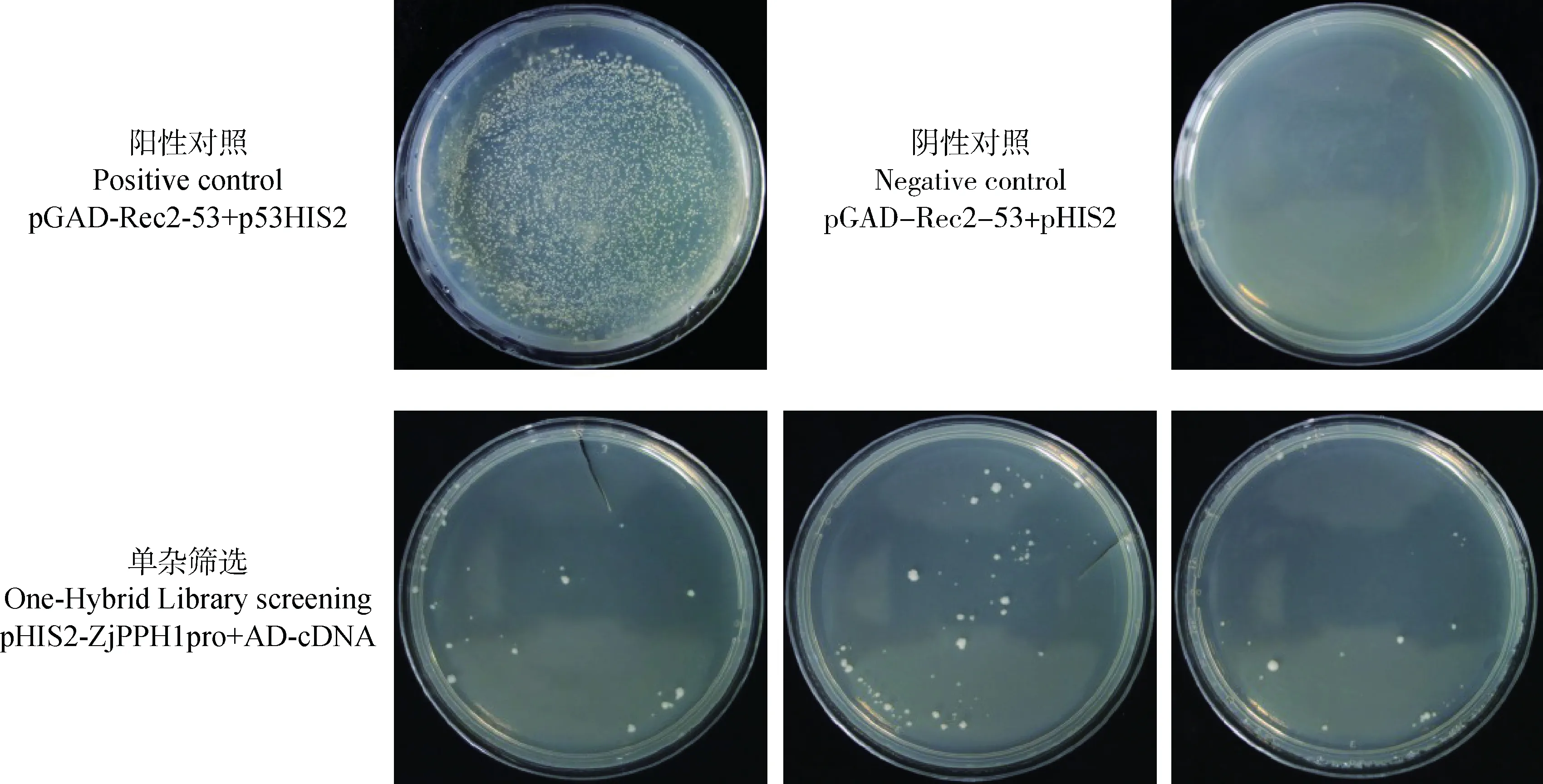
图7 酵母单杂交筛选Fig.7 Screening of yeast one-hybrid
2.8 候选基因的生物信息学分析
利用DNAMAN 8.0 去除PCR产物测序的载体序列片段后,利用百迈客云平台的基因功能注释功能对测序得到的20个候选基因序列进行注释,结果表明,20个候选基因均可注释到序列信息。Nr同源物种注释结果表明,20个注释到的基因中可比对到粟(Setariaitalica)的最多有7个,其次是玉米(Zeamays)和粳稻(OryzasativaJaponica Group)各3个。GO注释结果表明,这些基因可分为细胞组分、分子功能和生化过程3大类。在细胞过程、单一组织进程和代谢过程所含的基因数量最多。KEGG注释结果显示,这些候选基因富集到了糖酵解、柠檬酸循环、缬氨酸、亮氨酸和异亮氨酸合成、丙酮酸代谢路和丁酸甲酯代谢等通路。
图8 候选克隆的PCR检测Fig.8 PCR verification of the candidate clones注:1~48表示不同候选基因Note:1~48 representing different candidate genes
表1 候选基因的生物信息学分析Table 1 Bioinformatics analysis of candidate genes

NCBI登录号NCBI accession number候选蛋白Candidate protein比对物种Aligned speciesGO注释GO annotationMK283737假定蛋白Hypothetical protein 稻瘟菌(Magnaporthe oryzae)—MK283738线粒体丙酮酸载体Mitochondrial pyruvate carrier 二穗短柄草(Brachypodium distachyon)GO:0005743MK283739丙酮酸脱氢酶E1成分亚单位Beta 3 pyruvate dehydrogenase E1 component subunit beta-3 短花药野生稻(Oryza brachyantha)GO:0004739MK283741未知功能蛋白Uncharacterized protein 粟(Setaria italica)GO:0009941MK283742假定蛋白Hypothetical protein高粱(Sorghum bicolor)GO:0003677MK283743假定蛋白Hypothetical protein高粱(Sorghum bicolor)GO:0005840MK28374440S核糖体蛋白 40S ribosomal protein S14 粳稻(Oryza sativa Japonica Group)GO:0003735MK283740核定位序列结合蛋白Nuclear localization sequence-binding protein 粟(Setaria italica)GO:0000166MK283745重金属相关的异戊基植物蛋白Heavy metal-associated isoprenylated plant protein 粟(Setaria italica)GO:0000302MK283746依赖于ATP的锌金属蛋白酶ATP-dependent zinc metalloprotease 粟(Setaria italica)GO:0004222MK283747未知功能蛋白Uncharacterized protein粟(Setaria italica)GO:0005739MK283748假定蛋白Hypothetical protein石藻(Emiliania huxleyi)GO:0043231MG870086未知功能蛋白Uncharacterized protein 玉米(Zea mays)—MK283749SPX含蛋白结构域SPX domain-containing protein 粟(Setaria italica)GO:0005634MK283750未知功能蛋白Uncharacterized protein 玉米(Zea mays)—MK283751假定蛋白Hypothetical protein 玉米(Zea mays)GO:0016023MK283752假定蛋白Hypothetical protein 油菜(Brassica napus)GO:0005840MH428376NAC蛋白NAC superfamily protein粳稻(Oryza sativa Japonica Group)GO:0003677MH479420AP2/ERF蛋白AP2/ERF superfamily protein粟(Setaria italica)GO:0009693MK286467SOC1蛋白SOC1 protein粳稻(Oryza sativa Japonica Group)GO:0004739
3 讨论
构建高质量的cDNA文库是利用酵母系统进行单杂、双杂筛选互作蛋白的基础。坪观质量很大程度上决定了草坪草的市场价值,因此,衰老一直是草坪草研究的一个重要方向。本研究开展之前尚未发现有关构建日本结缕草衰老叶片cDNA文库的报道,而该cDNA文库的构建对揭示日本结缕草叶片衰老进程和叶绿素降解途径的研究十分重要。衡量一个cDNA文库的质量,滴度、重组率和插入片段的大小是主要的评价指标[16]。一般cDNA文库的滴度要求不少于1.0×106cfu·mL-1 [17]。cDNA插入片段的大小以及重组率是另外两个影响文库质量的重要标准,植物cDNA长度一般大于或者等于1 kb,文库平均插入片段过小会影响文库质量,而过度筛除cDNA片段则会造成初始文库的滴度过低,丢失部分基因信息[16]。本研究利用SMRT cDNA合成技术构建的cDNA文库滴度约2.0×106cfu·mL-1,插入片段平均长度大于1 kb,cDNA片段插入重组率为100%,表明该文库的完整性很高,可涵盖绝大多数基因信息,并且达到了高质量的标准,可满足后续酵母单杂交筛选的要求。
酵母单杂交技术是筛选与基因启动子特异结合转录因子的有效手段,也可以用于鉴定与DNA互作的蛋白。Chen等[18]利用酵母单杂交技术研究了拟南芥PPH基因的上游调控转录因子,并证实SOC1转录因子可直接结合PPH的CArG-box来调控叶绿素降解。Wang等[19]利用酵母单杂交筛选,发现苹果MYB22可结合UFGT和FLS基因启动子的MYBCORE元件来影响黄酮醇的合成。Ma等[20]通过酵母单杂交揭示了番茄SlNAP2通过结合SlSAG113,SlSGR1和SlPAO的启动子来调节叶片衰老。本研究利用pHIS2酵母单杂交系统,筛选到50余个可与日本结缕草ZjPPH1启动子结合的候选菌落,为研究ZjPPH1的转录调控提供了基础。
生物信息学分析表明,随机挑选测序的20个候选基因都可以在Nr数据库中获得注释。注释到的20个候选基因比对到粟(Setariaitalica)的最多。尽管结缕草基因组已公布(http://zoysia.kazusa.or.jp./),但此次分析结果显示并未比对到结缕草相关基因,这可能是由于此版本的Nr数据库不含结缕草的数据导致。根据已有研究报道并结合ZjPPH1启动子序列作用元件分析,本试验初步确定了NAC,AP2/ERF,SOC1,HGRP,CCH蛋白和40S核糖体合成蛋白作为后期试验的候选基因。前期利用PlantCARE(http://bioinformatics.psb.ugent.be)网站分析,在ZjPPH1的启动子序列中发现CGT[G/A],CArG作用元件,它们分别是NAC,AP2/ERF转录因子潜在的结合位点。此外,HGRP蛋白主要在维持细胞壁韧性和调节细胞生长中起作用[21]。CCH蛋白主要分布于衰老叶片的维管束和叶柄中,可调控铜元素在衰老组织和繁殖器官之间的转运[22]。40S核糖体合成蛋白在细胞增殖中可起到非常重要的作用,其功能的缺失可严重影响细胞的生长周期[23]。这些蛋白在日本结缕草叶片衰老和叶绿素降解中的作用和结合的作用元件仍不清楚,需要进一步的双荧光素酶系统和EMSA等试验的验证。GO注释结果显示,这些候选基因可能主要参与了细胞过程、单一组织进程和代谢等生物过程。KEGG注释结果表明,这些候选基因在糖酵解、柠檬酸循环、缬氨酸等通路中起调控作用。这些生物信息学分析结果为进一步挖掘候选基因提供了参考。
4 结论
本研究率先利用SMRT cDNA合成技术构建了日本结缕草衰老叶片的cDNA文库,其滴度为2.0×106cfu·mL-1,插入片段平均长度大于1 kb,cDNA片段插入重组率为100%。该文库的完整性较高,可涵盖绝大多数基因信息,为酵母单杂交筛选奠定了基础。
利用酵母单杂交技术,筛选到可与ZjPPH1基因启动子结合的候选基因42个,并对20个候选基因进行测序和生物信息学分析,初步筛选到6个参与细胞生长和衰老过程的重要候选基因。为进一步探索ZjPPH1基因在日本结缕草衰老和叶绿素降解中的上游调控机制提供了理论支撑。

