PCR-DGGE分析吉林市传统发酵豆酱中细菌的多样性
左丽丽,高永欣,王舒然,富校轶
(吉林医药学院 公共卫生学院,吉林 吉林 132013)
本研究以吉林市农家自制大酱为试材,在传统发酵豆酱发酵过程中,利用聚合酶链式反应-变性梯度凝胶电泳(polymerase chain reaction-denaturing gradient gel electrophoresis,PCR-DGGE)技术,结合分子生物学手段及相关软件对不同发酵周期大酱中细菌的变化规律进行分析,通过研究该发酵过程中的优势菌群及变化规律,为传统发酵大酱的制作工艺及进一步研究其品质及风味物质的形成机理提供理论支持。
1 材料与方法
1.1 材料与试剂
农家大酱:采用东北传统方法制成[13]。
甲酰胺、四甲基乙二胺(tetramethylethylene diamine,TEMED)、尿素、甲叉丙烯酰胺、过硫酸铵:美国AMRESCO公司;氨苄青霉素、X-Gal、异丙基硫代半乳糖苷(isopropyl β-D-thiogalactoside,IPTG):大连美仑生物技术有限公司;RNaseA:北京索莱宝科技有限公司;硫酸镁、丙三醇、Tris-Base、冰乙酸、氢氧化钠、氯化钠、碳酸钙:天津市大茂化学试剂有限公司;乙二胺四乙酸(ethylenediaminetetraacetic acid,EDTA):美国AMRESCO公司;琼脂:北京金泰宏达生物科技有限公司。实验试剂均为分析纯。
Ezup柱式细菌基因组DNA抽提试剂盒:生工生物工程(上海)股份有限公司;细菌通用引物27f、1495R、1492R、518R、338F(27f:5'-AGAGTTTGATCCTGGCTCAG-3';1495R:5'-CTACGGCTACCTTGTTACGA-3';1492R:5'-GGCTACCTTGTTACGACTT-3';GC-338F:5'-CGCCCGCCGCGCGCGGCGGGCGGGGCGGGGGCACGGGGGGACTCCTACGGGAGGCAGCAG-3';518R:5'-ATTACCGCGGCTGCTGG-3'):均由生工生物工程(上海)股份有限公司合成。2×TaqPCR MasterMix:购于天根生化科技(北京)有限公司。
1.2 仪器与设备
GI54DWS高压灭菌锅:致微(厦门)仪器有限公司;5430R型高速冷冻离心机、6331PCR仪:德国eppendorf公司;HZL-F100恒温双层振荡培养箱:太仓市强乐实验设备有限公司;FE20K pH计:METTLER TOLEDO公司;ChemiDoc XRS+凝胶成像系统、1645050电泳仪:美国BIO-RAD公司;GWA-UN4-C30超纯水处理系统:北京普析通用仪器有限责任公司;JNOEC XS-212-201生物显微镜:日本Olympus公司。
1.3 方法
1.3.1 样品的采集
采集1~10周不同发酵时期的传统发酵豆酱各50 g,记录发酵过程及时间等信息,置于10 mL灭菌离心管中,运回实验室于-80 ℃超低温冰箱保存备用。
随着我国经济的快速发展和人们生活水平的不断提高,人们对生活质量的要求越来越高,越来越多的人涌进城市,使得我国的城市化水平不断提高。青岛市作为我国经济发展迅速的城市,同时作为美丽的海滨城市,其人口城市化率每年都在提高(见图1)。
1.3.2 豆酱总DNA的提取
称取不同发酵周期一定质量的豆酱,置于2 mL离心管中,离心去除大部分水分,用土壤基因组DNA快速提取试剂盒提取豆酱总DNA,测定DNA的浓度,并用1%琼脂糖凝胶电泳进行检测[14-15],置于-20 ℃保存。
1.3.3 16S rDNA PCR扩增
以上述提取的豆酱总DNA进行16S rDNA扩增,豆酱总DNA为模板,27F/1492R为引物,产物长度约为1 500 bp。PCR扩增体系:DNA模板1 μL,引物27F 1 μL,引物1492R 1 μL、2×TaqPCR Master mix 12.5 μL,ddH2O 9.5 μL。PCR扩增程序:95 ℃预变性5 min;94 ℃变性1 min,58 ℃退火45 s,72 ℃延伸90 s,30个循环;72 ℃后延伸10 min[16-17]。产物经1%琼脂糖电泳验证后,作为巢式PCR的模板。
以上述的PCR产物为模板,GC338F/518R为引物PCR扩增细菌16S rDNA V3可变区间,产物长度约为250 bp。PCR扩增体系与上述相同。PCR扩增程序:94 ℃预变性5 min;94 ℃变性45 s,65~55 ℃退火45 s(每个循环降低0.5 ℃),72 ℃延伸30 s,20个循环,94 ℃变性40 s,55 ℃退火45 s,72 ℃延伸30 s,15个循环;72 ℃后延伸10 min。PCR产物经过2%琼脂糖凝胶电泳验证,作为变性梯度电泳样品。
1.3.4 变性梯度凝胶电泳
采用Bio-Rad公司的DGGE系统对上述PCR扩增产物进行电泳分离分析。聚丙烯酰胺浓度为8%,变性梯度为35%~65%,电泳条件为1×TAE 缓冲液,恒温60 ℃、恒压80 V,电泳720 min[18-19]。电泳结束后分别用固定液、银染液、显色液洗涤,使用凝胶成像系统拍照。
1.3.5 DGGE图谱分析
利用凝胶系统自带的软件quantityone对DGGE的图片进行分析,根据图片中条带的数量及灰度计算样品的Shannon-Wiener多样性指数(H),Margalef丰富度指数(D)以及Pielou均匀度指数(E),对豆酱中细菌的多样性进行分析[20]。
1.3.6 DGGE条带的回收、克隆及测序
分别切胶回收DGGE图谱中的优势条带,加适量超纯水过夜溶解片段,取上清液,以不带GC夹板的引物338F/518R扩增细菌16S rDNA V3区。反应体系及程序与带GC夹板的一致。目标片段经过2%琼脂糖凝胶电泳检测后,-20 ℃冰箱保存,作为后续克隆的目的片段。采用pMD18-T Vector Cloning Kit克隆目的片段,挑阳性克隆子培养,送生工生物工程(上海)股份有限公司测序。
1.3.7 乳酸菌16S rDNA同源性分析及种属鉴定
登录美国国立生物技术信息中心(national center of biotechnology information,NCBI)网站(http://www.ncbi.nlm.nih.gov/BLAST),对测序结果进行同源性分析,从数据库中得到相关种属的细菌16S rDNA序列信息。以16S rDNA的序列同源性>99%为标准进行属种归类。然后再对待测菌株与模式菌株16S rDNA序列利用程序MEGA7.0中的邻接(neighbor-joining,NJ)法构建系统发育树,进一步进行种属鉴定。
2 结果与分析
2.1 传统发酵大酱细菌16S rDNA、16S rDNA V3区PCR扩增结果
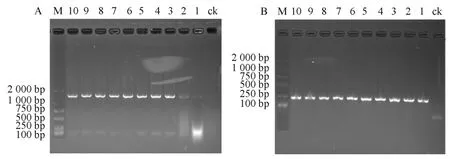
图1 细菌16S rDNA(A)、16S rDNA V3区(B)PCR扩增结果Fig.1 PCR amplification results of 16S rDNA (A) and 16S rDNA V3(B) regions gene of bacteria
以27F/1492R、GC-338F/518R 引 物PCR扩增细菌16SrDNA、16SrDNAV3区序列,分别得到大小约为1500bp、250 bp左右的目的片段,结果如图1所示,说明目的片段扩增成功。
2.2 DGGE指纹图谱分析
大酱发酵过程中1~10周细菌16S rDNA V3区变性梯度凝胶电泳结果如图2所示。

图2 豆酱发酵过程中1~10周细菌16S rDNA V3区DGGE指纹图谱Fig.2 DGGE fingerprint of bacterial 16S rDNA V3 regions during fermentation process of 1-10 weeks of soybean pastes
DGGE图谱中每一泳道条带的数量代表菌落的遗传多样性,亮度强弱代表细菌数量的多少。由图2可知,发酵前期1~5周各泳道的条带数目相近,但各条带的迁移率及亮度均有一定差异,说明发酵过程中,细菌的种类和数量发生了一定程度的变化。条带①、⑤、⑧、存在于发酵的整个周期中,且亮度比较大,对应的菌即为整个发酵过程中的优势菌。条带②、③、④只存在于泳道1、2和5中,其所对应的菌可能是发酵前期带入的杂菌,随着发酵的不断进行,菌体逐渐消失。条带出现在发酵中后期,对应的菌可能是发酵过程中产生的优势菌。
2.3 细菌多样性结果分析

表1 不同发酵周期豆酱中细菌菌群多样性分析Table 1 Bacterial diversity analysis of soybean pastes at different fermentation periods
电泳图谱中条带多少可直观反映豆酱样品中细菌种群的遗传多样性,多样性指数是研究群落物种数和个体数及其分布均匀度的综合指标。根据图谱条带信息,采用Shannon-Wiener多样性指数(H)、Margalef丰富度指数(D)以及Pielou均匀度指数(E)对细菌的多样性进行分析,结果如表1所示。
由表1可知,不同发酵周期豆酱中细菌的多样性指数、丰富度指数均表现出一定的差异,而均匀度则非常的接近。发酵第2周样品具有较高的细菌丰度及多样性,可能是由于发酵初期带入的杂菌所导致。随着发酵的进行,发酵中期(第5周后)时优势菌生长,保持菌群结构的稳定。发酵后期,细菌丰富度指数在第9周达最高,但和中期差别不显著。以上结果与DGGE图谱的直观结果一致。
2.4 测序及系统发育树的构建
细菌DGGE图谱中,选取15个标记的亮度高、分离清晰的条带,经过切胶回收、纯化连接、克隆转化、测序后,将测序结果在NCBI上进行BLAST比对,测序结果如表2所示。由表2可知,在所测的15个条带中得到的相似序列主要有戊糖片球菌(Pediococcus pentosaceu)、乳明串珠菌(Leuconostoc lactis)、葡萄球菌属(Staphylococcussp.)、屎肠球菌(Enterococcus faecium)、解淀粉芽孢杆菌(Bacillus amyloliquefaciens),是发酵周期的优势菌群,存在于发酵整个过程,而弗氏柠檬酸杆菌(Citrobacter freundii)、霍氏肠杆菌(Enterobacter hormaechei)、产酸克雷伯氏菌(Klebsiella oxytoca)、不可培养的杆菌(Uncultured bacterium)存在于发酵的中后期,对后期大酱风味的形成起到一定的改善作用。
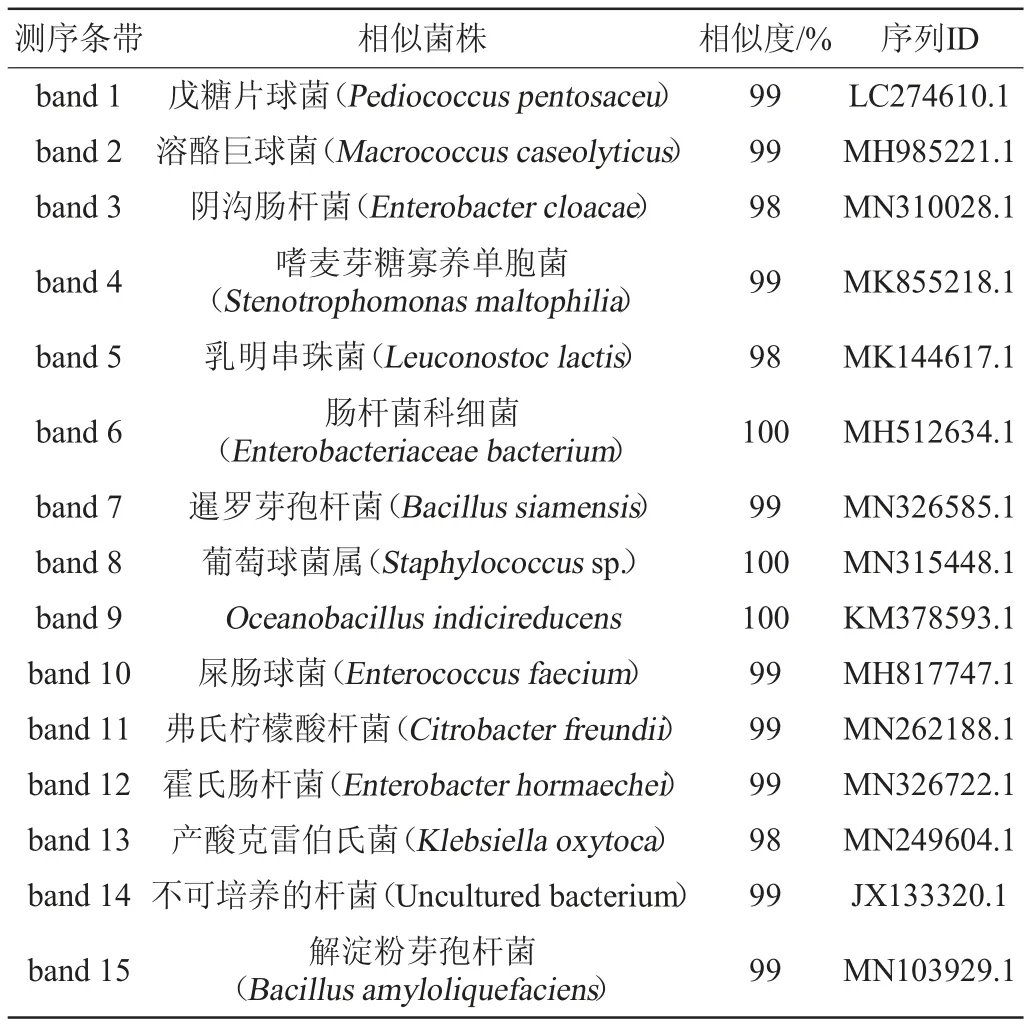
表2 DGGE优势条带测序比对结果Table 2 Sequencing alignments results of dominate DGGE bands
MEGA7.0软件构建细菌系统发育树,NJ法自展数(bootstrap)为1000,结果如图3所示,从系统发育树可以看出,所有分离菌株与相应的参考菌株同源性很高,并与其聚在一起,证明乳酸菌16S rDNA区域序列分析结果的准确性。

图3 豆酱中细菌的16S rDNA系统发育树Fig.3 Phylogenetic tree of bacteria in soybean pastes based on 16S rDNA
3 结论
本实验利用PCR-DGGE技术分析吉林市农家自制大酱不同发酵阶段细菌的生物多样性及变化规律,结果表明大酱发酵过程中细菌菌群种类较为丰富,有戊糖片球菌(Pediococcus pentosaceu)、乳明串珠菌(Leuconostoc lactis)、葡萄球菌属(Staphylococcussp.)、屎肠球菌(Enterococcus faecium)、解淀粉芽孢杆菌(Bacillus amyloliquefaciens)、弗氏柠檬酸杆菌(Citrobacter freundii)、霍氏肠杆菌(Enterobacter hormaechei)、产酸克雷伯氏菌(Klebsiella oxytoca)、不可培养的杆菌(Uncultured bacterium)。其中戊糖片球菌、乳明串珠菌、葡萄球菌属、屎肠球菌、解淀粉芽孢杆菌是发酵全程的优势菌,弗氏柠檬酸杆菌、霍氏肠杆菌、产酸克雷伯氏菌、不可培养的杆菌为发酵后期优势菌。在整个发酵过程中乳酸菌相对较多,为发酵的优势菌群,对豆酱的风味起着主要作用。结合相关分析软件表明,发酵过程中不同周期细菌的多样性指数和丰富度指数存在一定的差异,均匀度比较接近,通过系统发育树可知,各细菌之间具有一定的亲缘关系,这促进了传统发酵大酱制作工艺的发展,为进一步品质变化及风味物质的形成机理提供理论支持,同时也为大酱的进一步开发利用提供科学依据。

