基于高通量测序的棉花InDel标记开发及其应用
陆海燕,陈璐,王显生,赵涵,沈奇*
(1.江苏省农业科学院/江苏省农业生物学重点实验室,南京210014;2.盐城师范学院,江苏盐城224007)
棉花是世界上重要的纤维作物[1]。我国作为主要的棉花生产国家,棉花新品种数量持续增加,产量和纤维品质也有极大提高,促进了棉花生产的发展[2]。在新品种审定过程中,须通过田间测试评价品种的特异性、一致性和稳定性[3],工作量大,成本高,且同名异物和近似品种等问题,无形中增加了工作难度。此外,随着衍生品种的不断涌现,通过形态学鉴定棉花品种显得越来越难[4]。分子标记能有效地在DNA水平上区分材料之间的差异,反映亲缘关系,可用于辅助选择近似品种以及棉花品种的鉴定工作。
目前许多分子标记已用于棉花品种鉴定等研究中,例如随机扩增多态性DNA(Random amplified polymorphic DNA,RAPD)标记[5-8]、扩增片段长度多态性(Amplified fragment length polymorphism,AFLP)标记[9-11]、简单 重 复序列(Simple sequence repeat,SSR)[12-15]。 尽管这些标记能够在一定程度上揭示不同品种的基因变异,但是部分标记仍存在多态性低、基因型不易判别等局限性。而理想的分子标记应具有稳定性好、多态性高、操作简便、带型清晰易判别、在各基因组上分布均匀及开发使用成本低等特点[16]。
InDel(Insertion-deletion)是基于聚合酶链式反应(Polymerase chain reaction,PCR)扩增技术的碱基序列长度多态性标记[17]。大量研究表明,InDel标记不仅具有分布广、重复性好、开发成本低、结果准确等优点,而且基因型判别简单快速[18]。InDel标记已经被认定为1个重要的分子标记来源,并成功应用于水稻[19-20]、玉米[21-22]等作物的连锁图谱构建和品种鉴定。徐鹏等开发了陆地棉耐盐相关的功能InDel标记,能够用于辅助育种改良陆地棉等耐盐性[23];Zhang等[24]开发了一个能用于区分携带核不育修复基因的恢复系和不携带该基因等位基因的其他基因型的InDel-R标记,而InDel标记用于解析棉花的遗传组成差异、棉花品种鉴定和纯度检测等方面的报道较少。目前,棉花基因组二代测序数据提供了丰富的In-Del变异位点,为开发InDel标记提供了便利。通过生物信息学手段筛选、过滤测序数据,评价多态性,能够挖掘有效的InDel分子标记。
本研究拟采用来源不同的121份棉花全基因组信息,根据高多态性信息含量(Polymorphism information content,PIC)筛选多态性高的InDel位点,基于部分多态性高的InDel位点,开发二态性InDel标记,并在66个棉花主栽品种中进行验证;通过基因型数据构建系统发育树及对所用棉花品种的背景和来源进行遗传结构推断,以获得可以用于棉花品种鉴定和纯度检测的多态性In-Del标记,提高种子检验的精确度和效率,并在棉花的育种中发挥作用。
1 材料与方法
1.1 棉花基因组序列来源及InDel位点的筛选
从 NCBI(www.ncbi.nlm.nih.gov)下载 121 份棉花序列,删除经Q20过滤后保留的碱基数低于棉花理论基因组大小的材料。采用编写的Perl脚本和VCFtools软件计算分析最小等位基因频率(Minimum average allelic frequency,MAF)和最终保留PIC,选择MAF大于0.05的位点,删除基因型缺失率超过20%的位点,同时以哈迪温伯格平衡显著性阀值(Hardy-Weinberg equilibrium,HWE)P<0.001为标准再次过滤,最终保留PIC>0.4的二态性InDel变异位点[21]。
1.2 棉花品种来源
选取来源于我国黄河流域与长江流域棉区的66个主栽棉花品种为研究材料,于2018年种植于江苏省农业科学院实验基地,采集嫩叶提取DNA,用于 PCR 验证(表 1)。
1.3 InDel标记开发和筛选
InDel标记开发:根据InDel位点和侧翼序列设计PCR扩增引物,开发InDel分子标记。每个标记包含上下游(F、R)2条引物,引物设计由mInDel软件包[25]完成,设置参数Tm为(58±3)℃,引物长度为(20±3)bp(base paris),PCR 产物预测长度为100~450 bp,其他参数为默认。引物由上海生工生物科技有限公司合成。
棉花基因组DNA提取和PCR扩增:取棉花幼嫩叶,按照上海浦迪植物基因组提取试剂盒操作步骤提取基因组DNA。PCR反应总体系为12 μL, 包含上下游引物各 0.5 μL(浓度 为10 μmol·L-1)、6 μL 2 ×TaqMaster Mix、3.2 μL ddH2O、模板 DNA 1.8 μL。PCR反应程序:第 1步,94 ℃ 3 min; 第 2 步,94 ℃ 30 s,58 ℃ 30 s,72℃ 30 s,共进行 35个循环 ;第 3步:72℃ 5 min。PCR扩增产物在20 g·L-1的琼脂糖凝胶上进行电泳分离,经溴化乙锭(EB)染色,用凝胶成像仪拍照、记录。

表1 试验材料详细信息Table 1 Details of experimental materials
1.4 多态性InDel位点遗传多态性分析
凝胶上的带型读取方式:短片段读成A、长片段读成B、2条带型读成H。根据基因型分型结果,以及标记在棉花基因组染色体上的分布等信息,挑选有效的InDel标记。采用编写的Perl脚本和VCFtools软件计算分析二态性InDel标记的MAF、PIC[26]。利用TASSELV5.0软件的邻接算法(Neighbor-Joining,N-J)[27]计算品种之间的遗传距离并构建聚类图。
2 结果与分析
2.1 棉花多态性InDel位点信息分析
通过同源对比过滤121份棉花基因组序列,共筛选出10 967个二态性InDel位点(缺失率<20%,MAF>0.05)。 物理定位结果显示,InDel位点主要分布在基因间区。
2.2 棉花二态性InDel标记的开发和鉴定
为了便于电泳技术检测目标InDel位点差异,选择生物信息学分析获得的大于20 nt的In-Dels,并结合其在棉花基因组上的位置信息,在这些位点附近选择合适的位点转化为InDel标记。本研究选取85个位点合成引物,用于检测InDel标记的多态性,其中有52对引物是棉花基因组At亚组的特异引物,33对是Dt亚组的特异引物。用85对引物分别扩增66个主栽棉花品种DNA,结果显示:64对引物能扩增出清晰的带型(其中部分引物信息见表2),在不同品种之间有明显的多态性。以引物JSC009、JSC071为例,在66个品种DNA中,均能扩增出3种带型:A/B/H(图1),且产物大小与预测结果相吻合。
对扩增有效的64对引物位点的多态性信息统计结果:At亚组染色体的MAF变化范围为0.15~0.49,平均值为 0.45;Dt亚组染色体的MAF变化范围为0.14~0.49,平均值为0.32。At亚组染色体的PIC变化范围为0.16~0.49,平均值为 0.49;Dt亚组染色体的 PIC变化范围为0.18~0.49,平均值为 0.40(图 2)。 结果表明,64对引物具有较高的特异性和稳定性。

图1 高多态性InDel标记JSC009和JSC071对66个棉花品种DNA扩增产物的琼脂糖凝胶检测结果Fig.1 The agarose gel electrophoresis results of DNA PCR products with high polymorphism InDel markers JSC009 and JSC071 in 66 cotton cultivars

表2 部分InDel标记信息Table 2 The information of partial InDel markers

表2(续)Table 2(Continued)
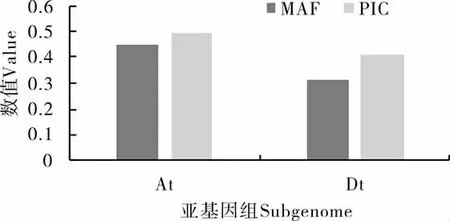
图2 64个InDel标记的遗传多态性Fig.2 The genetic polymorphisms of 64 InDels
2.3 InDel分子标记在棉花品种鉴定中的应用
基于64个InDel分子标记的电泳结果统计66个棉花品种的基因型,计算其遗传距离。结果(表3)表明,所用棉花品种的遗传距离范围是0.04~0.65 cM(centimorgan,厘摩),平均为 0.39 cM,表明所用的棉花品种具有丰富的遗传多样性,能够用来验证64个InDel分子标记特异性。遗传距离最大的2个品种是泗棉3号(编号为49)和中棉所 36(编号为 43),遗传距离为 0.65 cM,表明二者的遗传差异较大,亲缘关系较远;遗传距离最小的是徐棉18(编号为8)和徐杂3号(编号为9),遗传距离为0.04 cM,表明两者遗传差异较小。
66个棉花品种的系统发育树(图3)显示,在Group 1中,来自河南的新植5号、豫杂35、豫杂37、中棉所12、中棉所45聚在一起;鑫秋1号和鑫秋4号聚集,通过对这2个品种遗传系谱查询表明,两者均来自山东;同样来自河南的郑农棉4号、郑育棉2号、郑杂棉2号聚类在一起。Group 2群体结构显示,来自徐州的2个品种,徐棉18和徐杂3号聚类,来自湖南的湘K27、湘XP63、湘K26聚类在一起。在Group 3中,同样来自河南的银山2号、豫棉13、中棉所17、中棉所19聚
类在一起。具有相同地理来源的多数品种聚为一类,但也有例外,如安徽省的2个品种,绿亿棉19划分在Group 1中,思福棉1号划分为Group 2;江苏省的其他7个品种散落于3组。
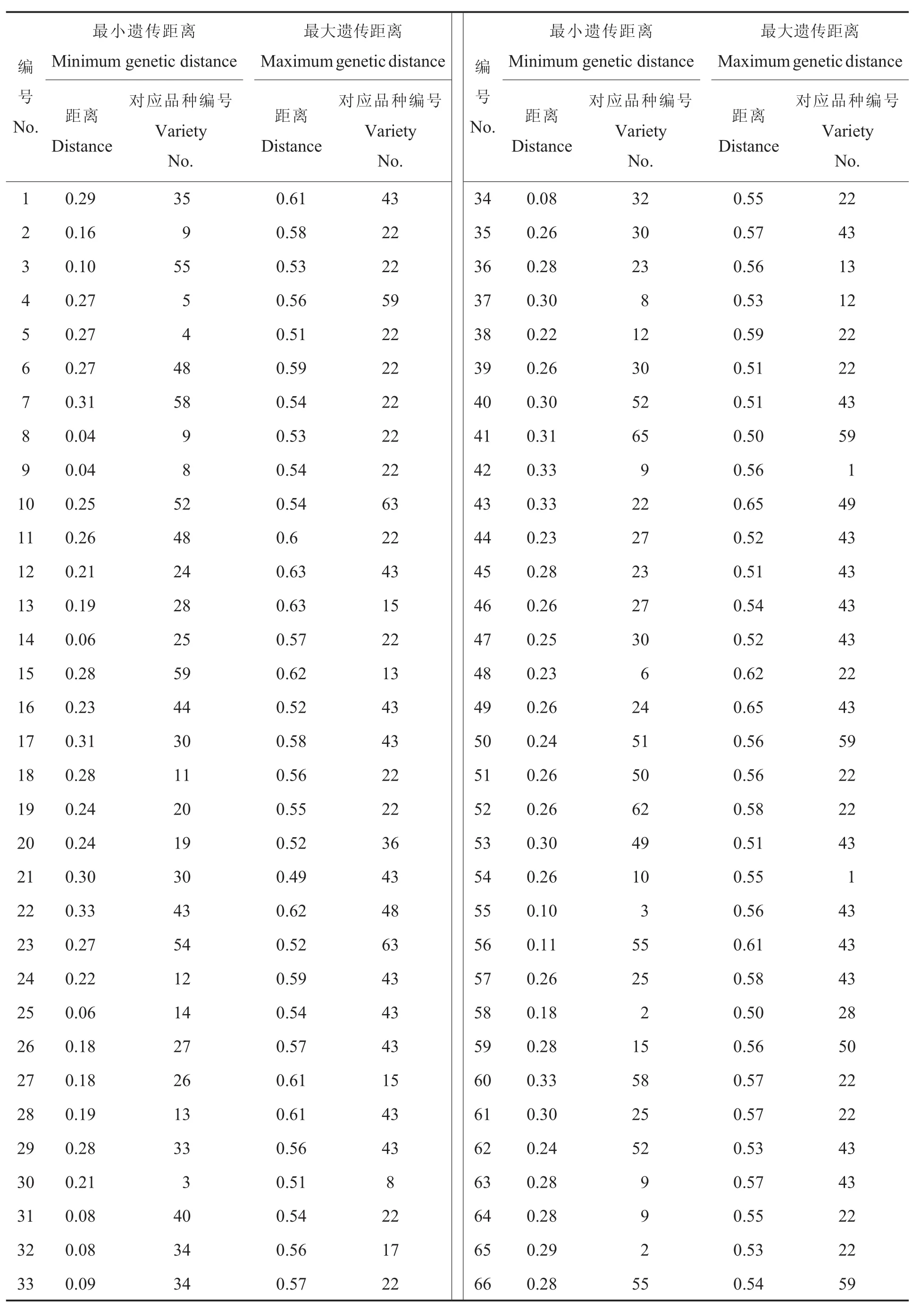
表3 遗传距离统计结果Table 3 Statistics of genetic distance
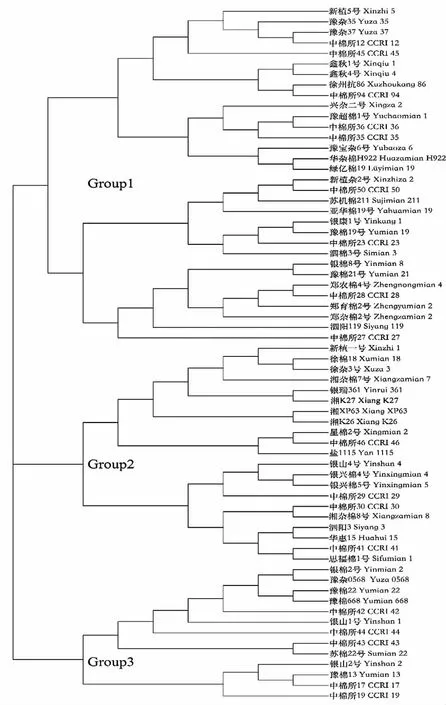
图3 基于邻接算法构建的66个棉花品种系统发育树Fig.3 Phylogenetic tree of 66 cotton cultivars based on the Neighbor-Joining method
3 讨论
在分子水平上揭示不同材料之间的遗传差异,方法易行,结果可靠,不受外界环境条件影响。分子标记技术可在棉花生育早期检测,准确鉴定棉花品种间的差异,对于提高种子纯度检测效率有重要意义。Jia等[28]利用80个全基因组SSR标记,对197份亚洲棉进行了基因分型,建立了亚洲棉的遗传多样性和种群结构模式。郭旺珍等[5]利用18个RAPD标记,对我国9个棉花主栽品种的基因组进行PCR扩增,有13个标记在品种间具有多态性,证明了RAPD标记在品种纯度鉴定的可行性;Abdalla等[11]利用16个AFLP引物组合对我国2种四倍体棉种和3种二倍体棉种进行了研究,结果表明,AFLP标记的多态性明显高于RAPD标记;匡猛等[1]用36对SSR引物扩增32个材料DNA,共扩增出142种基因型,平均每对引物扩增出3.94种基因型。本研究用64对InDel引物扩增66个材料的DNA,每对引物均呈现二态性,提高了基因型判读的准确性和效率。
目前,在水稻、人类等物种中,InDel的相关研究越来越受到关注[29-30]。InDel标记已被公认为重要的遗传标记资源,应用于高密度遗传图谱的构建、全基因组关联分析等研究中。二态性InDel标记具有稳定遗传、多态性高、共显性等优点,通过简单的PCR及凝胶电泳即可进行基因分型。但是二态性InDel标记在检测多倍体物种中,用一对引物不能实现该位点多种不同变异的检测,因此最终产生的二态性InDel标记组合需要覆盖整个基因组,才能保证品种鉴定的准确性。
本研究所用棉花是异源四倍体,基因组的结构变异更为复杂。棉花二态性InDel标记的获得对揭示棉花进化以及品种鉴定有着巨大的价值。本研究中,基于棉花基因组序列,采用生物学手段筛选,挖掘10 967个InDel位点,这些位点覆盖棉花基因组的关键区域。根据InDel标记基因分型特点及位点的多态性要求,合成85对InDel引物,根据基因分型结果,选择其中高PIC的引物,共64对。通过对66个棉花材料聚类分析发现,具有相同地理来源的品种有时会最先聚为一起,例如徐棉18和徐杂3号,这与Liu等[31]认为SSR聚类结果与地理分布成正相关基本一致。本研究表明,地理来源相近的多数品种聚为一类,来自同一种植区的栽培种亲缘关系较近,遗传距离和聚类分析结果基本上反映了品种之间的亲缘关系。棉花At、Dt基因亚组在驯化和选择过程中的进化是独立的,各基因组的变异能够反映整个棉花的基因组变异。
4 结论
本研究基于66个国内主要棉花品种,开发针对棉花At、Dt基因亚组的特异性InDel标记,不仅容易检测,而且能很好地反映棉花之间的差异,有助于棉花品种的检测以及能够在棉花分子育种方面提供DNA水平上的信息。

