棉花CLE多肽家族的全基因组鉴定与生物信息学分析
袁娜,李阳,杨郁文,张保龙,杜建厂
(江苏省农业科学院种质资源与生物技术研究所/江苏省农业生物学重点实验室,南京210014)
植物激素是植物生命活动中必不可少的内源物质,也是调控植物生长、发育、衰老等主要生理过程的一类重要有机分子。近年来,随着遗传学和分子生物学的发展,人们在传统植物激素的信号转导、激素间的相互作用和激素生物合成的调控等方面取得了较多的进展和突破[1]。自1991年在番茄中发现了植物第一个多肽激素——系统素(Systemin)后,越来越多的研究陆续从植物中分离出了多种多肽激素,并且发现它们参与植物的生长发育及抗逆等许多生命过程,特别是作为一种重要的信号分子,在细胞与细胞之间的短距离信息交流中起着关键作用[2]。
一般认为,植物多肽激素是一类长度小于100个氨基酸,具有特殊功能的成熟多肽分子,通常在植物体内含量较低[3]。到目前为止,在植物中已有几十类多肽激素被鉴定出来[4-5]。根据其来源方式,植物多肽可以分为非功能前体蛋白来源、功能前体蛋白来源和非前体来源[6]。例如目前研究较为广泛的系统素(Systemin)[7]、CLV3/Embryo Surrounding Region-Related(CLE)[8]、Tracheary Element Differentiation Inhibitory Factor(TDIF)[9]、Phytosulfokine(PSK)[10]、Plant Peptide Containing Sulfated Tyrosine1(PSY1)[11]、C-Terminally Encoded Peptide(CEP)[12]等,均来源于非功能前体蛋白,经过羟基化、糖基化和硫酸盐化等翻译后修饰形成的多肽。近年来,也有部分研究发现,功能蛋白也可以作为多肽分子的来源,例如Chen等[13]基于多肽组学在番茄中发现防御蛋白PR-1来源的CAPE1。此外,随着生物信息学的发展,Hanada等[14-15]利用其开发的sORF finder软件在拟南芥基因组中预测到近万条潜在编码多肽的分子序列,并且通过芯片验证,发现有2 099 sORFs是真实表达的。植物多肽激素作为信号分子在细胞与细胞之间短距离的信息传递过程中发挥着重要作用。细胞膜表面存在与多肽激素相对应的受体激酶,当感受到多肽激素信号时受体激酶会调控下游基因的表达从而引发相应的一系列信号转导过程[16]。目前研究发现,植物多肽类激素可以影响植物的生长发育,例如多肽PSK可以控制细胞的增殖;IDA可以调控根生长;CLV3则参与植物干细胞分化调控等[6]。同时,多肽激素也能在植物应对生物胁迫及非生物胁迫过程中起到重要的作用,例如CEP和CAPE作为负调控因子可以调节植物抗盐胁迫反应[17]。此外,还有研究发现多肽激素能够在体内长距离的调节组织与组织之间的交流,例如CEP1多肽能够在氮饥饿时调节植物根的生长[18]。
CLE家族是迄今为止最大的植物多肽分子家族,也是近十年来研究最为热门的植物多肽激素。其调节植物的各种生理和发育过程,尤其对调控分生组织中细胞的分裂和分化起关键作用[19]。1997年,CLE基因在玉米中作为胚乳特有的基因首次被报道,其名称源于拟南芥CLAVATA3(CLV3)和玉米Embryo Surrounding Region(ESR)这两个基因[20]。目前在拟南芥中已鉴定了34个CLE基因家族成员,其编码的CLE蛋白较小,通常小于100个氨基酸,N-端有一段分泌信号肽(SP),C-端有一段高度保守的包含14个氨基酸的 CLE 结构域(CLE domain)[21]。目前,CLV3(CLAVATA3)是在植物分生组织中研究最为清楚的一个调控植物发育的多肽激素,其终产物是一个含有12个氨基酸的多肽。遗传学与生物化学相关研究表明,CLV3作为配基激活CLV1/CLV2受体复合物后,将CLV3信号从一个细胞传递到邻近的细胞,通过一系列的中间过程,限制同源异型结构域转录因子WUS(WUSCHEL)的表达。与此同时,CLV信号复合体与WUS转录因子之间形成一个非常精确的反馈调节环,是控制茎端分生组织中干细胞活性及分生能力的关键因素[22-23]。根据表达模式和结构的差异,不同的CLE成员在拟南芥中发挥着不同的生物学功能。例如CLE3能够感受低氮信号,作用于下游受体CLV1进而抑制侧根的发生[24];CLE41经韧皮部细胞分泌后与原形成层的PXY结合,进而调控维管干细胞的分裂[25]。2018年,Shinozaki研究小组在Nature上发表论文,发现CLE25可以在根部感受缺水信号,并通过维管束长距离运输到叶片中,通过与叶片的BAM受体结合后,调控ABA的生成和气孔的关闭,首次证明了多肽能够作为长距离的分子信号调节非生物干旱胁迫[26]。总之,近年来若干里程碑式的研究使人们认识到,CLE多肽激素及其介导的信号通路在调控植物分生组织中起着重要作用。开展CLE多肽激素调控植物分生组织干细胞分裂与分化调控途径的研究,对于了解植物生长发育、组织器官发生以及形态建成等分子机制具有重要的理论意义。
棉花是世界上最重要的经济作物之一,是生产天然纤维的重要的纺织工业原料。然而,随着环境不断恶化,棉花在其生长区域经常遭受着干旱、高盐、低温等非生物以及病害等生物胁迫的侵袭,严重影响着棉花的生长发育和产量[27]。CLE多肽激素通过参与植物分生组织的分裂和分化,可以调控作物的株型,以及作物对生物和非生物胁迫的适应性等,对于作物产量的形成与品质的保持起着至关重要的作用。研究发现,植物寄生虫如囊状线虫,可以分泌类似寄主CLE多肽的效应蛋白,通过沉默植物体中的CLE多肽受体,从而增强植物对大豆囊状线虫的侵染[28]。水稻、玉米、番茄以及烟草等诸多重要的粮食和经济作物的基因组中均存在高度保守的CLE基因[29],然而目前在棉花中,尚未开展CLE基因家族的鉴定及相关研究。随着雷蒙德氏棉(Gossypium raimondiiUlbrich)[30]、亚洲棉(G.arboreumL.)[31]、陆地棉(G.hirsutumL.)[32-33]和海岛棉(G.barbadenseL.)[34]基因组测序的完成,为我们挖掘和鉴定多肽基因,也为全基因组水平分析棉花中重要基因家族的演化提供可能。因此,对棉花中CLE多肽家族的鉴定及演化研究,可以为进一步的挖掘植物多肽在棉花生长发育和应对环境胁迫过程中的功能及其信号转导研究提供一定的理论基础,最终为棉花分子育种工作提供有效的理论指导。
1 材料与方法
1.1 棉花CLE基因的查找与鉴定
在拟南芥TAIR10数据库(https://www.arabidopsis.org/)中,用CLE基因的相关注释“CLAVATA3”做关键词搜索,找出并核对全部拟南芥CLE基因(AtCLE),根据其基因的Locus Name,获得拟南芥的CLE peptide sequence。随后,利用AtCLE的peptide sequence分别对雷蒙德棉[30]、亚洲棉[31]、陆地棉[32]和 海岛棉[34]基因组(https://www.cottongen.org/)进行 BLASTP 检索,E值≤10-10的基因作为候选基因。同以上方法,再用候选基因的peptide sequence进行二次查找,确保没有遗漏。最后,根据候选基因的Locus Name,找出基因的Genomic sequence、Transcript sequence、CDS sequence 和 Peptide sequence, 同时获取基因的大小、染色体位置、内含子数量以及转录方向等信息,全部保存于文件。候选基因确定后,再根据CLE基因家族的结构特征,通过对候选基因的N端信号肽预测,C端CLE基序查找等最终确定棉花的CLE基因。
1.2 棉花CLE基因结构和蛋白序列分析
利用ExPASy Proteomics Server软件(http://www.expasy.org/)对已鉴定的棉花CLE基因家族成员的氨基酸序列进行理化性质分析;利用在线 预 测 工 具 SignalP4.1 Server(http://www.cbs.dtu.dk/services/Signal P/),对棉花CLE基因进行信号肽预测。利用在线预测工具GSDS v2.0(http://gsds.cbi.pku.edu.cn/)分析和绘制棉花CLE基因家族成员的基因结构图。利用在线软件MEME(http://meme-suite.org/)对CLE蛋白保守结构域进行分析。基序最大发现数量设置为5,其他参数为默认值。
1.3 CLE基因染色体定位及基因同源性分析
根据棉花CLE的基因位置信息和棉花染色体的长度,使用TBtools[35]和Adobe illustrator CS3[36]软件绘制其染色体分布图。根据蛋白序列比对结果识别同源基因对,经过比对后,如果短序列能覆盖超过长序列70%的区域,则认为这对同源基因为重复基因(duplication genes)。利用InParanoid[37]软件对4个棉种的CLE基因的直系同源性关系进行分析。利用KaKs_Calculator 2.0[38]软件计算同源基因对的Ka(非同义突变率)、Ks(同义突变率)值,明确其在演化过程中受何种选择。
1.4 多序列比对与系统进化树的构建
利用MEGA 7.0[39]软件对获得的CLE蛋白序列进行多重序列比对。以多序列比对结果为基础,采用邻接(Neighbor-Joining)算法构建系统进化树,Bootstrap 重复测试(Replications)设置为1 000。
1.5 CLE基因表达热图谱构建
在NCBI SRA数据库(http://www.ncbi.nlm.nih.gov/sra/)下载亚洲棉的叶片、幼苗,雷蒙德氏棉的叶片、花瓣,陆地棉的茎、叶片、花瓣、花托以及海岛棉的茎、叶片和花瓣转录组测序数据,下载 序 列 号 分 别 为 SRR530430,SRR952685,SRR389183,SRR943769,SRR1695174,SRR169-5175,SRR1695177,SRR1695176,SRR1652331,SRR1652333,SRR1652334。基于以上数据,利用Tophat[40]和Cufflink[41]软件包分析转录组测序数据并进行表达量计算,提取CLE基因家族成员的基因表达数据,将表达数据标准化后,使用HemI(Heatmap Illustrator,Version 1.0)[42]软件绘制热图将表达数据可视化。
2 结果与分析
2.1 棉花CLE基因家族成员的鉴定
利用拟南芥中已鉴定的CLE蛋白序列和上述方法,我们最终共获得148个棉花CLE基因,其中亚洲棉有21个、雷蒙德氏棉有26个,陆地棉和海岛棉分别有49和52个,分别命名为GaCLE1~GaCLE21、GrCLE1~GrCLE26、GhCLE1~Gh-CLE49和GbCLE1~GbCLE52(表1)。 对棉花CLE多肽基因家族的理化性质分析发现,该家族编码的蛋白长度在73~453个氨基酸,最大和最小分子量分别为47 996.26 Da和8 276.52 Da。GaCLE8的等电点最小,为 6.82;GbCLE23和GbCLE46的等电点最大,为 12.22,平均值为10.48,表明该家族蛋白偏碱性。信号肽预测发现148个棉花CLE多肽中,103个具有信号肽结构,45个没有预测到信号肽的存在。亚洲棉、雷蒙德氏棉、陆地棉中无信号肽的CLE多肽分别占各自总数的38.1%,42.3%和40.8%,海岛棉中无信号肽CLE最少,仅占总数的11.5%。

表1 棉花CLE基因家族成员信息Table 1 Information of the CLE genes identified in cotton

表1(续)Table 1(Continued)

表1(续)Table 1(Continued)

表1(续)Table 1(Continued)
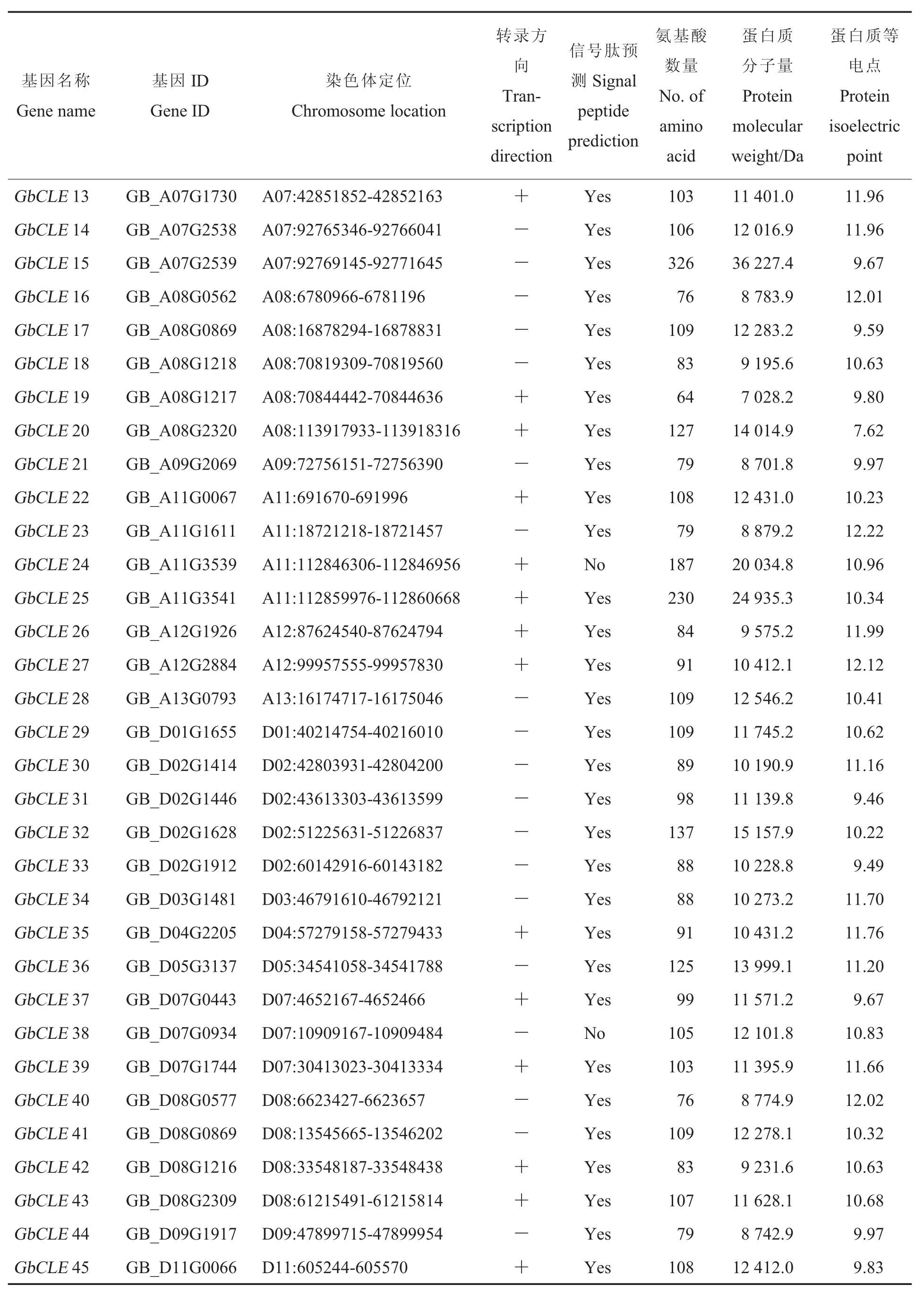
表1(续)Table 1(Continued)

表1(续)Table 1(Continued)
2.2 棉花CLE蛋白的系统进化分析
利用MEGA 7.0软件通过邻接法对34个拟南芥CLE蛋白和148个棉花CLE蛋白序列构建了系统进化树,按照拟南芥的CLE家族分类关系可将这些成员分成5个亚组,分别用Clade I、II、III、IV、V 表示不同的亚组(图 1A),此外棉花属内CLE蛋白系统进化树的分组结果也与该结果较为一致。Clade V和Clade IV中家族成员较多,分别含有45和35个家族成员。Clade III和Clade I其次,分别含有25和24个家族成员,Clade II家族成员最少,仅含有19个家族成员(图 2A)。
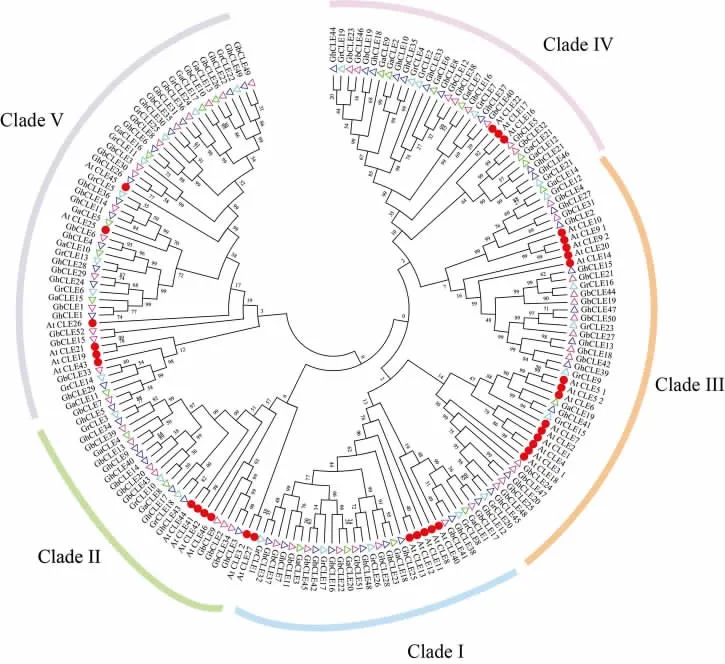
图1 拟南芥和棉花CLE多肽蛋白序列的系统进化树Fig.1 Phylogenetic tree of CLE protein sequences in Arabidopsis thaliana and four Gossypium species
基因家族结构特征分析显示,棉花CLE基因家族多为无内含子或少内含子的基因,Clade V中有50%的基因具有内含子,Clade II和Clade IV分别有47%和31%的基因具有内含子,而Clade III和Clade I只有3到5个基因有内含子(图2B)。这一结构特征与多肽家族基因长度普遍较小相契合,我们发现雷蒙德氏棉和陆地棉中,除GrCLE20,GhCLE36,GhCLE45,GhCLE47和GhCLE20以外,其他基因长度均小于500 bp(base pairs,碱基对)。海岛棉中CLE基因长度大于500 bp的基因数量最多,共有17个(Gb-CLE15,GbCLE52,GbCLE6,GbCLE5,GbCLE29,GbCLE1,GbCLE32,GbCLE48,GbCLE47,Gb-CLE36,GbCLE14,GbCLE25,GbCLE24,GbCLE-17,GbCLE41,GbCLE34,GbCLE2)。
采用MEME在线软件分析148个棉花CLE蛋白的序列特征,发现有5个保守基序(图2C)。包含motif1和motif2的基因数量最多,共有115个。包含motif3和motif4的基因多分别分布于Clade I和Clade V,包含motif5的基因则仅在Clade V有分布。通常每个基序在基因上仅有1到2个分布,但在Clade III中,我们发现motif1在单个基因上的分布数量高达14个(Gr-CLE20)。motif1位于基因的C端,其包含了CLE多肽的核心功能序列(RRVPTGSNPLHN)(图3)。由于植物多肽的表达量通常较低,这些基因是否通过增加序列中功能单位的数量从而提高多肽的表达量,在后续的基因功能研究需要进一步的验证。

图2 棉花CLE多肽蛋白序列的系统进化树(A),基因结构(B)和保守基序(C)分析Fig.2 Phylogenetic tree(A),gene structure(B)and conserved motif(C)of cotton CLE protein sequences in three Gossypium species
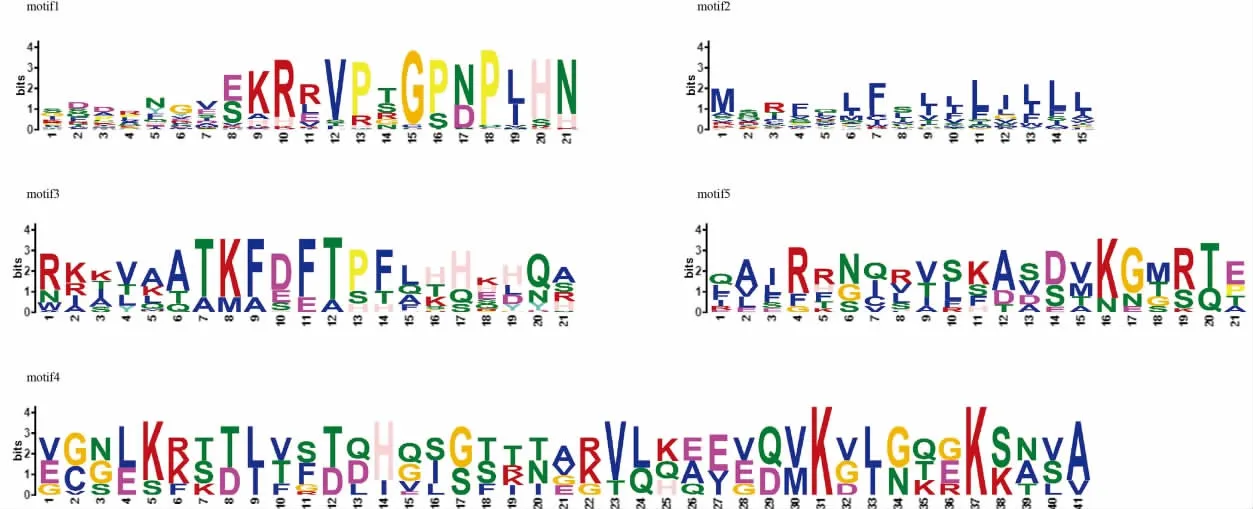
图3 棉花CLE蛋白保守基序Fig.3 Weblogo of conserved motifs identified in the cotton CLE proteins
2.3 棉花CLE基因家族染色体定位和同源性关系分析
根据位置信息共将146个CLE基因定位到棉花染色体上,GhCLE49和GbCLE52基因由于来自Scaffold,因此未被定位到染色体上。亚洲棉的21个CLE基因分布在9条染色体上,2号、8号、9号、12号均无CLE基因分布。其中1号染色体上分布较多,有6个基因;剩余染色体分别包含1~3个CLE基因(图4A)。雷蒙德氏棉的26个CLE基因分布在10条染色体上,3号、10号和11号染色体上均没有分布。1号染色体上有5个CLE基因,4号、5号和7号染色体各分布4个(图4B)。陆地棉的26条染色体中有19条染色体包含CLE基因,其中A亚组有9条,D亚组有10条。A亚组的7号染色体、11号染色体,D亚组2号和7号染色体上CLE基因最多,各有5个CLE基因(图4C)。海岛棉的26条染色体中有21条染色体包含CLE基因,其中A亚组有10条,D亚组有11条。A亚组的3号染色体上CLE基因最多,有6个CLE基因,7号和8号染色体各有5个CLE基因分布。D亚组的染色体上分别有1~4个CLE基因分布(图4C)。此外,在陆地棉中发现1个串联重复的基因簇(GhCLE18和GhCLE19),而在亚洲棉、雷蒙德氏棉和海岛棉中没有发现该串联重复现象。
利用InParanoid软件在4个棉种中共检测到112对直系同源基因对(图5),此外,我们还在陆地棉和海岛棉中分别检测到4对和2对旁系同 源 基 因(GhCLE38/GhCLE12,GhCLE13/Gh-CLE39,GhCLE16/GhCLE42,GhCLE19/GhCLE-18;GbCLE12/GbCLE38,GbCLE5/GbCLE32)(表2)。我们利用KaKs_Calculator对这些同源基因对进行Ka/Ks分析,结果显示约35%的直系同源基因的Ka/Ks值小于0.5,这表明这些基因经历了较强的负选择作用,暗示这些复制基因在进化中较为保守,结构比较稳定,功能具有一致性;13对直系同源基因的Ka/Ks值在0.5~1之间(表2)。另外,有11对直系同源基因的Ka/Ks值大于1(GhCLE15/GrCLE16,GhCLE11/GaCLE5,Gh-CLE45/GrCLE20,GhCLE29/GrCLE14,GaCLE11/GrCLE14,GaCLE2/GrCLE4,GhCLE32/GrCLE1,GbCLE36/GrCLE24,GbCLE38/GhCLE34,Gb-CLE37/GrCLE1,GbCLE51/GhCLE48),暗示这些基因在进化中受到了较强的正选择作用。此外,陆地棉和海岛棉中检测到的6对旁系同源基因的Ka/Ks值均小于1。
2.4 棉花CLE基因的组织表达分析
根据已有的亚洲棉叶片、幼苗,雷蒙德氏棉叶片、花瓣,陆地棉的茎、叶片、花瓣、花托以及海岛棉的茎、叶片和花瓣转录组数据库的分析,获得CLE基因表达量的FPKM(Fragments per kilobase of transcript per million fragments mapped)值,利用HemI 1.0软件绘制出基因表达模式图(图6)。结果显示,大部分CLE基因在组织中的
表达量均较低。在亚洲棉中的幼苗和叶片中,CaCLE4基因的表达量最高,其次为GaCLE8和GaCLE11;GaCLE13、GaCLE14、GaCLE16 和Ga-CLE21仅在叶片组织中有稍高的表达量。在雷蒙德氏棉中,仅有GrCLE14和GrCLE2在叶片中有稍高的表达量,其余基因的表达量均较低。在陆地棉中,GhCLE34和GhCLE9在叶片和茎中的表达量均较高,叶片中的GhCLE14和GhCLE40以及茎中GhCLE13的表达量次之。花瓣和花托中CLE基因的表达量均较低。在海岛棉中,GbCLE39在茎中的表达量最高,GbCLE13和GbCLE43次之,叶片和花瓣中CLE基因表达量均较低。
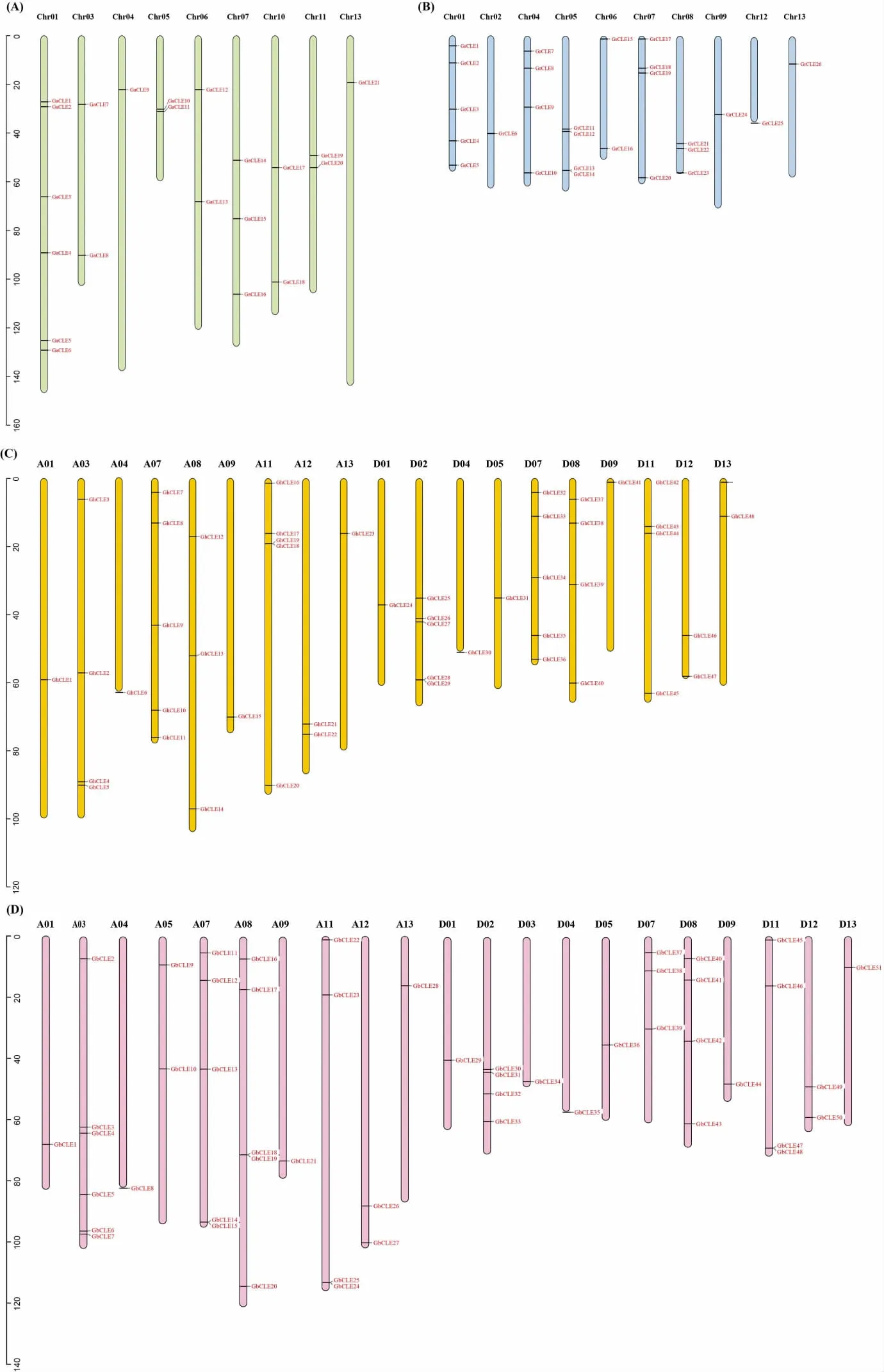
图4 亚洲棉(A)、雷蒙德氏棉(B)、陆地棉(C)和海岛棉(D)CLE基因在染色体上的位置Fig.4 Chromosomal distribution of CLE genes in G.arboreum L.(A), G.raimondii Ulbrich(B), G.hirsutum L.(C)and G.barbadense L.(D)
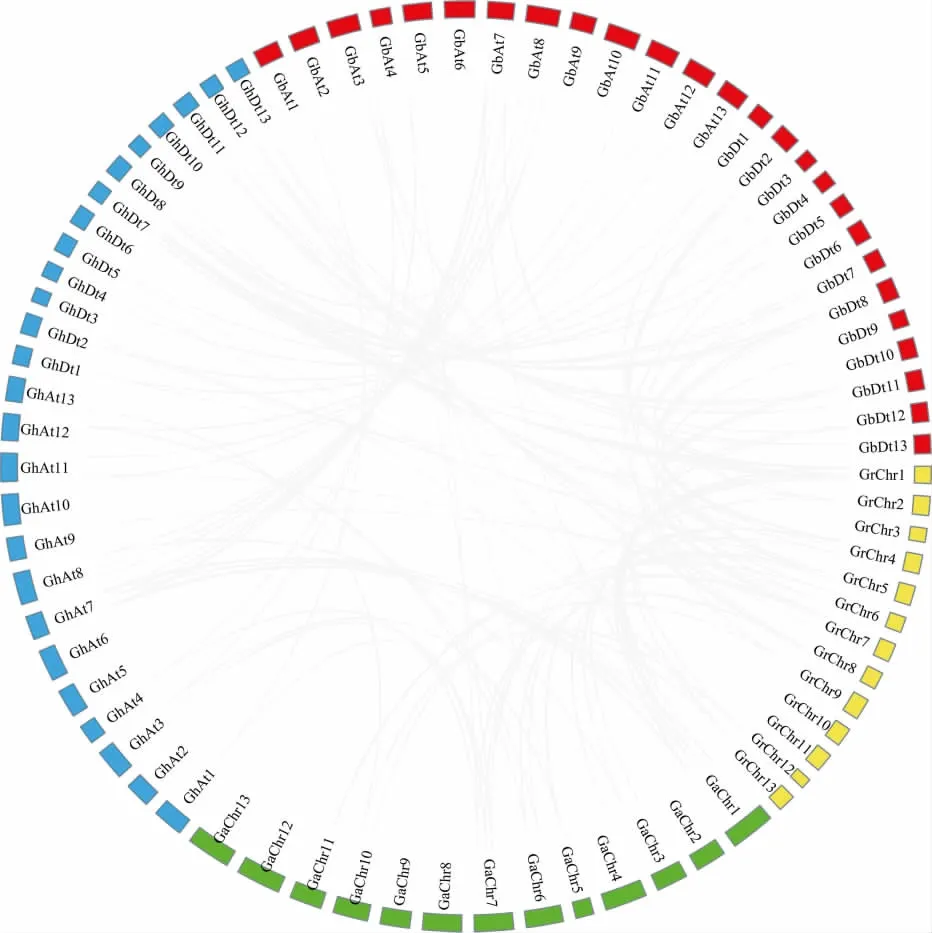
图5 棉花CLE基因家族成员的同源关系分析Fig.5 Homologous relationships of CLE gene family numbers in cotton
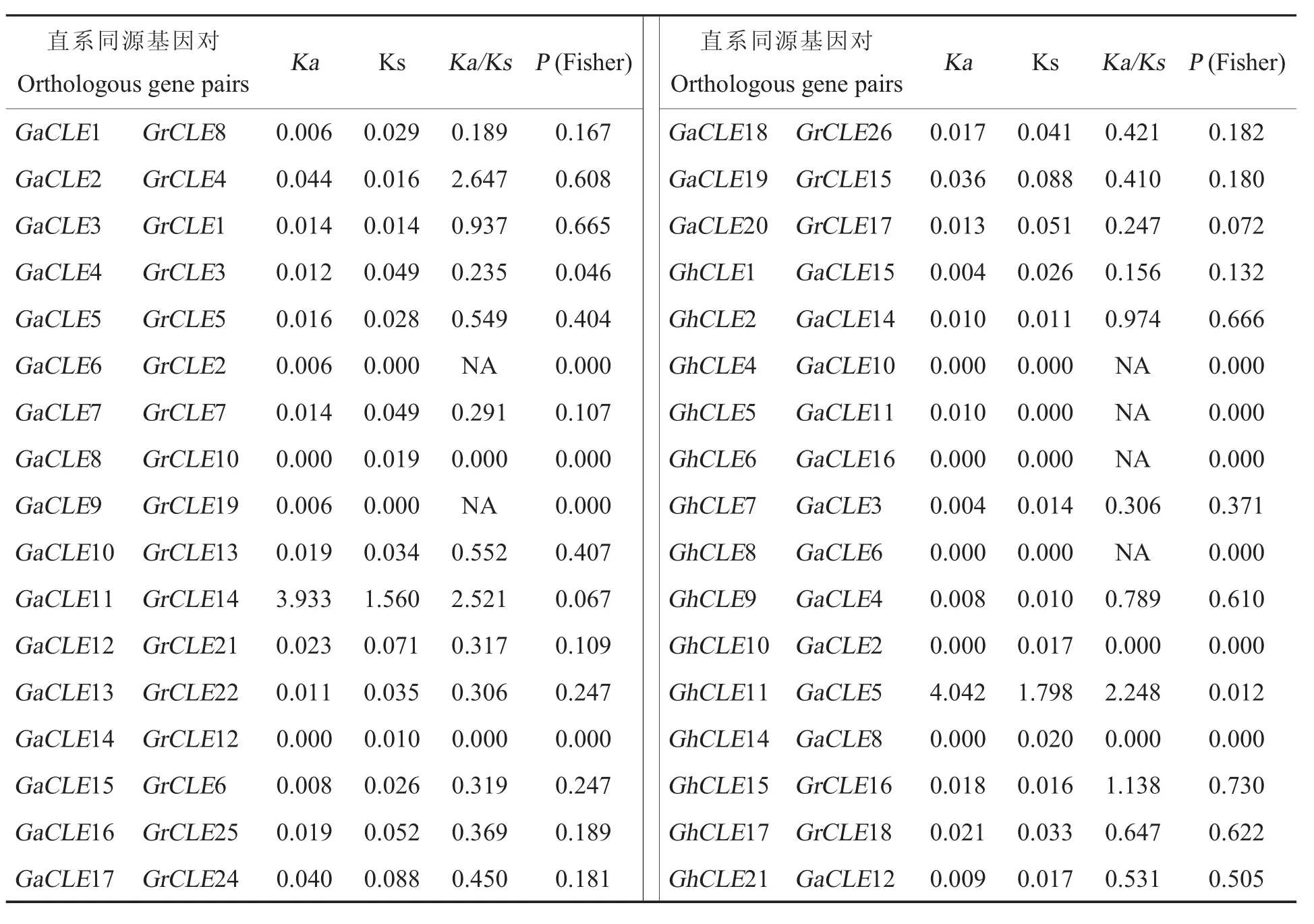
表2 直系同源基因对的 Ka/Ks Table 2 Ka/Ks ratio of orthologous gene pairs
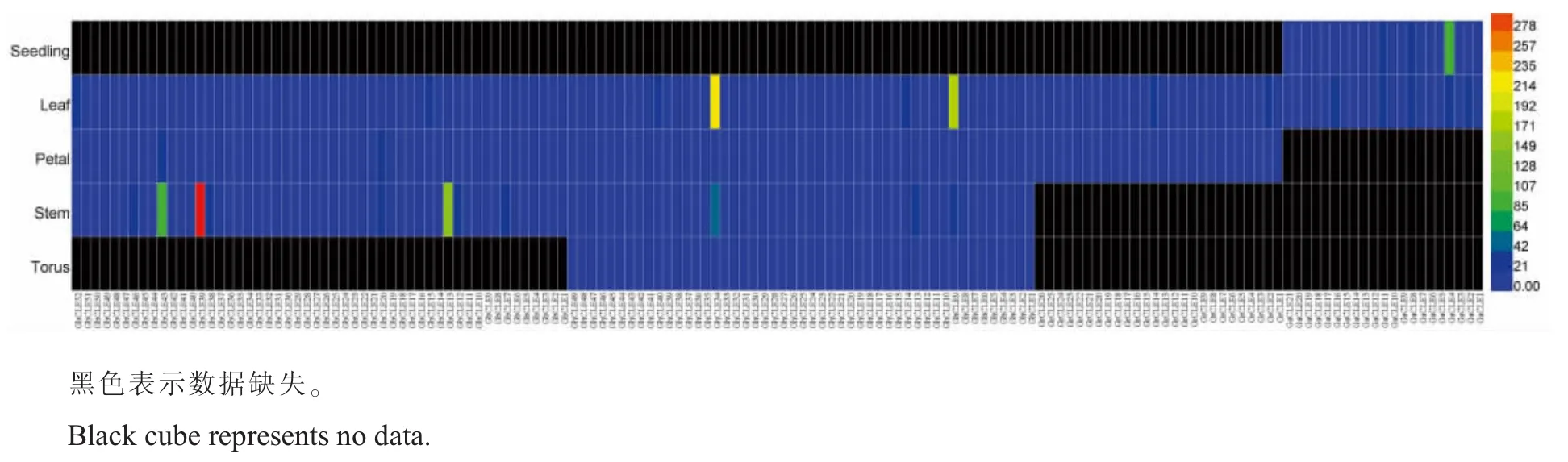
图6 CLE基因家族在棉花组织中的表达模式Fig.6 Expression patterns of CLE gene family numbers in different cotton tissues
3 讨论
相较于动物、真菌和微生物,尽管植物多肽研究较为滞后,但近20年来,随着遗传学和分子生物学等技术的进一步发展,人们逐步意识到植物多肽这一常被忽略的小分子物质,在植物的生长、发育、生殖以及对外界环境的响应中具有重要的调节作用[43]。目前,植物多肽信号转导已经成为植物科学研究的新热点。CLE多肽是迄今为止在植物中最庞大的多肽分子家族,能够通过编码分泌蛋白来参与细胞间的信号转导,在植物生长发育的调控过程中起着关键的作用。本文以重要经济作物棉花为研究对象,利用生物信息学手段分别在亚洲棉、雷蒙德氏棉、陆地棉和海岛棉中共鉴定出148个CLE基因,并进一步揭示CLE基因家族的特征与功能,从而弥补CLE基因在棉花研究中的空白。
亚洲棉的21个CLE基因分布在9条染色体上,雷蒙德氏棉的26个CLE基因分布在10条染色体上,陆地棉有19条染色体包含CLE基因,其中A亚组有9条、D亚组有10条。海岛棉中有21条染色体包含CLE基因,其中A亚组有10条、D亚组有11条。此外,陆地棉的CLE基因数量分别是亚洲棉和雷蒙德氏棉的2倍,并且在直系同源基因对检测时,除了ChCLE3和Gh- CLE25外,剩余基因均能在亚洲棉或雷蒙德氏棉中找到直系同源基因,这表明陆地棉在A、D亚组杂交加倍形成后,CLE基因没有发生大量丢失现象。在海岛棉中,少部分CLE基因没有检测到直系同源基因,揭示了在海岛棉在物种形成后,这些基因可能发生了独立演化事件。聚类分析显示棉花的148个CLE基因可以分为5个亚组,Clade V和Clade IV中家族成员较多,分别含有45和35个家族成员。四个棉种的CLE基因在这5个分组均有分布,表明棉花四倍体栽培种的CLE基因在异源多倍化过程中,没有发生大量的基因丢失,较大保留了二倍体栽培种的CLE基因来源。此外,我们发现棉花中大部分CLE基因只含有一个核心CLE保守基序(motif1:xRxcPs-GpDPIHHh),但是在CladeIII中,我们发现Gh-CLE45、GrCLE20、GbCLE48、GhCLE20、GbCLE25、GbCLE24、GbCLE47等6个基因分别包含多个motif1序列重复,其中GrCLE20中motif1重复高达14个。通常植物多肽分子的表达量较低,这些基因是否通过增加重复单元来提高多肽的表达量,在后续的功能研究中值得进一步探索和验证。每个基序被二聚脯氨酸分隔开,这与水稻中发现的 3个 CLE(OsCLE502、OsCLE504和 Os-CLE506)相似,这可能在CLE多肽信号分子成熟的过程中发挥作用,比如被不同类型肽酶所识别[44-45]。
组织表达分析表明,大多数CLE成员在四个棉种的组织中表达量均较低,亚洲棉中表达量最高的基因为GaCLE4。陆地棉中GhCLE9、Gh-CLE34, 海岛棉中GbCLE39、GbCLE13和Gb-CLE43表达量相对较高。雷蒙德棉中的CLE基因表达量均较低。GaCLE4与GhCLE9为直系同源基因,在不同物种中保持着较为一致的高表达量,暗示其功能和演化的保守性。而表达量较高的GhCLE34,与其直系同源基因GrCLE3表达模式差异较大,表明该直系同源基因在不同物种的功能或许存在差异。基于Ka/Ks值,本研究发现11对直系同源基因在演化中受到了较强的正选择作用。通过序列同源比对,发现上述大部分基因功能及信号途径仍然不清晰。
本研究将棉花CLE基因与拟南芥CLE基因进行系统发育分析,发现与拟南芥的分组较为一致。通过比对拟南芥CLE家族中CLE基序(CLE motif)发现其具有高度的保守特性[21]。Fiers等研究显示,CLV3基因中CLE域的大部分侧翼序列被删除后,不影响CLV3的功能,进一步研究显示,用人工合成的CLE保守基序处理拟南芥幼苗,会减小根端分生组织,抑制根的生长,表现出类似CLE基因过表达的表型,CLV3多肽同时也能恢复clv3-2突变体的表型[46]。此外,研究发现拟南芥根部异位表达的CLE19和CLE40能够促进根顶端分生组织干细胞分化,而体外施加多肽 CLE19-CLE-motif和 CLE40-CLE-motif,均同样能够促进根顶端分生组织干细胞分化[47-48],这些研究表明CLE基序中12个氨基酸的核心序列才是CLE多肽的真正活性形式,也是其行使功能所需的最小结构形式[49-51]。因此,本研究通过将棉花CLE的12个氨基酸核心基序与其他物种比对,发现10种CLE-motif序列较为保守,在其他物种中均能找到一致序列,其中包括一些研 究 较 为 透 彻 的 CLE25、CLE41、CLE44、FON1等成员。因此,在后续的功能验证中,可以参考这些CLE的功能及信号转导途径研究,验证其在棉花中是否存在功能保守性。此外,我们还发现12种棉花特有CLE-motif,其中包括来源于受到强烈正选择作用的GhCLE15/GrCLE16和Gh-CLE45/GrCLE20同源基因。本研究结果可以为后续进一步深入分析这些高表达量或者棉花特异的CLE多肽提供相应的参考依据,为丰富棉花多肽的功能研究及信号调控网络研究提供了一定的理论基础,在后续研究中,我们可以重点关注这些棉花特有多肽,以利于棉花的生长发育调控研究。
多肽激素的发现改变了人们对植物分子调控的传统认识,开辟了植物科学研究的新领域[43]。尽管目前一些信号多肽和受体已得到了鉴定,但还有更多多肽在植物的生长发育等方面的重要性还没有被挖掘。例如,多肽信号的产生和加工、信号多肽与受体的相互作用以及信号分子调控植物生长、发育和对环境响应的机制等。多肽基因通常较小及表达丰度较低,这为多肽研究带来一定的难度,但随着质谱技术及CRISPR-Cas9等分子技术的发展、完善,以及借鉴动物多肽信号的研究成果,这些问题将有望逐步解决。多肽激素的功能鉴定及调控解析,不仅可以扩展我们对植物信号调控网络的认识,丰富植物肽类激素相关理论知识,更可为未来调控植物,特别是作物的生长发育和对环境的响应提供更绿色和有效的生物技术手段。
4 结论
采用全基因组分析,我们在二倍体棉种中鉴定出21个亚洲棉GaCLE基因和26个雷蒙德氏棉GrCLE基因,在四倍体陆地棉和海岛棉中分别鉴定出49和52个CLE基因。聚类分析显示棉属的CLE基因可以分为5个亚组。选择压力分析显示大部分CLE基因都经历了负选择作用并且在棉花组织中的表达量均较低。海岛棉中的GbCLE39、GbCLE13、GbCLE43, 陆 地 棉 中Gh-CLE34、GhCLE9以及亚组棉中的GaCLE4在棉花组织中具有相对较高的表达量。根据保守基序分析发现了12个棉花特有的CLE多肽。

