粪肠球菌β-N-乙酰氨基葡萄糖苷酶基因的克隆表达及酶学性质表征
陈宝莉, 秦 臻, 赵黎明
华东理工大学生物工程学院, 发酵工业分离提取技术研发中心, 生物反应器工程国家重点实验室, 上海 200237
N-乙酰氨基葡萄糖是生物体内许多重要糖类的组成成分,也是甲壳类动物的骨骼和真菌细胞壁的最小组成单位。N-乙酰氨基葡萄糖通过β-1,4糖苷键连接形成自然界中来源最广泛的天然聚糖之一——几丁质(chitin)[1~3]。其年储量仅次于纤维素[4]。几丁质水溶性差、不利于吸收,因而难以被直接使用于食品和药品中,而几丁质的水解产物易溶于水、生物相容性好[5],具有更大的应用潜力。研究发现N-乙酰氨基葡萄糖具有许多重要的生理功能,例如:改善皮肤水合程度、加速伤口愈合、提高人体免疫力、改善骨关节炎和抗肿瘤等,因此被广泛应用于医疗、食品和化妆品等多个领域[6~8]。在工业生产中,N-乙酰氨基葡萄糖目前主要通过降解几丁质制备得到。常用的降解方法包括化学法和生物法。其中化学降解主要为酸水解和氧化降解,存在副产物较多、污染严重的问题;相比之下,生物降解法产物纯度高,环境污染小且操作简单,成为近年来的研究热点[9, 10]。生物法降解几丁质涉及多种糖苷水解酶的作用,根据作用底物和产物类型的不同可分为内切型几丁质酶(endochitinase,EC 3.2.1.14)、外切型几丁质酶(exochitinase,EC 3.2.1.29)、几丁二糖酶(chitobiosidase, EC 3.2.1.29)和β-N-乙酰氨基葡萄糖苷酶(β-N-acetylglucosaminidase,EC 3.2.1.52)[11]。其中内切型几丁质酶可随机水解几丁质糖链,得到低聚合度寡糖;外切型几丁质酶和几丁二糖酶可特异性作用于几丁质的非还原端,得到几丁二糖或单糖;β-N-乙酰氨基葡萄糖苷酶可作用于几丁寡糖的非还原端,最终水解得到N-乙酰氨基葡萄糖。
β-N-乙酰氨基葡萄糖苷酶在自然界中的来源较为丰富,存在于多种动植物、细菌和真菌体内。CAZy数据库(http://www.cazy.org)依据蛋白质序列进化关系对糖苷水解酶进行归类,β-N-乙酰氨基葡萄糖苷酶主要分布在GH3、GH20、GH84和GH116家族中。本研究涉及的粪肠球菌是人和动物肠道内重要的菌群之一,其内存在丰富的碳水化合物代谢酶系[12~14],是发掘高效β-N-乙酰氨基葡萄糖苷酶的潜在微生物来源。本研究克隆得到了粪肠球菌来源的β-N-乙酰氨基葡萄糖苷酶基因EfNagase,成功进行了原核异源表达,并对其酶学性质进行了初步研究,为开发高稳定性的β-N-乙酰氨基葡萄糖苷酶奠定了理论基础。
1 材料与方法
1.1 菌株与质粒
粪肠球菌(Enterococcusfaecalis,CGMCC 1.2135)由本实验室保存;大肠杆菌感受态细胞EscherichiacoliDH5α、BL21(DE3)均购于康为世纪生物科技有限公司(上海);pET-28a(+)载体由本实验室保存。DNA Marker DL2000、蛋白Marker均购自TaKaRa公司(大连);细菌基因组DNA快速抽提试剂盒,SanPrep柱式质粒DNA小量抽提试剂盒、DNA胶回收试剂盒、PCR产物纯化试剂盒均购自生工生物工程(上海)股份有限公司;TaqDNA聚合酶购自Vazyme生物科技有限公司(南京);限制性内切酶NheⅠ和XhoⅠ购自TaKaRa公司(大连);T4 连接酶购自New England Biolabs(美国);对硝基苯基-N-乙酰β-D-氨基葡萄糖苷(pNP-GlcNAc)购自Sigma公司(美国);几丁二糖购自Megazyme公司(爱尔兰);其他试剂均为国产或进口分析纯。
1.2 EfNagase序列分析
根据NCBI数据库提供的粪肠球菌来源的GH20家族潜在糖苷水解酶基因序列(GenBank登录号:AAO79989.1),使用DNAMAN软件分析核酸序列并确定酶切位点;利用Primer Premier 5软件设计引物。利用BlastP(https://blast.ncbi.nlm.nih.gov/Blast.cgi)分析与其他蛋白的序列相似性。使用MEGA 7.0软件按照Neighbor-joining运算方法构建系统发育树[15~18]。
1.3 EfNagase基因扩增与原核表达
序列分析表明粪肠球菌来源的AAO79989.1基因为多结构域糖苷水解酶,包含一个GH18家族结构域(N端)及GH20家族结构域(C端)。为了获得特异性GH20家族β-N-乙酰氨基葡萄糖酶,本研究选取该基因中GH20家族催化区进行进一步的克隆表达研究。
按照细菌基因组DNA快速抽提试剂盒说明书,提取Enterococcusfaecalis基因组DNA,并以此为模板,利用聚合酶链式反应扩增GH20家族结构域基因(命名为EfNagase),上下游引物分别为EfNagase-F:5′-ATTCATGCTAGCAAAAGTGTCTT-TTCCATTGATGC-3′和EfNagase-R:5′-ATTCCGCTCGAGTTATTTAACCACCATATACTCACGAT-3′(下划线部分别为酶切位点NheⅠ和XhoⅠ)。PCR程序为:94℃ 2 min;94℃ 30 s,52℃ 30 s,72℃ 2 min,共30个循环;72℃ 10 min。用1%琼脂糖凝胶电泳鉴定PCR产物并胶回收。胶回收片段与载体pET-28a(+)经限制性内切酶酶切后,用T4连接酶连接。将连接产物热激转化至E.coliDH5α感受态细胞中,挑单菌落验证阳性克隆,重组质粒命名为pET-28a-EfNagase。将重组质粒热激转化至E.coliBL21(DE3)感受态细胞中,挑取阳性克隆在LB液体培养基中,37℃恒温震荡培养至OD600达到0.6~0.8后,转移至30℃恒温摇床培养,用1 mmol/L的IPTG诱导表达10 h。用超声波破碎细胞获得粗酶液,离心收集上清液,上样至已预平衡的Ni-IDA亲和层析柱中,继续用平衡缓冲液(20 mmol/L Tris-HCl,500 mmol/L NaCl,20 mmol/L咪唑)冲洗至OD280小于0.05,再用洗脱缓冲液(20 mmol/L Tris-HCl,500 mmol/L NaCl,40 mmol/L咪唑)洗至OD280小于0.05,除去非特异性结合蛋白,最后用洗脱缓冲液(20 mmol/L Tris-HCl,500 mmol/L NaCl,200 mmol/L咪唑)洗脱目的蛋白并收集目标组分,用SDS-PAGE检测蛋白纯度。
1.4 酶活力的测定
蛋白含量测定:采用Bradford法进行测定[19]。取100 μL适当稀释的纯酶液,加入1 mL 1×Bradford工作液,迅速混匀后室温反应3 min。以蒸馏水作空白对照,于595 nm波长下测定吸光值。以牛血清白蛋白(BSA)为标准品绘制标准曲线,计算纯酶液的蛋白含量。
β-N-乙酰氨基葡萄糖苷酶酶活的测定(pNPG法)[20]:取100 μL 2 mmol/L pNP-GlcNAG与50 μL 0.2 mol/L柠檬酸盐缓冲液(pH 6.0)混匀,50℃预热10 min,加入50 μL适当稀释的纯酶溶液,迅速混匀50℃反应10 min,最后加入200 μL 0.5 mol/L NaOH溶液终止反应。冷却至室温,以蒸馏水作空白对照,于410 nm波长下测定吸光值。酶活力单位(U)定义:在上述条件下,每分钟生成1 μmoL对硝基苯酚所需的β-N-乙酰氨基葡萄糖苷酶酶量为1个酶活力单位。
β-N-乙酰氨基葡萄糖苷酶酶活的测定(3,5-二硝基水杨酸,DNS法)[21]:取350 μL溶解在0.2 mol/L柠檬酸盐缓冲液(pH 6.0)中,质量分数1%的几丁二糖溶液,再加入50 μL适当稀释的纯酶溶液,迅速混匀50℃反应10 min,最后加入600 μL DNS溶液,沸水浴煮沸10 min终止反应。冷却至室温,10 000 r/min离心5 min,取上清液。以蒸馏水作空白对照,于540 nm波长下测定吸光值。酶活力单位(U)定义:在上述条件下,每分钟生成1 μmoL N-乙酰氨基葡萄糖所需的β-N-乙酰氨基葡萄糖苷酶酶量为1个酶活力单位。
1.5 EfNagase的酶学性质研究
1.5.1最适温度及温度稳定性的测定 将纯酶溶液溶于0.2 mol/L pH 6.0的柠檬酸盐缓冲液并稀释至适当浓度,按1.4中的酶活测定方法测定该酶于不同反应温度(35~70℃)下的酶活,以最高酶活点为100%,确定其最适反应温度。
将纯酶溶液溶于0.2 mol/L pH 6.0的柠檬酸盐缓冲液并稀释至适当浓度,于不同温度条件下(35~60℃)水浴保温30 min,冰浴冷却10 min后,按1.4中的酶活测定方法测定不同温度条件下该酶的残余酶活,以未经处理的酶活为100%,确定其温度稳定性。
1.5.2最适pH及pH稳定性的测定 将纯酶溶液溶于不同pH缓冲体系(柠檬酸盐缓冲液:2.5~6.5,磷酸盐缓冲液:5.5~8.0,Tris-HCl:7.5~9.0,甘氨酸-NaOH:9.0~13.0)中并稀释至适当浓度,按1.4中的酶活测定方法测定该酶于不同pH缓冲体系下的酶活,以最高酶活点为100%,确定其最适反应温度。
将纯酶溶液分别溶于不同pH缓冲体系中,置于50℃水浴中保温30 min,冰浴冷却10 min后,按1.4中酶活测定方法测定不同pH缓冲体系中该酶的残余酶活力,以未经处理的酶活为100%,确定其pH稳定性。
1.5.3EfNagase底物特异性 分别以天然糖类(壳二糖、壳聚糖、几丁二糖和几丁质)和人工合成的糖苷(pNP-β-D-葡萄糖苷、pNP-GlcNAc)为底物,由于EfNagase对上述天然糖类的水解活性较低,为了探索EfNagase对于上述底物是否具有催化能力,采用TLC法分析产物。将适量的天然糖类与纯酶溶液混匀50℃水浴保温1 d,用薄层层析法分析产物组成(展开剂为异丙醇:水:氨水=15∶1∶7.5)。按照1.4中酶活测定方法测定EfNagase水解人工合成糖苷的能力,以水解释放对硝基苯酚的速率来计算酶活力。
1.5.4EfNagase动力学参数 用50 mmol/L pH 6.0的柠檬酸盐缓冲液配制11个不同浓度(0.02 mmol/L、0.025 mmol/L、0.04 mmol/L、0.05 mmol/L、0.067 mmol/L、0.1 mmol/L、0.2 mmol/L、0.25 mmol/L、0.5 mmol/L、1 mmol/L、2 mmol/L)的pNP-GlcNAc底物,加入适宜浓度的β-N-乙酰氨基葡萄糖苷酶酶液,50℃分别反应5 min、10 min、15 min、30 min、60 min、120 min,利用Sigma Plot软件计算Vmax和Km值。
2 结果与分析
2.1 EfNagase序列分析
Enterococcusfaecalis来源的GH20家族潜在的β-N-乙酰氨基葡萄糖苷酶基因全长1 101 bp,编码367个氨基酸。通过NCBI数据库中Blast结果显示EfNagase氨基酸序列与GH20家族中的StreptococcuspneumoniaeTIGR4(AAK76198.1)来源的β-N-乙酰氨基葡萄糖苷酶序列相似度最高,为52.08%。用MEGA 7.0软件以Neighbor-joining运算方法构建系统发育树,分析结果(图1)同样显示EfNagase与StreptococcuspneumoniaeTIGR4具有最近的进化关系。

图1 EfNagase与GH20家族不同来源的糖苷水解酶系统发育树分析Fig.1 Phylogenetic analysis of β-N-acetylglucosaminidase fromEnterococcus faecalisand GH20 family.注:基于GH20家族中不同来源的糖苷水解酶氨基酸序列,利用MEGA 7.0软件采用Neighbor-joining法构建系统发育树,序列编号附于括号中。
2.2 EfNagase基因扩增与原核表达
以Enterococcusfaecalis基因组DNA为模板,通过PCR扩增得到EfNagase基因片段,1%琼脂糖凝胶电泳鉴定(图2A)得到一条大小约为1 100 bp具有特异性的核酸条带,符合理论预期。通过限制性内切酶双酶切反应、目的基因与载体的连接反应,挑取单菌落测序为阳性克隆,得到成功构建的重组质粒pET-28a-EfNagase。将其转入E.coliBL21(DE3)感受态细胞中后,用1 mmol/L IPTG诱导,30℃、200 r/min培养10 h。超声波破碎细胞壁,离心取上清液,利用Ni-IDA亲和层析柱分离纯化。通过SDS-PAGE检测纯化产物的纯度,结果(图2B)显示成功获得可溶性表达的高纯度的目的蛋白条带,分子量约为40 kDa,符合预期。以pNP-GlcNAG为底物检测纯化后的EfNagase酶学性质,EfNagase的比活力为3 606.1 U/mg;以几丁二糖为底物检测到EfNagase的比活力为352.60 U/mg。
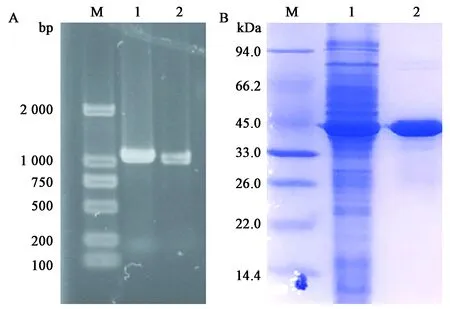
图2 PCR扩增产物琼脂糖凝胶电泳(A)及重组蛋白SDS-PAGE电泳(B)结果Fig.2 Agarose gel electrophoresis(A)and SDS-PAGE(B)of recombinant protein fromEnterococcus faecalis.A:M:DNA Marker;1, 2:EfNagase基因PCR产物;B:M:蛋白Marker;1:粗酶液;2:纯酶液。
2.3 EfNagase酶学性质研究
2.3.1最适温度和温度稳定性 在相同pH条件下,测定EfNagase在不同反应温度下(35~70℃)的酶活力。结果如图3A所示,EfNagase为中温酶,在50℃时酶活最高;当温度高于70℃时,EfNagase失去活性。将酶液分别孵育于不同温度(35~70℃)条件下30 min,按标准酶活测定方法测其残余酶活。结果如图3B所示,EfNagase在孵育温度低于50℃时表现出极高的热稳定性,酶活基本没有损失,延长孵育时间至一周,其残余酶活仍高于80%;当孵育温度高于50℃,EfNagase的稳定性开始降低。
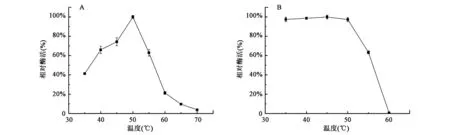
图3 重组蛋白EfNagase的最适温度(A)与温度稳定性(B)Fig.3 Optimum temperature(A)and thermostablity(B)of purified recombinant EfNagase.
2.3.2最适pH和pH稳定性 在50℃,不同pH缓冲液体系中测定EfNagase的酶活力。结果如图4A所示,EfNagase的最适pH为6.0;当pH在5.0~7.0之间时,相对酶活高于50%,且当pH 5.0~6.5时,EfNagase在柠檬酸盐缓冲体系中的相对酶活比在磷酸盐缓冲体系中的更高;当EfNagase在pH高于7.5的条件下,酶活明显降低。将酶液分别溶于不同pH缓冲体系中50℃孵育30 min,按标准酶活测定方法测定残余酶活。结果如图4B所示,EfNagase具有极高的pH稳定性,在很宽的pH范围内(pH 3.5~11.5)残余酶活均高于90%;当pH低于3.0时,残余酶活仍维持在70%以上;当pH高于12.5,残余酶活出现了大幅度下降。
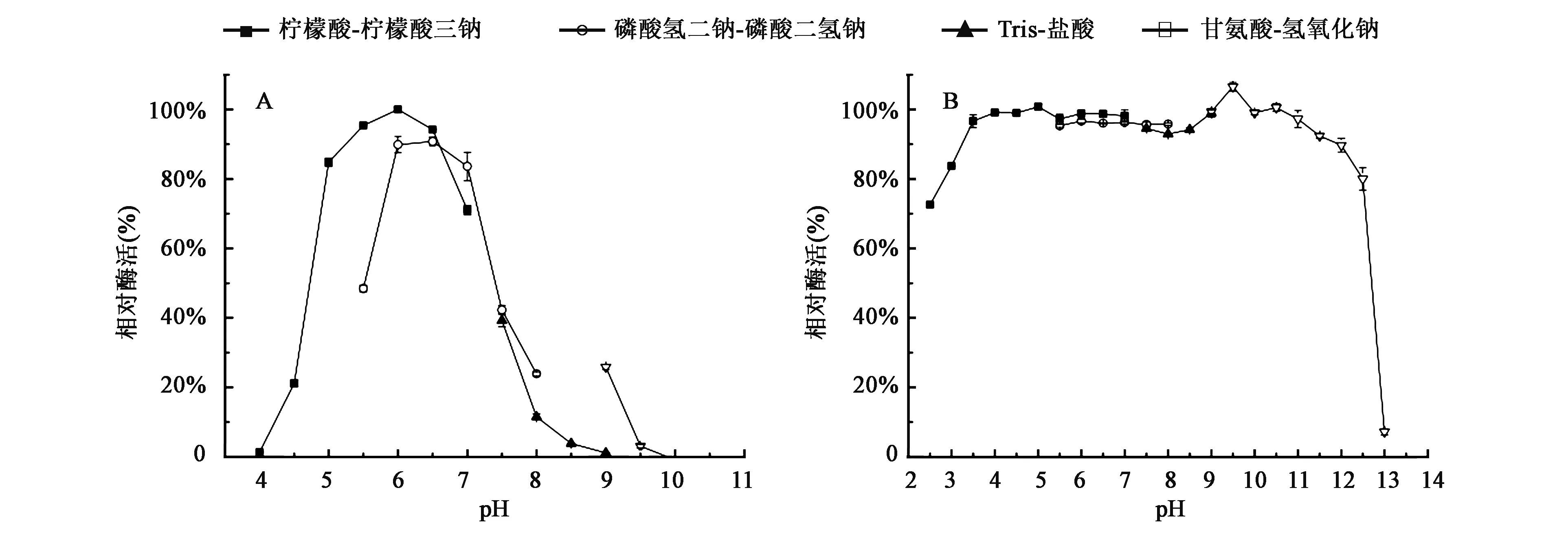
图4 重组蛋白EfNagase的最适pH(A)与pH稳定性(B)Fig.4 Optimum pH(A)and pH stability(B)of purified recombinant EfNagase.
2.3.3EfNagase的底物特异性及动力学参数 分别以天然糖类(壳二糖、壳聚糖、几丁二糖和几丁质)和人工合成的pNP-β-D-葡萄糖苷、pNP-GlcNAc为底物。用TLC检测EfNagase水解几丁二糖的反应历程,结果如图5所示,EfNagase可在60 min内将几丁二糖完全水解成为N-乙酰氨基葡萄糖。而EfNagase对几丁质的水解率很低,且不能水解壳二糖、壳聚糖。用pNPG法检测发现EfNagase对pNP-GlcNAc表现出高的水解活性,不能水解其他人工合成的pNP底物。结果表明EfNagase对几丁二糖和pNP-GlcNAc有更高的特异性,对氨基葡萄糖衍生物没有催化活性。以pNP-GlcNAc为底物,检测得到EfNagase的Vmax及Km分别为520.37 μmol/min·mg和0.599 mmol/L。
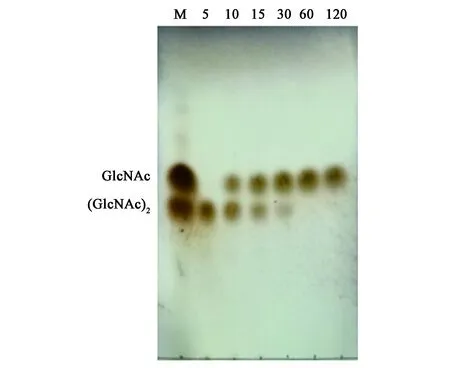
图5 薄层层析检测EfNagase水解几丁二糖的反应历程Fig.5 TLC analysis of the time course of chitin disaccharides hydrolyzed by purified EfNagase.M:N-乙酰氨基葡萄糖与几丁二糖标准品; 5,10,15,30,60,120:分别表示EfNagase水解反应了5 min,10 min,15 min,30 min,60 min,120 min。
3 讨论
N-乙酰氨基葡萄糖由于其众多重要的生理活性而备受关注,β-N-乙酰氨基葡萄糖苷酶作为酶法绿色制备N-乙酰氨基葡萄糖中具有重要意义的水解酶之一,也受到学者的广泛研究。本研究从粪肠球菌中克隆得到的β-N-乙酰氨基葡萄糖苷酶(EfNagase),通过相似度比对和构建系统进化树,确定其应归属GH20家族糖苷水解酶。目前,β-N-乙酰氨基葡萄糖苷酶主要分布在GH3、GH20、GH84和GH116家族中,其中GH20家族的发现和研究最多。虽然目前已有报道的β-N-乙酰氨基葡萄糖苷酶很多,但普遍存在酶活较低的缺点,大约超过70%的β-N-乙酰氨基葡萄糖苷酶酶活低于100 μmol/min·mg[22]。相对于其他家族的β-N-乙酰氨基葡萄糖苷酶,GH20家族的酶活更高,Microbacteriumsp.来源[23]的β-N-乙酰氨基葡萄糖苷酶水解pNP-GlcNAc的比活力为1 773.1 μmol/min·mg,水解几丁二糖的比活为481.4 μmol/min·mg;Shinellasp.来源[24]的β-N-乙酰氨基葡萄糖苷酶水解pNP-GlcNAc的比活力为538.8 μmol/min·mg,水解几丁二糖的比活为35.4 μmol/min·mg。EfNagase以pNP-GlcNAc为底物的比活为3 606.10 U/mg,以几丁二糖为底物的比活力为352.60 U/mg,已远高于多数其他来源的β-N-乙酰氨基葡萄糖苷酶。EfNagase在温度50°C、pH 6.0的条件下活性最高,这和其他多数β-N-乙酰氨基葡萄糖苷酶性质相近。近60%的β-N-乙酰氨基葡萄糖苷酶的最适温度在37~50℃,最适pH多在5~8之间[25]。EfNagase展现出了良好的温度稳定性,在温度低于50℃的条件下孵育一周,其残余酶活仍高于80%;且具有良好的pH稳定性,在pH 3.5~11.5的范围内展现出高的稳定性(残余酶活>90%)。适用于大部分食品、化学和医药工业的生产条件。研究还表明EfNagase对pNPG和几丁二糖有较强的水解活性和底物特异性。EfNagase的优良性质为其在食品、化学、医药工业以及环境生物技术等领域中的应用奠定了基础。

