棉花线粒体基因cRT-PCR改良及其在寻找CMS相关基因中的应用
王裴林, 周利利, 梁成真, 孟志刚, 郭三堆, 张 锐
中国农业科学院生物技术研究所, 北京 100081
线粒体是细胞内氧化磷酸化和合成三磷酸腺苷(ATP)的主要场所,为细胞的活动提供了能量。细胞生命活动所需的能量95%来自线粒体,所以其又有“细胞动力工厂”之称[1]。除了为细胞供能外,线粒体还参与诸如细胞分化、细胞信息传递和细胞凋亡等过程,并有调控细胞生长和细胞周期的能力。因此,线粒体基因组中基因的异常表达常常会影响细胞的能量代谢,从而引发一系列的表型变化,如抗病性降低或细胞质雄性不育的发生等。对线粒体基因组编码基因的全长转录本进行分析,有利于深入研究细胞质基因的功能以及某些与细胞质关联性状的分子机理。
细胞质雄性不育(cytoplasmic male sterility,CMS)是由细胞质基因控制的并可被核恢复基因抑制的母性遗传性状。近几年,棉花功能基因组学进展良好,但棉花细胞质雄性不育机理的相关研究报道不是很多,尤其是胞质不育的机理,尚缺乏足够的分子生物学证据[1,2]。基于目前的研究进展,多数科学家认为CMS不育发生的原因大多是由于线粒体基因的重组重排导致,所以对线粒体基因全长转录本的获得显得尤为重要。
在传统方法中,cDNA末端快速扩增(rapid amplification of cDNA ends, RACE)方法往往被用于精确定位RNA分子的5′或3′末端,但由于线粒体基因大部分不存在转录后加工的过程,即不会在转录后进行5′加帽子结构和3′端加polyA序列,也没有内含子,而是采用边转录边翻译的方式,所以不适用于RACE技术。为克服这一弊端,Kuhn和Binder于2002年提出环式反转录聚合酶链式反应(cyclic reverse transcription polymerase chain reaction, cRT-PCR),该技术极大简化了操作步骤,成功实现一次性确定目的RNA的5′和3′末端的设想,具有明显的高效与便捷性[3]。
cRT-PCR技术现已广泛应该用于确定基因的转录起始位点和启动子、鉴定核糖体RNA前体的5′和3′末端以及评估成熟mRNA的3′末端的长度和转录后延伸。目前,cRT-PCR技术应用于水稻和拟南芥等植物的细胞基因研究,但研究报道中均是利用该方法对特异基因进行研究[4,5],尚未见从转录组的水平获得全部线粒体基因全长转录本的应用。
目前,根据诸多的文献报道,CMS基因通常有以下五种特点:①不同的CMS之间通常不具备同源性;②CMS的位点经常出现在ATP基因的上下游;③它们往往通过重组产生嵌合的ORF;④CMS编码的蛋白通常具有细胞毒性;⑤CMS编码蛋白通常会形成跨膜结构域。尤其是嵌合ORF的形成是不育基因目前的重点研究方向,通过cRT-PCR技术可以较为简单的获得靶标基因的UTR区,为寻找嵌合的ORF提供了一个新的方向[6~8]。
近几年棉花功能基因组学进展良好,但棉花细胞质雄性不育机理的相关研究报道不是很多,同其他作物雄性不育机理研究相比有所滞后。因此,我们希望通过该方法的改良及应用能为寻找不育基因带来新的方法。本研究在获得高质量RNA的基础上,对RNA进行高效环化,并通过随机引物对线粒体全基因组基因进行cRT-PCR,获得了全部线粒体基因的全长转录本,并以ATP6基因为例,比较了利用特异引物和随机引物进行cRT-PCR后所获得的全长转录本的差异。棉花细胞质雄性不育的研究对于更好地利用棉花杂种优势,以及加深对植物不育机理的认识,具有重要的理论和现实意义。
1 材料与方法
1.1 材料与试剂
1.1.1实验材料 本实验所用材料为中国农业科学院抗虫棉课题组实验室培育的不育系P30A。
1.1.2试剂 原平皓RNA提取试剂盒、RNA酶抑制剂、DNA酶、Superstar max超保真mix购自Genestar公司;反转录试剂盒购自全式金生物科技有限公司;T4 RNA连接酶购自Promega公司;DNA琼脂糖凝胶回收试剂盒购自Axygen生化科技有限公司;T载体、感受态购自擎科新业生物技术有限公司;高温灭菌冷却至室温的DEPC水。
1.2 实验方法
1.2.1引物设计 ①RT-PCR引物使用本实验室发表的PrimerServer: PCR Primers Batch Design & Specificity Check引物设计网站[9],对参考基因序列设计普通引物,引物位置在参考序列两边留出100~300 bp为宜,并由网站检测为特异引物。
②cRT-PCR引物由RT-PCR引物反向互补得到,估计其大小,约为原始序列两边预留的200 bp加上可能的UTR区域(约100~600 bp)(表1)。
1.2.2RNA提取 按照原平皓试剂盒RNA提取说明书提取RNA,并在加入去蛋白液前增加DNA酶,去除DNA,为后续实验去除DNA污染。提取完成后通过琼脂糖凝胶电泳检测RNA的完整度;并通过Nano-drop对RNA的浓度和纯度进行测量。

表1 4类线粒体基因cRT-PCR引物信息Table1 Four gene families mitochondrial gene cRT-PCR primer information.
1.2.3RNA环化 体系:10 μg总RNA,1 μL RNA酶抑制剂,2 μL T4 RNA连接酶Buffer,1 μL T4 RNA连接酶,1 μL ATP, DEPC水补足至20 μL。将混合好的样品置于金属浴上,16℃连接过夜。在连接液的每管中加入10 μL 氯仿并用移液器反复吹打;加入100 μL无水乙醇移液器吹打混匀,冰浴15 min;将混合物12 000 r/min,10 min离心,弃上清;最后,加入14 μL DEPC水溶解[11,12]。
1.2.4反转录 按照全式金生物科技有限公司反转录试剂盒对产物进行反转录,完成后用Nano测定浓度,ddH2O稀释至终浓度200 ng/μL ,冻存备用。
1.2.5cRT-PCR程序 我们在棉花线粒体基因的主要4种类型的基因ATP、COX、RPS、NAD基因中随机选择7个作为实验对象,并根据普通PCR引物反向互补链设计相应的cRT-PCR引物,其特点是仅能以环化RNA为模板获得扩增条带。扩增体系:25 μL Superstar max超保真mix(Genestar),1 μL cDNA,正反向引物各1 μL,ddH2O 22 μL。扩增程序:94℃预变性5 min;94℃变性30 s;54℃(取决于引物)退火30 s,72℃延伸40 s,35~40个循环;72℃延伸10 min,4℃保存。完成PCR之后,将PCR产物于1%琼脂糖凝胶中上样,120 V,30 min。电泳结束后,按照DNA琼脂糖凝胶回收试剂盒(Axygen生化科技有限公司)回收条带。
1.2.6ATP6的测序分析 为进一步分析测序结果的准确性,从上述7个棉花线粒体基因中随机选取ATP6为代表进行分析。对ATP6基因的cRT-PCR引物进行扩增,然后将该序列回收后连接T载体,挑单克隆进行测序,最终挑选出一个一致性最好的克隆子进行分析,并与NCBI上的序列进行比对。然后采用随机引物和特异引物共同对其进行cRT-PCR(图1)。
2 结果与分析
2.1 线粒体基因cRT-PCR电泳分析
对棉花线粒体基因的主要4种类型的基因中随机选择的7个基因的扩增结果显示,7个线粒体基因的电泳条带清晰(图2),表明RNA模板已经环化,为后续的实验分析提供必要的前提。

图1 随机引物和特异引物ATP6全长转录本对比Fig.1 Comparison of random primer and specific primer ofATP6gene full-length transcript.
我们对棉花线粒体的33编码蛋白基因(ATP1、ATP4、ATP6、ATP8、ATP9;COX1、COX2、COX3;RPL2、RPL5、RPL10、RPL16;NAD3、NAD4、NAD4L、NAD6、NAD7、NAD9;RPS3、RPS4、RPS7、RPS10、RPS12、RPS14;CCMB、CCMC、CCMFC、CCMFN;SDH3、SDH4;MATR、COB、MTTB)进行了cRT-PCR,发现大多数的基因UTR都在300~500 bp左右。我们使用特异引物和随机引物同时进行扩增,发现两者所获得结果基本保持一致,通过测序验证,仅有个别基因出现了极少数的SNP。
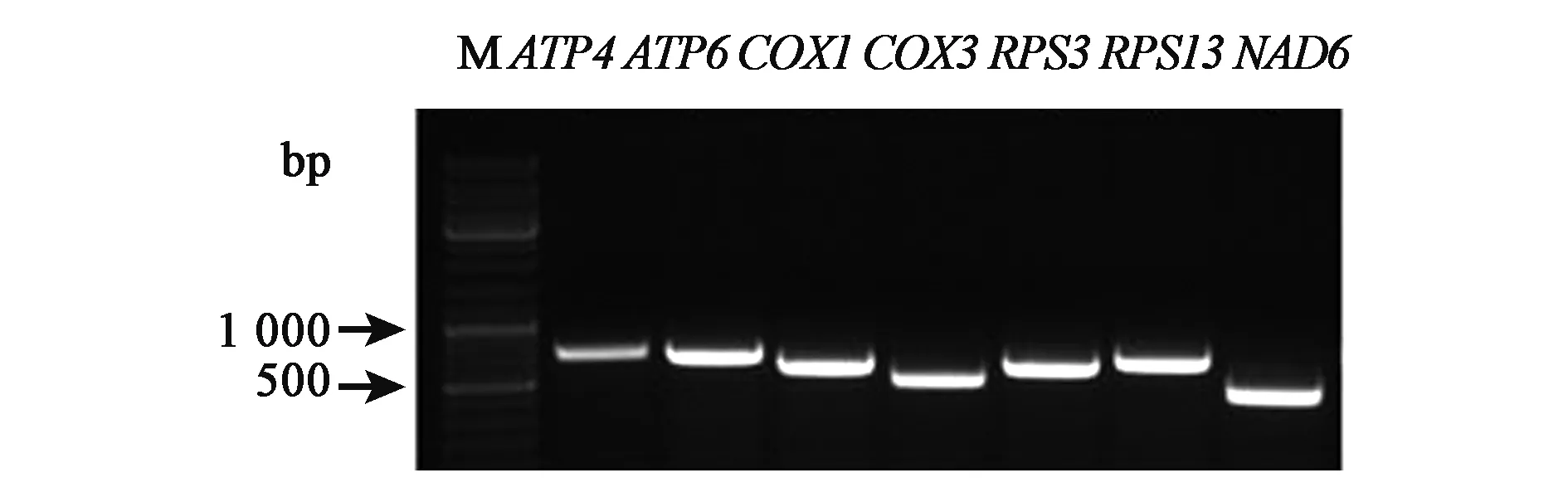
图2 线粒体基因cRT-PCR电泳分析Fig.2 Analysis of mitochondrial gene by cRT-PCR.
2.2 ATP6基因测序结果分析
通过ATP6基因的cRT-PCR引物进行扩增,获得一段全长567 bp的序列(图3),测序并与NCBI上的序列进行比对,我们获得了284 bp的5′UTR和22 bp的3′UTR,并且原基因上的一段重复区域也完全匹配,获得了ATP6的完整全长转录本。测序结果与参考序列保持一致,之后,采用随机引物和特异引物共同对其进行cRT-PCR,发现两者获得条带一致。测序后,两者序列仅出现了一个SNP。其余基因验证测序结果同样保持一致,所以我们认为采用随机引物替代靶基因特异引物的方法在保证了正确率的情况下,更为方便快速。
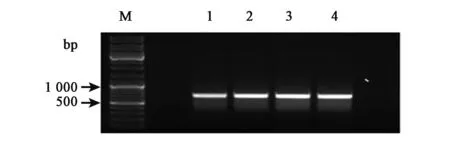
图3 ATP6基因cRT-PCR扩增结果Fig.3 cRT-PCR results ofATP6gene.注:M:Marker;1,2:随机引物;3,4:特异引物。
综上所述,通过随机引物进行反转录cRT-PCR同特异引物一样能获得目的基因的全长转录本,但在比对序列时也发现极少部分基因的测序结果存在基因的不完整性和突变性,主要表现为基因两端碱基缺失与单核苷酸多态性。前者的发生取决于RNA的完整度以及环化效率,而单核苷酸多态性的发生则多半是由于特异引物和随机引物的差异造成的。因此,在方便快捷的基础上,首先采用随机引物对大批量的基因进行cRT-PCR,如若所获基因转录本的序列异常,为保证准确性,可辅助设计特异引物进行反转录。
2.3 cRT-PCR在寻找不育基因中的应用
通过对本课题组繁育的不育系和保持系同时进行线粒体基因的批量cRT-PCR,根据条带大小以及测序结果对线粒体编码蛋白基因进行考量,找到一些具有差异的候选基因,认为可能是嵌合ORF的插入导致了结果的差异。
如图4所示,差异表达出现了四种情况,一是不育系和保持系条带完全一致;二是不育系和保持系条带存在明暗亮度差异;三是不育系有而保持系没有条带;四是在不育系和保持系中条带大小不一致的情况。
另外有的基因在电泳条带上表现一致,但通过测序出现了几个氨基酸的缺失,或是出现一些SNP的情况,结果如表2所示: 线粒体编码蛋白基因中ATP1、ATP4、ATP6、COX1在不育系和保持系的扩增中出现了明显差异,尤其是ATP1、ATP6、COX1,在两段序列中出现了大段的插入或者缺失,我们认为这些差异片段可能是由于线粒体的某种重组或者重排造成,或可能引起作物的不育。除此之外,线粒体的其他基因ATP8、COX2、NAD3等出现了在不育系和保持系中序列有SNP的情况,可能是由于PCR或者测序的原因,也有可能是线粒体的某种突变引起,但这种方式引起不育的可能性过低。
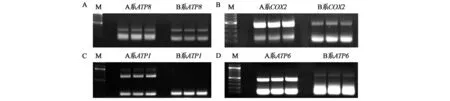
图4 线粒体基因在不育系与保持系中的扩增条带Fig.4 Amplification bands of mitochondrial genes in sterile line and maintainer line.注:A:ATP8;B:COX2;C:ATP1;D:ATP6;M:Marker;A系:不育系;B系:保持系。

基因名称差异情况基因名称差异情况ATP1A系260 bp,B系没有RPS3SNPATP4A系:5′-AACGCGTTTAGGTAAGACTT-3′B系:5′-AAC--G---A-------CTT-3′RPS4RPS7-ATP6A系360 bp,B系526 bp,SNPRPS10SNPATP8SNPRPS12-ATP9-RPS14SNPCOX1A系B系161 bp序列差异,SNPCCMB-COX2SNPCCMCSNPCOX3-CCMFC-NAD3SNPCCMFNSNPNAD4-SDH3SNPNAD4LSNPSDH4未获条带NAD6-MATRSNPNAD7SNPCOB未获条带NAD9SNPMTTBSNP
3 讨论
长期以来,cRT-PCR技术已广泛应用于拟南芥、水稻等植物的细胞研究中,但鲜有涉及棉花细胞线粒体基因的研究。在本研究中,我们对转录本序列分析发现,cRT-PCR技术适用于获得棉花线粒体目的基因的全长转录本,为后续技术改良奠定了实验基础。在传统cRT-PCR技术的基础上,我们优化了RNA提取方法和环化连接体系,显著提高了环化效率。反转录阶段,在保证与特异引物获得相同完整转录本的基础上,创新性地采用随机引物进行大批量的基因检测,操作更方便快捷。
基于本研究中改良的cRT-PCR技术存在上述提及的基因的不完整性和突变性现象,本研究提出如下针对性措施:首先保证RNA提取方法的科学准确性,鉴于RNA的降解起始于5′和3′末端,直接决定环化效率并影响扩增中基因序列的完整性,因此反应试剂中应保证一定浓度的RNA酶抑制剂,以从源头上确保基因序列的准确性;其次,我们发现环化的效率极大的影响了扩增的结果,无论是特异引物还是随机引物,都会出现区段缺失的情况,但在必要时将随机引物改为传统的特异性引物,以从反应途径上确保降低单核苷酸多态性的发生,从而提升基因序列的特异性。在本研究中,需要综合考虑影响实验的诸多环节,其中需要格外关注的是RNA环化连接的质量。过夜可以为充分环化提供必要的时间保障,但鉴于单链RNA的易降解性,保证充分环化时间的前提是确保所加入的RNA酶抑制剂达到一定浓度,能够保障RNA在环化期间的稳定性。此外,在过夜后环化效率依然不高的特殊情形下,加入适量ATP可以明显加速RNA环化,提高环化效率。
我们通过改良的cRT-PCR技术对不育基因进行探索,通过对不育系和保持系的扩增,发现在ATP和COX家族中存在UTR区有差异的基因(在其他线粒体基因中大多是SNP的差异)。在此,我们暂不讨论条带明暗的差异,这可能和扩增效率或是拷贝数相关。差异基因中有无的差异、条带大小的差异,都很可能是由于线粒体内部重排或者ORF的插入导致的,而这些差异可能会引起作物的CMS。根据本实验结果,发现线粒体编码蛋白基因中ATP1、ATP4、ATP6、COX1在不育系和保持系的扩增中出现了明显差异,我们认为这些差异片段可能是由于某种重组或者插入造成,或可能引起作物的不育。UTR区段的差异倾向于ATP和COX基因,这和目前发表的CMS相关文献保持一致,提升了作为CMS相关基因的可能性,因此我们认为该方法用来寻找不育基因是可行的。当然,这些差异的出现不一定是CMS基因造成的嵌合ORF,具体结果如何还需要进一步的实验验证。但是,通过cRT-PCR技术,为寻找CMS基因提供了新的方法和思路。

