5S rRNA互作蛋白组研究揭示5S rRNA参与mRNA剪接调控*
周祥明,杨兵,赖巧,李乐,董军,廖建友,朱爽
(1.广东药科大学生命科学与生物制药学院,广东 广州 511400; 2.中山大学孙逸仙纪念医院,广东 广州 510030)
非编码RNA(non-coding RNA,ncRNA)是指不编码蛋白质的RNA,包括长链非编码RNA(long non-coding RNA,lncRNA)、核糖体RNA(ribosomal RNA,rRNA)、核小RNA(small nuclear RNA,snRNA)和microRNA 等等[1-4]。它们参与调控了几乎所有的细胞生命活动过程。ncRNA主要通过与其结合蛋白的互作来发挥调控功能。例如,各种lncRNA都是通过与常规的和非常规的RNA结合蛋白(RNA binding protein,RBP)的互作来调控各种生命活动过程[5],rRNA通过与蛋白质翻译机器蛋白的互作来行使翻译功能,而microRNA则通过与AGO蛋白的结合来抑制mRNA翻译或者促进mRNA降解来调控基因表达[6]。因此确定ncRNA的RBP对理解ncRNA功能至关重要。由于每个ncRNA都不止与一种蛋白发生互作,极大地增加了ncRNA功能和调控机制的研究难度。例如,近期研究显示U1 snRNA在体内与超过400种RNA结合蛋白互作[7]。而目前绝大多数ncRNA的互作蛋白都是未知的,严重阻碍了我们对ncRNA在细胞生命活动中所扮演角色的理解,因此亟需系统地去鉴定各种ncRNA的互作蛋白组。
5S rRNA是一个长度为120 nt的小RNA,在原核生物和真核生物中都具有高保守性的二级和三级结构。在1960年,5S rRNA在核糖体中首次被发现[8]。在1971年,首次报道了大肠杆菌在缺失5S rRNA的情况下不能合成多肽从而导致翻译终止,证明5S rRNA是核糖体的重要组成部分。在真核生物细胞中,5S rRNA与eL5/L18家族蛋白,23S rRNA结合构成了核糖体的主要成分[9]。进一步研究表明,5S rRNA除了参与蛋白质翻译调控外,还可能具有其它的调控功能。例如,Andria Pelava等[10]发现5S rRNA可以通过结合肿瘤抑制因子p53来参与肿瘤发生发展的调控。然而目前还缺乏对5S rRNA相互蛋白的系统研究工作,不清楚5S rRNA的互作蛋白组全貌是怎样的,所以对5S rRNA的互作蛋白组系统解析将可以帮助我们更全面地认识5S rRNA这种细胞生命活动过程中最重要调控因子之一的功能和调控机制。
本研究将利用国际上最先进的ncRNA互作蛋白组研究技术ChIRP-MS[7](chromatin isolation by RNA purification-mass spectrometry,图1A)首次鉴定出5S rRNA互作蛋白组,并采用系统的生物信息学分析方法(图1B)探究5S rRNA 新的调控功能和分子机制。
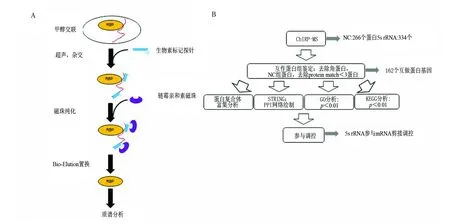
图1 ChIRP-MS流程及生物信息学分析流程Fig.1 Procedures of ChIRP-MS and Bioinformatics analysis
1 材料与方法
1.1 ChIRP-MS实验
1.1.1 探针设计 在UCSC网站上查询目的基因序列。根据目的基因序列设计探针,并在上海生工合成生物素标记探针。
1.1.2 收集细胞并交联 收细胞后计数。加入φ=3%甲醛溶液(用PBS按照107个细胞10 mL配置)重悬细胞,颠倒混匀,在摇床上室温混匀30 min。加入1/10体积的1.25 mol/L甘氨酸(glycine),混匀后在摇床上室温摇5 min。4 ℃离心弃上清。预冷PBS洗一次,4 ℃离心弃上清。按每2×107个细胞的1 mL PBS的量加入预冷PBS并重悬。每个1.5 mL管加入1 mL细胞悬浮液分装,4 ℃离心弃上清。
1.1.3 裂解细胞 细胞称重,加入Lysis Buffer重悬细胞(0.05 mol/L Tris-HCl pH7.0,10 mmol/L EDTA,w=1% SDS。按照每100 mg细胞添加1 mL Lysis Buffer。Lysis Buffer中提前加入蛋白酶抑制剂、PMSF和RNasin)。
1.1.4 超声打断DNA和RNA 4 ℃水浴中用最高能量以30 s ON,45 s OFF条件超声细胞。取5 μL检测超声效果,如果DNA大小在100~500 bp,说明超声完成,如果不是,则继续超声。
1.1.5 杂交 将超声样品4 ℃离心取上清,取细胞裂解液体积的1/100作为Input对照。取10 μL链霉亲和素磁珠用100 μL Lysis Buffer洗三次(该磁珠用于去除非特异结合的蛋白)。将细胞裂解液转移到15 mL管中,加入Hybridization Buffer(750 mmol/L NaCl,w=1% SDS,50 mmol/L Tris-HCl pH7.0,1 mmol/L EDTA pH 8.0,w=15% Formamide。每1 mL Lysis Buffer添加2 mL Hybridization Buffer,并提前加入蛋白酶抑制剂、PMSF和RNasin)。加入10 μL洗过的磁珠,在杂交炉中37 ℃洗30 min后弃磁珠。每2×107个细胞加入1 μL 100 μmol/L探针,在杂交炉中37 ℃孵育过夜。取100 μL磁珠,用Lysis Buffer洗三次。用裂解细胞相同体积的Lysis Buffer(加入蛋白酶抑制剂、PMSF和RNasin)来将磁珠悬浮。在杂交结束后,将磁珠悬浮液加到每个管中,在杂交炉中37 ℃旋转混匀30 min。用1 mL Wash Buffer(2×SSC,w=0.5% SDS)洗5次磁珠。在最后一次洗脱完成后,先加入与磁珠等体积的Wash Buffer,取10 μL磁珠用于提RNA,剩下所有磁珠用于提蛋白。
1.1.6 qRT-PCR检测ncRNA是否被探针捕获 在10 μL RNA Input中加入85 μL RNA PK Buffer pH7.0(100 mmol/L NaCl,10 mmol/L Tris-HCl pH.7.0,1 mmol/L EDTA pH8.0,w=0.5% SDS),加入5 μL Proteinease K。10 μL磁珠中加入95 μL的RNA PK Buffer pH7.0,加入5 μL Proteinease K。在杂交炉中50 ℃孵育45 min。加入100 μL DEPC水,加入200 μL酚氯仿(苯酚∶氯仿=1∶1),剧烈涡旋5 s。4 ℃离心取上清加入等体积氯仿,剧烈涡旋5 s。4 ℃离心取上清,加入1/10体积3 mol/L NaAc,0.5 μL 糖元(glycogen blue),3倍体积无水乙醇,-20 ℃沉淀过夜。4 ℃离心弃上清,真空抽干后用10 μL DEPC处理水回溶。qRT-PCR检测目标ncRNA和GAPDH mRNA的丰度。
1.1.7 回收蛋白用于MS 弃上清留磁珠,加入与磁珠等体积的Biotin elution Buffer(12.5 mmol/L D-biotin,7.5 mmol/L HEPES pH7.5,75 mmol/L NaCl,1.5 mmol/L EDTA pH8.0,w=0.15% SDS,w=0.075% sarkosyl,w=0.02%Na-Deoxycholate,6.4×10-3mol/L NaOH),涡旋混匀后室温静置20 min,65 ℃干浴10 min,收集上清。再次加入与磁珠等体积的Biotin elution Buffer,室温静置20 min,65 ℃干浴10 min,合并两次上清。加入4倍体积预冷丙酮,-20 ℃沉淀过夜。4 ℃离心弃上清,室温静置1 min,立刻加入30 μL 1×laemmli sample buffer (biorad)。95 ℃干浴30 min,每5 min混匀一次,最终获得蛋白样品。蛋白样品跑w=10% SDS-PAGE胶后银染并用于质谱检测。
1.2 蛋白序号转换基因名
Uniport(The Universal Protein Resource)是一个蛋白序列和蛋白功能的综合性数据库[11],并且是目前信息最丰富、资源最广的免费蛋白质数据库。本研究利用uniprot在线转换工具将蛋白序号(protein accession)转换成基因名(gene symbol)。
1.3 蛋白复合体富集分析与PPI网络构建
了解细胞内的不同分子间的互作有助于健康和疾病的深入研究,所以急需一个可以将不同分子互作数据库整合起来的综合性数据库。ConsensusPathDB就是这样的一个数据库,它整合了超过30个分子互作相关的人、鼠和酵母数据库,总共包括了215 541个分子互作(蛋白与蛋白互作、生化反应、基因调控互作)和4 601条通路信息,并可将这些信息可视化[12-13]。文章中利用ConsensusPathDB分析获得5S rRNA互作蛋白组的蛋白复合体富集信息。
STRING 数据库(Search Tool for the Retrival of Interacting Genes/Proteins Database) 是一个搜寻蛋白质之间互作的数据库。它能够帮助用户轻松获取独特的、覆盖范围广的实验以及预测的互作关系信息,既包括蛋白质之间的直接物理互作,也包括蛋白质之间的间接功能相关性。它除了包含有实验数据、从PubMed摘要中挖掘的结果和综合其他数据库数据外,还可利用生物信息学方法预测结果。Cytoscape 是一款图形化显示网络并进行分析和编辑的软件,我们利用Cytoscap 将STRING数据库查询的蛋白互作信息可视化并构建蛋白与蛋白互作(protein-protein interaction network,PPI)网络。
1.4 GO 注释富集和KEGG信号通路富集
GO(Gene Ontology)是一个提供基因及产物功能注释信息的数据库。KEGG(Kyoto Encyclopediaof Gene and Genome)是一个包含基因、通路、疾病、药物的综合数据库[12]。利用GO和KEGG对目标基因进行分析,从而预测该基因参与的生物过程(biological process,BP)、细胞分布(components of cells,CC)、分子功能(molecular function,MF)及参与通路(pathway)。
2 结果与分析
2.1 内源5S rRNA互作蛋白的捕获和检测
ChIRP-MS技术是近年发展起来的一种系统鉴定特定RNA内源互作蛋白组的技术。它首先利用甲醛交联细胞,再用生物素修饰DNA 探针捕获目标RNA,然后经过链霉亲和素磁珠吸附纯化和洗脱,结合高效液相色谱-质谱联用(HPLC-MS)技术,最终获得目标RNA内源互作蛋白组[14](图1A)。使用人模式细胞Hela来研究5S rRNA的内源互作蛋白组,细胞培养后利用φ=3%甲醛对细胞做了交联处理并进行超声。利用在线ChIRP探针设计软件ChIRP-Designer,设计了针对5S rRNA的特异探针(表1)。为了减少质谱结果的非特异性,我们同时设计了一条靶向细菌RNA的NC对照组探针(表1)。在利用所设计的探针捕获5S rRNA互作蛋白后,取一部分去提RNA。通过实时定量PCR(quantitative real-time polymerase chain reaction,qRT-PCR)检测,发现5S rRNA探针捕获到的5S rRNA占input 5S rRNA的(23±2.3)%(图2A),而5S rRNA探针几乎没有捕获到GAPDH mRNA。NC对照组探针几乎没有捕获到5S rRNA和GAPDH mRNA。该结果说明我们成功地利用所合成的5S rRNA探针特异地捕获到了5S rRNA,且NC对照组探针的特异性是非常高的。将探针捕获到的5S rRNA互作蛋白跑SDS-PAGE胶后银染。银染结果显示5S rRNA互作蛋白分布于整个泳道,相比之下NC对照组探针泳道没有明显信号,这说明5S rRNA在细胞内可能结合有大量的蛋白,而这里面的绝大部分蛋白可能都是未知的(图2B)。

表1 本研究中的序列Table 1 The sequences in this study
虽然qRT-PCR结果显示此次实验所使用的5S rRNA探针捕获到的5S rRNA达到了input 5S rRNA的(23±2.3)%(图2A)。但是在以后的ChIRP-MS实验中,需要进一步提高5S rRNA探针的捕获效率从而提高实验成功率。可考虑同时设计多条探针并比较他们捕获效率后,再选取捕获效率最高的探针进行实验。
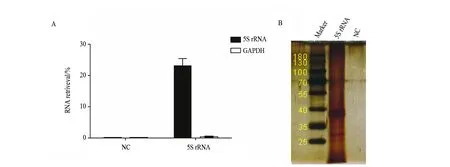
图2 5S rRNA捕获效率检测及银染Fig.2 5S rRNA captured efficiency detection and silver staining
2.2 HPLC-MS鉴定5S rRNA互作蛋白组
随后用HPLC-MS系统鉴定了NC对照组探针和5S rRNA特异探针捕获到的蛋白。NC对照组共检测出266个蛋白质,5S rRNA组共检测出334个蛋白质。为了进一步减少质谱结果的非特异性,提高质谱结果可信度,首先去除两组蛋白中质谱信号低的蛋白,也即protein match<3的蛋白;随后再去除样品组和NC对照组中所有的角蛋白;最后在5S rRNA的蛋白列表中移除NC中同样存在的蛋白;最终获得了162个高可信度的5S rRNA内源互作蛋白,并用uniprot在线转换工具将protein accession转换为gene symbol。表2展示了5S rRNA互作蛋白组中质谱信号排名前20的蛋白。
表2 5S rRNA互作蛋白组中质谱信号排名前20的蛋白
Table 2 The top 20 proteins of MS signal in the 5S rRNA interactome
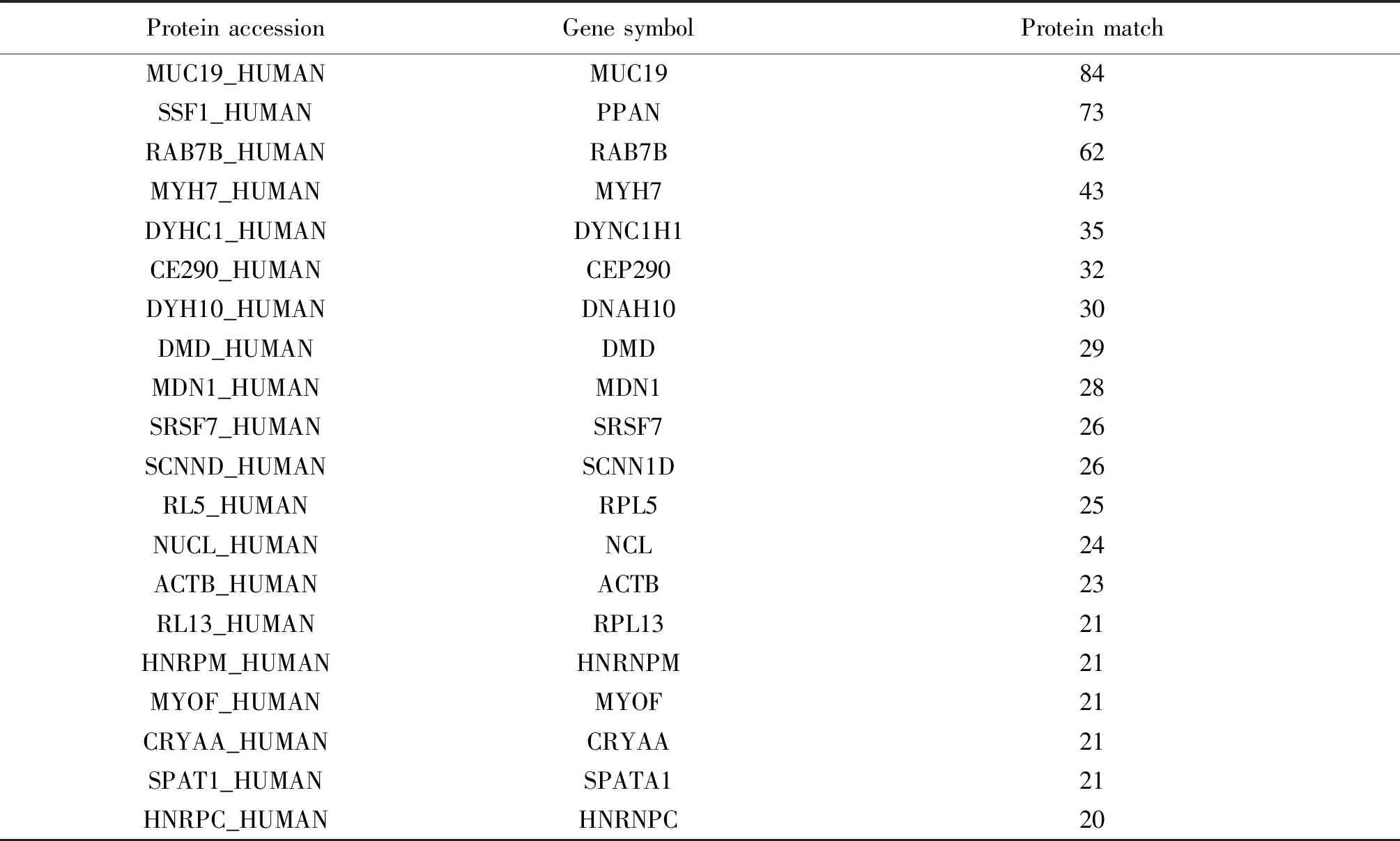
Protein accessionGene symbolProtein matchMUC19_HUMANMUC1984SSF1_HUMANPPAN73RAB7B_HUMANRAB7B62MYH7_HUMANMYH743DYHC1_HUMANDYNC1H135CE290_HUMANCEP29032DYH10_HUMANDNAH1030DMD_HUMANDMD29MDN1_HUMANMDN128SRSF7_HUMANSRSF726SCNND_HUMANSCNN1D26RL5_HUMANRPL525NUCL_HUMANNCL24ACTB_HUMANACTB23RL13_HUMANRPL1321HNRPM_HUMANHNRNPM21MYOF_HUMANMYOF21CRYAA_HUMANCRYAA21SPAT1_HUMANSPATA121HNRPC_HUMANHNRNPC20
2.3 5S rRNA互作蛋白组复合体富集分析和PPI网络构建
利用ConsensusPathDB数据库中的复合体富集分析(protein complex-based gene sets analysis)对5S rRNA的互作蛋白组成分进行了系统分析(图3A)。分析结果显示5S rRNA的互作蛋白组显著富集了核糖体、蛋白质翻译相关的蛋白复合体。例如,Nop56p-associated pre-rRNA complex[15]、PGC-1-SRp40-SRp55-SRp75 complex[16]、Ribosome, cytoplasmic、60S ribosomal subunit,cytoplasmic、40S ribosomal subunit,cytoplasmic等。这与人们熟知的5S rRNA蛋白翻译调控功能相一致。此外,还可以看到5S rRNA互作蛋白组中还包含其它蛋白复合体,包括:TRBP containing complex[17]、TNF-α/NF-κB signaling complex 6、Spliceosome、capped, methylated pre-mRNP: CBC complex[18]、DGCR8 multiprotein complex[19]、Large Drosha complex[20]、TLE1 corepressor complex[21]、p54nrb/PSF/Matrin3/inosine RNA[22]、H2AX complex, isolated from cells without IR exposure[23]、Emerin complex 25[24]、SNW1 complex[25]等等。以上结果说明5S rRNA可能不仅仅参与了蛋白质翻译调控,可能还参与了microRNA加工成熟调控和RNA转录调控、mRNA剪接调控、细胞信号通路调控、DNA损伤修复等过程。进一步利用STRING数据库对5S rRNA互作蛋白组中质谱信号排名前50的蛋白绘制了PPI网络(图3B)。结果显示,5S rRNA互作蛋白组间也有复杂的互作模式,并且其构成的PPI网络是由HNRNP家族[27]、SR蛋白家族[27]、核糖体蛋白RPL家族与DDX5蛋白[28-29]构成网络主体。这些蛋白都是我们所熟知的,其在细胞中发挥着重要生物学功能。这些功能包括:核糖体构成和mRNA剪接。这些互作网络在5S rRNA的参与下很有可能会变的更复杂。
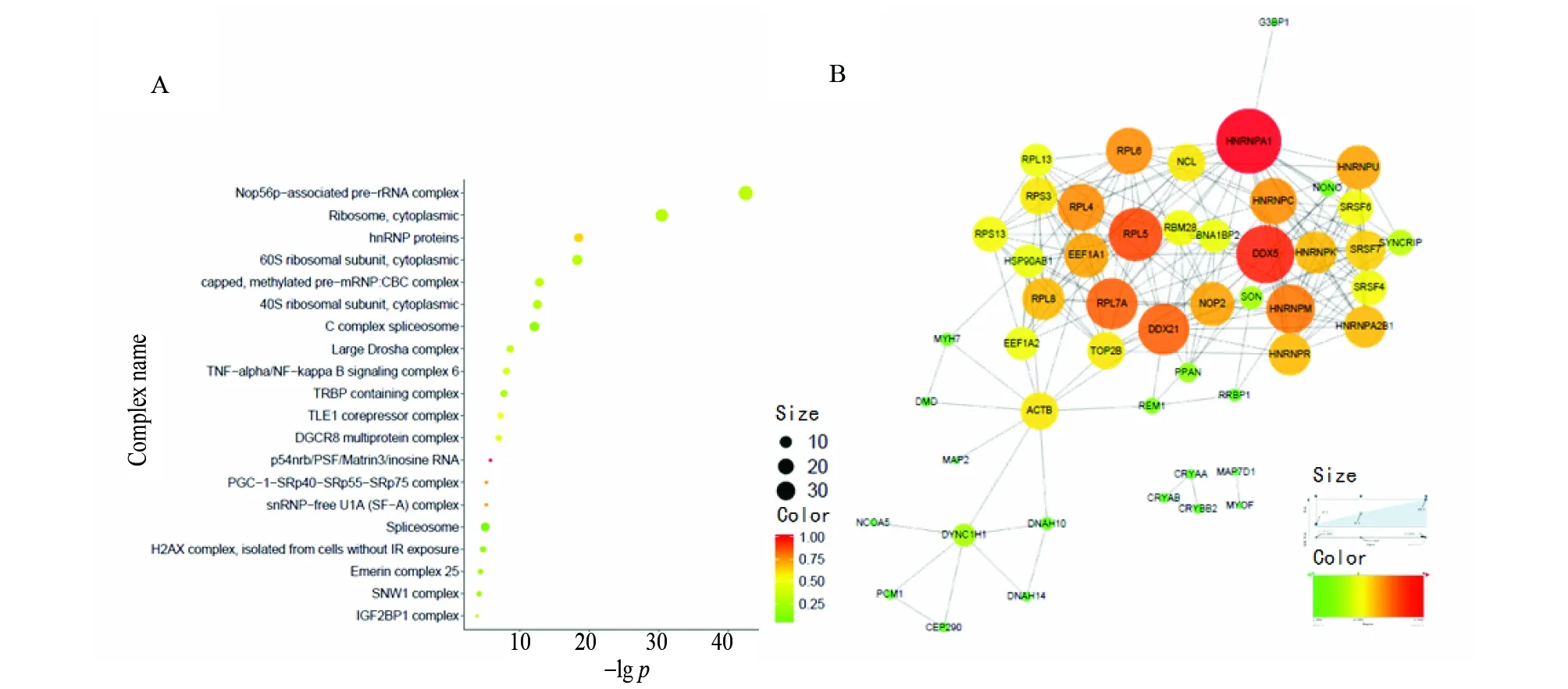
图3 5S rRNA互作蛋白组复合体富集及其蛋白与蛋白互作网络Fig.3 Protein complex enrichment and PPI network of 5S rRNA interactome
2.4 GO和KEGG通路富集显示5S rRNA参与mRNA剪接调控
将5S rRNA互作蛋白组在ConsensusPathDB数据库中进行GO分析,设定p<0.01,并用R软件作图(图4)。GO的BP分析结果显示:5S rRNA互作蛋白组显著富集于核糖体蛋白复合体起源、翻译启动(图4),此外还富集多种复合物代谢调控过程和染色质调控过程。GO细胞成分(CC)分析显示5S rRNA与大量细胞核蛋白(nuclear part)有互作。这些蛋白可能跟5S rRNA的加工成熟相关,但也有可能意味着5S rRNA不仅在细胞质中参与蛋白质的翻译调控,还可能进入细胞核中参与各种基因表达或者加工的调控。可以预计5S rRNA互作蛋白组中应该主要为核酸结合蛋白,这点得到了GO分子功能(MF)分析结果的支持。MF分析显示5S rRNA互作蛋白组主要富集了核酸结合蛋白,这说明我们的数据可信度较高。MF分析也显示5S rRNA也结合了其它功能的蛋白,这些功能蛋白有可能是非典型的RNA结合蛋白(图4)。对5S rRNA互作蛋白组进行KEGG通路富集分析(表3),并根据p值排序(p值越小,富集该通路的可信度越高),5S rRNA相互作用蛋白组富集到的通路可信度最高的是核糖体通路,与蛋白复合体富集分析结果一致。值得注意的是mRNA剪接通路富集可信度排名第二,仅次于核糖体通路。5S rRNA与包括各类SRSF系列蛋白在内的14个mRNA剪接相关蛋白有互作,表明mRNA剪接调控功能可能是5S rRNA翻译调控功能之外的又一重要功能。除了核糖体通路和mRNA剪接通路外,5S rRNA相互作用蛋白组还富集了单纯疱疹病毒感染和内质网蛋白加工通路。随着p值的增大,5S rRNA互作蛋白组富集到单纯疱疹病毒感染和内质网蛋白加工通路可信度变低,富集到这两个通路中的蛋白也随之变少。
综上所述,结合蛋白复合体富集分析、PPI网络构建、GO分析、KEGG通路富集分析,我们得出5S rRNA参与mRNA剪接调控的结论。
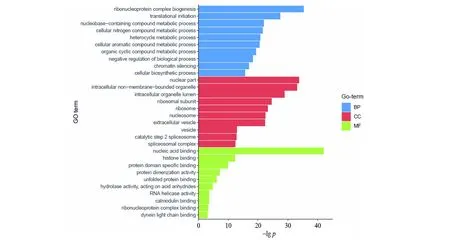
图4 5S rRNA互作蛋白组的GO分析Fig.4 Genetic ontology analysis of 5S rRNA interactome

p1)q2)TermGenes1.04E-244.46E-23hsa03010:Ribosome-Homo sapiens (human)RPS13; RPL37A; RPL8; RPS14; RPL6; RPL7; RPL4; RPS18; RPS15A; RPS3; RPS8; RPL7A; RPL29; RPL23A; RPL24; RPL5; RPL14; RPL17; RPL11; RPL13; RPS4X; RPL18; RPL19; RPL26L1; RPS3A; RPL10L3.13E-143.37E-13hsa03040:Spliceosome-Homo sapiens (human)SRSF5; SRSF4; SRSF7; SRSF6; SRSF1; HNRNPA3; HNRNPA1; HNRNPU; SRSF8; RBMXL1; DDX5; RB-MXL3; HNRNPK; HNRNPM; RBMX; HNRNPC; TRA2B0.003 075 650.022 042hsa05168:Herpes simplex in-fection-Homo sapiens (hu-man)SRSF5; SRSF4; SRSF7; SRSF6; SRSF1; SRSF8; HNRN-PK0.007 294 180.039 206hsa04141:Protein processing in endoplasmic reticulum-Ho-mo sapiens (human)SYVN1; CRYAB; CRYAA; HSP90AB1; HSP90AA1; RRNP1
1),2)E-n: ×10-n
3 讨 论
RNA互作蛋白组研究是RNA研究的前沿领域之一,迅速确定RNA内源互作蛋白组图谱可以帮助我们全面理解RNA的功能和调控机制。采用传统体外的标记RNA Pull-Down方法对RNA互作蛋白进行研究具有通量低、实验繁琐耗时和效率低等缺点,并且一次只能鉴定一到几种RNA结合蛋白。利用传统的RNA结合蛋白鉴定方法是很难对RNA的结合蛋白谱有全面的了解。例如,从mRNA发现后的几十年间,人们总共才鉴定出了500多种mRNA结合蛋白。而在2012年,Castello等[26]利用和我们使用的类似策略系统分离了mRBP,并利用HPLC-MS做鉴定,将mRNA结合蛋白成员迅速增加到了接近900种,极大地提高了RNA结合蛋白的鉴定效率。再比如调控X染色体失活的lncRNA Xist,通过ChIRP-MS技术将其互作蛋白从不到10种一下增加到了81种[7],使得Xist的功能和调控机制得以重新认识。
在本研究中,利用ChIRP-MS这种RNA互作蛋白组高通量鉴定技术系统地鉴定了5S rRNA的互作蛋白组图谱。共鉴定出162个5S rRNA互作蛋白,极大地拓展了对5S rRNA互作蛋白的认识。结果显示ChIRP-MS不仅鉴定到了大量已知的5S rRNA互作蛋白(表2,图2,3),还鉴定出了大量新的5S rRNA互作蛋白。这些新的5S rRNA互作蛋白来自各种细胞功能蛋白复合体,包括mRNA剪接复合体、TNF-α信号通路调控复合体、microRNA加工成熟相关的DGCR8复合体等等(图3A)。5S rRNA的这些相互作用蛋白暗示5S rRNA可能参与了mRNA剪接调控、TNF-α信号通路调控和microRNA加工成熟调控等正常生理调控过程。此外还发现5S rRNA可能参与了一些病理过程的调控,比如病毒感染(表3)。这些发现极大地扩展了5S rRNA的可能功能范围,为未来5S rRNA的具体功能和调控机制研究指明了新的方向。
基于蛋白复合体富集分析、PPI网络构建、GO分析、KEGG通路富集分析,我们发现5S rRNA与mRNA剪接复合体有明显的互作,暗示它可能参与了mRNA剪接调控。在5S rRNA互作蛋白组中大量存在SR蛋白家族、HNRNP蛋白家族和DDX5等跟mRNA剪接密切相关的蛋白[27-29],结果暗示这些蛋白可能需要通过与5S rRNA的互作来发挥正常的功能。此外,SR蛋白家族、HNRNP蛋白家族除了与mRNA剪接相关外,还参与调控RNA稳定性、翻译和蛋白降解等等[27],这也暗示了5S rRNA可能也具有同样的功能。综上所述,我们的5S rRNA互作蛋白组研究表明5S rRNA的功能可能远不局限于蛋白质翻译调控,可能还参与了mRNA剪接、信号通路调控等过程。研究也说明互作蛋白组研究是ncRNA功能和调控机制研究的有力工具,能帮助揭示ncRNA在生命活动过程中所扮演角色的全貌。基于本文的内容,我们计划更深层次研究5S rRNA如何参与mRNA剪接调控,并将在相关已知发生mRNA剪接生物学表型的病理细胞或组织样品进行一系列工作。这样会使我们的工作更有临床价值和生物学意义。接下来的工作可以包括:对可信度较高的蛋白进行细胞内RNA免疫沉淀(RNA immunoprecipitation,RIP)与免疫共沉淀(protein complex immunoprecipitation,Co-IP)实验以证明该组合的有效性,截断实验确定5S rRNA与其互作蛋白的结合位点,RNA干扰(RNA interference,RNAi)实验研究5S rRNA调控其互作蛋白发挥功能的模式。

