网络药理的大黄素及其类似物抗肿瘤分子机制研究
秦春明
大黄素是大黄、何首乌等常见中药材中的一个活性成分,具有良好的抗肿瘤、抗炎和抗微生物等方面作用[1]。因为大黄素具有多种生物活性,同时在合成的大黄素类似物中也表现出不同的生物活性,特别是在抗肿瘤活性方面有着巨大地开发潜力[2]。然而,大黄素及其类似物的药理及分子机制的研究需深入开展,大黄素及其类似物的药物靶点特别是基因靶点的作用环节十分复杂,常规药理和分子机制研究方法很难全面揭示抗肿瘤药理和分子机制[3]。本研究同时借助多个在线数据库资源,分析大黄素及 其类似药物中已知基因靶点的生物学信息,借助相互作用网络,通过关键基因通路分析,建立“药物-靶点-通路”网络,为进一步了解大黄素及其类似物抗肿瘤活性、分子机制提供资料和思路。
1 资料与方法
1.1 大黄素类似物筛选
药物数据DrugBank(https://www.drugbank.ca/)是世界最大、最完整的药物资源库,由加拿大卫生研究院提供支持。通过检索全医药学大词典,获取 大黄素化学名称列表,将该列表导入DrugBank 数据库中在线检索,基于化合物结构相似、生物活性相近的原则,以相似度评分为标准筛选药物,以系统默认的相似度评分阈值>0.7 为标准[4],获取高相似度药物的编号及药物名称。
1.2 基因靶点预测
生物医学文本挖掘的一个关键任务是发现各种生物医学实体之间的潜在关联,Polysearch 2.0(http: //polysearch.cs.ualberta.ca/)是一个在线文本挖掘系统,可以用于识别人类疾病、基因、蛋白质、药物等之间关系的生物数据库。将大黄素及其类似物列表,通过Polysearch 2.0 数据库检索,获取大黄素及其类似物已知靶点列表,该列表导入可视化软件Cytoscape 3.6.1,将结果可视化输出,大黄素及其类似物和靶点由节点表示,2 个节点之间的相互作用由边表示。节点的度数是与节点相连边的数量,度数越大,说明网络中与该节点直接相关的节点数越多,表明该节点在网络中越重要。
1.3 蛋白-蛋白相互作用网络(PPI)分析
String 数据库(https://string-db.org/)主要是一个搜寻蛋白质之间相互作用的数据库,是一个检索相互作用基因的搜索工具,可以用于预测蛋白质相互作用信息的生物数据库。将上述所得基因信息导入到String 数据库中,将置信度评分≥0.7 定义为显著,通过可视化工具Cytoscape 3.6.1 构建PPI 网络,以阈值degree>10 为标准确定中心基因。使用Cytoscape插件MCODE(Molecular Complex Detection,MCODE)对PPI 网络的模块进行分析,筛选显著模块,条件设置为:degree cutoff=2,node score cutoff=0.2,k-core=2,maximum depth=100[5]。
1.4 KEGG 通路分析
生物学信息注释数据库(DAVID,https://david.ncifcrf.gov/)可以提供系统综合的生物功能注释信息,能够找出最显著富集的生物学注释,通过David 数据库对上述显著模块中基因KEGG(Kyoto Encyclopedia of Genes and Genomes)通路分析,设定阈值P<0.05,筛选具有显著性差异的生物学过程和通路[5]。
2 结果
2.1 大黄素类似物筛选结果
通过DrugBank 数据库筛选,以大黄素相似度评分为1,相似度评分>0.7 的共找到7 个,根据相似度评分对大黄素类似物行降序排列,有1,2,5,8-四羟基蒽醌-9,10-二酮(1,2,5,8-tetrahydroxy anthracene- 9,10-dione)、二羟蒽醌(Dantron)、大黄酸(Rhein)、大黄酸蒽酮(Rheinanthrone)、蒽啉(Anthralin)、氧化乙酰基蒽三酚(Oxidized Acetyl Dithranol)、乙酰基蒽三酚(Acetyl Dithranol)共7 个。
2.2 大黄素及7 个类似物潜在的基因靶点
通过Polysearch 2.0 数据库检索,收集大黄素及7 个类似物潜在的相关基因靶点,其中大黄素有36 个基因靶点,大黄酸有30 个基因靶点,蒽啉有3 个靶点,二羟蒽醌有1 个基因靶点,其余化合物在Polysearch 2.0 数据库均未检索到相关基因靶点。通过资料汇总,剔除重复基因靶点,共收集到60 个基因靶点,运用Cytoscape 3.6.1 软件构建化合物-基因靶点网络,如图1所示(方形节点为药物,椭圆形节点为基因靶点)。图中共64 个节点(4 个大黄素及其类似物和60 个潜在基因靶点),其中大黄素与大黄酸沟通作用基因靶点有7 个,大黄素与蒽啉沟通作用基因靶点有2 个,BARS 基因则是大黄素、大黄酸、大黄酸蒽酮共同作用的靶点。
2.3 基因靶点相互作用网络分析
基因靶点的PPI 网络由String 数据库中构建的60 个节点和168 条边组成,PPI 富集P=1.0E-16。将String 数据库中所得数据导入Cytoscape 3.6.1 软件进行可视化(图2A),以阈值degree>10 为标准,确定的中心基因总共13 个,分别有VEGFA、BCL2、IL8、IL1B、CXCL12、PTGS2、HRAS、CDH1、FGF2、TLR4、HSPA4、VIM、KIT。通过Cytoscape 3.6.1软件插件MCODE 在构建的PPI 图中筛选出显著模块,显著模块中有13 个节点和70 条边组成,且筛选出模块中包含所有中心基因(图2B)。
2.4 KEGG 通路富集分析
与相关的13 个基因靶点进行KEGG 通路注释,得到P<0.05 的通路有24 条,根据P值大小排序列出前5 的KEGG 通路(表1)主要涉及肿瘤过程、NF-kappa B 信号通路、PI3K-Akt 信号通路、miRNA肿瘤过程和Rap1 信号通路,涉及基因包括HRAS、PTGS2、BCL2、VEGFA、CDH1、KIT、FGF2、CXCL12、IL1B、TLR4、KIT、FGF2 等,其中NF-B 信号通路、PI3K-Akt 信号通路和Rap1 信号通路均与肿瘤关系密切。
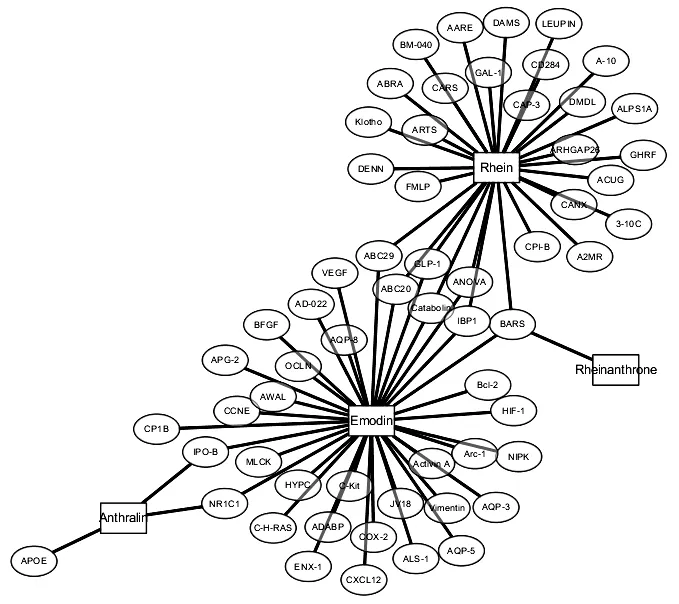
图1 药物-基因靶点网络图
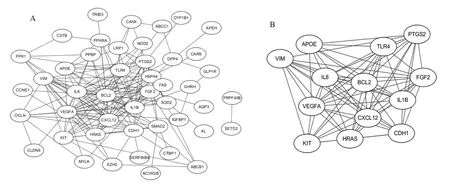
图2 相互作用网络图(A 为PPI 网络图,B 为显著表达基因靶点网络图)

表1 Top 5 KEGG 通路富集分析
3 讨论
大黄素是中药中重要的单体化合物,具有多种功效,包括抗炎、抗肿瘤和抗微生物、保护肝肾、改善微循环等作用,其衍生物也报道均有多种生物活性,包括抗肿瘤生物活性[6]。多数研究是基于传统的药理学方法,对于传统药理学方法揭示药物的药理作用机制有较完善和成熟的研究思路和方法,但是,这些研究方法很难全面描述药物与机体直接错综复杂的关系及分子机制,而网络药理学以系统生物学为基础,系统阐述了机体与药物相互作用的原理与规律[7]。
本研究通过已有的数据库资源(DrugBank、Polysearch 2.0、String、DAVID),运用网络药理学的方法,对大黄素及其类似物整理分析已知基因靶点信息,构建了药物-基因靶点效应网络图,同时通过运用PPI 网络找寻网络中的显著基因,获得了大黄素及其类似物所对应的基因靶点之间的相互关系,通过富集分析,获取基因所涉及通路。研究结果显示,大黄素类似物具有潜在的抗肿瘤活性,有可能通过影响NF-κB 信号通路、PI3K-Akt 信号通路和Rap1信号通路而发挥抗肿瘤活性。
大黄素及其类似物通过多靶点和多种通路协调发挥抗肿瘤作用。大黄素及其类似物涉及与抗肿瘤密切相关的生物学过程有细胞分裂,涉及基因包括VEGFA、CXCL12 等多种基因,Lin 等[8]研究报道大黄素具有抑制细胞的分裂增殖,诱导细胞凋亡,同时通过调节VEGFR 等发挥抑制血管生成的作用,具有双重抗肿瘤作用。大黄素也可以下调肝癌细胞中CXCL12-CXCR4 信号,CXCL12-CXCR4 信号级联在肝癌患者侵袭转移过程中发挥重要作用,抑制CXCL12 诱导的肝癌细胞系迁移和侵袭[9]。
本研究中通过KEGG 通路分析发现,大黄素及其类似物涉及NF-κB 通路、PI3K/Akt 信号通路等。NF-κB 通路在体内是一条经典的通路,NF-κB 信号的失调与肿瘤、炎症、自身免疫性疾病密切相关[10],在肺癌细胞中,大黄素通过对TRIB3 的下调,通过NF-κB 信号通路,影响诱导的细胞凋亡,从而抑制肺癌生长[11]。PI3K/Akt 信号通路是络氨酸激酶级联信号传导通路,与肿瘤增殖、生长、凋亡、参与血管生成等密切相关,在肿瘤的发生、发展、预后等过程中具有重要作用[12];PI3K/Akt 信号通路异常活化,磷酸化酶激酶-3β,进而降低抗凋亡蛋白BCL2的降解,从而抑制凋亡,大黄素可诱导细胞外信号调节激酶(ERK)和p38 的磷酸化,同时轻度抑制p-c-Jun-n 端激酶(p-JNK)的表达,从而诱导癌细胞发生凋亡[13]。
总之,本研究通过大黄素类似物筛选、靶点预测、网络构建、KEGG 通路分析对大黄素一类药物的抗肿瘤潜在作用机制进行探讨,为了解抗肿瘤机制提供了理论依据,为今后研究这一类药物的共同作用机制提供依据。

